修回日期: 2005-04-14
接受日期: 2005-07-05
在线出版日期: 2005-10-15
目的: 应用抑制性消减杂交技术及生物信息学技术筛选并克隆乙型肝炎病毒DNAPTP1反式激活的新型靶基因.
方法: 以HBV DNAPTP1表达质粒pcDNA3.1(-)- DNAPTP1转染HepG2细胞, 以空载体pcDNA3.1(-)为对照; 制备转染后的细胞裂解液, 提取mRNA并逆转录为cDNA, 经Rsa I酶切后, 将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制性聚合酶链反应(PCR), 将产物与pGEM-Teasy载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR扩增后进行测序及同源性分析.
结果: 成功构建DNAPTP1反式激活基因差异表达的cDNA消减文库. 文库扩增后得到60个白色克隆, 经菌落PCR分析, 得到32个200-1 000 bp插入片段. 对所得片段测序, 并进行同源性分析, 获得3个差异表达的已知蛋白基因和4个未知功能的染色体序列.
结论: 筛选到的cDNA全长序列, 包括一些与细胞生长调节、物质代谢、免疫及细胞凋亡密切相关的蛋白编码基因, 推测了DNAPTP1可能存在的调控机制的线索.
引文著录: 高学松, 成军, 甄真, 郭江, 张黎颖, 陶明亮. 应用抑制性消减杂交技术克隆乙型肝炎病毒DNAPTP1的反式调节基因. 世界华人消化杂志 2005; 13(19): 2371-2374
Revised: April 14, 2005
Accepted: July 5, 2005
Published online: October 15, 2005
AIM: To clone and identify the new target genes transactivated by DNAPTP1 of the hepatitis B virus with the suppression subtractive hybridization technique and bioinformatics.
METHODS: The mRNA was isolated from HepG2 cells transfected with pcDNA3.1 (-)-DNAPTP1, and then it was transcribed into the cDNA. The pcDNA3.1 (-) empty vector was used as control. After digested by Rsa I, the tester cDNA was then divided into two groups and ligated with two different adaptors. After the tester cDNA was hybridized twice with the driver cDNA and underwent two times of nested polymerase chain reaction (PCR), the amplified cDNA fragments were subcloned into pGEM-Teasy plasmid vectors to construct the subtractive library. The library was amplified by E. coli strain DH5a. The randomly picked cDNA was sequenced and analyzed in the GenBank after the PCR.
RESULTS: The cDNA subtractive library of HBV PTP1 transactivating genes was successfully constructed. The amplified library contained 60 positive clones, from which 32 inserts with 200-1?000 bp in length were obtained. Three differentially expressed protein genes and 4 sequences with unknown function were found by sequence analysis.
CONCLUSION: The obtained genes may code the proteins involved in the regulation of cell cycle, metabolism, immunity and cell apoptosis.
- Citation: Gao XS, Cheng J, Zhen Z, Guo J, Zhang LY, Tao ML. Cloning of hepatitis B virus DNAPTP1 transactivating genes by suppression subtractive hybridization technique. Shijie Huaren Xiaohua Zazhi 2005; 13(19): 2371-2374
- URL: https://www.wjgnet.com/1009-3079/full/v13/i19/2371.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v13.i19.2371
乙型肝炎病毒(HBV)是嗜肝DNA病毒的一种, HBV DNA的长度为3.2 kb, 具有4个开放读码框架(ORF), 分别编码HBV的表面抗原蛋白、核心/e抗原蛋白、X蛋白以及HBV DNA聚合酶(HBV DNA P)[1-3]. HBV DNA P基因产物是一种多蛋白, 参与病毒复制的全过程, 每一成分蛋白分别在不同环节发挥作用. 目前只知道HBV DNA P在HBV的复制过程中具有聚合酶/逆转录酶的活性, 但是, HBV DNA P在肝细胞中表达之后, 对于肝细胞本身有什么样的影响一直没有系统的研究. HBV DNA P蛋白在肝细胞中产生之后, 通过对于肝细胞信号转导的影响, 改变肝细胞的基因表达谱, 这是HBV感染肝细胞的重要的致病机制.
我们已经应用抑制性消减杂交(suppression subtra-ctive hybridization, SSH)技术[4,5]研究了HBV DNA P的反式激活靶基因, 筛选到了一系列未知功能基因, 其中一个命名为乙型肝炎病毒DNA聚合酶反式激活蛋白1(human gene 1 transactivated by hepatitis B virus DNA polymerase, DNAPTP1)[6]. 为进一步研究DNAPTP1与乙型肝炎病毒致病性的关系, 我们应用抑制性消减杂交技术, 筛选DNAPTP1蛋白反式激活作用相关的靶基因, 为研究DNAPTP1蛋白的生物学功能及乙型肝炎病毒致病机制提供理论依据.
HepG2细胞及感受态大肠杆菌DH5a(本室保存), pcDNA3.1(-)真核表达载体(Invitrogen), FuGENE6转染试剂(Roche), mRNA纯化试剂盒(Amersham Pharmacia Biot ech), PCR-Select cDNA Subtraction试剂盒(Clontech), 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech), High Pure PCR Product纯化试剂盒(Boehringer Mannheim), T7、SP6通用引物及pGEM-Teasy载体(Promega). HBV DNAPTP1真核表达质粒pcDNA3.1(-)-DNAPTP1由本室构建.
1.2.1 真核表达载体的细胞转染: 用FuGENE6转染试剂将2 mg pcDNA3.1(-)-DNAPTP1及pcDNA3.1(-)空载体分别转染35 mm平皿HepG2细胞, 48 h后收获细胞.
1.2.2 细胞mRNA提取: 用mRNA纯化试剂盒, 直接提取转染了重组表达质粒及空载体的HepG2细胞mRNA, 经琼脂糖凝胶电泳及分光光度计进行定性、定量分析.
1.2.3 消减杂交文库的建立: 采用PCR-Select cDNA Subtraction Kit, 常规SSH方法按说明书进行: 转染了重组表达质粒及空载体的HepG2细胞cDNA分别标记为Tester和Driver, 经Rsa Ⅰ消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的cDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver cDNA进行杂交; 合并两种杂交产物后再与Driver cDNA做第2次杂交; 最后将杂交产物做选择性PCR扩增, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增.
1.2.4 消减文库扩增及克隆鉴定分析: 扩增产物与pGEM-Teasy载体连接, 转化DH5a感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养16-18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 产物经20 g/L琼脂糖凝胶电泳鉴定, 证明含有插入片段(200-1 000 bp)后, 测序(上海联合基因公司). 应用生物信息学将测得序列与GenBank数据库进行同源性分析.
使用高质量的mRNA是保证cDNA高产量的前提. 紫外分光检测显示, 转染了真核表达质粒及空载体的HepG2细胞mRNA分别为3.74 mg和4.22 mg, A260/A280 = 2.17, 10 g/L琼脂糖凝胶电泳见 mRNA为>0.5 kb清晰慧尾片状条带.
将连接有Adaptor l和Adaptor 2的两组dscDNA分别用不同的特异性引物(看家基因甘油三磷酸脱氢酶G3PDH引物)进行28个循环扩增, 产物用20 g/L琼脂糖凝胶电泳鉴定. 结果显示两组dscDNA扩增产物浓度相当, 说明dscDNA已与接头高效率连接.
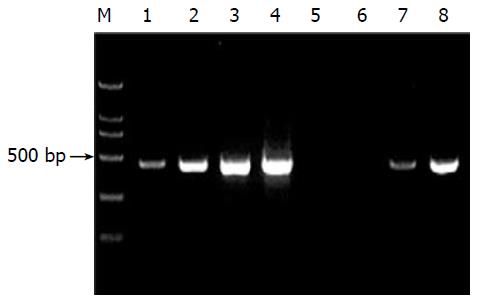
分别以消减及未消减PCR产物为模板, 用G3PDH引物进行PCR扩增, 分别在18, 23, 28, 33次循环结束时从体系中吸取5 mL进行电泳鉴定. 结果显示: 与未消减组PCR产物相比, 消减组PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图1).
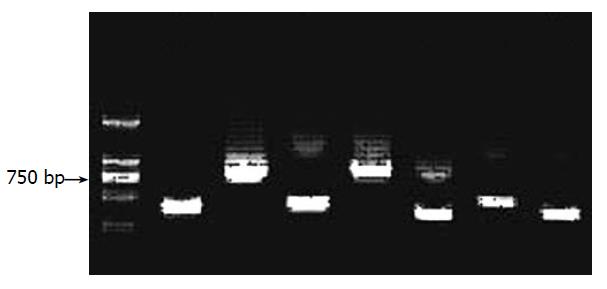
杂交产物经两轮PCR扩增后, 凝胶电泳显示部分差异表达的cDNA条带, 这些条带可能代表差异表达的基因片段. 对差异表达基因片段进行菌落PCR扩增, 结果显示为200-1 000 bp大小不等的插入片段, 所获得的60个克隆中几乎均含有插入片段(图2).
对32个克隆测序, 与GenBank数据库进行初步比较. 其中19个克隆分别与5条未知功能染色体序列同源, 可能代表了某些新基因. 其余31个均与已知基因的部分序列高度同源(85-100%)(表1)
| 已知的同源序列编码蛋白 | 相同克隆数 | 同源性(%) |
| 甲胎蛋白 | 1 | 100 |
| 细胞分裂周期素26 (CDC26) | 2 | 100 |
| 原胶原脯氨酸a多肽1 (脯氨酰-4羟化酶a多肽1 P4HA1) | 1 | 96 |
| 酪蛋白激酶2b多肽 (CK2B) | 1 | 99 |
| 线粒体39S核糖体蛋白L27 | 1 | 100 |
| 细胞色素C氧化酶8A亚单位(COX8A) | 1 | 99 |
| 丝氨酸/苏氨酸激酶25 | 1 | 100 |
| 帕金森病蛋白7 | 1 | 98 |
| 核糖体蛋白质S3 | 1 | 100 |
| 苏氨酰基tRNA合成酶 | 1 | 100 |
| 人类蛋白酶体a亚型7 | 1 | 88 |
| 未知功能序列 | 12 | 95-100 |
HBV DNA P存在于Dane颗粒核心内, 是一种依赖于DNA的DNA聚合酶, 其功能与修补及延伸双链DNA的短链有关. HBV DNA P基因在ORF中最长, 并且与C、S、X基因区有重叠, 其编码的P蛋白含有3个功能域和1个无意义的间隔区, 分别是末端蛋白(TP)、反转录酶(RT)和RNA酶H(RNase H), 各区段分别在2 307-2 840 nt、133-1 128 nt、1 129-1 621 nt, 其间的2 841-0-132 nt为不编码的间隔区. 在4个区域中, 末端蛋白作用独特, 末端蛋白内的第96位酪氨酸残基可引导DNA合成, 并使多聚酶与病毒DNA共价结合, 间隔区无已知的功能, 只是将末端蛋白和其他分子连接起来, 逆转录酶和RNase H包含2个已知的酶活性位点, 后两者与其他相关的逆转录病毒和逆转录因子的多聚酶是一致的[7-11]. 既往的研究多集中于HBV DNA P在病毒生活周期中的作用机制, 但随着对HBV致病机制研究的深入, 发现许多病毒基因组的反式激活蛋白, 以其表达产物的间接方式参与另外一些基因的功能调节. 由于受到不能从病毒体直接纯化和在不同的系统中表达有活性酶的限制, 目前对聚合酶及其所编码的各个功能区域蛋白质的功能的认识还不是很清楚. 肝细胞内表达的DNAPTP1是否也能激活某些特定基因的表达? 这些表达产物对细胞的损害、增生分化的作用如何? 基于上述研究目的, 我们采用抑制性消减杂交方法, 克隆DNAPTP1基因片段转染细胞, 反式激活差异表达的基因, 构建其cDNA消减文库, 为今后进一步分析和研究病毒蛋白的致病机制奠定基础.
与传统的方法比较, SSH方法具有实验周期短、易操作、可靠性高、假阳性率低等特点, 能有效地分离扩增低丰度特异表达的基因, 可以在较短的时间内获得较理想的实验结果. 实验应用SSH方法成功地构建了DNAPTP1反式激活相关基因差异表达的cDNA消减文库, 随机挑选32个克隆测序, 并与GenBank数据库进行同源性比较分析, 其中包括一些细胞内结构与细胞生长相关蛋白, 如线粒体蛋白、核糖体蛋白, 在细胞生长、分化、黏附中发挥重要作用; 甲胎蛋白与肝细胞恶性增生相关, 作为肝细胞癌的血清标志物已被公认; 分裂后期促进复合物(APC)是一种细胞周期调节泛素-蛋白连接酶, 调节有丝分裂中的重要事件, 例如分裂后期的起始与末期的退出. 细胞分裂周期素26(CDC26)是其家族成员, 对于细胞分裂、分化起重要作用[12]. 细胞色素C氧化酶是一种线粒体呼吸链的末端转移酶, 细胞色素C氧化酶8对于细胞内最高呼吸水平和细胞色素C氧化酶的活性至关重要[13]. 原胶原脯氨酸a多肽1(脯氨酰-4羟化酶a多肽1, P4HA1)在胶原合成中发挥重要作用. 它通过使脯氨酸羟基化来催化4-羟脯氨酸的形成. 与肝纤维化、肝硬化形成过程密切相关[14]. Sakaida et al[15]研究脯氨酰-4羟化酶抑制剂(HOE 077)在小鼠的肝纤维化的形成中的作用时发现, 该抑制剂可以抑制脯氨酸羟基化和星状细胞的激活, 从而降低原胶原和组织基质金属蛋白酶抑制剂1(TIMP-1) mRNA的表达. 有学者研究发现, 细胞缺氧导致细胞外基质主要是胶原的合成与分解的平衡被打破, 可以上调原胶原-脯氨酸a多肽在转录和翻译的水平, 在纤维肉瘤细胞HT1080中, 短期的缺氧有利于原胶原-脯氨酸a多肽的表达和抑制MMPs的合成. 但在长期缺氧时, 抗纤维化的机制占了主导作用, 虽然原胶原-脯氨酸a多肽仍在高水平表达, 但胶原形成是由MMPs合成与活性抑制的解除决定的[16]. 胶原病理性沉积, 肝小叶结构紊乱, 肝细胞功能受损最终导致肝纤维化、肝硬化的形成. 脯氨酰-4羟化酶是细胞内胶原处理的关键酶. 使用其抑制剂S 4682可以在培养的肝星状细胞中降低羟脯氨酸的合成. 使用C14标记脯氨酸, 可以发现S 4682可以抑制肝内胶原的羟基化. 在CCl4造成的肝损伤模型中, 血清Ⅲ型原胶原N肽(PⅢNP)显著升高, 使用S 4682降低胶原沉积和PⅢNP水平, 减少了腹水的发生[17]. 蛋白激酶CK2是一种普遍存在的丝氨酸/苏氨酸激酶, 是一种稳定的a2b2的四聚物. a为接触反应亚基, b亚基为调节亚基. 在体外CK2b亚基在调节CK2的接触反应中发挥核心作用. CK2b亚基的N末端包含的最小功能域: 两个锌指结构域是CK2b二聚体结构的中枢, 特异序列是核运输的工具. 在这个区域包含核运输的信号系统[18]. 通过CK2b介导, 蛋白激酶CK2可以使CDC25B磷酸化, 调节CDC25B的活性, 在有丝分裂的起始阶段发挥重要作用[19]. Shimada et al[20]在使用酵母双杂交筛选人类疱疹病毒6直接早期(immediate early, IE)蛋白2结合蛋白时发现CK2b可与HHV-6 IE2特异性结合, 并被体外免疫共沉淀等试验证实, 说明CK2b在病毒的复制中可能影响病毒和细胞RNA的转录和翻译. 人免疫缺陷病毒-1(HIV-1)Rev蛋白是转录后调节蛋白, 穿梭于感染细胞的细胞核和细胞质之间, 通过控制mRNAs的核浆转运的速率调节HIV蛋白的表达, HIV Rev蛋白的磷酸化严格依赖CK2b, 磷酸化位点外的底物(substrate)的构象改变可以明显改变HIV Rev蛋白的磷酸化. 同时Rev调节CK2的多种属性, 如自磷酸化作用、对钙调蛋白的接触反应活性和聚阳离子效应剂的灵敏度, 他们的共同特性就是都包含有b亚单位[21]. 以上提示DNAPTP1可能与病毒感染后的转录与翻译有关. 通过对DNAPTP1的上述反式激活基因的分析, 我们发现其在细胞内表达后, 部分涉及细胞生长、分化、物质和能量代谢、信号转导、肿瘤发生等方面的基因表达增加, 提示DNAPTP1对体内多个系统都存在影响.
电编: 张敏 编辑:潘伯荣 审读:张海宁
| 4. | Diatchenko L, Lau YF, Campbell AP, Chenchik A, Moqadam F, Huang B, Lukyanov S, Lukyanov K, Gurskaya N, Sverdlov ED. Suppression subtractive hybridization: a method for generating differentially regulated or tissue-specific cDNA probes and libraries. Proc Natl Acad Sci USA. 1996;93:6025-6030. [PubMed] |
| 5. | Rebrikov DV, Desai SM, Siebert PD, Lukyanov SA. Suppression subtractive hybridization. Methods Mol Biol. 2004;258:107-134. [PubMed] [DOI] |
| 6. | 张 黎颖, 成 军, 邓 红, 王 春花, 刘 妍. 乙型肝炎病毒DNA聚合酶逆转录酶蛋白反式调节基因1的克隆化研究. 胃肠病学和肝病学杂志. 2004;13:466-470. |
| 8. | Stuyver LJ, Locarnini SA, Lok A, Richman DD, Carman WF, Dienstag JL, Schinazi RF. Nomenclature for antiviral-resistant human hepatitis B virus mutations in the polymerase region. Hepatology. 2001;33:751-757. [PubMed] [DOI] |
| 9. | zu Putlitz J, Lanford RE, Carlson RI, Notvall L, de la Monte SM, Wands JR. Properties of monoclonal antibodies directed against hepatitis B virus polymerase protein. J Virol. 1999;73:4188-4196. [PubMed] |
| 10. | Wang X, Hu J. Distinct requirement for two stages of protein-primed initiation of reverse transcription in hepadnaviruses. J Virol. 2002;76:5857-5865. [PubMed] [DOI] |
| 11. | 王 春花, 郎 振为, 成 军, 吴 煜, 杨 艳杰, 张 黎颖, 党 晓燕. 应用抑制性消减杂交技术筛选HBV DNA聚合酶中RNase H的反式调节基因. 世界华人消化杂志. 2004;12:1564-1568. [DOI] |
| 12. | Gmachl M, Gieffers C, Podtelejnikov AV, Mann M, Peters JM. The RING-H2 finger protein APC11 and the E2 enzyme UBC4 are sufficient to ubiquitinate substrates of the anaphase-promoting complex. Proc Natl Acad Sci USA. 2000;97:8973-8978. [PubMed] [DOI] |
| 13. | Hüttemann M, Schmidt TR, Grossman LI. A third isoform of cytochrome c oxidase subunit VIII is present in mammals. Gene. 2003;312:95-102. [PubMed] [DOI] |
| 14. | Helaakoski T, Veijola J, Vuori K, Rehn M, Chow LT, Taillon-Miller P, Kivirikko KI, Pihlajaniemi T. Structure and expression of the human gene for the alpha subunit of prolyl 4-hydroxylase. The two alternatively spliced types of mRNA correspond to two homologous exons the sequences of which are expressed in a variety of tissues. J Biol Chem. 1994;269:27847-27854. [PubMed] |
| 15. | Sakaida I, Uchida K, Hironaka K, Okita K. Prolyl 4-hydroxylase inhibitor (HOE 077) prevents TIMP-1 gene expression in rat liver fibrosis. J Gastroenterol. 1999;34:376-377. [PubMed] [DOI] |
| 16. | Fähling M, Perlewitz A, Doller A, Thiele BJ. Regulation of collagen prolyl 4-hydroxylase and matrix metalloproteinases in fibrosarcoma cells by hypoxia. Comp Biochem Physiol C Toxicol Pharmacol. 2004;139:119-126. [PubMed] [DOI] |
| 17. | Bickel M, Baringhaus KH, Gerl M, Günzler V, Kanta J, Schmidts L, Stapf M, Tschank G, Weidmann K, Werner U. Selective inhibition of hepatic collagen accumulation in experimental liver fibrosis in rats by a new prolyl 4-hydroxylase inhibitor. Hepatology. 1998;28:404-411. [PubMed] [DOI] |
| 18. | Filhol O, Nueda A, Martel V, Gerber-Scokaert D, Benitez MJ, Souchier C, Saoudi Y, Cochet C. Live-cell fluorescence imaging reveals the dynamics of protein kinase CK2 individual subunits. Mol Cell Biol. 2003;23:975-987. [PubMed] [DOI] |
| 19. | Theis-Febvre N, Filhol O, Froment C, Cazales M, Cochet C, Monsarrat B, Ducommun B, Baldin V. Protein kinase CK2 regulates CDC25B phosphatase activity. Oncogene. 2003;22:220-232. [PubMed] [DOI] |
| 20. | Shimada K, Kondo K, Yamanishi K. Human herpesvirus 6 immediate-early 2 protein interacts with heterogeneous ribonucleoprotein K and casein kinase 2. Microbiol Immunol. 2004;48:205-210. [PubMed] [DOI] |
| 21. | Meggio F, Marin O, Boschetti M, Sarno S, Pinna LA. HIV-1 Rev transactivator: a beta-subunit directed substrate and effector of protein kinase CK2. Mol Cell Biochem. 2001;227:145-151. [PubMed] [DOI] |