修回日期: 2016-02-23
接受日期: 2016-02-29
在线出版日期: 2016-04-08
目的: 探究微小染色体维持蛋白7(mini-chromosome maintenance protein 7, MCM7)基因在调节肝癌细胞生长的相关机制.
方法: 通过siRNA干扰技术沉默人肝癌SMMC-7721细胞中的MCM7基因表达, 采用人全基因组表达谱芯片, 筛查MCM7沉默后差异表达基因, 进行生物信息学分析, 并对部分差异表达基因进行蛋白免疫印迹法(Western blot)验证.
结果: 芯片筛选出在人肝癌SMMC-7721细胞中沉默MCM7基因后差异表达基因1010个, 其中上调基因391个, 下调基因619个. 这些基因主要涉及生物大分子代谢、细胞周期调控、细胞增殖、细胞凋亡等方面, 参与胞吞、肿瘤相关通路、P53信号通路、mTOR信号通路等通路的改变. Western blot对部分差异基因表达验证的结果显示CCND1、SKP2和JUP蛋白表达均下调, 与芯片检测结果一致.
结论: 应用人基因表达谱芯片, 成功筛选出沉默MCM7基因后人肝癌细胞SMMC-7721差异表达基因, 为探究MCM7基因影响肝癌细胞生长提供了有效线索.
核心提示: 本研究通过筛选沉默MCM7基因后人肝癌细胞SMMC-7721中差异表达的基因, 发现差异表达基因涉及肿瘤相关通路、细胞周期通路等信号通路, 经Western blot证实了CCND1、SKP2、JUP等蛋白差异表达, 为揭示MCM7基因影响肝癌细胞生长的作用机制提供了新的思路.
引文著录: 罗旺, 曹骥, 杨香娣, 邓玲, 王谷洋, 杨春, 李科志, 李瑗. 沉默MCM7基因后肝癌SMMC-7721细胞差异表达基因的筛选. 世界华人消化杂志 2016; 24(10): 1492-1500
Revised: February 23, 2016
Accepted: February 29, 2016
Published online: April 8, 2016
AIM: To explore the mechanisms of mini-chromosome maintenance protein 7 (MCM7) for regulating the growth of liver cancer cells.
METHODS: The expression of MCM7 gene in SMMC-7721 cells was silenced with small interfering RNA (siRNA). Human genome-wide expression profile chip was then employed to screen the differentially expressed genes, and bioinformatics analysis of the differentially expressed genes was performed. Finally, part of these differentially expressed genes were confirmed by Western blot assay.
RESULTS: In total there were 1010 genes that were differentially expressed in SMMC-7721 cells after the expression of MCM7 was silenced, including 391 up-regulated and 619 down-regulated ones. Bioinformatics analysis showed that these differentially expressed genes are involved in many cellular biological processes such as macromolecular metabolism, cell cycle regulation, cell proliferation regulation, apoptosis, endocytosis, P53 and mTOR signaling pathways. The down-regulation of CCND1, SKP2 and JUP was confirmed by Western blot, which was consistent with the results of the genome-wide expression profile chip.
CONCLUSION: The differentially expressed genes after silencing the gene MCM7 in liver cancer cells SMMC-7721 might provide some clues for understanding the mechanism by which MCM7 affects the growth of liver cancer cells.
- Citation: Luo W, Cao J, Yang XD, Deng L, Wang GY, Yang C, Li KZ, Li Y. Screening of differentially expressed genes after silencing MCM7 in liver cancer cell line SMMC-7721. Shijie Huaren Xiaohua Zazhi 2016; 24(10): 1492-1500
- URL: https://www.wjgnet.com/1009-3079/full/v24/i10/1492.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i10.1492
微小染色体维持蛋白7(mini-chromosome maintenance protein 7, MCM7)在细胞周期中作为DNA复制的准许因子, 可启动和参与DNA复制, 确保DNA的复制在每个细胞周期仅发生1次, 参与调节细胞增殖[1]. 研究发现MCM7基因的扩增和高表达与肝癌[2]、前列腺癌[3]、结直肠癌[4]、霍奇金淋巴瘤[5]等多种肿瘤的发生及预后有关. 本课题组前期实验采用RNA干扰技术沉默人肝癌SMMC-7721细胞中MCM7基因后, 发现SMMC-7721细胞周期进程得到明显阻滞, 凋亡大幅增加, 其生长增殖功能明显受到抑制[6]. 沉默MCM7基因后, SMMC-7721细胞裸鼠移植瘤的生长速度明显低于对照组移植瘤的生长速度[7]. 研究表明MCM7基因与肝癌细胞周期相关, 而目前关于MCM7基因影响肿瘤细胞周期的具体调控机制仍未完全清楚. 本研究通过Affymetrix芯片分别对沉默和未沉默MCM7基因的人肝癌SMMC-7721细胞进行检测, 寻找差异表达基因, 初步探究MCM7基因对肿瘤细胞周期的作用机制.
人肝癌SMMC-7721细胞购自中国科学院细胞研究所, 同时带有绿色荧光标记蛋白和嘌呤霉素抗性筛选标记的MCM7基因沉默重组慢病毒颗粒LV-shRNA-MCM7及对照慢病毒颗粒LV-shRNA-NC由上海吉凯基因公司构建提供, 其中shRNA靶点已在前期试验中验证[6]. DMEM、胎牛血清、PBS粉剂购自Hyclone公司; 总RNA提取试剂TRIzol购自Invitrogen公司; 逆转录试剂盒购自Fermentas公司; 实时定量PCR检测(real-time quantitative PCR, qRT-PCR)试剂盒购自TaKaRa宝生物工程(大连)有限公司; GeneChip®PrimeViewTM Human Gene Expression Array由Affymitrix提供; RIPA细胞裂解液、苯甲基碘酰氟(phenylmethanesulfonyl fluoride, PMSF)、SDS-PAGE蛋白上样缓冲液(5×)、BCA蛋白浓度测试试剂盒(增强型)、20×TBS缓冲液等均购自江苏碧云天生物技术研究所; 蛋白质预染Marker购自美国Fermentas(MBI)公司; PVDF膜购自美国Millipore公司; 兔抗人CCND1单克隆抗体、鼠抗人JUP单克隆抗体、鼠抗人SKP2单克隆抗体购自美国Abcam公司; Anti-rabbit IgG及Anti-mouse IgG二抗购自Santa Cruz公司.
1.2.1 细胞培养与慢病毒转染: 人肝癌细胞株SMMC-7721在37 ℃、50 mL/L CO2的条件下, 置于10%胎牛血清的DMEM培养基中培养. 随机分成2组, 即实验组(LV-shRNA-MCM7慢病毒沉默组)和对照组(LV-shNRA-NC空载慢病毒组). 转染过程如前期实验操作[6].
1.2.2 总RNA提取: 在25 cm2培养瓶中常规培养实验组和对照组细胞, 按照Invitrogen公司TRIzol试剂使用说明书抽提总RNA, 采用Thermo NanoDrop 2000检测其浓度和纯度, 并采用凝胶电泳系统Agilent-2100检测总RNA的完整性.
1.2.3 qRT-PCR: 应用软件Primer5.0设计引物. MCM7基因上游引物序列为: 5'-ACATCACAGCAGCATACG-3', 下游引物序列为5'-TCCACCACATCCACCATT-3', 扩增片段为139 bp, 内参GAPDH基因上游引物序列: 5'-TGACTTCAACAGCGACACCCA-3', 下游引物序列为: 5'-CACCCTGTTGCTGTAGCCAAA-3', 扩增片段大小为121 bp. 引物由TaKaRa宝生物工程(大连)有限公司合成. 将TRIzol法分别提取得到的2组细胞总RNA, 逆转录成cDNA. 设置反应条件为: 95 ℃预变性30 s, 95 ℃变性5 s, 60 ℃退火30 s, 共45个循环, 同时扩增各个样本的目的基因和内参基因, 每组细胞设计3个重复孔. 通过GAPDH基因水平校正, 采用2-△△Ct分析法验证MCM7 mRNA的相对表达量.
1.2.4 基因芯片检测及分析: 本研究采用Affymitrix公司GeneChip® PrimeViewTM Human Gene Expression Array, 包含36000多个转录本. 首先将抽提到的实验组和对照组(每组包含3个重复样本)的总RNA通过GeneChip 3' IVT Expression Kit制备成aRNA, 然后用试剂盒内的纯化试剂将aRNA纯化, 配置aRNA片段化反应液, 加入纯化的aRNA进行片段化. 然后将片段化后的aRNA与Affymitrix芯片探针杂交, 杂交完成后, 取出芯片, 用GeneChip Fluidics Station450仪器对芯片进行自动洗染, 最后采用GeneChip Scanner 3000扫描芯片, 并采用Expression console软件对扫描结果进行解析获得注释结果, 根据Ration值筛选出差异表达基因(上调或下调倍数>1.5, P<0.05), 采用GSEA软件对差异基因进行GO-Analysis(基于Gene Ontology数据库)和Pathway-Analysis(基于KEGG数据库)分析基因相关功能及生物学通路. 对根据分析结果选取显著通路中的差异表达基因采用IPA软件进行网络调控分析, 使用R-Project软件中的gplots包对差异基因进行聚类分析.
1.2.5 Western blot验证部分差异表达基因: 提取实验组和对照组各组细胞总蛋白, 采用BCA法测定蛋白浓度, -80 ℃保存备用. 蛋白变性后每孔上样20 µg, 经10%SDA-PAGE胶上电泳, 经300 mA, 150 min将蛋白转移到PVDF膜上. 用含5%脱脂牛奶的TBST溶液室温封闭PVDF膜1 h. 用封闭液稀释一抗(差异蛋白: CCND1为1:200, JUP为1:1000, SKP2为1:200; 内参蛋白: GAPDH为1:750), 然后与封闭好的PVDF膜4 ℃孵育过夜, 再用TBST洗膜4次, 每次8 min. 室温下孵育相应的二抗(稀释浓度均为1:2000)1.5 h, 再用TBST洗膜4次, 每次8 min. 采用Thermo公司PierceTM ECL. Western blot Substrate试剂盒进行X光显影.
统计学处理 采用SPSS16.0软件进行统计学分析, 结果以mean±SD表示. 计量资料两组之间比较采用独立样本t检验, P<0.05为差异具有统计学意义.
实验组与对照组细胞提取总RNA, 其A260-280介于2.01-2.09之间, 浓度>153.0 ng/µL. Agilent 2100 Bioanalyzer检测实验组RIN值>6.7, 阴性对照组RIN值>7.0, 总RNA质量符合实验要求.
qRT-PCR检测MCM7 mRNA表达情况, 结果显示, 相对于对照组, 实验组细胞的MCM7 mRNA表达水平明显下降(0.414±0.077 vs 1.001±0.057, P<0.01), 与前期实验结果一致[6].
通过siRNA干扰技术使MCM7基因表达下调后, 发现显著差异表达基因1010个, 其中上调391个, 下调619个, 涉及蛋白质代谢、氧化应激、细胞周期调控、细胞增殖、细胞凋亡等重要功能. 其中部分肿瘤相关的重要基因的差异表达情况如表1.
| 基因名称 | 基因ID | 基因描述 | 变化倍数 | P值 |
| 上调基因 | ||||
| CCL20 | 6364 | CC亚族趋化因子配体20 | 3.63 | 0.000 |
| FABP4 | 2167 | 脂肪酸结合蛋白4 | 2.90 | 0.000 |
| IL8 | 3576 | 白介素8 | 2.55 | 0.000 |
| MET | 4233 | MET原癌基因, 酪氨酸激酶受体 | 2.48 | 0.019 |
| RAB5A | 5868 | Ras致癌基因家族成员 | 1.82 | 0.007 |
| CDK6 | 1021 | 周期蛋白依赖激酶6 | 1.78 | 0.002 |
| IL6 | 3569 | 白介素6 | 1.71 | 0.007 |
| PTEN | 5728 | 同源性磷酸酶张力蛋白 | 1.64 | 0.000 |
| CDC25A | 993 | 细胞分裂周期25A | 1.59 | 0.023 |
| CDC42 | 998 | 细胞分裂周期蛋白42 | 1.52 | 0.001 |
| 下调基因 | ||||
| PPP6C | 5537 | 蛋白磷酸酶6, 催化亚单位 | -3.63 | 0.003 |
| JUP | 3728/3872 | 盘状球蛋白 | -2.64 | 0.000 |
| WARS | 7453 | 色氨酸转移核糖体合成酶 | -2.45 | 0.000 |
| FAS | 355 | 细胞表面死亡受体 | -1.83 | 0.006 |
| SKP2 | 6502 | S期激酶相关蛋白2 | -1.81 | 0.003 |
| CCND1 | 595 | 细胞周期蛋白D1 | -1.77 | 0.000 |
| SUFU | 51684 | 抑制基因的融合同旋体 | -1.75 | 0.003 |
| MAPK9 | 5601 | 有丝分裂原激活蛋白激酶 | -1.66 | 0.001 |
| TP53 | 7157 | 肿瘤蛋白p53 | -1.54 | 0.002 |
| CDC45 | 8318 | 细胞周期分裂蛋白45 | -1.51 | 0.001 |
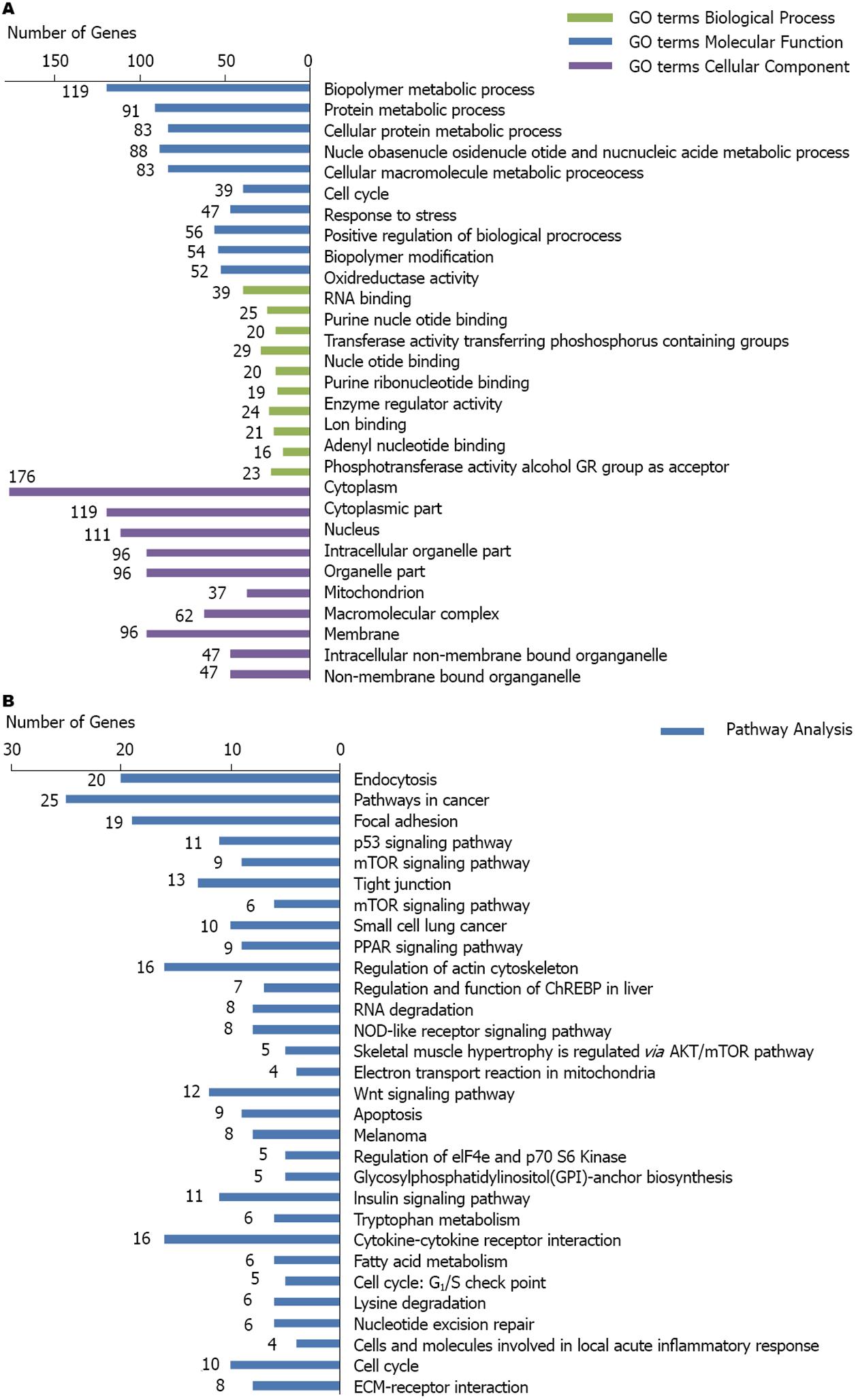
基于Gene Ontology数据库富集生物学过程、分子功能、细胞组分, 按富集指数(enrichment score, ES)由高到低排列出部分显著富集条目及富集差异表达基因数目(图1A), 其中, 生物学过程富集条目涉及生物大分子代谢过程、细胞周期、应激反应、生物过程的正性调节、分子聚合物的修饰等; 分子功能富集条目主要涉及氧化还原酶活性、RNA结合、酶调节活性、离子结合、腺嘌呤核苷酸结合等; 细胞组分富集条目主要涉及细胞质组分、细胞核、细胞内细胞器、线粒体、大分子复合物等.
基于KEGG数据库富集信号通路, 按ES由高到低排列出部分显著富集的信号通路及各通路所富集差异表达基因数目(图1B), 富集到的通路涉及胞吞、肿瘤相关通路、P53信号通路、小细胞肺癌、PPAR信号通路等.
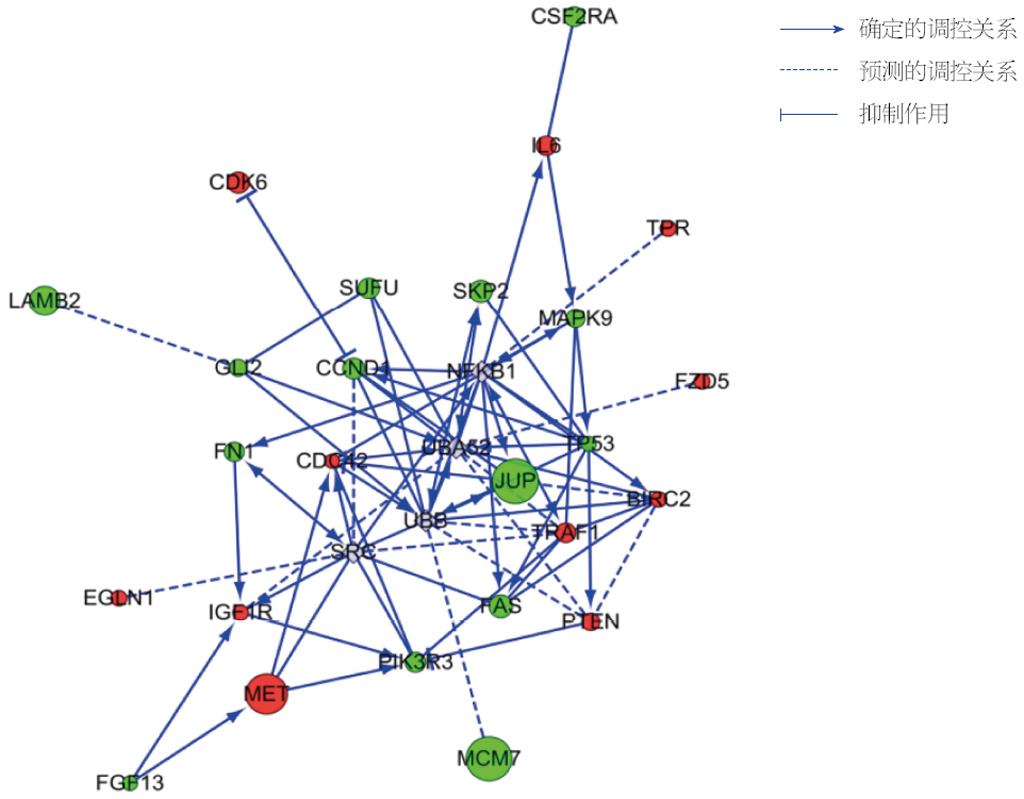
根据Pathway分析结果, 选取Pathways in cancer富集所得的基因, 绘制基因关系网络图(图2), 从预测关系网络图中, 未发现与MCM7直接作用的差异表达基因. MCM7基因有可能是通过人泛素启动子UBB调节肿瘤相关通路中部分差异蛋白的表达, 如下调CCND1、JUP、SUFU等.
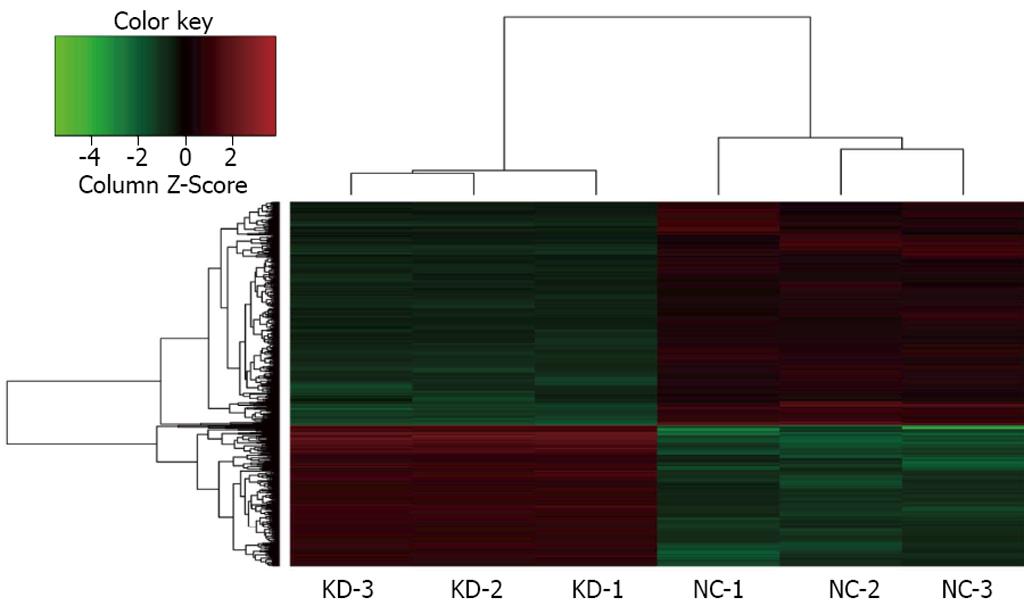
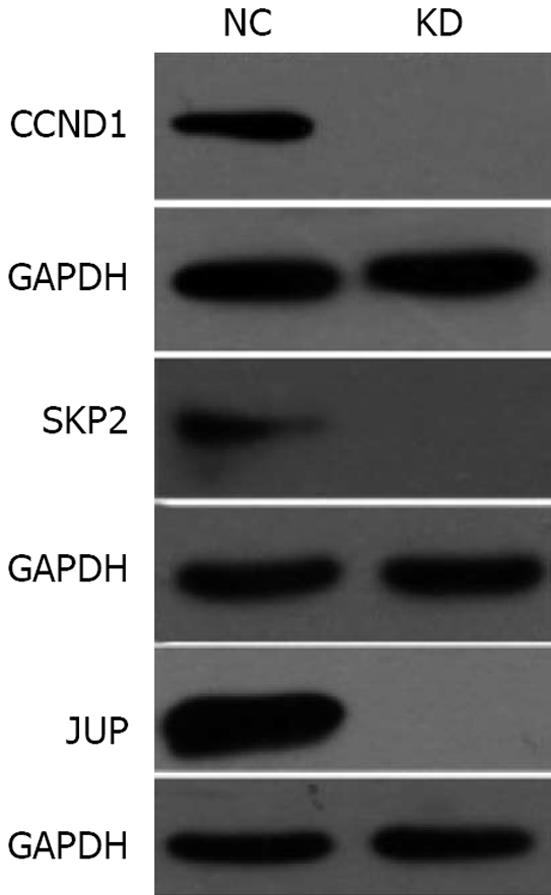
对所有差异表达基因进行分层聚类分析(图3), 其中实验组样本与对照组样本分别聚类在一起, 说明聚类结果可靠, 样本组内基因表达差异小, 组间基因表达差异大. 采取Western blot技术对部分差异基因进行验证, 结果(图4)显示: CCND1、SKP2、JUP蛋白表达均下调. 与芯片检测结果一致.
MCM7是MCM蛋白家族的成员之一, 也是DNA复制的关键蛋白. 研究[8,9]表明, MCM4,6,7形成的复合物具有DNA解旋酶活性. 在真核生物细胞周期的G1期MCM2-7形成多聚复合物, 进入S期, 与CDC45和GINS蛋白结合, 形成CMG复制叉解旋酶, 启动DNA复制[10]. 而在S期末期, MCM7蛋白泛素化促使CMG复制叉解旋酶复合体解离[11]. MCM7在保证细胞周期中DNA仅发生1次复制起到非常重要的作用. 相关研究[6,12]已经证实干扰MCM7基因后, 能明显抑制人肝癌细胞的增殖, 使细胞停滞在G1期, 并且诱导细胞的凋亡.
为更加系统全面探究MCM7基因在影响细胞周期进程和肿瘤发病中的作用机制, 本研究通过Affymitrix人类基因表达谱芯片筛选经RNA干扰技术沉默和未沉默MCM7的SMMC-7721细胞的差异表达基因, 并对其进行生物信息学分析. 在筛选出的1010个差异表达基因中, 上调基因391个, 下调基因619个, 通过GO分析发现这些差异基因主要涉及生物大分子代谢、细胞周期、应激反应等生物学过程, 其中富集到细胞周期相关的差异表达基因39个, 包含CCND1、SKP2、CDC45等关键基因. 在KEGG通路分析中发现所筛选出的差异表达基因主要参与肿瘤相关通路、P53信号通路、mTOR信号通路、WNT信号通路、细胞周期通路等通路的改变. 其中, P53信号通路、mTOR信号通路、WNT信号通路及细胞周期均已被认定与肝癌的发生密切相关[13,14]. 在诸通路中, 以Pathways in cancer所富集的差异表达基因数最多, 也是与肿瘤相关变化最显著的信号通路.
我们选取了部分差异变化较明显的基因做了Western blot验证, 发现CCND1、SKP2蛋白表达下调, 这些基因均是细胞周期通路中重要的参与者, 其对于肿瘤发生的影响机制, 均有相关研究报道证实. CCND1是细胞周期蛋白(cyclins)的重要成员, 可与CDK4、CDK6形成cyclinD1/CDK4/6复合物, 使Rb蛋白磷酸化, 诱导E2F1表达, 从而诱导细胞从G1期向S期转化, 促进细胞周期的进展[15,16]. CCND1基因在肝癌[17]、小细胞肺癌[18]、乳腺癌[19]、浆细胞性骨髓瘤[20]等多种肿瘤组织中的存在过表达现象, 该基因通过对细胞周期的影响在多种肿瘤的发生中起到重要的作用. SKP2基因被称作S期激酶相关蛋白基因, 其表达的SKP2蛋白参与形成SCF-SKP2复合物及Myc-SKP2-Mizl复合物, 参与细胞周期进程的调节、肿瘤的转移等过程[21,22]. SKP2基因在肝癌[23]、非小细胞肺癌[24]、胆管癌[25]、卵巢癌[26]、结直肠癌[27]中均呈现高表达. 本研究发现在沉默MCM7基因的人肝癌SMMC-7721细胞中CCND1、SKP2蛋白表达下调, MCM7与CCND1、SKP2之间有可能存在某种调控关系, 通过影响细胞周期而干扰肝癌细胞的生长和增殖.
本研究针对Pathways in cancer通路所富集到的差异基因进行相互作用网络图绘制, 未发现与MCM7直接作用的差异表达基因, 预测出MCM7基因可能通过人泛素启动子UBB与其他基因发生间接调控关系, 尤其是与JUP基因的显著差异表达相关. UBB是泛素编码基因之一, 其与UBC基因编码多聚泛素前体, 与蛋白质结合, 参与蛋白质的泛素化修饰[28]. 泛素分子通过泛素-蛋白酶体系参与几乎所有细胞调控过程, 包括细胞周期进程的重要环节[29]. JUP是细胞内糖蛋白catenin家族成员, 与β-catenin具有高度同源性, 但其作用机制或与之不同. JUP可通过激活c-myc基因的高表达而参与肿瘤的发生发展[30]. 有研究[31]报道称, 在人肝癌组织中检测到JUP的异常表达, 并发现其高表达与肿瘤的包膜转移和卫星结节相关. 而有研究[32]报道, JUP的蓄积受泛素-蛋白酶体途径的调控. 本研究发现下调MCM7基因后, 人肝癌SMMC-7721细胞中JUP呈明显低表达. MCM7与JUP在肝癌细胞中的具体调控机制有待进一步探究.
本研究发现下调MCM7基因后, 人肝癌细胞出现细胞周期阻滞, 凋亡增加, 其作用机制可能是: 通过影响CMG复制叉解旋酶复合体的形成, 阻滞细胞周期G1/S期的转换; 通过影响CCND1、SKP2等细胞周期关键基因的表达, 进而阻滞细胞周期的进程; 而MCM7基因对于相关基因的调控则可能是通过人泛素启动子UBB而间接实现的, 具体过程和机制仍需进一步研究.
MCM7基因的高表达与肝癌等多种肿瘤的发生及预后有关, 但目前关于MCM7基因具体调控机制仍未完全清楚. 本研究通过Affymetrix芯片分别对沉默和未沉默MCM7基因的人肝癌SMMC-7721细胞进行检测, 寻找差异表达基因, 探讨MCM7基因在肝癌发生发展中的作用机制.
高英堂, 研究员, 天津市第三中心医院; 伦永志, 副教授, 大连大学医学院; 赵鹏伟, 讲师, 内蒙古医科大学基础医学院
MCM7基因如何通过调控肿瘤细胞周期, 从而影响肿瘤的发生发展, 成为目前研究的热点.
本课题组前期实验结果表明, MCM7在人肝癌组织中呈高表达, 且与患者肝癌分期、转移、复发相关. 在沉默MCM7基因后, 人肝癌细胞周期进程受阻, 凋亡大幅增加, 裸鼠移植瘤模型也证实沉默组瘤体生长速度明显减慢, 提示MCM7基因与肝癌发生有关.
本文利用siRNA干扰技术结合人基因表达谱芯片, 通过高通量数据分析研究MCM7基因表达下调后, 对肝癌细胞基因表达谱及信号通路的影响, 探讨MCM7基因对人肝癌细胞生长的可能调控机制.
本文揭示了人肝癌中沉默MCM7基因后差异表达的基因, 探讨相关的信号通路和调控基因, 为肝癌的发病机制研究提供新的思路.
基因表达谱芯片: 是采用cDNA或寡核苷酸片段作探针, 固化在芯片上; 将待测样品(处理组)与对照样品的mRNA以两种不同的荧光分子进行标记, 然后同时与芯片进行杂交, 通过分析2种样品与探针杂交的荧光强度的比值, 来检测基因表达水平的变化, 属于高通量筛选的技术手段之一.
本文选题新颖, 技术路线清晰, 研究方法可靠, 结果合理, 讨论深入, 对相关基础研究有较高的科学意义, 尤其基于大数据分析思维对于基础及临床研究具有较明显的指导意义.
编辑: 于明茜 电编:闫晋利
| 1. | Walters AD, Chong JP. Methanococcus maripaludis: an archaeon with multiple functional MCM proteins? Biochem Soc Trans. 2009;37:1-6. [PubMed] [DOI] |
| 3. | Ren B, Yu G, Tseng GC, Cieply K, Gavel T, Nelson J, Michalopoulos G, Yu YP, Luo JH. MCM7 amplification and overexpression are associated with prostate cancer progression. Oncogene. 2006;25:1090-1098. [PubMed] [DOI] |
| 4. | Nishihara K, Shomori K, Fujioka S, Tokuyasu N, Inaba A, Osaki M, Ogawa T, Ito H. Minichromosome maintenance protein 7 in colorectal cancer: implication of prognostic significance. Int J Oncol. 2008;33:245-251. [PubMed] |
| 5. | Marnerides A, Vassilakopoulos TP, Boltetsou E, Levidou G, Angelopoulou MK, Thymara I, Kyrtsonis MC, Pappi V, Tsopra O, Panayiotidis P. Immunohistochemical expression and prognostic significance of CCND3, MCM2 and MCM7 in Hodgkin lymhoma. Anticancer Res. 2011;31:3585-3594. [PubMed] |
| 6. | 孙 雯, 曹 骥, 卢 晓旭, 朱 伶群, 杨 春, 欧 超, 骆 成飘, 李 瑗, 苏 建家. RNA干扰技术沉默MCM7基因对人肝癌SMMC-7721细胞生物学行为的影响. 世界华人消化杂志. 2014;22:553-562. [DOI] |
| 7. | 孙 雯, 曹 骥, 卢 晓旭, 朱 伶群, 杨 春, 欧 超, 骆 成飘, 李 瑗, 苏 建家. RNAi沉默MCM7基因对人肝癌细胞SMMC-7721裸鼠移植瘤影响研究. 中华肿瘤防治杂志. 2015;22:7-12. |
| 8. | Kanter DM, Bruck I, Kaplan DL. Mcm subunits can assemble into two different active unwinding complexes. J Biol Chem. 2008;283:31172-31182. [PubMed] [DOI] |
| 9. | You Z, Ishimi Y, Masai H, Hanaoka F. Roles of Mcm7 and Mcm4 subunits in the DNA helicase activity of the mouse Mcm4/6/7 complex. J Biol Chem. 2002;277:42471-42479. [PubMed] [DOI] |
| 10. | Bruck I, Perez-Arnaiz P, Colbert MK, Kaplan DL. Insights into the Initiation of Eukaryotic DNA Replication. Nucleus. 2015;6:449-454. [PubMed] [DOI] |
| 11. | Maric M, Maculins T, De Piccoli G, Labib K. Cdc48 and a ubiquitin ligase drive disassembly of the CMG helicase at the end of DNA replication. Science. 2014;346:1253596. [PubMed] [DOI] |
| 12. | Liu J, Tian L, Chen BA, Xia JR. Biological effects of lentivirus-mediated silencing of minichromosome maintenance protein 7 with shRNA on the liver cancer MHCC-97H cells. Int J Clin Exp Med. 2015;8:8433-8441. [PubMed] |
| 13. | Zender L, Villanueva A, Tovar V, Sia D, Chiang DY, Llovet JM. Cancer gene discovery in hepatocellular carcinoma. J Hepatol. 2010;52:921-929. [PubMed] [DOI] |
| 14. | Lu X, Sun W, Tang Y, Zhu L, Li Y, Ou C, Yang C, Su J, Luo C, Hu Y. Identification of key genes in hepatocellular carcinoma and validation of the candidate gene, cdc25a, using gene set enrichment analysis, metaanalysis and crossspecies comparison. Mol Med Rep. 2016;13:1172-1178. [PubMed] [DOI] |
| 16. | Imoto M, Doki Y, Jiang W, Han EK, Weinstein IB. Effects of cyclin D1 overexpression on G1 progression-related events. Exp Cell Res. 1997;236:173-180. [PubMed] |
| 17. | Xu JM, Wen JM, Zhang M, Lü GL, Wu LZ, Wang WS. [A study of gene amplification and expression of cyclin D1 in hepatocellular carcinoma]. Zhonghua Binglixue Zazhi. 2004;33:26-30. [PubMed] |
| 18. | George J, Lim JS, Jang SJ, Cun Y, Ozretić L, Kong G, Leenders F, Lu X, Fernández-Cuesta L, Bosco G. Comprehensive genomic profiles of small cell lung cancer. Nature. 2015;524:47-53. [PubMed] [DOI] |
| 19. | Vranic S, Marchiò C, Castellano I, Botta C, Scalzo MS, Bender RP, Payan-Gomez C, di Cantogno LV, Gugliotta P, Tondat F. Immunohistochemical and molecular profiling of histologically defined apocrine carcinomas of the breast. Hum Pathol. 2015;46:1350-1359. [PubMed] [DOI] |
| 20. | Sun Q, An G, Liu E, Li Z, Zhang H, Yang Q, Sun F, Ma Y, Xian M, Zhang P. [The clinic and pathologic significance of plasma cell myeloma with CCND1]. Zhonghua Xueyexue Zazhi. 2015;36:775-779. [PubMed] [DOI] |
| 21. | Chan CH, Lee SW, Li CF, Wang J, Yang WL, Wu CY, Wu J, Nakayama KI, Kang HY, Huang HY. Deciphering the transcriptional complex critical for RhoA gene expression and cancer metastasis. Nat Cell Biol. 2010;12:457-467. [PubMed] [DOI] |
| 22. | Nakayama K, Nagahama H, Minamishima YA, Miyake S, Ishida N, Hatakeyama S, Kitagawa M, Iemura S, Natsume T, Nakayama KI. Skp2-mediated degradation of p27 regulates progression into mitosis. Dev Cell. 2004;6:661-672. [PubMed] |
| 23. | Lee SW, Li CF, Jin G, Cai Z, Han F, Chan CH, Yang WL, Li BK, Rezaeian AH, Li HY. Skp2-dependent ubiquitination and activation of LKB1 is essential for cancer cell survival under energy stress. Mol Cell. 2015;57:1022-1033. [PubMed] [DOI] |
| 24. | Ding M, Li X, Qiu T. Combination of multiple gene markers to detect circulating tumor cells in the peripheral blood of patients with non-small cell lung cancer using real-time PCR. Genet Mol Res. 2015;14:13033-13040. [PubMed] [DOI] |
| 25. | Sanada T, Yokoi S, Arii S, Yasui K, Imoto I, Inazawa J. Skp2 overexpression is a p27Kip1-independent predictor of poor prognosis in patients with biliary tract cancers. Cancer Sci. 2004;95:969-976. [PubMed] [DOI] |
| 26. | Hafez MM, Alhoshani AR, Al-Hosaini KA, Alsharari SD, Al Rejaie SS, Sayed-Ahmed MM, Al-Shabanah OA. SKP2/P27Kip1 pathway is associated with Advanced Ovarian Cancer in Saudi Patients. Asian Pac J Cancer Prev. 2015;16:5807-5815. [PubMed] |
| 27. | Bochis OV, Irimie A, Pichler M, Berindan-Neagoe I. The role of Skp2 and its substrate CDKN1B (p27) in colorectal cancer. J Gastrointestin Liver Dis. 2015;24:225-234. [PubMed] [DOI] |
| 28. | Hicke L. Protein regulation by monoubiquitin. Nat Rev Mol Cell Biol. 2001;2:195-201. [PubMed] [DOI] |
| 29. | McBride WH, Iwamoto KS, Syljuasen R, Pervan M, Pajonk F. The role of the ubiquitin/proteasome system in cellular responses to radiation. Oncogene. 2003;22:5755-5773. [PubMed] [DOI] |
| 30. | Zhurinsky J, Shtutman M, Ben-Ze'ev A. Plakoglobin and beta-catenin: protein interactions, regulation and biological roles. J Cell Sci. 2000;113:3127-3139. [PubMed] |
| 31. | Endo K, Ueda T, Ueyama J, Ohta T, Terada T. Immunoreactive E-cadherin, alpha-catenin, beta-catenin, and gamma-catenin proteins in hepatocellular carcinoma: relationships with tumor grade, clinicopathologic parameters, and patients' survival. Hum Pathol. 2000;31:558-565. [PubMed] [DOI] |
| 32. | Salomon D, Sacco PA, Roy SG, Simcha I, Johnson KR, Wheelock MJ, Ben-Ze'ev A. Regulation of beta-catenin levels and localization by overexpression of plakoglobin and inhibition of the ubiquitin-proteasome system. J Cell Biol. 1997;139:1325-1335. [PubMed] [DOI] |