修回日期: 2013-10-15
接受日期: 2013-10-17
在线出版日期: 2013-11-18
目的: 越来越多的研究发现粪便中基因异常甲基化可作为生物标志来检测结直肠肿瘤. 为了系统评价粪便甲基化基因作为生物标志物来检测结直肠肿瘤的可行性.
方法: 以"colorectal cancer/colorectal adenoma/colorectal polyps"、"methylation"、"stool/fecal DNA"作为关键词, 检索PubMed、Web of Kowledge和OVID(On-line Visual Display Unit Interrogation of Databases)数据库纳入研究甲基化基因作为生物标志物来检测结直肠癌(colorectal cancer, CRC)或腺瘤的相关文献. 该荟萃分析是使用了敏感性, 特异性和95%CI作为影响测量结果, 采用Statal1.0软件进行统计学分析.
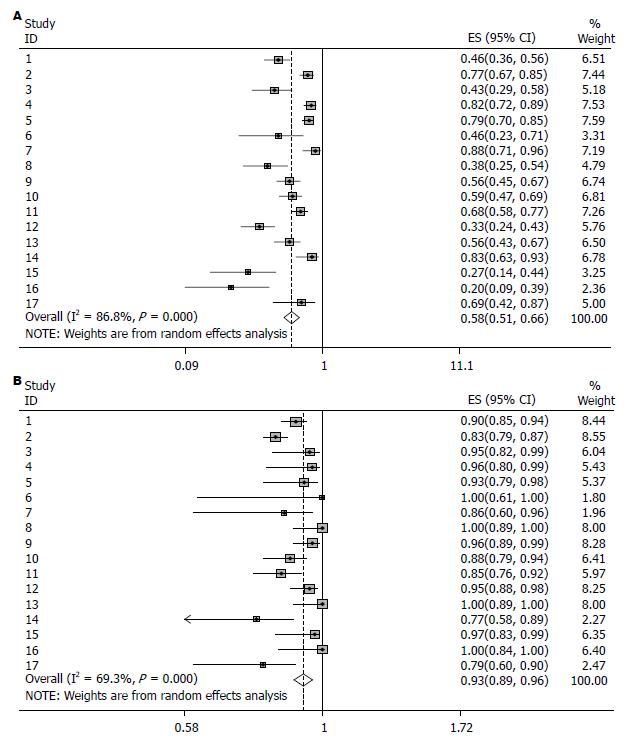
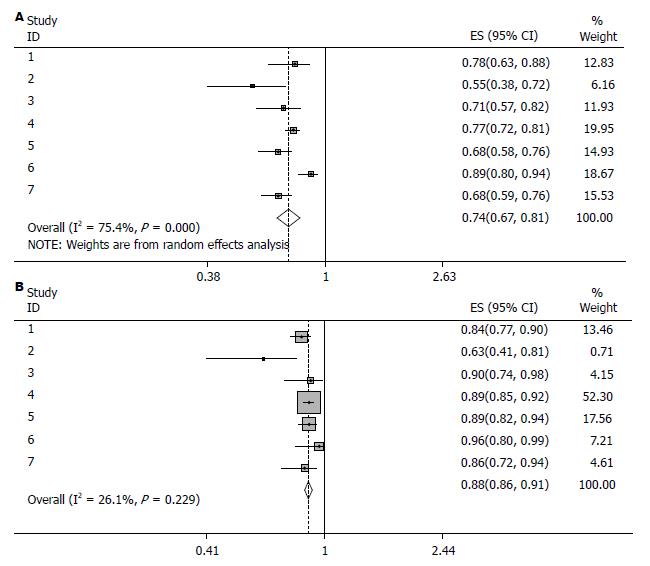
结果: 一共有24个研究, 3555例患者被纳入. 统计结果显示粪便DNA中检测单基因甲基化筛查结直肠肿瘤(CRC和腺瘤)的敏感性和特异性分别为0.58(95%CI: 0.51-0.66)和0.93(95%CI: 0.89-0.96), 联合检测多基因甲基化筛查结直肠肿瘤的敏感性和特异性分别为0.79(95%CI: 0.67-0.81)和0.88(95%CI: 0.86-0.91).
结论: 通过本次荟萃分析, 我们可以发现粪便基因甲基化检测筛查CRC具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景, 而且联合检测多基因甲基化方法筛查结直肠肿瘤优于单基因检测方法.
核心提示: 本次荟萃分析发现粪便基因甲基化检测筛查结直肠癌(colorectal cancer, CRC)具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景, 而且, 联合检测多基因甲基化方法筛查结直肠肿瘤优于单基因检测方法.
引文著录: 陆宏娜, 张谢, 王丹萍, 董显文, 黄志刚. 粪便中基因甲基化检测筛查结直肠肿瘤的荟萃分析. 世界华人消化杂志 2013; 21(32): 3585-3591
Revised: October 15, 2013
Accepted: October 17, 2013
Published online: November 18, 2013
AIM: To systemically evaluate the feasibility of using methylated genes as biomarkers for the detection of colorectal neoplasia.
METHODS: We searched PubMed, the Web of Science and On-line Visual Display Unit Interrogation of Data Bases (OVID) with the key words "colorectal cancer/colorectal adenoma/colorectal polyps", "methylation", "stool/fecal DNA" for studies that used hypermethylated genes as biomarkers for the detection of colorectal cancer (CRC) or adenomas. This meta-analysis used the sensitivity, specificity and 95% confidence intervals (95%CI) as effect measurements, and the Stata statistical software (version 11.0) was used to perform statistical analyses.
RESULTS: A total of 24 studies including 3555 patients were selected into the final analysis. The sensitivity and specificity of detecting single methylated gene in fecal DNA for the detection of colorectal neoplasia (CRC and adenomas) were 0.58 (95%CI: 0.51-0.66) and 0.93 (95%CI: 0.89-0.96), respectively. The sensitivity and specificity of detecting multiple methylated genes in fecal DNA for the detection of colorectal neoplasia were 0.79 (95%CI: 0.67-0.81) and 0.88 (95%CI: 0.86-0.91), respectively.
CONCLUSION: Analysis of stool DNA methylation has a reasonable sensitivity and specificity for the detection of CRC, and may provide a promising and noninvasive screening method for colorectal cancer. Combined detection of multiple methylated genes in fecal DNA is superior to the single gene detection method for colorectal neoplasia screening.
- Citation: Lu HN, Zhang X, Wang DP, Dong XW, Huang ZG. Detection of fecal DNA methylation for colorectal neoplasia screening: A meta-analysis. Shijie Huaren Xiaohua Zazhi 2013; 21(32): 3585-3591
- URL: https://www.wjgnet.com/1009-3079/full/v21/i32/3585.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i32.3585
结直肠癌(colorectal cancer, CRC)是全球发病率和死亡率都排名第3的恶性肿瘤[1]. 在过去的二十年中, 我国的CRC的发病率和死亡率迅速增加, 成为癌症死亡的第5大原因[2]. 由于CRC早期临床表现不典型, 因此对CRC的早期筛查有助于降低CRC的发病率及死亡率. 目前已有很多筛查CRC的方法, 包括粪便潜血试验(fecal occult-blood test, FOBT)、软式乙状结肠镜、钡剂灌肠和结肠镜检查. 其中, 结肠镜检查及粪便潜血试验是使用最频繁的, 前者是敏感度高, 但存在需清洁灌肠、痛苦、检查时间较长、有一定并发症等缺点, 而后者虽然相对简单易用, 但敏感性和特异性不高[3]. 粪便DNA检测相对于这些筛选方式的优势在于他是非侵入性的, 肿瘤细胞不断脱落进入肠腔, 并与粪便混合, 我们可以从粪便标本中分离出人类的DNA, 并进行生物标志物分析, 在理论上, 可用于CRC或腺瘤的早期检测.
在过去的几年中, 越来越多的研究发现粪便标本中异常甲基化的基因可以作为诊断标志物来检测CRC或腺瘤. 但由于各个研究所选择的基因类型及基因甲基化检测方法不同, 无法评估粪便DNA基因甲基化检测筛查CRC的精确性, 因此, 对之前研究检测粪便DNA生物标志物的敏感性和特异性进行荟萃分析可为这些测试的诊断价值提供更精确的估计. 此次研究的目的是利用荟萃分析, 以明确检测粪便DNA甲基化诊断CRC或腺瘤的准确性.
以"colorectal cancer/colorectal adenoma/colorectal polyps "、"methylation"、"stool/fecal DNA"作为检索词, 检索PubMed、Web of Science和OVID(On-line Visual Display Unit Interrogation of Databases)数据库, 检索从建库至2012-12为止的英文文献, 纳入使用粪便标本中一个或多个甲基化基因作为生物标志物来检测CRC或腺瘤的相关文献.
1.2.1 纳入标准: (1)各研究假设和研究方法相似, 文献提供病例入选标准, 有严格的对照组; (2)原始研究的病例组是组织病理检测确诊的结直肠癌或腺瘤患者, 且病例组样本为CRC或腺瘤患者粪便; (3)文献中至少有一个或多个基因甲基化研究, 并提供敏感性及特异性.
1.2.2 排除标准: (1)重复报告, 存在研究设计缺陷, 质量差; (2)原始资料数据不完整, 不提供或无法计算敏感性及特异性的文献; (3)非病例对照研究, 文献综述等.
统计学处理 (1)数据收集: 收集各个研究中检测的敏感性, 特异性, 95%CI; (2)异质性检验: 用χ2评估是否存在统计学异质性, I2检验评估异质性大小, 若P>0.05, I2<50%为异质性不显著, P<0.05, I2>50%为异质性显著; (3)荟萃分析: 若数据间异质性不显著, 则采用固定效应模型, 否则采用随机效应模型; (4)敏感性分析: 应用固定效应模型和随机效应模型分别进行荟萃分析, 两种模型结果的一致程度可在一定程度上反映合并结果的可靠性; (5)评估潜在偏倚: 采用漏斗图分析法, Begg的秩相关检验法, 如果Z>1.98, P<0.05, 提示可能存在发表偏倚; 如果Z<1.98, P>0.05, 则认为没有发表偏倚; (6)所有数据的统计分析使用STATA11.0软件完成.
共检索到215篇相关文献, 其中有189篇文献不符合上述的纳入标准, 一篇数据重复, 另一篇的原始资料数据不完整, 无法计算敏感性及特异性. 经过筛选, 共有24篇文献, 3555例患者被纳入. 研究源于11个国家或地区(包括美国, 中国、德国、荷兰、西班牙、日本、加拿大、伊朗、香港、奥地利和韩国), 发表在2004-2011年间. 研究的样本量范围为41-639例. 其中8项仅研究CRC患者粪便的基因甲基化状态[4-11], 3项仅研究腺瘤[12-14], 13项研究了CRC和腺瘤患者[15-27]. 24篇文献中有17项研究仅用单个甲基化基因作为诊断标志物进行研究[4-6,8-12,14-19,21-23], 另外7项研究使用多基因甲基化进行研究[7,13,20,24-27]. 研究的基因甲基化检测方法也有所不同, 11项研究使用甲基化特异性PCR(methylation-specific PCR, MSP)[5-7,11,13,16,18-20,26,27], 6项使用定量甲基化特异性PCR(quantitative methylation specific PCR, qMSP)[8,9,14,21-23], 3项使用甲基化荧光定量PCR(MethyLight)[4,12,17], 1项使用结合重亚硫酸盐的限制性内切酶法(combined bisulfite restriction analysis, COBRA)[25], 1项使用溶解曲线分析法(melting curve analysis, MC)[10], 1项使用数字PCR-流式技术(methyl-BEAMing)[5], 1项研究中使用QuARTS技术(quantitative allele-specific real-time target and signal amplification)[24]. 研究对象的基本情况如表1.
| 编号 | 入选研究 | 国家 | 结直肠癌 | 腺瘤 | 正常 | 方法 | 基因 | |||
| 阳性 | 阴性 | 阳性 | 阴性 | 阳性 | 阴性 | |||||
| 1 | Chen等[5]2005 | USA | 43 | 51 | 20 | 178 | MSP | vimentin | ||
| 2 | Itzkowitz等[6]2008 | USA | 63 | 19 | 62 | 301 | MSP | vimentin | ||
| 3 | Itzkowitz等[7]2007 | USA | 9 | 31 | 19 | 103 | MSP | vimentin, HLTF | ||
| 4 | Li等[15]2009 | USA | 9 | 13 | 9 | 11 | 2 | 36 | Methyl-BEAMing | vimentin |
| 5 | Huang等[26]2007 | China | 49 | 3 | 11 | 10 | 1 | 23 | MSP | SFRP2 |
| 6 | Wang[17]2008 | China | 60 | 9 | 21 | 13 | 2 | 28 | MethyLight | SFRP2 |
| 7 | Oberwalder等[12]2007 | Austria | 6 | 7 | 0 | 6 | MethyLight | SFRP2 | ||
| 8 | Zhang等[18]2007 | Germany | 16 | 3 | 7 | 0 | 2 | 12 | MSP | SFRP1 |
| 9 | Lenhard等[19]2005 | Germany | 11 | 15 | 4 | 9 | 0 | 32 | MSP | HIC1 |
| 10 | Petko等[13]2005 | USA | 16 | 13 | 7 | 12 | MSP | CDKN2, MGMT, MLH1 | ||
| 11 | Leung等[20]2007 | Hongkong | 15 | 5 | 17 | 8 | 3 | 27 | MSP | APC, ATM, hMLH1, SFRP2, HLTF, MGMT |
| 12 | Melotte等2009[8] | Netherlands | 42 | 33 | 3 | 72 | qMSP | NDRG4 | ||
| 13 | Hellebrekers等2009[9] | Netherlands | 44 | 31 | 9 | 66 | qMSP | GATA4 | ||
| 14 | Glöckner等2009[21] | USA | 59 | 14 | 4 | 15 | 11 | 64 | qMSP | TFPI2 |
| 15 | Kim等[22]2009 | USA | 26 | 43 | 2 | 14 | 4 | 77 | qMSP | OSMR |
| 16 | Bosch等[23]2011 | Netherlands | 29 | 15 | 6 | 13 | 0 | 30 | qMSP | PHACTR3 |
| 17 | Ahlquist等[24]2011 | Canada | 214 | 38 | 51 | 43 | 32 | 261 | QuARTS | BMP3, NDRG4, VIMETIN, TFPI2 |
| 18 | Muller等[4]2004 | Austria | 19 | 4 | 6 | 20 | MethyLight | SFRP2 | ||
| 19 | Nagasaka等[25]2009 | Japan | 63 | 21 | 12 | 15 | 12 | 101 | COBRA | RASSF2, SFRP2 |
| 20 | Huang等[16]2007 | China | 50 | 2 | 15 | 6 | 1 | 23 | MSP | SFRP2, HPP1, MGMT |
| 21 | Baek等[27]2009 | South Korea | 45 | 15 | 31 | 21 | 5 | 32 | MSP | MGMT, hMLH1, Vimentin |
| 22 | Mayor等[10]2009 | Spain | 8 | 22 | 1 | 29 | Melting Curve Analysis(MC) | EN1 | ||
| 23 | Abbaszadegan等[11]2007 | Iran | 5 | 20 | 0 | 20 | MSP | p16 | ||
| 24 | Ausch等[14]2009 | USA | 9 | 4 | 6 | 22 | qMSP | IGTA4 | ||
荟萃分析显示, 粪便DNA中检测单基因甲基化筛查CRC、腺瘤、结直肠肿瘤的敏感性和筛查CRC, 结直肠肿瘤的特异性以及联合检测多基因筛查CRC, 结直肠肿瘤敏感性的异质性检验结果为P<0.05, I2>50%, 提示研究间异质性显著, 采用随机效应模型进行Meta分析, 其余数据分析结果为P>0.05, I2<50%, 提示研究间异质性不显著, 采用固定效应模型.
统计结果显示粪便DNA中检测单基因甲基化筛查CRC、腺瘤、结直肠肿瘤(CRC和腺瘤)的总敏感性分别为0.62(95%CI: 0.53-0.71)、0.48(95%CI: 0.35-0.65)、0.58(95%CI: 0.51-0.66); 特异性分别0.93(95%CI: 0.90-0.96)、0.96(95%CI: 0.93-0.98)、0.93(95%CI: 0.89-0.96)(图1).
联合检测多基因甲基化筛查CRC、腺瘤、结直肠肿瘤的总敏感性分别为0.82(95%CI: 0.75-0.90)、0.58(95%CI: 0.52-0.64)、0.79(95%CI: 0.67-0.81); 特异性分别0.89(95%CI: 0.86-0.91)、0.89(95%CI: 0.86-0.92)、0.88(95%CI: 0.86-0.91)(图2).
我们还对结直肠肿瘤患者粪便样本中单基因及多基因甲基化检测的敏感性和特异性进行了偏倚评估. 单基因甲基化检测敏感性的Begg的秩相关检验法显示Z = 3.01>1.98, P<0.05提示可能存在发表偏倚, 特异性的Begg的秩相关检验法显示Z = 0.91<1.98, P>0.05,认为没有发表偏倚; 多基因甲基化检测敏感性的Begg的秩相关检验法显示Z = 0.90>1.98, P>0.05提示没有发表偏倚, 特异性的Begg的秩相关检验法显示Z = 0.90<1.98, P>0.05,认为没有发表偏倚.
尽管对50岁以上的人进行CRC的筛查, 可以降低CRC的发病率[1], 但只有少数人能够接受以结肠镜检查为基础的筛查. FOBT是目前使用最广泛的非侵入性的大肠癌筛查方法, 然而, 研究表明FOBT的灵敏度大概只有14%, 部分是因为无出血的肿瘤未能被发现[28]. 因此, 我们需要寻找一种非侵入性的高敏感性和特异性的方法来筛查结直肠肿瘤.
从癌症的病因学角度分析, 良性肿瘤如息肉发展到恶性肿瘤是遗传学和表遗传学改变积累的过程. 表观遗传学变异是在细胞分裂过程中可遗传的不涉及DNA序列改变的基因组修饰作用[29]. 其中基因启动子上的甲基化可以导致基因失活, 且往往发生在肿瘤早期, 其检测有助于肿瘤的早期诊断及评价肿瘤的发展、转移及预后, 对指导临床工作有重要意义. 由于肿瘤细胞不断脱落进入肠腔并与粪便混合, 因此在粪便DNA中检测基因遗传学或表观遗传学改变可以作为筛查方法来早期发现结直肠肿瘤[30].
在过去的研究中, 很多高甲基化基因已经被用作生物标志物来检测CRC或腺瘤, 包括P16、MutL同源蛋白1(MutL homolog 1, MLH1)、O型6-甲基鸟嘌呤-DNA甲基转移酶(O-6-methylguanine-DNA methyltransferase, MGMT)、分泌型卷曲相关蛋白2(secreted frizzled-related protein 2, SFRP2)、N-Myc下游调节基因4(N-myc downstream-regulated gene 4, NDRG4)和波形蛋白(vimentin)等[6,8,11,20,26], 但这些研究的甲基化检测的敏感性和特异性差异很大, 例如联合检测SFRP2、HPP1、MGMT基因甲基化的敏感性高达96%[26], 而检测P16的敏感性却只有20%[11]. 单基因甲基化检测筛查CRC和腺瘤的敏感性分别为20%-94%和13%-100%, 特异性为77%-100%[4-6,8-12,14-19,21-23]; 联合检测多基因甲基化筛查CRC和腺瘤的敏感性分别为75%-96%和44%-71%, 特异性为63%-96%[7,13,20,24-27], Ahlquist等[24]在研究粪便DNA的骨形态发生蛋白3(bone morphogenetic protein 3, BMP3)、NDRG4、vimentin、组织因子途径抑制因子2(tissue factor pathway inhibitor 2, TFPI2)4个基因的甲基化状态中发现, 联合检测这4个基因的敏感性比单独各项检测指标高.
本荟萃分析显示, 粪便单基因及多基因甲基化检测筛查CRC的敏感性分别为62%和82%, 特异性分别为93%和89%, 这些结果表明, 检测粪便基因甲基化发现CRC具有很高的精确度, 检测腺瘤的敏感性分别为48%和58%, 特异性分别为96%和89%, 粪便基因甲基化检测方法对大肠癌的敏感性大于腺瘤. 粪便DNA中联合检测多基因甲基化筛查结直肠肿瘤的总敏感性(79%)明显高于单基因甲基化检测的敏感性(58%), 联合检测多基因甲基化方法筛查结直肠肿瘤优于单基因检测方法. 此研究表明, 与传统的FOBT相比, 粪便基因甲基化检测能够更加精确诊断CRC及腺瘤, 虽然此方法的敏感性不如诊断CRC的"金标准"-结肠镜, 但是, 粪便基因甲基化检测方法易于取样, 无需肠道准备且无痛苦, 更容易让患者接受.
统计学分析结果显示, 结直肠肿瘤患者粪便样本中单基因甲基化检测的敏感性存在发表偏倚, 这可能与纳入文献所研究的患者的种族, 性别, 样品例数, 研究人员技术差异, 所研究的基因以及检测基因甲基化的方法不同有关. 通过本次荟萃分析, 我们可以发现粪便基因甲基化检测筛查CRC具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景. 但目前, 尚缺乏理想的粪便甲基化基因标志物, 能够单独用于结直肠肿瘤的筛查中. 其次, 甲基化检测的方法学及灵敏度需要进一步改进和标准化, 将有助于粪便甲基化检测能够尽早应用于临床.
结直肠癌是目前发病率和死亡率都排名第3的恶性肿瘤, 绝大部分来自于癌前疾病-结肠腺瘤, 结肠腺瘤到癌变过程大约需5-15年, 因此, 针对普通人群的结直肠癌筛查可显著降低结直肠癌死亡率.
吴建兵, 教授, 主任医师, 南昌大学第二附属医院肿瘤科
通过本次荟萃分析, 我们可以发现粪便基因甲基化检测筛查CRC具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景, 而且, 联合检测多基因甲基化方法筛查结直肠肿瘤优于单基因检测方法.
Muller等检测结肠癌患者粪便中SFRP2基因甲基化情况, 发现与对照组相比灵敏性为90%, 特异性77%. Lenhard等报道在结肠癌及大肠腺瘤患者中高甲基化HIC1率分别为42%和31%. 对于联合检测多个基因甲基化研究, Beak等联合检测MGMT、hMLH1、vimentin基因甲基化, 发现对结直肠癌检测阳性率可达75%, 结直肠腺瘤检测率为59.6%.
本文综合分析了近十年国内外关于粪便中基因甲基化检测筛查结直肠肿瘤的研究, 并比较单基因检测及多基因检测筛查结直肠肿瘤的敏感性和特异性, 我们可以发现粪便基因甲基化检测筛查CRC具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景.
通过本次荟萃分析, 我们可以发现粪便基因甲基化检测筛查CRC具有较高的敏感性和特异性, 具有作为无创性CRC筛查方法的前景. 其次, 甲基化检测的方法学及灵敏度需要进一步改进和标准化, 将有助于粪便甲基化检测能够尽早应用于临床.
DNA甲基化: 是指生物体在DNA甲基转移酶(DMT) 的催化下, 以s-腺苷甲硫氨酸(SAM)为甲基供体, 将甲基转移到CpG胞嘧啶的5'碳原子, 形成5-甲基胞嘧啶的过程;
甲基化特异性PCR: 是一种特异位点甲基化检测技术. 其基本原理是用亚硫酸氢钠处理基因组DNA, 未甲基化的胞嘧啶变成尿嘧啶, 而甲基化的胞嘧啶不变. 因此从理论上讲, 用不同的引物做PCR, 即可检测出这种差异, 从而确定基因有无CpG岛甲基化.
粪便中基因甲基化检测筛查结直肠肿瘤的荟萃分析一文分析数据科学, 得出结论可靠, 是一篇较好的荟萃分析文章.
编辑: 郭鹏 电编:闫晋利
| 1. | Jemal A, Murray T, Ward E, Samuels A, Tiwari RC, Ghafoor A, Feuer EJ, Thun MJ. Cancer statistics, 2005. CA Cancer J Clin. 2005;55:10-30. [PubMed] [DOI] |
| 2. | Sung JJ, Lau JY, Goh KL, Leung WK. Increasing incidence of colorectal cancer in Asia: implications for screening. Lancet Oncol. 2005;6:871-876. [PubMed] [DOI] |
| 3. | Jenkinson F, Steele RJ. Colorectal cancer screening - methodology. Surgeon. 2010;8:164-171. [PubMed] [DOI] |
| 4. | Müller HM, Oberwalder M, Fiegl H, Morandell M, Goebel G, Zitt M, Mühlthaler M, Ofner D, Margreiter R, Widschwendter M. Methylation changes in faecal DNA: a marker for colorectal cancer screening? Lancet. 2004;363:1283-1285. [PubMed] [DOI] |
| 5. | Chen WD, Han ZJ, Skoletsky J, Olson J, Sah J, Myeroff L, Platzer P, Lu S, Dawson D, Willis J. Detection in fecal DNA of colon cancer-specific methylation of the nonexpressed vimentin gene. J Natl Cancer Inst. 2005;97:1124-1132. [PubMed] [DOI] |
| 6. | Itzkowitz S, Brand R, Jandorf L, Durkee K, Millholland J, Rabeneck L, Schroy PC, Sontag S, Johnson D, Markowitz S. A simplified, noninvasive stool DNA test for colorectal cancer detection. Am J Gastroenterol. 2008;103:2862-2870. [PubMed] [DOI] |
| 7. | Itzkowitz SH, Jandorf L, Brand R, Rabeneck L, Schroy PC, Sontag S, Johnson D, Skoletsky J, Durkee K, Markowitz S. Improved fecal DNA test for colorectal cancer screening. Clin Gastroenterol Hepatol. 2007;5:111-117. [PubMed] [DOI] |
| 8. | Melotte V, Lentjes MH, van den Bosch SM, Hellebrekers DM, de Hoon JP, Wouters KA, Daenen KL, Partouns-Hendriks IE, Stessels F, Louwagie J. N-Myc downstream-regulated gene 4 (NDRG4): a candidate tumor suppressor gene and potential biomarker for colorectal cancer. J Natl Cancer Inst. 2009;101:916-927. [PubMed] [DOI] |
| 9. | Hellebrekers DM, Lentjes MH, van den Bosch SM, Melotte V, Wouters KA, Daenen KL, Smits KM, Akiyama Y, Yuasa Y, Sanduleanu S. GATA4 and GATA5 are potential tumor suppressors and biomarkers in colorectal cancer. Clin Cancer Res. 2009;15:3990-3997. [PubMed] [DOI] |
| 10. | Mayor R, Casadomé L, Azuara D, Moreno V, Clark SJ, Capellà G, Peinado MA. Long-range epigenetic silencing at 2q14.2 affects most human colorectal cancers and may have application as a non-invasive biomarker of disease. Br J Cancer. 2009;100:1534-1539. [PubMed] [DOI] |
| 11. | Abbaszadegan MR, Tavasoli A, Velayati A, Sima HR, Vosooghinia H, Farzadnia M, Asadzedeh H, Gholamin M, Dadkhah E, Aarabi A. Stool-based DNA testing, a new noninvasive method for colorectal cancer screening, the first report from Iran. World J Gastroenterol. 2007;13:1528-1533. [PubMed] |
| 12. | Oberwalder M, Zitt M, Wöntner C, Fiegl H, Goebel G, Zitt M, Köhle O, Mühlmann G, Ofner D, Margreiter R. SFRP2 methylation in fecal DNA--a marker for colorectal polyps. Int J Colorectal Dis. 2008;23:15-19. [PubMed] [DOI] |
| 13. | Petko Z, Ghiassi M, Shuber A, Gorham J, Smalley W, Washington MK, Schultenover S, Gautam S, Markowitz SD, Grady WM. Aberrantly methylated CDKN2A, MGMT, and MLH1 in colon polyps and in fecal DNA from patients with colorectal polyps. Clin Cancer Res. 2005;11:1203-1209. [PubMed] |
| 14. | Ausch C, Kim YH, Tsuchiya KD, Dzieciatkowski S, Washington MK, Paraskeva C, Radich J, Grady WM. Comparative analysis of PCR-based biomarker assay methods for colorectal polyp detection from fecal DNA. Clin Chem. 2009;55:1559-1563. [PubMed] [DOI] |
| 15. | Li M, Chen WD, Papadopoulos N, Goodman SN, Bjerregaard NC, Laurberg S, Levin B, Juhl H, Arber N, Moinova H. Sensitive digital quantification of DNA methylation in clinical samples. Nat Biotechnol. 2009;27:858-863. [PubMed] [DOI] |
| 16. | Huang Z, Li L, Wang J. Hypermethylation of SFRP2 as a potential marker for stool-based detection of colorectal cancer and precancerous lesions. Dig Dis Sci. 2007;52:2287-2291. [PubMed] [DOI] |
| 17. | Wang DR, Tang D. Hypermethylated SFRP2 gene in fecal DNA is a high potential biomarker for colorectal cancer noninvasive screening. World J Gastroenterol. 2008;14:524-531. [PubMed] [DOI] |
| 18. | Zhang W, Bauer M, Croner RS, Pelz JO, Lodygin D, Hermeking H, Stürzl M, Hohenberger W, Matzel KE. DNA stool test for colorectal cancer: hypermethylation of the secreted frizzled-related protein-1 gene. Dis Colon Rectum. 2007;50:1618-1626; discussion 1626-1627. [PubMed] |
| 19. | Lenhard K, Bommer GT, Asutay S, Schauer R, Brabletz T, Göke B, Lamerz R, Kolligs FT. Analysis of promoter methylation in stool: a novel method for the detection of colorectal cancer. Clin Gastroenterol Hepatol. 2005;3:142-149. [PubMed] [DOI] |
| 20. | Leung WK, To KF, Man EP, Chan MW, Hui AJ, Ng SS, Lau JY, Sung JJ. Detection of hypermethylated DNA or cyclooxygenase-2 messenger RNA in fecal samples of patients with colorectal cancer or polyps. Am J Gastroenterol. 2007;102:1070-1076. [PubMed] [DOI] |
| 21. | Glöckner SC, Dhir M, Yi JM, McGarvey KE, Van Neste L, Louwagie J, Chan TA, Kleeberger W, de Bruïne AP, Smits KM. Methylation of TFPI2 in stool DNA: a potential novel biomarker for the detection of colorectal cancer. Cancer Res. 2009;69:4691-4699. [PubMed] [DOI] |
| 22. | Kim MS, Louwagie J, Carvalho B, Terhaar Sive Droste JS, Park HL, Chae YK, Yamashita K, Liu J, Ostrow KL, Ling S. Promoter DNA methylation of oncostatin m receptor-beta as a novel diagnostic and therapeutic marker in colon cancer. PLoS One. 2009;4:e6555. [PubMed] [DOI] |
| 23. | Bosch LJ, Oort FA, Neerincx M, Khalid-de Bakker CA, Terhaar sive Droste JS, Melotte V, Jonkers DM, Masclee AA, Mongera S, Grooteclaes M. DNA methylation of phosphatase and actin regulator 3 detects colorectal cancer in stool and complements FIT. Cancer Prev Res (Phila). 2012;5:464-472. [PubMed] [DOI] |
| 24. | Ahlquist DA, Zou H, Domanico M, Mahoney DW, Yab TC, Taylor WR, Butz ML, Thibodeau SN, Rabeneck L, Paszat LF. Next-generation stool DNA test accurately detects colorectal cancer and large adenomas. Gastroenterology. 2012;142:248-256; quiz e255-256. [PubMed] [DOI] |
| 25. | Nagasaka T, Tanaka N, Cullings HM, Sun DS, Sasamoto H, Uchida T, Koi M, Nishida N, Naomoto Y, Boland CR. Analysis of fecal DNA methylation to detect gastrointestinal neoplasia. J Natl Cancer Inst. 2009;101:1244-1258. [PubMed] [DOI] |
| 26. | Huang ZH, Li LH, Yang F, Wang JF. Detection of aberrant methylation in fecal DNA as a molecular screening tool for colorectal cancer and precancerous lesions. World J Gastroenterol. 2007;13:950-954. [PubMed] |
| 27. | Baek YH, Chang E, Kim YJ, Kim BK, Sohn JH, Park DI. Stool methylation-specific polymerase chain reaction assay for the detection of colorectal neoplasia in Korean patients. Dis Colon Rectum. 2009;52:1452-1459; discussion 1459-1463. [PubMed] [DOI] |
| 28. | Imperiale TF, Ransohoff DF, Itzkowitz SH, Turnbull BA, Ross ME. Fecal DNA versus fecal occult blood for colorectal-cancer screening in an average-risk population. N Engl J Med. 2004;351:2704-2714. [PubMed] [DOI] |
| 29. | Kim MS, Lee J, Sidransky D. DNA methylation markers in colorectal cancer. Cancer Metastasis Rev. 2010;29:181-206. [PubMed] [DOI] |
| 30. | Ahlquist DA, Shuber AP. Stool screening for colorectal cancer: evolution from occult blood to molecular markers. Clin Chim Acta. 2002;315:157-168. [PubMed] [DOI] |