修回日期: 2013-04-01
接受日期: 2013-04-12
在线出版日期: 2013-05-08
目的: 筛选与胰腺癌干细胞相关的差异表达基因, 并进行生物信息学分析.
方法: 以MIA-PaCa2(TIChigh)与BxPc-3(TIClow)为研究胰腺癌干细胞的工具细胞制备总RNA, 经质量鉴定后进行荧光标记; 采用Agilent人全基因4*44K芯片进行杂交实验, 获得mRNA表达谱; 以Gene Spring Software 11.0软件和Quantile算法分析芯片实验数据, 筛选与胰腺癌干细胞相关的差异mRNAs; 基于Gene Ontology数据库进行GO注释, 基于KEGG数据库进行Pathway注释.
结果: 获得符合基因芯片实验质量标准的RNA样品, 基因芯片杂交及数据分析得到差异表达mRNAs 7059个(P<0.05, fold change≥2), 其中MIA-PaCa2(TIChigh)中上调表达3440个, 下调表达3619个. GO注释显示他们主要涉及多胺代谢过程、含碱基的小分子相互转换、组蛋白H4乙酰化、细胞分裂、细胞周期、凋亡、Toll样受体信号转导通路等. Pathway注释主要涉及癌症的途径、内吞作用、O-糖基化生物合成、嘧啶代谢、谷胱甘肽代谢等. 信号通路调控网络图显示多条信号通路存在交叉对话.
结论: 筛选出的差异表达基因可能参与胰腺癌干细胞的发生发展, 参与不同信号通路的调控, 并有可能成为胰腺癌新的治疗靶点.
核心提示: 本文通过通路分析, 发现差异表达的基因显著性影响许多信号通路, 特别是涉及癌症的途径、嘧啶代谢. 通过信号通路调控网络图分析, 显示上述信号通路存在交叉对话, 这有可能从整体角度揭示胰腺癌干细胞的发生与调控机制.
引文著录: 江建新, 高珊, 王敏, 李旭, 孙诚谊. 胰腺癌干细胞差异基因的表达及生物信息学分析. 世界华人消化杂志 2013; 21(13): 1218-1225
Revised: April 1, 2013
Accepted: April 12, 2013
Published online: May 8, 2013
AIM: To identify differentially expressed genes in pancreatic cancer stem cells and to analyze them by bioinformatics.
METHODS: MIA-PaCa2 (TIChigh) and BxPc-3 (TIClow) were used as tool cells in this study. Total RNA was prepared from MIA-PaCa2 (TIChigh) and BxPc-3 (TIClow) cells. After quality identification and fluorescent labeling, RNA samples were hybridized with the Agilent human gene expression microarrays. Raw data were normalized using Quantile algorithm and Gene Spring Software 11.0. Gene Ontology database and KEGG database were used for GO and Pathway comments on differentially expressed genes, respectively.
RESULTS: A total of 7059 differentially expressed genes were identified (P < 0.05, fold change ≥ 2), including 3440 up-regulated and 3619 down-regulated genes in MIA-PaCa2 (TIChigh) cells. These differentially expressed genes were mainly involved in polyamine metabolic process, nucleobase-containing small molecule interconversion, histone H4 acetylation, cell division, cell cycle, apoptotic process, and toll-like receptor signaling pathway. GO analysis demonstrated they are involved in cancer, endocytosis, O-glycan biosynthesis, pyrimidine metabolism, and glutathione metabolism. Pathway analysis showed the presence of crosstalk among several signaling pathways.
CONCLUSION: Differentially expressed genes identified in this study may be involved in the regulation of different signaling pathways and the genesis of pancreatic cancer stem cells. They might become new therapeutic targets for pancreatic cancer.
- Citation: Jiang JX, Gao S, Wang M, Li X, Sun CY. Identification and bioinformatic analysis of differentially expressed genes in pancreatic cancer stem cells. Shijie Huaren Xiaohua Zazhi 2013; 21(13): 1218-1225
- URL: https://www.wjgnet.com/1009-3079/full/v21/i13/1218.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i13.1218
胰腺癌是高度恶性、预后最差的恶性肿瘤之一, 5年生存率不到5%[1]. 大多数患者因确诊时伴有局部和/或远处的侵袭、转移而丧失手术时机[2], 即使临床手术根治性切除的胰腺癌患者术后也大多数因出现复发和远处转移预后极差, 其原因为术前就有微转移灶形成及其化疗抵抗[3]. 胰腺癌的早期转移、对传统的治疗抵抗以及较易复发是其致死的关键因素. 肿瘤干细胞理论解释了上述现象. 胰腺癌干细胞研究表明[4]: 在原发肿瘤里存在一个亚群细胞, 这群细胞具有自我更新、分化潜能以及在异位重新成瘤能力. 同时, 这群细胞还具有侵袭转移和化疗抵抗特性. 最新的研究表明: 胰腺癌在形成之前通过上皮-间质转化(epithelial-to-mesenchymal transition, EMT)形成胰腺癌干细胞, 是其早期发生转移, 治疗抵抗, 复发的根本原因[5]. 已有的研究表明: 具有CD44+/CD24-表型的胰腺癌细胞具有肿瘤干细胞特性, 胰腺癌细胞系MIA-PaCa2的CD44+/CD24-表达率95.5%, 而BxPc-3的表达率17.5%. 因此, MIA-PaCa2(TIChigh)与BxPc-3(TIClow)可以直接作为研究胰腺癌干细胞的工具细胞, 不需要进一步富集[6]. 本研究采用miRNA芯片技术, 以MIA-PaCa2(TIChigh)和BxPc-3(TIClow)为研究对象, 筛选胰腺癌干细胞相关的mRNAs, 并对其进行生物信息学分析, 为进一步研究胰腺癌干细胞的发生和调控机制, 寻找新的治疗靶点奠定基础.
人胰腺癌细胞株BxPc-3(TIClow)和MIA-PaCa2(TIChigh)由本室保存; 细胞培养按照文献[7,8]采用DMEM培养基(Gibic公司), 含10%的56 ℃热灭活胎牛血清, 25 mmol/L HEPES, 2 mmol/L L-谷氨酰胺, 青霉素50 U/mL及链霉素50 µg/mL.
1.2.1 RNA抽提、纯化和质检: 采用mirVana™ miRNA Isolation Kit(Ambion, Austin, TX, US) (专用于抽提普通组织、细胞的miRNA)并且根据生产厂商提供的标准操作流程进行样品总的RNA抽提, 抽提所得总RNA经Agilent Bioanalyzer 2100(Agilent technologies, Santa Clara, CA, US)电泳质检合格后备用.
1.2.2 芯片杂交、扫描及数据提取(由上海生物芯片工程有限公司完成): 样品RNA采用Agilent表达谱芯片配套试剂盒, Low Input Quick Amp Labeling Kit, One-Color(Agilent technologies, Santa Clara, CA, US)和标准操作流程对样品总RNA中的mRNA进行放大和标记, 并用RNeasy mini kit(QIAGEN, GmBH, Germany)纯化标记好的cDNA. 按照Agilent表达谱芯片配套提供的杂交标准流程和配套试剂盒(Agilent technologies, Santa Clara, CA, US), 用1.65 μg Cy3标记的cDNA与Agilent人全基因4*44K芯片滚动杂交, 65 ℃, 10 r/min, 滚动杂交17 h. 杂交后的芯片清洗扫描, 洗片所用的试剂为Gene Expression Wash Buffer Kit(Agilent technologies, US), 芯片结果采用Agilent Microarray Scanner(Agilent technologies, US)进行扫描, 软件设置Scan resolution = 5 μm, PMT 100%, 10%, 16 bit. 用Feature Extraction software 10.7(Agilent technologies, US)读取数据, 采用Gene Spring Software 11.0(Agilent technologies, US)进行归一化处理, 所用的算法为Quantile, 杂交信号以数据形式输出. 差异mRNA使用了limma进行显著性水平的计算, 设置参数P<0.05, fold change≥2[Foldchange = Signal MIA-PaCa2(TIChigh)/Signal BxPc-3(TIClow)].
1.2.3 生物信息学分析: (1)功能分析(GO-Analysis): 将上述基因差异表达的mRNA基于Gene Ontology数据库进行GO注释, 得到基因参与的所有的GO. 采用基于超几何分布(hypergeometric distribution)的Fisher exact test进行计算每个GO的显著性水平(P值), 并对多重假设检验的结果进行校正并获得误判率(false discovery rate, FDR), 筛选出差异基因所体现的显著性GO. 显著性筛选的标准: P<0.01. 上调和下调差异基因分别进行显著性功能分析; 然后上调和下调差异基因进行显著性功能富集; (2)通路分析(pathway-analysis): 将上述基因差异表达的mRNA基于基于KEGG数据库进行Pathway注释, 得到差异基因所参与的所有的Pathway. 利用Fisher精确检验计算每个Pathway的显著性水平, 并对多重假设检验的结果进行校正并获得FDR, 筛选出差异基因所显著参与的pathway, 显著性筛选的标准: P<0.05; (3)信号通路调控网络(path-act-network): 基于KEGG数据库对显著pathway之间的关系进行整理, 从宏观方面得到显著性pathway之间的信号转导关系, 然后构建显著性pathway之间的信号转导关系网络. 该网络中Pathway显著性选择标准为P<0.05.
统计学处理 数据用mean±SD表示. 应用SPSS17.0统计软件分析, 基因芯片分析进行t检验.
所抽提的总RNA, 经分光光度计检测A260/280的吸光度, 确定样品的浓度和纯度; 运用Agilent2100系统进行电泳质检. 经测定, RNA的A260/280比值介于2.03-2.09, 18S、28S电泳条带清晰, 28S条带无明显降解; RNA完整性评分(RNA integrity number)介于8.7-9.3, RNA样品平均浓度>60 mg/L, 总RNA质量完好, 结果合格, 可以进行芯片实验.
mRNA芯片杂交代表性图像见图1, 显示荧光信号分布均匀, 信号点饱满, 背景较低, 杂交效果理想. 将芯片扫描图像导入Feature Extraction software 10.7读取数据, 从中共鉴定到41001个mRNAs, 将数据导人Gene Spring Software 11.0分析, 得到差异表达mRNAs 7059个(P<0.05, fold change≥2), 其中MIA-PaCa2(TIChigh)中上调表达3440个, 下调表达3619个. 其中部分涉及EMT相关差异表达基因见表1.
| GeneSymbol | P值 | 改变倍数 |
| S100A4 | 0.0004723 | 161.8347 |
| VIM | 2.75E-05 | 59.54529 |
| SNAI1 | 3.16E-05 | 18.09616 |
| CDH1 | 2.64E-05 | 0.001399 |
| ACTA2 | 0.25197 | 0.64338 |
| TJP1 | 9.83E-05 | 0.268177 |
| CTNNB1 | 2.41E-05 | 0.406122 |
| NFYA | 3.67E-06 | 1.954545 |
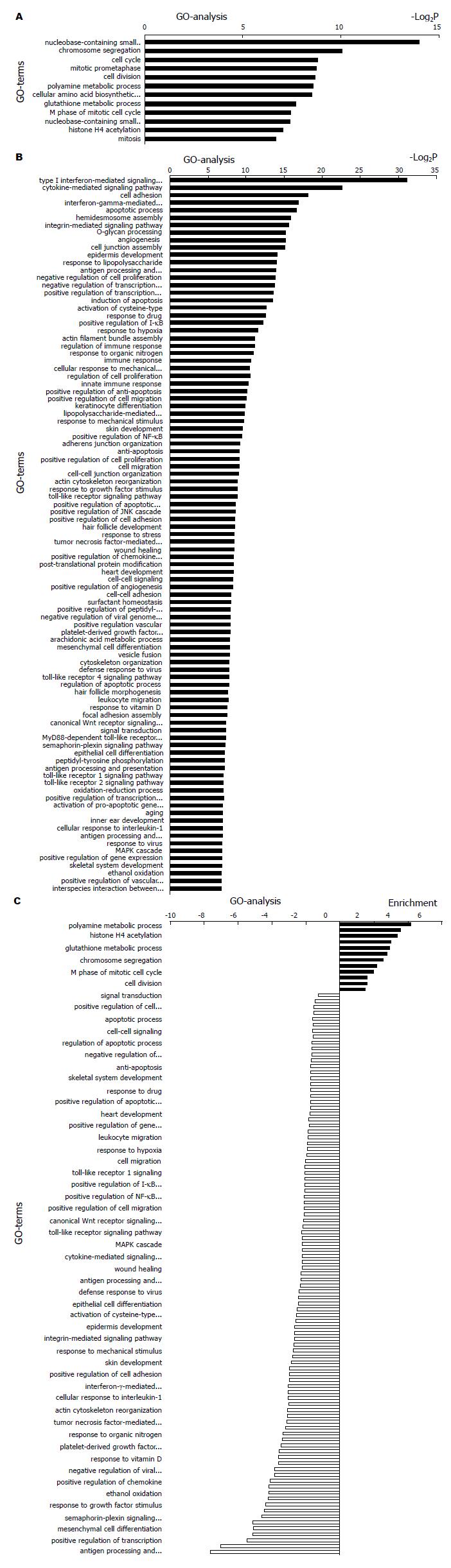
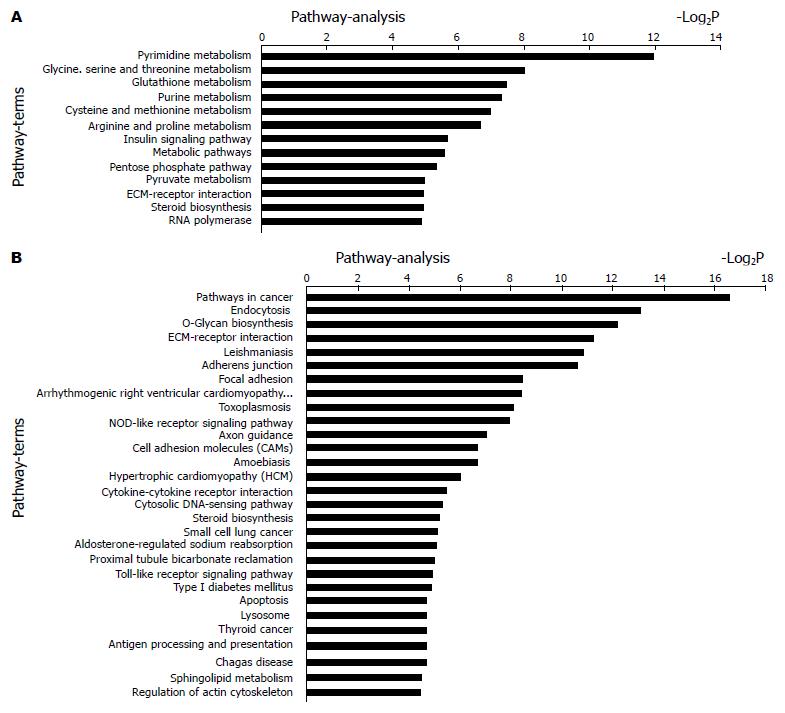
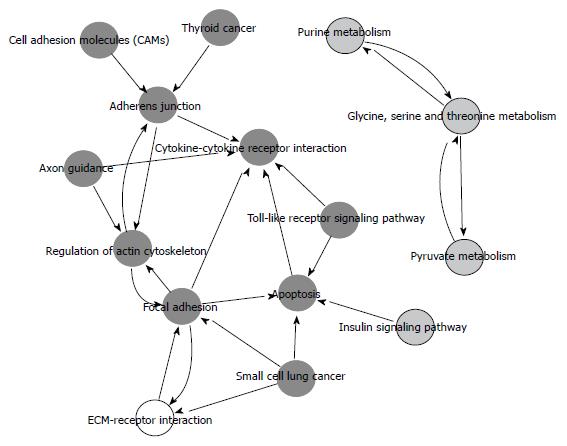
上述筛选的差异表达基因基于Gene Ontology数据库进行GO注释, 其中, 其生物学功能主要涉及多胺代谢过程、含碱基的小分子相互转换、组蛋白H4乙酰化、细胞氨基酸生物合成过程、谷胱甘肽的代谢过程、染色体分离、有丝分裂早中期、有丝分裂细胞周期的M期、细胞分裂、细胞周期、信号转导、凋亡、Toll样受体信号转导通路、Ⅰ型干扰素介导的信号转导通路、semaphorin-plexin信号通路、血小板衍生生长因子受体信号转导途径、肿瘤坏死因子介导的信号转导通路、间充质干细胞分化、半桥粒装配、不依赖TAP的MHC Ⅰ类外源性多肽抗原加工与提呈等. 上调和下调差异基因分别进行显著性功能分析, 显著性功能富集等见图2. 上述筛选的差异表达基因基于KEGG数据库进行Pathway注释, 上调和下调差异基因所显著参与的信号通路见图3. 主要涉及癌症的途径、内吞作用、O-糖基化生物合成、嘧啶代谢、谷胱甘肽代谢等. 基于KEGG数据库对显著pathway之间的关系进行整理, 得到信号通路调控网络见图4.
肿瘤发起细胞, 又称肿瘤干细胞, 具有成瘤、侵袭转移及治疗抵抗作用[9]. Kallifatidis等[6]从胰腺癌细胞标记分子CD44(+)/CD24(-), CD44(+)/CD24(+)或CD44(+)/CD133(+)的表达与裸鼠成瘤能力、分化程度、克隆性生长、肿瘤微球体形成、ALDH活性以及治疗抵抗等肿瘤干细胞特性的关系进行系列研究, 认为具有CD44+/CD24-表型的胰腺癌细胞具有肿瘤干细胞特性; 胰腺癌细胞系MIA-PaCa2的CD44+/CD24-表达率95.5%, 而BxPc-3的表达率17.5%; 并认为MIA-PaCa2(TIChigh)与BxPc-3(TIClow)可以直接作为研究胰腺癌干细胞的工具细胞, 不需要进一步富集. 因此, 本研究基于上述研究基础, 直接以MIA-PaCa2(TIChigh)与BxPc-3(TIClow)作为研究胰腺癌干细胞的工具细胞, 未做CD44(+)/CD24(-)标志物筛选.
新近的研究表明: 胰腺癌在形成之前, 源于具有潜在癌变分子基础的上皮细胞(如: PanIN 2和3级中胰腺上皮细胞), 在各种因素的作用(如: 慢性胰腺炎)下发生EMT, 导致细胞内相关基因发生重新编程, 获得侵袭转移能力和肿瘤干细胞的特性, 形成胰腺癌干细胞[5]. 其他许多研究也表明: EMT涉及肿瘤干细胞(cancer stem cells, CSCs)形成, 肿瘤干细胞具有EMT特性. 例如, Gioffi等[10]研究永生化的人乳腺上皮细胞(human mammary epithelial cell, HMLE)在外源性导入Twist或Snail转录因子, 或通过TGF-β1诱导EMT产生过程中, 获得了正常乳腺干细胞表型CD44high/CD24low, 并且具有干细胞的特性(如较强的自我更新能力、克隆微球的形成、成瘤能力等); 此外, 通过对CD44low/CD24high表型的HMLEs诱导EMT产生过程中, 获得了同样的结果; 这表明EMT能够产生干细胞, 这些细胞也表达EMT相关标志分子(如N-cadherin、Fibronectin、vimentin、FOXC2、SIP1和Snail). 在HMLE中外源性导入HER2/neu癌基因(HMLEN cells), 再外源性导入Twist或Snail基因, 诱导EMT发生, 这些细胞能导致肿瘤形成, 而对照组(未导入Twist或Snail基因)不能成瘤; 这表明EMT能促进CSCs形成. Xie等[11]研究IL-6介导的EMT导致了乳腺癌干细胞的形成, 认为EMT是癌细胞获得"干性"的关键机制, 非"干性"的癌细胞的EMT是CSCs的一个来源. Morel等[12]研究证实表型CD44+CD24-/low的乳腺癌干细胞能够来源于表型CD44lowCD24+非致瘤性的乳腺上皮细胞, 其机制是通过激活Ras/MAPK信号途径和EMT. Kasimir-Baver等[13]研究表明: 侵入到循环系统中的乳腺癌细胞(circulating tumor cells, CTC)干细胞标志物和EMT分子标志物过表达, 具有肿瘤干细胞和EMT特性. 此外, 许多实验证明在头颈部鳞状癌干细胞、宫颈癌干细胞以及肝癌干细胞与EMT紧密相关[14-16]. Shah等[17]通过对吉西他滨耐药的胰腺癌细胞研究发现, 这群细胞侵袭转移能力更强, EMT标志分子表达升高, 干细胞表型分子CD24、CD44和ESA表达升高. 这表明化疗药物富集的胰腺癌干细胞具有EMT表型的分子基础. Bao等[18,19]研究证实: 在胰腺癌细胞株AsPC-1中, 过表达Notch-1和FoxM1诱导EMT发生, 不仅上调了EMT标志分子(ZEB1、CD44、EpCAM、Hes-1), 也上调了CSC表面标志物(CD44和EpCAM)的表达, 增强了肿瘤细胞的侵袭转移、克隆形成、自我更新等能力; 上述这些研究表明EMT的发生与CSC表型的相关基因重新编程相关, 并有助于肿瘤的复发和耐药. 我们运用芯片技术筛选高表达胰腺癌干细胞MIA-PaCa2(TIChigh)和低表达胰腺癌干细胞BxPc-3(TIClow)的差异基因表达谱表明前者较后者, 与EMT相关的基因表达显著升高, 不仅证实了上述研究结论, 也从一个分子机制层面证实了MIA-PaCa2(TIChigh)和BxPc-3(TIClow)作为胰腺癌干细胞的研究工具细胞具有一定的可靠性.
此外, 我们采用生物信息学方法对上述芯片结果进行了分析, 差异表达的基因显著性影响许多生物学功能, 特别是多胺代谢过程和不依赖TAP的MHC Ⅰ类外源性多肽抗原加工与提呈, 表明影响上述两个生物学功能的差异表达基因更有可能参与胰腺癌干细胞的表型与发生. 通过通路分析, 发现差异表达的基因显著性影响许多信号通路, 特别是涉及癌症的途径、嘧啶代谢, 表明影响上述两个通路的差异表达基因更有可能揭示胰腺癌干细胞的发生与调控参与胰腺癌干细胞的发生与调控机制. 我们基于KEGG数据库对显著pathway之间的关系进行整理, 得到信号通路调控网络图, 该图显示上述信号通路的交叉对话可能从整体角度揭示胰腺癌干细胞的发生与调控机制.
总之, 我们通过芯片技术筛选了7059条与胰腺癌干细胞相关的差异表达基因, 并对其进行了生物信息学分析, 这些基因的分子功能和参与的信号通路可能涉及调控胰腺癌干细胞的各种生物学特性, 进一步对其生物学功能和调控机制进行研究, 有助于从整体上分析胰腺癌干细胞的发生机制, 寻找并设计新的治疗靶点.
胰腺癌进展快、预后差的主要原因是其早期转移、对传统的治疗抵抗以及较易复发. 当前的研究表明胰腺癌干细胞是上述现象的理论基础.
王钦红, 副教授, 美国杜克大学医学院肿瘤生物系
目前关于胰腺癌干细胞相关基因差异表达谱可见部分文献报道, 作者的课题组也曾利用胰腺癌组织分选的原代胰腺癌干细胞裸鼠模型进行基因芯片分析过胰腺癌干细胞差异基因表达谱.
Kallifatidis等研究, 认为具有CD44+/CD24-表型的胰腺癌细胞具有肿瘤干细胞特性; 胰腺癌细胞系MIA-PaCa2的CD44+/CD24-表达率95.5%, 而BxPc-3的表达率17.5%; 并认为MIA-PaCa2(TIChigh)与BxPc-3(TIClow)可以直接作为研究胰腺癌干细胞的工具细胞. 秦仁义等采用分选的原代胰腺癌干细胞裸鼠模型进行基因芯片分析过胰腺癌干细胞差异基因表达谱, 其中差异基因涉及细胞凋亡、细胞周期、代谢、细胞线粒体结构和耐药等多个方面.
目前关于采用胰腺癌细胞系MIA-PaCa2(TIChigh)与BxPc-3(TIClow)作为研究胰腺癌干细胞的工具细胞, 采用基因芯片技术筛选、鉴定胰腺癌干细胞相关差异基因表达谱尚未见报道. 本研究结合文献研究有助于发现新的与胰腺癌干细胞发生、发展相关的基因及其调控机制.
转录组学: 一门在整体水平上研究细胞中基因转录的情况及转录调控规律的学科, 是从RNA水平研究基因表达的情况, 是研究细胞表型和功能的一个重要手段.
本文选题新颖, 研究方法合理, 对胰腺癌干细胞相关差异基因表达谱的探讨具有一定的创新, 有重大科研意义.
编辑: 田滢 电编: 鲁亚静
| 1. | Siegel R, Naishadham D, Jemal A. Cancer statistics, 2012. CA Cancer J Clin. 2012;62:10-29. [PubMed] [DOI] |
| 2. | Tuveson DA, Neoptolemos JP. Understanding metastasis in pancreatic cancer: a call for new clinical approaches. Cell. 2012;148:21-23. [PubMed] [DOI] |
| 3. | Lim KH, Chung E, Khan A, Cao D, Linehan D, Ben-Josef E, Wang-Gillam A. Neoadjuvant therapy of pancreatic cancer: the emerging paradigm? Oncologist. 2012;17:192-200. [PubMed] [DOI] |
| 4. | Rasheed ZA, Matsui W. Biological and clinical relevance of stem cells in pancreatic adenocarcinoma. J Gastroenterol Hepatol. 2012;27 Suppl 2:15-18. [PubMed] [DOI] |
| 5. | Rhim AD, Mirek ET, Aiello NM, Maitra A, Bailey JM, McAllister F, Reichert M, Beatty GL, Rustgi AK, Vonderheide RH. EMT and dissemination precede pancreatic tumor formation. Cell. 2012;148:349-361. [PubMed] [DOI] |
| 6. | Kallifatidis G, Rausch V, Baumann B, Apel A, Beckermann BM, Groth A, Mattern J, Li Z, Kolb A, Moldenhauer G. Sulforaphane targets pancreatic tumour-initiating cells by NF-kappaB-induced antiapoptotic signalling. Gut. 2009;58:949-963. [PubMed] [DOI] |
| 7. | Rausch V, Liu L, Kallifatidis G, Baumann B, Mattern J, Gladkich J, Wirth T, Schemmer P, Büchler MW, Zöller M. Synergistic activity of sorafenib and sulforaphane abolishes pancreatic cancer stem cell characteristics. Cancer Res. 2010;70:5004-5013. [PubMed] [DOI] |
| 8. | Zhang C, Kolb A, Büchler P, Cato AC, Mattern J, Rittgen W, Edler L, Debatin KM, Büchler MW, Friess H. Corticosteroid co-treatment induces resistance to chemotherapy in surgical resections, xenografts and established cell lines of pancreatic cancer. BMC Cancer. 2006;6:61. [PubMed] [DOI] |
| 9. | Mani SA, Guo W, Liao MJ, Eaton EN, Ayyanan A, Zhou AY, Brooks M, Reinhard F, Zhang CC, Shipitsin M. The epithelial-mesenchymal transition generates cells with properties of stem cells. Cell. 2008;133:704-715. [PubMed] [DOI] |
| 10. | Cioffi M, Dorado J, Baeuerle PA, Heeschen C. EpCAM/CD3-Bispecific T-cell engaging antibody MT110 eliminates primary human pancreatic cancer stem cells. Clin Cancer Res. 2012;18:465-474. [PubMed] [DOI] |
| 11. | Xie G, Yao Q, Liu Y, Du S, Liu A, Guo Z, Sun A, Ruan J, Chen L, Ye C. IL-6-induced epithelial-mesenchymal transition promotes the generation of breast cancer stem-like cells analogous to mammosphere cultures. Int J Oncol. 2012;40:1171-1179. [PubMed] [DOI] |
| 12. | Morel AP, Lièvre M, Thomas C, Hinkal G, Ansieau S, Puisieux A. Generation of breast cancer stem cells through epithelial-mesenchymal transition. PLoS One. 2008;3:e2888. [PubMed] [DOI] |
| 13. | Kasimir-Bauer S, Hoffmann O, Wallwiener D, Kimmig R, Fehm T. Expression of stem cell and epithelial-mesenchymal transition markers in primary breast cancer patients with circulating tumor cells. Breast Cancer Res. 2012;14:R15. [PubMed] [DOI] |
| 14. | Zhang Z, Filho MS, Nör JE. The biology of head and neck cancer stem cells. Oral Oncol. 2012;48:1-9. [PubMed] [DOI] |
| 15. | López J, Poitevin A, Mendoza-Martínez V, Pérez-Plasencia C, García-Carrancá A. Cancer-initiating cells derived from established cervical cell lines exhibit stem-cell markers and increased radioresistance. BMC Cancer. 2012;12:48. [PubMed] [DOI] |
| 16. | Kim HM, Haraguchi N, Ishii H, Ohkuma M, Okano M, Mimori K, Eguchi H, Yamamoto H, Nagano H, Sekimoto M. Increased CD13 expression reduces reactive oxygen species, promoting survival of liver cancer stem cells via an epithelial-mesenchymal transition-like phenomenon. Ann Surg Oncol. 2012;19 Suppl 3:S539-S548. [PubMed] [DOI] |
| 17. | Shah AN, Summy JM, Zhang J, Park SI, Parikh NU, Gallick GE. Development and characterization of gemcitabine-resistant pancreatic tumor cells. Ann Surg Oncol. 2007;14:3629-3637. [PubMed] [DOI] |
| 18. | Bao B, Wang Z, Ali S, Kong D, Li Y, Ahmad A, Banerjee S, Azmi AS, Miele L, Sarkar FH. Notch-1 induces epithelial-mesenchymal transition consistent with cancer stem cell phenotype in pancreatic cancer cells. Cancer Lett. 2011;307:26-36. [PubMed] [DOI] |
| 19. | Bao B, Wang Z, Ali S, Kong D, Banerjee S, Ahmad A, Li Y, Azmi AS, Miele L, Sarkar FH. Over-expression of FoxM1 leads to epithelial-mesenchymal transition and cancer stem cell phenotype in pancreatic cancer cells. J Cell Biochem. 2011;112:2296-2306. [PubMed] [DOI] |