修回日期: 2012-01-04
接受日期: 2012-02-06
在线出版日期: 2012-02-08
目的: 应用表面增强激光解吸电离飞行时间质谱蛋白质芯片(SELDI-TOF-MS)联合激光显微切割(LCM)技术筛选大肠癌及其肝转移标志蛋白.
方法: 采用LCM技术获取24例大肠癌肝转移患者正常大肠、原发灶及肝转移灶癌细胞; 应用SELDI-TOF-MS技术对其行蛋白质谱分析; 采用Biomarker Wizard软件分析差异蛋白; 通过查询蛋白库对特定分子质量所对应的标志蛋白进行初步确定.
结果: 比较3组细胞间的SELDI质谱图, 发现大肠癌原发灶与正常大肠两组间存在15个标志蛋白, 12个表达上调, 3个表达下调; 大肠癌肝转移灶与原发灶两组间存在9个标志蛋白, 5个表达上调, 4个表达下调. 其中质荷比4 676.63 Da, 11 740.87 Da, 21 063.59 Da和22 783.36 Da, 蛋白峰差异性最明显(P<0.01). 通过查询ExPasy蛋白库筛选出20个差异蛋白, 包括整合膜蛋白2C、DNA修复蛋白RAD51同系物4、细胞周期检查点蛋白RAD1、人附睾蛋白4、着丝粒蛋白R、Pleckstrin同源结构域家族成员3等. 其中细胞凋亡调节Bax蛋白γ亚型、蛋白质S100A11(Protein S100-A11)、Raf激酶抑制蛋白(RKIP)和热休克蛋白27(HSP-27)在正常大肠、原发灶及肝转移灶癌细胞中均呈差异性表达, 并且差异性最明显(P<0.01).
结论: SELDI蛋白质芯片联合LCM技术有可能筛选出敏感性高、特异性强的大肠癌标志蛋白, 筛选出的差异蛋白可能是大肠癌及其肝转移特异性生物标志物.
引文著录: 赵渊博, 王云海, 阿不都外力•吾守尔. SELDI-TOF-MS联合LCM技术筛选大肠癌肝转移早期诊断标志蛋白. 世界华人消化杂志 2012; 20(4): 296-303
Revised: January 4, 2012
Accepted: February 6, 2012
Published online: February 8, 2012
AIM: To screen protein markers for liver metastases of colorectal cancer using surface-enhanced laser desorption/ionization time-of-flight mass spectrometry protein chip (SELDI-TOF-MS) in combination with laser capture microdissection (LCM) technology.
METHODS: Normal colorectal cells, primary tumor cells and liver metastatic cells were obtained from 24 colorectal cancer patients with liver metastases using the LCM technology. Protein profiling was performed using SELDI-TOF-MS technology. Differentially expressed proteins were analyzed using Biomarker Wizard software and identified by querying the database.
RESULTS: Fifteen differentially expressed proteins were found between primary tumor cells and normal colorectal cells, of which 12 were up-regulated and 3 down-regulated. Nine differentially expressed proteins were found between liver metastatic cells and primary tumor cells, of which 5 were up-regulated and 4 down-regulated. Twenty differentially expressed proteins were identified by querying ExPasy protein database, including integral membrane protein 2C, DNA repair protein RAD51 homolog 4, cell cycle checkpoint protein RAD1, human epididymis protein 4, centromere protein R, and pleckstrin homology domain family member 3. Apoptosis regulator Bax-γ, S100A11, Raf kinase inhibitor protein (RKIP) and heat shock protein 27 (HSP-27) displayed most obvious differential expression among normal colorectal cells, primary tumor cells and liver metastatic cells (all P < 0. 01).
CONCLUSION: SELDI-TOF-MS technology combined with LCM may allow to screen highly sensitive and specific protein markers for colorectal cancer and liver metastases of colorectal cancer.
- Citation: Zhao YB, Wang YH, Abuduwaili•Wushour. Application of SELDI-TOF-MS and LCM to screen protein markers for early diagnosis of liver metastasis of colorectal carcinoma. Shijie Huaren Xiaohua Zazhi 2012; 20(4): 296-303
- URL: https://www.wjgnet.com/1009-3079/full/v20/i4/296.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i4.296
大肠癌肝转移是一个复杂的多基因参与的多步骤过程, 明确大肠癌肝转移相关基因及蛋白质的作用, 筛选和鉴定肿瘤生物学标志物是大肠癌研究的最前沿领域和热点之一. SELDI-TOF-MS是一种集蛋白质分离、纯化及数据处理为一体的蛋白质组学技术, 其敏感性和特异性高、需要样本少[1]. 从复杂、不均一的样品中获得高度同质细胞是大肠癌组织蛋白质组学研究中需解决的主要问题之一[2]. LCM技术可有效剔除间质细胞和其他可能影响结果分析的杂质成分纯化细胞, 从而保证研究组分的可比性[3]. 本研究应用SELDI-TOF-MS结合LCM技术筛选大肠癌组织差异蛋白, 旨在发现并鉴定参与大肠癌发生及与肝转移有关的各种蛋白, 并探讨其在临床诊断、预后以及治疗上的意义.
24例大肠癌标本来自于2010-03/12新疆医科大学第一附属医院胃肠外科手术切除标本, 所有患者术前均经病理检查证实已发生肝转移, 且未行放化疗. 男15例, 女9例, 年龄39-72岁, 平均59岁. 手术标本取出后取新鲜大肠癌组织与对应正常组织和肝转移癌组织, 用4 ℃冰盐水冲洗3-5次, 冲净血液; 分割成1.0 cm×1.0 cm大小, 分装于标志清楚的1.8 mL冻存管置液氮中速冻, -80 ℃冰箱保存备用. 整个采集过程控制于30 min内. 羟乙基哌嗪乙磺酸(HEPES)、尿素(Urea)、3-环乙胺-1-丙磺酸(CHAPS)、白芥子酸(SPA)均购自美国Sigma公司; 冰冻切片机(Leica, Germany); 激光捕获显微切割系统(Arcturus, USA); Protein Chip Biology System(PBS II-C)质谱仪、弱阳离子交换蛋白芯片(CM10蛋白芯片, Ciphergen Biosystems, USA), Biomarker Wizard软件(Ciphergen, USA).
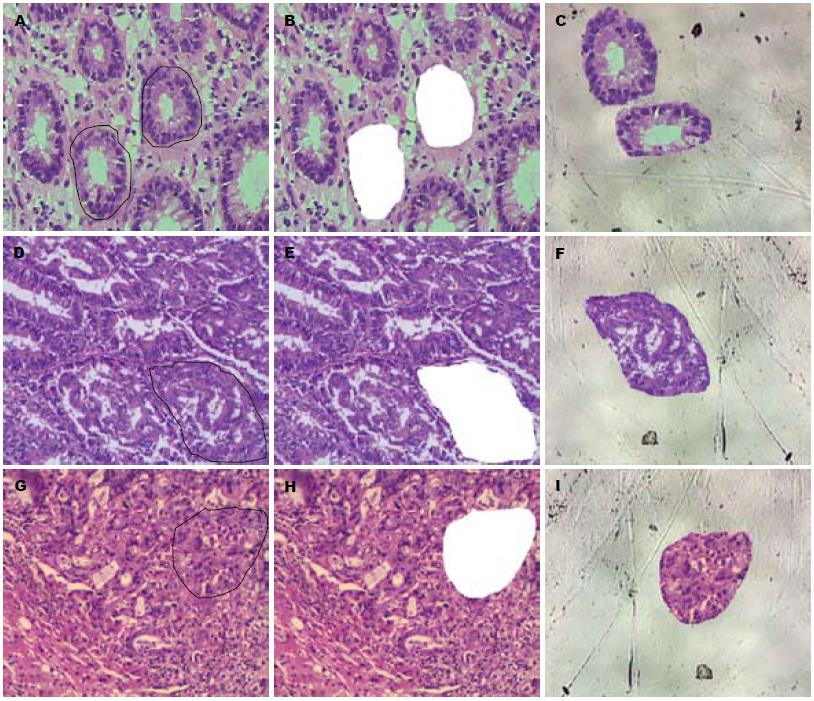
1.2.1 LCM获取目的细胞: 将保存在-80 ℃条件下的标本制成6 μm厚的冰冻切片及HE染色. 采用LCM技术(激光脉冲为7 000), 在合适倍率下找到细胞密集、染色满意区域选择癌巢细胞和正常黏膜腺体组织分类切割, 并收集至不同收集管装载细胞收集帽子.
1.2.2 总蛋白质提取及点样: 样品加裂解液(9 mol/L Urea, 2%CHAPS, 1%DTT)30 μL冰上裂解30-60 min, 超声振荡20 min; 10 000 r/min、4 ℃离心30 min. 将已加样处理的CM10芯片装入Bioprocessor中, 每孔加入200 μL结合缓冲液, 室温振荡洗涤2次, 每次5 min, 甩干. 每孔加入样品混合液100 μL, 振荡孵育1 h, 甩去样品, 用洗脱缓冲液(100 mmol/L, NaAc, pH4.0)200 μL室温振荡洗涤2次, 每次5 min, 甩干; 再用HPLC H2O洗涤1次, 立即甩干. 取出芯片晾干后, 每点加2次SPA 0.5 μL, 晾干后上机测试.
1.2.3 数据统计分析和蛋白质数据库搜索: 用All-in-one标准蛋白质校正质谱仪使系统的质量偏差≤0.1%. 原始数据先以Proteinchip Software 3.0软件校正, 使总离子强度及分子量达到均一. 应用Biomarker Wizard软件初步筛选差异蛋白质, 所有样品质谱图在2 500-25 000质荷比(m/z)范围进行2次信噪比过滤, 初始噪音过滤值为5, 二次噪音过滤值为2; 过滤掉m/z在2 500以下及信噪比小于4的蛋白峰. 采集的数据应用ZUCI-ProteinChip Data Analyze System软件包将各样本中差异<0.5%的峰聚类; Wilconxon秩和检验方法筛选各组间表达差异有统计学意义的蛋白质峰(P<0.01). 对差异蛋白质进行ExPasy蛋白数据库搜索(http://www.expasy.org), 寻找与他们分子量最为接近的蛋白质, 检索时分子质量设定为质荷比±0.1%, PI值9.0±5.0.
在设定捕获条件下, 根据癌巢在切片中的位置和大小调整视野, 激光束沿间质细胞一侧切割. 切割的细胞被收集入收集管帽(图1).
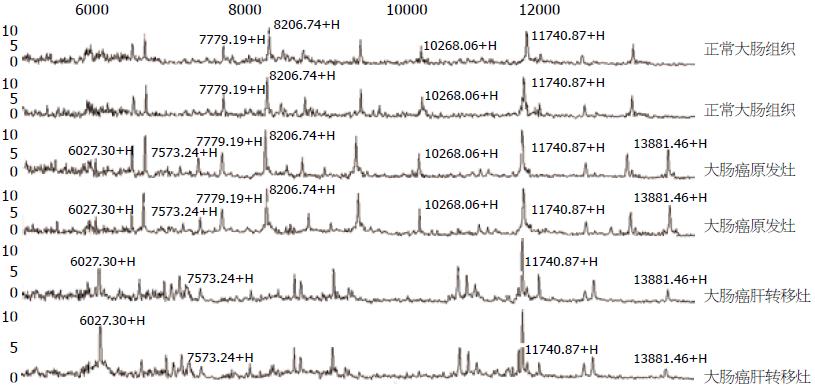
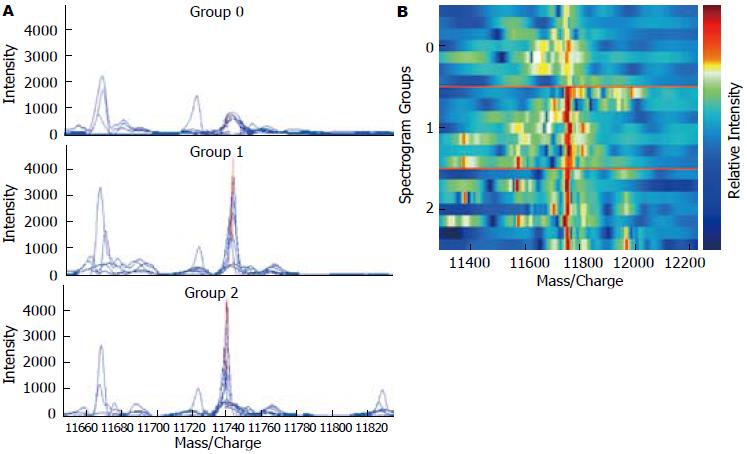
Ciphergen proteinChip 3.1软件分别对3组检测质谱图进行分析, 筛选显著性差异蛋白质峰(P<0.01). 发现大肠癌原发灶与正常大肠两组间存在15个标志蛋白, 其中12个在大肠癌原发灶中表达上调, 3个在大肠癌原发灶中表达下调, 大肠癌肝转移灶与原发灶两组间存在9个标志蛋白, 其中5个在大肠癌肝转移灶中表达上调, 4个在大肠癌肝转移灶中表达下调(图2). 其中质荷比(m/z)为4 676.63 Da、11 740.87 Da、21 063.59 Da和22 783.36 Da的4种蛋白峰差异性最明显(P<0.01). 质荷比(m/z)为11 740. 87 Da的蛋白峰在3组中的表达情况见图3.
通过网上对比ExPasy蛋白数据库共检测出20个差异蛋白, 一些蛋白或肽只有一个候选结果, 可初步判定检索结果即为该分子质量标志蛋白(表1, 2), 其他多候选结果者根据其组织来源、生物学功能, 初步确定为相应蛋白或多肽, 表达上调的有15个蛋白, 表达下调的有5个蛋白, 差异蛋白中有6种蛋白在大肠癌蛋白组学之前的研究中未见报道, 他们是: 整合膜蛋白2C(Integral membrane protein 2C)、DNA修复蛋白RAD51同系物4(DNA repair protein RAD51 homolog 4)、细胞周期检查点蛋白RAD1(Cell cycle checkpoint protein RAD1)、人附睾蛋白4(Human epididymis protein 4, HE4)、着丝粒蛋白R(Centromere protein R)、Pleckstrin同源结构域家族成员3(Pleckstrin homology-like domain family A member 3). 20个差异蛋白中细胞凋亡调节Bax蛋白γ亚型(Apoptosis regulator Bax-γ)、蛋白质S100A11(Protein S100-A11)、Raf激酶抑制蛋白(RKIP)和热休克蛋白27(HSP-27)在正常大肠、原发灶及肝转移灶癌细胞中均呈差异性表达, 并且差异性最明显(P<0.01, 表3).
| 差异蛋白质(Da) | Swiss-Prot 代码 | 等电点 | 分子量(Da) | 蛋白质名称 |
| 2735.04 | Q9NQX7 | 6.74 | 2735 | Integral membrane protein 2C precursor |
| 4628.76 | O00453-7 | 9.49 | 4629 | Leukocyte-specific transcript 1 protein (Isoform7) |
| 4676.63 | Q07812-3 | 11.22 | 4678 | Apoptosis regulator Bax (Isoform Gamma) |
| 5386.36 | O75771-2 | 6.52 | 5387 | DNA repair protein RAD51homolog 4 (Isoform2) |
| 6439.23 | Q538Z0 | 9.69 | 6437 | Leucine zipper protein 6 |
| 6636.59 | O14548 | 9.77 | 6636 | Cytochrome C oxidase subunit 7A-related protein precursor |
| 7779.19 | P78563-5 | 10.40 | 7778 | Double-stranded RNA-specificeditase1 (Isoform 5) |
| Q06643-2 | 11.71 | 7780 | Tumor necrosis factor C (Isoform 2) | |
| 8206.74 | O43521-15 | 5.25 | 8202 | Bcl-2-like protein 11 (Isoform Bim-beta6 ) |
| Q14508-4 | 4.24 | 8214 | Human Epididymis Protein 4 (Isoform 4) | |
| 9503.81 | P35222-2 | 4.88 | 9501 | Catenin beta-1 (Isoform 2) |
| 10268.06 | O95777 | 4.34 | 10266 | N-alpha-acetyltransferase 38 |
| P02775 | 8.94 | 10271 | Platelet basic protein precursor | |
| 11740.87 | P31949 | 6.55 | 11741 | Protein S100-A11 |
| 12612.62 | Q04609-3 | 8.60 | 12603 | Glutamate carboxypeptidase 2 (Isoform 3 ) |
| Q13352-3 | 7.83 | 12624 | Centromere protein R (Isoform 3) | |
| 13244. 73 | P06702 | 5.71 | 13246 | Protein S100-A9 |
| 21063. 59 | P30086 | 7.43 | 21057 | Raf kinase inhibitor protein(RKIP) |
| 22783. 36 | P40198-3 | 5.97 | 22783 | Heat shock protein 27(HSP-27) |
| 差异蛋白质(Da) | Swiss-Prot 代码 | 等电点 | 分子量(Da) | 蛋白质名称 |
| 4676.63 | Q07812-3 | 11.22 | 4678 | Apoptosis regulator Bax (Isoform Gamma) |
| 5914.77 | P05067 | 7.01 | 5911 | Gamma-secretase C-terminal fragment 50 |
| 6027.30 | Q13794 | 10.31 | 6030 | PMA-induced protein 1 |
| P02776 | 10.03 | 6033 | Platelet factor 4 precursor | |
| 7573.24 | O60671-3 | 6.00 | 7576 | Cell cycle checkpoint protein RAD1 (Isoform 3) |
| P53634 | 4.76 | 7578 | Dipeptidyl peptidase 1 light chain | |
| 9296.24 | P02775 | 7.85 | 9292 | Connective tissue-activating peptide III |
| Q9NQW1 | 6.55 | 9294 | SEC31-related protein B (Isoform 3) | |
| 11740.87 | P31949 | 6.55 | 11741 | Protein S100-A11 |
| 13881.46 | Q9Y5J5 | 9.72 | 13891 | Pleckstrin homology-like domain family A member 3 |
| 21063.59 | P30086 | 7.43 | 21057 | Raf kinase inhibitor protein(RKIP) |
| 22783.36 | P40198-3 | 5.97 | 22783 | Heat shock protein 27(HSP-27) |
| 分子量(Da) | P值 | 表达 | 蛋白质名称 | 功能 |
| 2735.041 | 0.002547 | ↑ | 整合膜蛋白2C前体 | 调节β-淀粉样肽生产, 抑制APP的加工 |
| 4628.762 | 0.004613 | ↓ | 白细胞特异性转录1蛋白亚型7 | 调节免疫反应 |
| 4676.633 | 0.006648 | ↓ | 细胞凋亡调节Bax蛋白γ亚型 | 加速程序性细胞死亡 |
| 5386.361 | 0.009294 | ↑ | DNA修复蛋白RAD51同系物4亚型2 | 涉及DNA的同源重组修复 |
| 5914.771 | 0.001434 | ↑ | γ-分泌C-端片段50 | 细胞表面受体, 转录调控 |
| 6027.301 | 0.000824 | ↑ | 血小板因子4前体 | 抑制血管内皮细胞增殖, 趋化因子 |
| 6439.232 | 0.005498 | ↑ | 亮氨酸拉链蛋白6 | 未知 |
| 6636.592 | 0.004415 | ↑ | 细胞色素C氧化酶亚基7A相关蛋白前体 | 调节靶细胞的能源生产 |
| 7573.241 | 0.000525 | ↓ | 细胞周期检查点蛋白RAD1亚型3 | 在DNA修复中发挥重要作用 |
| 7779.192 | 0.000126 | ↑ | 肿瘤坏死因子-C亚型2 | 免疫应答的调节和促进细胞增殖 |
| 8206.742 | 0.000182 | ↑ | 人附睾蛋白4亚型4 | 免疫调节和促进细胞增殖 |
| 9296.241 | 0.009294 | ↑ | 结缔组织激活肽III | 刺激纤溶酶原激活物的形成和分泌 |
| 9503.812 | 0.001613 | ↑ | β-连环蛋白-1亚型2 | 激活Wnt信号反应基因, 调控细胞黏附 |
| 10268.062 | 0.000545 | ↑ | 血小板碱性蛋白前体 | 刺激DNA的合成, 有丝分裂, 糖酵解 |
| 11740.873 | 0.000041 | ↑ | 蛋白质S100A11 | Ca2+结合, 信号传导, 促进细胞分化 |
| 12612.622 | 0.000419 | ↑ | 着丝粒蛋白R亚型3 | 转录, 细胞周期蛋白A相关激酶抑制剂 |
| 13244.732 | 0.000434 | ↑ | 蛋白质S100A9 | Ca2+结合, 信号传导 |
| 13881.461 | 0.003230 | ↓ | Pleckstrin同源结构域家族成员3 | 肿瘤抑制基因, 抑制Akt/AKT1信号传导 |
| 21063.593 | 0.000062 | ↓ | Raf激酶抑制蛋白 | 调节ERK和NF-κB信号转导而诱导凋亡 |
| 22783.363 | 0.000263 | ↑ | 热休克蛋白27 | 参与抗逆性, 伴侣蛋白 |
SELDI-TOF-MS技术是近年来新兴的一种蛋白质组学技术, 具有简单、快捷、灵敏等特点, 这种技术通过高通量检出蛋白质的表达图谱, 寻找患者和健康人之间存在的差异蛋白, 从中找到促使细胞发生癌变的预警分子, 为研究肿瘤标志物与肿瘤发生发展提供了良好的技术平台, 目前已广泛应用于多种肿瘤的研究中[4-9].
Engwegen等[10]应用SELDI技术比较了40名结直肠癌患者与49名正常对照的血清蛋白质谱, 筛选出5个差异表达的蛋白; Ward等[11]用此技术发现血清中去除C末端精氨酸的去精C3a、α1-抗胰蛋白酶和转铁蛋白, 对于结直肠癌具有诊断意义. 目前研究以血清标志物为主, 而应用SELDI技术进行大肠癌及其肝转移组织差异蛋白分析少见. 本研究应用SELDI-TOF-MS联合LCM技术筛选大肠癌及其肝转移组织差异蛋白, 比较3组细胞间的SELDI质谱图, 发现大肠癌原发灶与正常大肠两组间存在15个标志蛋白, 12个表达上调, 3个表达下调; 大肠癌肝转移灶与原发灶两组间存在9个标志蛋白, 5个表达上调, 4个表达下调. 其中质荷比4676.63 Da、11740.87 Da、21063.59 Da和22783.36 Da蛋白峰差异性最明显(P<0. 01), 并且在正常大肠、原发灶及肝转移灶癌细胞中呈差异性表达. 通过查询ExPasy蛋白库并进行筛选, 初步确定分子质量所对应的蛋白分别是细胞凋亡调节Bax蛋白γ亚型、蛋白质S100A11、RKIP和HSP-27, 其中蛋白质S100A11和HSP-27表达上调, 而细胞凋亡调节Bax蛋白γ亚型和RKIP蛋白表达下调.
细胞凋亡调节Bax蛋白是Bcl-2家族成员之一, 两者表达水平高低与凋亡调控直接相关: Bax增高, 促进细胞凋亡; Bcl-2增高, 抑制细胞凋亡; Bcl-2/Bax是决定对细胞凋亡抑制作用强弱的关键因素[12]. RKIP蛋白属于磷脂酞乙醇胺结合蛋白家族, 可以和Raf-1结合而抑制MARK信号转导通路, 并参与对G蛋白偶联受体信号通路和NF-κB信号通路的调控, 从而具有促进细胞凋亡和抗肿瘤转移等作用[13]. Lee等[14]发现RKIP在肝癌中的表达明显低于邻近的正常肝组织, 增加RKIP的表达可以减少肝癌细胞的增殖和迁移. 相似的结果亦见于黑色素瘤的研究[15]. 目前, 多数的研究发现[16,17]: RKIP的表达在正常组织中高于肿瘤组织中, 在肿瘤组织中原发灶高于转移灶, RKIP的低表达或缺失的肿瘤转移性、侵袭性强, 预后差, 故认为RKIP是肿瘤转移抑制基因.
S100A11属于分子量10-14 kDa的S100蛋白家族成员之一. Torres等[18]发现在良性病变中定位于细胞核, 而在恶性中定位于细胞质中的S100A11蛋白在肿瘤进展中的具体作用虽然还不是很清楚, 但有研究表明可能与肾癌、乳腺癌、结肠直癌、膀胱癌、前列腺癌、肺癌、胃癌的发生发展相关[19]. 研究发现, S100A11与子宫平滑肌瘤的生长相关, 其过度表达能够抑制子宫平滑肌瘤细胞的凋亡, 提示其具有抗凋亡作用[20]. Shankar等[21]研究发现S100A11表达降低或缺失, 会减少细胞间的互动及E-钙粘蛋白的表达, 并降低肿瘤的迁移和侵袭. 此外, S100A11还可作为P21的通路蛋白, 引起细胞生长负性调控因子P21CIP1/WAF1表达增强, 抑制细胞内DNA的合成, 从而间接抑制肿瘤的发生发展[22]. Ohuchida等[23]发现S100A11的表达在胰腺癌的早期致癌阶段是上升的, 但随着发展在后期是下降的, 这些研究结果表明, S100A11蛋白在肿瘤发生和发展, 其功能的复杂性.
HSP-27属于热休克蛋白家族, 相对分子质量27 kDa, 依据不同的磷酸化状态, 其在细胞内存在不同的聚合体状态, 如单聚体、二聚体或多聚体, 磷酸化则发生于丝裂原依赖的蛋白激酶/p38途径. HSP-27在细胞生物学过程中参与细胞信号转导, 从而影响细胞的分化、凋亡等. HSP-27在大肠癌的致癌过程中所起的作用仍然不清, 但其表达水平在包括神经母细胞瘤[24]、肝癌[25]、食管腺癌[26]和胃癌[27]中增加. 作为不依赖ATP的分子伴侣, 通过抑制应激诱导及死亡受体诱导的细胞凋亡途径而实现抗细胞凋亡作用, 表达水平增加可能引起肿瘤的抗宿主防御机制, 从而提高肿瘤细胞转移潜力[28]. Parcellier等[29]研究结果提示HSP-27能够促进磷酸化IκBa水解和增强NF-κB的活性, 这可能说明其具有抗凋亡的特性.
本研究中, 我们通过蛋白组学技术建立了大肠癌原发灶、肝转移灶与正常大肠组织之间的差异表达蛋白谱, 并且检测出了20个差异蛋白, 大多数分子参与细胞的生物学过程, 包括细胞增殖、转化、凋亡、代谢、蛋白折叠、构建细胞骨架等功能. 其中以上4种蛋白在正常大肠、原发灶及肝转移灶癌细胞表达差异性最明显, 提示可能与大肠癌的发生发展及肝转移有关, 但具体作用机制尚不清楚, 认为可能通过细胞凋亡调节Bax蛋白和RKIP蛋白表达下调及蛋白质S100A11和HSP-27表达上调, 抑制细胞凋亡并使细胞间黏附作用减弱, 从而促进大肠癌细胞的增殖及肝转移.
由于肿瘤细胞的功能分子是各种蛋白质, 所以蛋白分析比cDNA芯片更具临床价值[30]. 对于肿瘤和正常组织, 比较蛋白组学能检测出候选差异蛋白分子, 有助于肿瘤的诊断、治疗等. 本研究应用SELDI蛋白芯片联合LCM进行大肠癌蛋白质诊断标志的初步筛选, 获得了20个差异蛋白质, 其中细胞凋亡调节Bax蛋白γ亚型、S100A11、RKIP和HSP-27这4种差异最明显, 他们很可能是大肠癌特异性生物标志物, 参与了大肠癌的发生发展及肝转移过程. 我们下一步将对上述4种标志蛋白从血清ELISA、组织免疫组化和Western blot 3个方面进行验证, 进一步探讨其在大肠癌及其肝转移中的作用机制, 为大肠癌及其肝转移的早期诊断、发生发展机制的研究及寻找大肠癌新的治疗靶点奠定基础.
SELDI-TOF-MS是集蛋白分离、纯化、数据处理为一体的蛋白组学技术, 其敏感性、特异性高. LCM是从组织中纯化细胞最好的方法之一, 可为蛋白组学研究提供最佳的生物样本.
王娅兰, 教授, 重庆医科大学基础医学院病理教研室; 王健生, 副教授, 西安交通大学医学院第一附属医院
SELDI-TOF-MS作为一种较新的质谱技术, 目前在美国已应用于卵巢癌和前列腺癌的诊断, 其特异性和敏感性远远高于传统方法.
Engwegen等应用SELDI技术比较了40名结直肠癌患者与49名正常对照的血清蛋白质谱, 筛选出5个差异表达的蛋白; Ward等用此技术发现血清中去除C末端精氨酸的去精C3a、α1-抗胰蛋白酶和转铁蛋白对于结直肠癌具有诊断意义.
本研究应用SELDI蛋白芯片联合LCM技术可为蛋白组学研究提供最佳的生物样本, 并为大肠癌肝转移分子机制的阐明提供了实验依据.
本研究应用联合LCM进行大肠癌蛋白质诊断标志的初步筛选, 获得的标志蛋白很可能参与了大肠癌的发生发展及肝转移过程. 为大肠癌及其肝转移的早期诊断、发生发展机制的研究及寻找大肠癌新的治疗靶点奠定基础.
表面增强激光解吸电离飞行时间质谱(SELDI-TOF-MS): 该技术主要是在飞行质谱的检测系统中, 芯片特异性地与被测标本中蛋白结合, 再通过选择性清洗, 获得高分辨率的保留蛋白质谱. 蛋白在激光照射下解离, 带电分子在通过电场时加速, 检测仪记录飞行时间的长短, 质荷比(M/Z)越小, 飞行时间越短, 就会被最先检测到. 被测定蛋白质以一系列峰值的形式呈现.
本研究采用LCM和SELDI-TOF-MS技术筛选大肠癌及其肝转移标志蛋白. 该选题具有重要临床意义, 但还需进一步验证.
编辑: 张姗姗 电编:闫晋利
| 1. | Kikuchi T, Carbone DP. Proteomics analysis in lung cancer: challenges and opportunities. Respirology. 2007;12:22-28. [PubMed] [DOI] |
| 2. | Posadas EM, Simpkins F, Liotta LA, MacDonald C, Kohn EC. Proteomic analysis for the early detection and rational treatment of cancer--realistic hope? Ann Oncol. 2005;16:16-22. [PubMed] [DOI] |
| 3. | Gutstein HB, Morris JS. Laser capture sampling and analytical issues in proteomics. Expert Rev Proteomics. 2007;4:627-637. [PubMed] [DOI] |
| 4. | Kanmura S, Uto H, Kusumoto K, Ishida Y, Hasuike S, Nagata K, Hayashi K, Ido A, Stuver SO, Tsubouchi H. Early diagnostic potential for hepatocellular carcinoma using the SELDI ProteinChip system. Hepatology. 2007;45:948-956. [PubMed] [DOI] |
| 5. | Hellström M, Lexander H, Franzén B, Egevad L. Proteomics in prostate cancer research. Anal Quant Cytol Histol. 2007;29:32-40. [PubMed] |
| 6. | Engwegen JY, Mehra N, Haanen JB, Bonfrer JM, Schellens JH, Voest EE, Beijnen JH. Validation of SELDI-TOF MS serum protein profiles for renal cell carcinoma in new populations. Lab Invest. 2007;87:161-172. [PubMed] [DOI] |
| 7. | Zheng GX, Wang CX, Qu X, Deng XM, Deng BP, Zhang J. Establishment of serum protein pattern for screening colorectal cancer using SELDI-TOF-MS. Exp Oncol. 2006;28:282-287. [PubMed] |
| 8. | Wu SP, Lin YW, Lai HC, Chu TY, Kuo YL, Liu HS. SELDI-TOF MS profiling of plasma proteins in ovarian cancer. Taiwan J Obstet Gynecol. 2006;45:26-32. [PubMed] [DOI] |
| 9. | Zeidan BA, Townsend PA. SELDI-TOF proteomic profiling of breast carcinomas identifies clinicopathologically relevant groups of patients similar to previously defined clusters from cDNA expression. Breast Cancer Res. 2008;10:107. [PubMed] [DOI] |
| 10. | Engwegen JY, Helgason HH, Cats A, Harris N, Bonfrer JM, Schellens JH, Beijnen JH. Identification of serum proteins discriminating colorectal cancer patients and healthy controls using surface-enhanced laser desorption ionisation-time of flight mass spectrometry. World J Gastroenterol. 2006;12:1536-1544. [PubMed] |
| 11. | Ward DG, Suggett N, Cheng Y, Wei W, Johnson H, Billingham LJ, Ismail T, Wakelam MJ, Johnson PJ, Martin A. Identification of serum biomarkers for colon cancer by proteomic analysis. Br J Cancer. 2006;94:1898-1905. [PubMed] [DOI] |
| 12. | Neuzil J, Wang XF, Dong LF, Low P, Ralph SJ. Molecular mechanism of 'mitocan'-induced apoptosis in cancer cells epitomizes the multiple roles of reactive oxygen species and Bcl-2 family proteins. FEBS Lett. 2006;580:5125-5129. [PubMed] [DOI] |
| 13. | Zhang Z, Li M, Zhang G, Fang P, Yao H, Xiao Z, Chen Z. Identification of human gastric carcinoma biomarkers by differential protein expression analysis using 18O labeling and nanoLC-MS/MS coupled with laser capture microdissection. Med Oncol. 2010;27:296-303. [PubMed] [DOI] |
| 14. | Lee HC, Tian B, Sedivy JM, Wands JR, Kim M. Loss of Raf kinase inhibitor protein promotes cell proliferation and migration of human hepatoma cells. Gastroenterology. 2006;131:1208-1217. [PubMed] [DOI] |
| 15. | Houben R, Vetter-Kauczok CS, Ortmann S, Rapp UR, Broecker EB, Becker JC. Phospho-ERK staining is a poor indicator of the mutational status of BRAF and NRAS in human melanoma. J Invest Dermatol. 2008;128:2003-2012. [PubMed] [DOI] |
| 16. | Zeng L, Imamoto A, Rosner MR. Raf kinase inhibitory protein (RKIP): a physiological regulator and future therapeutic target. Expert Opin Ther Targets. 2008;12:1275-1287. [PubMed] [DOI] |
| 17. | Dangi-Garimella S, Yun J, Eves EM, Newman M, Erkeland SJ, Hammond SM, Minn AJ, Rosner MR. Raf kinase inhibitory protein suppresses a metastasis signalling cascade involving LIN28 and let-7. EMBO J. 2009;28:347-358. [PubMed] [DOI] |
| 18. | Torres-Cabala C, Bibbo M, Panizo-Santos A, Barazi H, Krutzsch H, Roberts DD, Merino MJ. Proteomic identification of new biomarkers and application in thyroid cytology. Acta Cytol. 2006;50:518-528. [PubMed] [DOI] |
| 19. | Salama I, Malone PS, Mihaimeed F, Jones JL. A review of the S100 proteins in cancer. Eur J Surg Oncol. 2008;34:357-364. [PubMed] [DOI] |
| 20. | Kanamori T, Takakura K, Mandai M, Kariya M, Fukuhara K, Sakaguchi M, Huh NH, Saito K, Sakurai T, Fujita J. Increased expression of calcium-binding protein S100 in human uterine smooth muscle tumours. Mol Hum Reprod. 2004;10:735-742. [PubMed] [DOI] |
| 21. | Shankar J, Messenberg A, Chan J, Underhill TM, Foster LJ, Nabi IR. Pseudopodial actin dynamics control epithelial-mesenchymal transition in metastatic cancer cells. Cancer Res. 2010;70:3780-3790. [PubMed] [DOI] |
| 22. | Sakaguchi M, Miyazaki M, Takaishi M, Sakaguchi Y, Makino E, Kataoka N, Yamada H, Namba M, Huh NH. S100C/A11 is a key mediator of Ca(2+)-induced growth inhibition of human epidermal keratinocytes. J Cell Biol. 2003;163:825-835. [PubMed] [DOI] |
| 23. | Ohuchida K, Mizumoto K, Ohhashi S, Yamaguchi H, Konomi H, Nagai E, Yamaguchi K, Tsuneyoshi M, Tanaka M. S100A11, a putative tumor suppressor gene, is overexpressed in pancreatic carcinogenesis. Clin Cancer Res. 2006;12:5417-5422. [PubMed] [DOI] |
| 24. | Pei H, Zhu H, Zeng S, Li Y, Yang H, Shen L, Chen J, Zeng L, Fan J, Li X. Proteome analysis and tissue microarray for profiling protein markers associated with lymph node metastasis in colorectal cancer. J Proteome Res. 2007;6:2495-2501. [PubMed] [DOI] |
| 25. | Zanini C, Pulerà F, Carta F, Giribaldi G, Mandili G, Maule MM, Forni M, Turrini F. Proteomic identification of heat shock protein 27 as a differentiation and prognostic marker in neuroblastoma but not in Ewing's sarcoma. Virchows Arch. 2008;452:157-167. [PubMed] [DOI] |
| 26. | Luk JM, Lam CT, Siu AF, Lam BY, Ng IO, Hu MY, Che CM, Fan ST. Proteomic profiling of hepatocellular carcinoma in Chinese cohort reveals heat-shock proteins (Hsp27, Hsp70, GRP78) up-regulation and their associated prognostic values. Proteomics. 2006;6:1049-1057. [PubMed] [DOI] |
| 27. | Doshi BM, Hightower LE, Lee J. The role of Hsp27 and actin in the regulation of movement in human cancer cells responding to heat shock. Cell Stress. Chaperones. 2009;14:445-457. [PubMed] [DOI] |
| 28. | Mosser DD, Morimoto RI. Molecular chaperones and the stress of oncogenesis. Oncogene. 2004;23:2907-2918. [PubMed] [DOI] |
| 29. | Parcellier A, Schmitt E, Gurbuxani S, Seigneurin-Berny D, Pance A, Chantôme A, Plenchette S, Khochbin S, Solary E, Garrido C. HSP27 is a ubiquitin-binding protein involved in I-kappaBalpha proteasomal degradation. Mol Cell Biol. 2003;23:5790-5802. [PubMed] [DOI] |
| 30. | Giaginis C, Daskalopoulou SS, Vgenopoulou S, Sfiniadakis I, Kouraklis G, Theocharis SE. Heat Shock Protein-27, -60 and -90 expression in gastric cancer: association with clinicopathological variables and patient survival. BMC Gastroenterol. 2009;9:14. [PubMed] [DOI] |