修回日期: 2012-06-30
接受日期: 2012-07-20
在线出版日期: 2012-08-08
目的: 观察siRNA沉默Cyclin E基因表达对肝癌HepG2、SMMC-7721和BEL-7402细胞增殖和侵袭能力的影响.
方法: 构建2个靶向Cyclin E基因siRNA载体, 转染人肝癌HepG2、SMMC-7721和BEL-7402细胞. RT-PCR、Western blot检测转染后HepG2、SMMC-7721和BEL-7402细胞Cyclin E基因mRNA和蛋白表达水平. CCK-8试验、软琼脂克隆形成实验检测HepG2、SMMC-7721和BEL-7402细胞增殖、克隆形成能力. 流式细胞术、transwell试验分别检测HepG2、SMMC-7721和BEL-7402细胞周期和侵袭能力.
结果: 构建的2个Cyclin E基因siRNA载体插入序列与所设计序列均一致; 转染HepG2、SMMC-7721和BEL-7402细胞后, 干扰1组、干扰2组与空白对照组和阴性对照组比较, Cyclin E mRNA和蛋白表达量均显著降低(P<0.05), 细胞生长速度延缓, 软琼脂细胞集落形成数、穿透细胞数均显著降低(P<0.05), S和G2/M期细胞比例减少, G0/G1期细胞比例增加.
结论: 沉默肝癌细胞Cyclin E表达水平, 可有效抑制细胞生长、增殖和侵袭能力.
引文著录: 刘慧涛, 王娜, 李敏, 臧文巧, 吴睿, 赵国强. siRNA沉默Cyclin E基因对肝癌HepG2、SMMC-7721和BEL-7402细胞增殖和侵袭能力的影响. 世界华人消化杂志 2012; 20(22): 2086-2091
Revised: June 30, 2012
Accepted: July 20, 2012
Published online: August 8, 2012
AIM: To observe the effect of RNAi-mediated silencing of the Cyclin E gene on the proliferation and invasion of HepG2, SMMC-7721 and BEL-7402 cells.
METHODS: Two vectors carrying siRNA targeting the Cyclin E gene were constructed and transfected into HepG2, SMMC-7721 and BEL-7402 cells. The mRNA and protein expression of Cyclin E were measured by RT-PCR and Western blot, respectively. CCK-8 assay and colony formation assay were employed to assess the proliferation and colony-forming ability of transfected HepG2, SMMC-7721 and BEL-7402. Flow cytometry (FCM) and transwell migration assay were used to evaluate cell cycle progression and migration of transfected cells.
RESULTS: Compared to the blank control group and negative control group, the expression of Cyclin E mRNA and protein was significantly decreased, the proliferation, colony-forming ability, and migration were suppressed significantly, and cell cycle was arrested in G0/G1 phase in two experimental groups.
CONCLUSION: Down-regulation of Cyclin E expression significantly inhibits the proliferation and migration of HepG2, SMMC-7721 and BEL-7402 cells.
- Citation: Liu HT, Wang N, Li M, Zang WQ, Wu R, Zhao GQ. RNAi-mediated silencing of the Cyclin E gene inhibits the proliferation and invasion of HepG2, SMMC-7721 and BEL-7402 cells. Shijie Huaren Xiaohua Zazhi 2012; 20(22): 2086-2091
- URL: https://www.wjgnet.com/1009-3079/full/v20/i22/2086.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v20.i22.2086
细胞周期调控机制紊乱导致的细胞生长失控是肿瘤发生的重要机制之一. Cyclin E是周期蛋白的一种, 在细胞的增殖分化及肿瘤发生中起重要作用[1]. 他能够与CDK2一起促进细胞周期的G1/S转换, 进而促进细胞分裂[2]. 研究发现, 异常情况下由于Cyclin E无顺序无规律的过度表达, 不断激活其下游蛋白, 驱使细胞异常增殖, 引起肿瘤[3]. 有研究表明Cyclin E过表达与肝癌的发生密切相关[4]. 因此, 本文构建靶向Cyclin E的siRNA载体, 转染肝癌HepG2、SMMC-7721和BEL-7402细胞, 观察沉默Cyclin E基因表达对细胞增殖、分裂和侵袭能力的影响.
人肝癌细胞株HepG2、SMMC-7721和BEL-7402(中国科学院上海生命科学研究院细胞资源中心); pGFP-V-RS载体(美国OriGene Technologies公司); 质粒DNA提取纯化试剂盒(北京天恩泽基因科技有限公司); 小量总RNA提取试剂盒(德国Qiagen公司); RPMI 1640培养基、小牛血清(美国GIBCO公司); 荧光定量PCR试剂盒(One Step SYBR® PrimeScript® RT-PCR Kit, 大连宝生物公司); 鼠抗Cyclin E单克隆抗体、辣根过氧化物酶标记的山羊抗鼠IgG抗体、鼠抗GAPDH抗体(美国Santa Cruz公司); LipofectAmineTM 2000(美国Invitrogen公司).
1.2.1 靶向Cyclin E基因siRNA载体的构建: 依据Cyclin E mRNA序列(NM_057182.1), 利用Dharmacon公司提供的在线软件siDESIGN筛选确定靶向Cyclin E基因的两个siRNA序列, 即951-969位(GCAAAAGGTTTCAGGGTAT)、1122-1140位(GGACAAAGCCCGAGCAAAG), 分别合成发卡样单链DNA, 退火后分别与siRNA载体pGFP-V-RS重组. 随机选取两个转化菌隔夜培养, 抽提质粒, 对插入序列进行测序. 得到重组载体pGFP-V-RS-siCE951和pGFP-V-RS-siCE1122.
1.2.2 肝癌细胞株的转染: 提取纯化GFP-V-RS-siCE951、pGFP-V-RS-siCE1122和pGFP-V-Con(无关序列siRNA阴性对照载体)质粒. 复苏HepG2、SMMC-7721和BEL-7402细胞, 按Invitrogen公司转染试剂LipofectAmineTM2000操作说明分别转染HepG2、SMMC-7721和BEL-7402细胞. 转染分4组: 干扰1组(转染pGFP-V-RS-siCE951)、干扰2组(转染pGFP-V-RS-siCE1122)、阴性对照组(转染pGFP-V-Con)和空白对照组(不转染任何质粒, 仅用脂质体处理). 转染12 h后换液, 加入含有抗生素的完全培养基37 ℃、50 mL/L CO2培养24 h进行以下实验.
1.2.3 RT-PCR检测Cyclin E mRNA表达: 使用Qiagen公司小量总RNA提取试剂盒提取各组转染细胞总RNA, AMV逆转录成cDNA. 使用宝生物公司的荧光定量PCR试剂盒扩增, Cyclin E扩增引物5'CGGGTCCACAGGGATGCGAAGGA3'和5'CAGGTGTGGGGATCAGGGAGCA3'. 内参GAPDH扩增引物5'GCCTTCCGTGTCCCCACTGC3'和5'CAATGCCAGCCCCAGCGTCA3'. 扩增反应条件: 94 ℃预变性20 s, 60 ℃ 60 s, 共40个循环. 每个标本均扩增5管, 以Cyclin E的表达量与内参GAPDH表达量的比值为Cyclin E的相对表达量.
1.2.4 Western blot检测Cyclin E蛋白表达: 裂解各组细胞, 用Bradford法测定上清中蛋白浓度以调整上样体积和上样量. SDS-PAGE凝胶100 V, 电泳1 h, 转膜, 置膜于25 mL封闭缓冲液中1 h. 1:800鼠抗Cyclin E单克隆抗体及鼠抗GAPDH, 室温孵育1-2 h. 1:2 000辣根过氧化物酶标记的山羊抗鼠IgG抗体, 室温孵育1 h, 显色, 成像扫描分析系统保存图像.
1.2.5 细胞增殖检测: 取各组细胞, 以1×104个细胞/孔细胞浓度接种各组细胞于96孔细胞培养板, 各组细胞均设置5个复孔, 接种后的1-5 d内, 采用CCK-8细胞增殖检测试剂盒, 测定各组细胞孔内的吸光度值(A), 记录数据, 计算平均值, 绘制各组细胞生长曲线.
1.2.6 流式细胞术分析细胞周期: 取各组对数生长期细胞, 胰酶消化, 用注射器将细胞用力打入5 mL 70%(预冷)乙醇中, 封口膜封口. 4 ℃固定过夜. 800 r/min 15 min收集细胞, PBS洗2次. 用0.4 mL PBS重悬细胞并转至Tube中轻轻吹打. 加RNase-A至终浓度50 μg/mL, 37 ℃水浴消化30 min. 加PI至终浓度65 μg/mL, 在冰浴中避光染色30 min. 300目尼龙网过滤, 上机检测. 每组细胞重复3次.
1.2.7 软琼脂克隆形成实验: 1:1混合1.2%的琼脂糖和2×RPMI 1640培养基, 加入2×抗生素和20%的小牛血清, 平皿中冷却凝固. 1:1混合0.7%的琼脂糖和2×DMEM培养基, 加入0.2 mL浓度为5×103个细胞/mL的细胞悬液, 注入上述平皿, 培养10-14 d取出培养板, 每组细胞随机选取10个低倍视野, 镜下计数形成的细胞克隆数(细胞数>50计为一个克隆)[5].
1.2.8 Transwell试验: 转染后48 h, 取各组细胞调整为2×105/mL, 在上室中加入200 μL细胞悬液, 下室加入500 μL含趋化因子的培养基, 每组设5个复孔. 37 ℃、50 mL/L CO2培养48 h后取出, 小心用棉签擦去基质胶和上室内的细胞, 结晶紫染色, 显微镜观察、取10个视野, 计数取平均值.
统计学处理 所有实验数据均使用SPSS16.0统计软件进行分析处理, 所有数据用mean±SD表示, 结果采用单因素方差(One-way ANOVA)分析, 以P<0.05为有显著性差异.
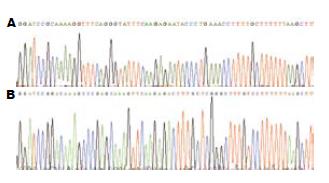
两种退火产物与载体重组转化后均得到大量转化菌, 对重组质粒中目的基因进行测序, 结果与所设计的发卡样单链DNA序列一致(图1). 说明构建得重组载体pGFP-V-RS-siCE951和pGFP-V-RS-siCE1122.
3种肝癌细胞的干扰1组、干扰2组、阴性对照组和空白对照组细胞Cyclin E mRNA的相对表达量见表1. 比较3种肝癌细胞的干扰1组、干扰2组与对照组, Cyclin E mRNA的相对表达量都显著降低, 差异均有非常显著性(HepG2: P<0.01, F = 27.39; SMMC-7721: P<0.01, F = 21.04; BEL-7402: P<0.01, F = 18.62). 说明干扰1组、2组肝癌HepG2、SMMC-7721和BEL-7402细胞中Cyclin E mRNA表达均受到显著抑制.
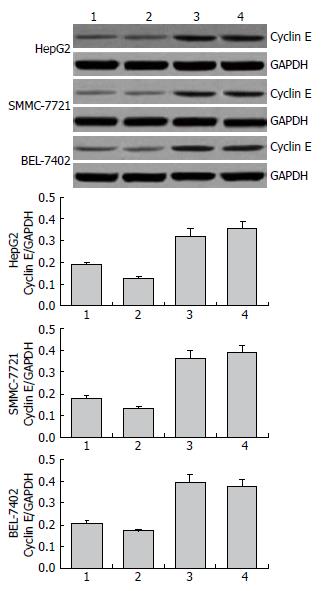
干扰1组、干扰2组、空白对照组和阴性对照组细胞在大约54 kDa处均有免疫印迹出现, 其中空白对照组和阴性对照组的免疫印迹显著强于干扰1组和干扰2组(图2). 说明设计靶向Cyclin E基因siRNA载体能够有效干扰肝癌HepG2、SMMC-7721和BEL-7402细胞Cyclin E蛋白的表达.
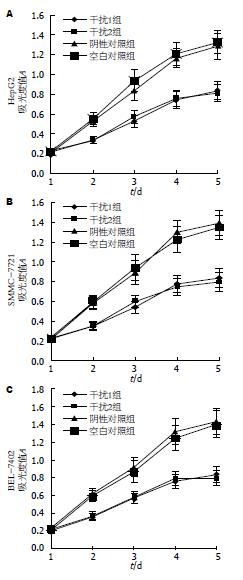
各组细胞的生长曲线见图3, 统计学分析显示, 比较空白对照组与阴性对照组细胞A值得出差异无显著性(P>0.05); 干扰1组、干扰2组与2个对照组比较, 在3、4、5 d细胞孔内的A值显著减少, 差异有显著性(P<0.05). 说明抑制肝癌HepG2、SMMC-7721和BEL-7402细胞Cyclin E基因表达, 可以降低这些细胞的生长速度.
细胞周期检测数据见表2. 3种肝癌细胞干扰1组和干扰2组各期细胞比例与2个对照组比较, S和G2/M期细胞比例均减少, G0/G1期细胞比例均增加, 差异有显著性(P<0.05). 说明在肝癌HepG2、SMMC-7721和BEL-7402细胞中抑制Cyclin E基因表达可以改变细胞周期, 使其S和G2/M期细胞比例减少, G0/G1期细胞比例增加.
| 分组 | HepG2 | SMMC-7721 | BEL-7402 | ||||||
| G0-G1 | S | G2-M | G0-G1 | S | G2-M | G0-G1 | S | G2-M | |
| 干扰1组 | 74.37±2.51b | 20.62± 0.41b | 5.01±0.17b | 68.27±2.36b | 24.48±0.38b | 7.25±0.14b | 70.26±2.66b | 22.05±0.56b | 7.69±0.21b |
| 干扰2组 | 70.49±2.31b | 22.42± 0.54b | 7.09±0.35b | 69.28±2.31b | 23.71±0.54b | 7.01±0.21b | 71.39±2.92b | 21.12±0.64b | 7.49±0.27b |
| 阴性对照组 | 61.22± 1.99 | 28.15± 0.53 | 10.63±0.21 | 58.19±2.07 | 30.29±0.51 | 11.52±0.22 | 62.09±3.15 | 27.15±0.42 | 10.76±0.32 |
| 空白对照组 | 62.69± 3.67 | 27.43± 0.57 | 9.88±0.44 | 57.35±2.28 | 29.81±0.62 | 12.84±0.33 | 60.15±2.33 | 28.43±0.64 | 11.42±0.39 |
3种肝癌细胞干扰1组、干扰2组、阴性对照组和空白对照组平均细胞集落形成数见表3. 空白对照组与阴性对照组平均集落形成数比较, 差异均无显著性(P>0.05); 干扰1组和干扰2组与2个对照组比较, 平均集落形成数均显著减少, 差异有显著性(P<0.05). 说明抑制肝癌HepG2、SMMC-7721和BEL-7402细胞Cyclin E基因表达, 可以降低细胞软琼脂集落形成能力.
干扰1组、干扰2组、阴性对照组和空白对照组平均穿透细胞数见表4. 空白对照组与阴性对照组平均穿透细胞数比较, 差异无显著性(P>0.05); 干扰1组和干扰2组与2个对照组比较, 平均穿透细胞数显著减少, 差异有显著性(P<0.01). 说明抑制肝癌HepG2、SMMC-7721和BEL-7402细胞Cyclin E基因表达, 可以降低细胞侵袭转移能力.
肿瘤的发生发展是一个多因素参与、多阶段发生和多基因改变的过程. 研究表明, 细胞周期调控机制紊乱导致的细胞增殖失控被认为是肿瘤发生发展的重要机制之一. 细胞周期是一个高度有序的进程, 受到各种蛋白质的严格调控, 周期蛋白(cyclin)保证细胞周期的正常进行具有重要作用[6,7].
Cyclin E是一种G1期周期蛋白, 1991年由美国学者Koff首先发现[8]. Cyclin E促进肿瘤的发生与其在细胞周期中的调控作用有关. Cyclin E是促进细胞周期G1/S转换的重要调节因子, Cyclin E的过度表达, 导致细胞异常增殖, 引起肿瘤[9-11]. RNA干扰技术可以特异阻断或降低其靶向基因的表达, 在抑制肿瘤生长的基础研究方面取得了一定的进展[12-14]. Liang等[15]报道, siRNA靶向沉默Cyclin E基因, 可以有效阻止乳腺癌细胞在裸鼠中的生长. Li等[16]报道, siRNA靶向Cyclin E可以抑制肝癌细胞Cyclin E的表达, 且可以加速体外培养的肝癌细胞系凋亡, 阻止细胞的分裂.
本研究采用RNA干扰技术观察了Cyclin E沉默对肝癌细胞株HepG2、SMMC-7721和BEL-7402增殖的影响. 实验结果显示, 构建的Cyclin E基因siRNA载体转染HepG2、SMMC-7721和BEL-7402细胞后, 干扰1组、干扰2组与空白对照组和阴性对照组比较, Cyclin E mRNA的相对表达量显著降低, CyclinE蛋白表达量明显下降, 细胞生长速度延缓, S、G2、M期细胞比例减少, G0、G1期细胞比例增加, 软琼脂细胞集落形成数、穿透细胞数显著减少.
分析试验结果, 我们认为沉默Cyclin E可使肝癌细胞Cyclin E mRNA和蛋白表达均显著下降, 周期阻滞于G0/G1期, 阻止了肝癌细胞的分裂速度, 进而延缓肝癌细胞生长. Transwell试验还表明, 沉默Cyclin E还降低了细胞的侵袭能力, Cyclin E和细胞侵袭能力的关系还有待进一步的研究. 本研究为深入探索肝癌的发病机制和靶向治疗积累实验资料.
Cyclin E蛋白是一种重要的周期蛋白, 是促进细胞周期G1/S转换的重要调节因子. Cyclin E在多种恶性肿瘤中存在过表达, 阻断其表达可抑制肿瘤细胞的生长,有望成为肿瘤治疗的候选靶点.
代智, 副研究员, 复旦大学附属中山医院肝癌研究所
研究发现, 异常情况下由于Cyclin E无顺序无规律的过度表达, 不断激活其下游蛋白, 驱使细胞异常增殖, 引起肿瘤. RNA干扰技术可以特异阻断或降低其靶向基因的表达, 在抑制肿瘤生长的基础研究方面取得了一定的进展.
Liang等报道, siRNA靶向沉默Cyclin E基因, 可以有效阻止乳腺癌细胞在裸鼠中的生长. Li等报道, siRNA靶向Cyclin E可以抑制肝癌细胞Cyclin E的表达, 且可以加速体外培养的肝癌细胞系凋亡, 阻止细胞的分裂.
Cyclin E蛋白的RNAi在肝癌SMMC-7721、BEL-7402中未有文献报道. 本研究采用Cyclin E基因siRNA载体转染人肝癌HepG2、SMMC-7721和BEL-7402细胞, RT-PCR、Western blot、CCK-8试验、软琼脂克隆形成实验、流式细胞术、transwell试验等来检测Cyclin E基因沉默后, 对目的细胞生长、增殖和侵袭能力的影响, 具有一定的创新性.
siRNA可以有效抑制肝癌细胞株HepG2、SMMC-7721和BEL-7402中Cyclin E的表达, 有效抑制细胞生长、增殖和侵袭能力.
本文选题尚可, 具有一定的学术价值.
编辑: 曹丽鸥 电编:闫晋利
| 1. | Stamatakos M, Palla V, Karaiskos I, Xiromeritis K, Alexiou I, Pateras I, Kontzoglou K. Cell cyclins: triggering elements of cancer or not? World J Surg Oncol. 2010;8:111. [PubMed] [DOI] |
| 2. | Freemantle SJ, Dmitrovsky E. Cyclin E transgenic mice: discovery tools for lung cancer biology, therapy, and prevention. Cancer Prev Res (Phila). 2010;3:1513-1518. [PubMed] [DOI] |
| 3. | Santamaria D, Ortega S. Cyclins and CDKS in development and cancer: lessons from genetically modified mice. Front Biosci. 2006;11:1164-1188. [PubMed] [DOI] |
| 4. | Tsuji T, Miyazaki M, Fushimi K, Mihara K, Inoue Y, Ohashi R, Ohtsubo M, Hamazaki K, Furusako S, Namba M. Cyclin E overexpression responsible for growth of human hepatic tumors with p21WAF1/CIP1/SDI1. Biochem Biophys Res Commun. 1998;242:317-321. [PubMed] [DOI] |
| 5. | 胡 志亮, 谭 德明, 侯 周华, 谢 萍, 刘 国珍, 欧阳 奕, 刘 菲, 刘 洪波. HBx基因缺失突变体HBx-d382对L02细胞增殖及非锚定依赖生长能力的影响. 世界华人消化杂志. 2010;18:1109-1114. [DOI] |
| 6. | Mazumder S, Plesca D, Almasan A. A jekyll and hyde role of cyclin E in the genotoxic stress response: switching from cell cycle control to apoptosis regulation. Cell Cycle. 2007;6:1437-1442. [PubMed] [DOI] |
| 7. | Lopez-Beltran A, MacLennan GT, Montironi R. Cyclin E as molecular marker in the management of breast cancer: a review. Anal Quant Cytol Histol. 2006;28:111-114. [PubMed] |
| 8. | Koff A, Cross F, Fisher A, Schumacher J, Leguellec K, Philippe M, Roberts JM. Human cyclin E, a new cyclin that interacts with two members of the CDC2 gene family. Cell. 1991;66:1217-1228. [PubMed] [DOI] |
| 9. | Bahnassy AA, Zekri AR, Loutfy SA, Mohamed WS, Moneim AA, Salem SE, Sheta MM, Omar A, Al-Zawahry H. The role of cyclins and cyclin dependent kinases in development and progression of hepatitis C virus-genotype 4-associated hepatitis and hepatocellular carcinoma. Exp Mol Pathol. 2011;91:643-652. [PubMed] [DOI] |
| 10. | Schildkraut JM, Moorman PG, Bland AE, Halabi S, Calingaert B, Whitaker R, Lee PS, Elkins-Williams T, Bentley RC, Marks JR. Cyclin E overexpression in epithelial ovarian cancer characterizes an etiologic subgroup. Cancer Epidemiol Biomarkers Prev. 2008;17:585-593. [PubMed] [DOI] |
| 11. | Zhou YJ, Xie YT, Gu J, Yan L, Guan GX, Liu X. Overexpression of cyclin E isoforms correlates with poor prognosis in rectal cancer. Eur J Surg Oncol. 2011;37:1078-1084. [PubMed] [DOI] |
| 12. | Kim DH, Rossi JJ. Strategies for silencing human disease using RNA interference. Nat Rev Genet. 2007;8:173-184. [PubMed] [DOI] |
| 13. | Jankovic R, Radulovic S, Brankovic-Magic M. siRNA and miRNA for the treatment of cancer. J BUON. 2009;14 Suppl 1:S43-S49. [PubMed] |
| 14. | Gondi CS, Rao JS. Concepts in in vivo siRNA delivery for cancer therapy. J Cell Physiol. 2009;220:285-291. [PubMed] [DOI] |
| 15. | Liang Y, Gao H, Lin SY, Goss JA, Brunicardi FC, Li K. siRNA-based targeting of cyclin E overexpression inhibits breast cancer cell growth and suppresses tumor development in breast cancer mouse model. PLoS One. 2010;5:e12860. [PubMed] [DOI] |
| 16. | Li K, Lin SY, Brunicardi FC, Seu P. Use of RNA interference to target cyclin E-overexpressing hepatocellular carcinoma. Cancer Res. 2003;63:3593-3597. [PubMed] |