修回日期: 2010-12-14
接受日期: 2010-12-21
在线出版日期: 2011-02-08
目的: 研究胃癌细胞SGC7901中RUNX2的表达, 以及siRNA沉默RUNX2的表达后, 对人胃癌细胞SGC7901的RUNX2的表达、细胞增殖和凋亡的影响.
方法: 将细胞分为以下3组: 空白对照组、空载体组、实验组(即转染RUNX2 siRNA组). 用针对RUNX2特异靶点的siRNA转染胃癌细胞SGC7901, 运用RT-PCR、Western blot检测RUNX2 mRNA和蛋白水平的变化; MTT检测细胞增殖的影响; 流式细胞仪检测细胞凋亡的变化. 所有实验均重复3次.
结果: 在胃癌细胞SGC7901中存在RUNX2的表达, siRNA沉默RUNX2的表达后, 与空白对照组相比, RUNX2在mRNA(0.27±0.068 vs 0.45±0.058, F = 75.6, P<0.01)和蛋白水平(F = 123.8, P<0.001)的表达均下降, 差异有显著统计学意义. siRNA转染24、48、72 h, MTT检测胃癌细胞SGC7901的增殖率与空白对照组相比明显下降(0.23±0.039 vs 0.32±0.012; 0.31±0.037 vs 0.45±0.074; 0.52±0.021 vs 0.72±0.006; F = 173.744、14.012、253.145; 均P<0.001), 流式细胞术检测RUNX2 siRNA组72 h细胞凋亡明显高于空白对照组及空载体组(45.65%±0.64% vs 4.46%±0.27%, 4.23%±0.33%, 均P<0.01).
结论: 胃癌细胞SGC7901中存在RUNX2的表达, siRNA抑制RUNX2的表达可抑制胃癌细胞的增殖, 促进凋亡, RUNX2可望成为胃癌基因治疗的新的靶点.
引文著录: 向春香, 陈锋, 袁静萍, 黄晓东. RUNX2-siRNA对胃癌细胞SGC7901增殖和凋亡的影响. 世界华人消化杂志 2011; 19(4): 338-343
Revised: December 14, 2010
Accepted: December 21, 2010
Published online: February 8, 2011
AIM: To detect the expression of runt-related transcription factor gene 2 (RUNX2) in human gastric cancer cell line SGC7901 and to investigate the influence of small interfering RNA (siRNA)-mediated silencing of the RUNX2 gene on the proliferation and apoptosis of SGC7901 cells.
METHODS: SGC7901 cells were divided into three groups: blank control group, negative control group (transfected with an empty vector), and experiment group (transfected with RUNX2 siRNA). After SGC7901 cells were transfected with RUNX2 siRNA, the mRNA and protein expression of RUNX2 was examined by RT-PCR and Western blot, respectively; cell proliferation was evaluated by MTT assay; and cell apoptosis was detected by flow cytometry (FCM).
RESULTS: Compared with cells of the blank control group, the expression of RUNX2 mRNA (0.27 ± 0.068 vs 0.45 ± 0.058, F = 75.6, P < 0.01) and protein (F = 123.8, P < 0.001) was down-regulated in cells transfected with RUNX2 siRNA. At 24, 48, and 72 h after transfection, the proliferation rates of SGC7901 cells transfected with RUNX2 siRNA were significantly lower than those of non-transfected cells (0.23 ± 0.039 vs 0.32 ± 0.012; 0.31 ± 0.037 vs 0.45 ± 0.074; 0.52 ± 0.021 vs 0.72 ± 0.006; F = 173.744, 14.012, 253.145; all P < 0.001). The apoptosis rate of SGC7901 cells transfected with RUNX2 siRNA was significantly higher than those of cells of the blank control group and negative control (45.65% ± 0.64% vs 4.46% ± 0.27%, 4.23% ± 0.33%, both P < 0.01).
CONCLUSION: RUNX2 expression was detected in SGC7901 cells. SiRNA-mediated silencing of the RUNX2 gene can inhibit proliferation and induce apoptosis in SGC7901 cells. RUNX2 may be a new gene therapy target for gastric cancer.
- Citation: Xiang CX, Chen F, Yuan JP, Huang XD. SiRNA-mediated silencing of the RUNX2 gene inhibits proliferation and induces apoptosis in human gastric cancer cell line SGC7901. Shijie Huaren Xiaohua Zazhi 2011; 19(4): 338-343
- URL: https://www.wjgnet.com/1009-3079/full/v19/i4/338.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i4.338
胃癌作为严重威胁人类健康的恶性肿瘤之一, 据估计, 其破坏力在世界范围内居恶性疾病的第2位[1], 其发生具有明显的地理分布, 中国是高发的国家之一[2,3]. 同其他肿瘤一样, 胃癌在发生、发展和转移的过程中与多种基因有关, 包括癌基因激活和抑癌基因失活[4,5]. 迄今, 有关胃癌的发生与发展的机制尚未取得突破性的进展. 人类相关转录基因2(runt-related transcription factor 2, RUNX2), 又名核心结合因子(core banding factor 1, Cbfa 1)[6], 是RUNX转录因子家族中的成员之一, 他调控骨组织的分化和形成[7]及正常发育中T淋巴细胞的分化[8,9]. 近来发现, 其在乳腺癌、黑色素瘤及前列腺癌中表达[10-12], 但在胃癌组织中关于他的表达并未见相关文献报道. 本研究拟通过细胞生物学和分子病理学的方法, 观察胃癌细胞株SGC-7901中RUNX2的表达, 并研究其对胃癌细胞增殖和凋亡的影响.
胃癌SGC-7901细胞购自武汉大学细胞库, 由本实验室保存. RMPI 1640、小牛血清及DMEM培养基购自大连宝生物工程有限公司; TRIzol购自Invitrogen公司; RT-PCR试剂盒(TaKaRa)购自大连宝生物工程有限公司; TRANSfection转染试剂购自TIANGEN公司; siRNA及引物序列由赛百胜引物合成公司合成; RUNX2单克隆抗体购自美国Santa Cruz公司; 持久型ECL化学发光显影试剂盒购自上海杰美基因医药科技有限公司; 膜封闭液购自北京天为时代生物技术有限责任公司; MTT购自武汉凌飞科技有限公司; 凋亡检测试剂盒购自碧云天生物技术有限公司.
1.2.1 细胞培养基及培养条件: 在37 ℃、50 mL/L CO2、饱和湿度条件下用含100 mL/L FBS的RPMI 1640培养基培养, 细胞传代采用2.5 g/L的胰酶+0.02%的EDTA, 每3-5 d传代1次.
1.2.2 分组: 本实验所有指标检测都按如下分组: 正常对照组, 空载体组, 实验组(即转染RUNX2 siRNA组), 转染上述研究对象的同时另转染一组FAM荧光标记的无义序列siRNA, 以测定转染效率. 所使用的RUNX2 siRNA序列为Sense: 5'-CAAGGACAGAGUCAGAUUAUU-3'; Antisense: 5'-UAAUCUGACUCUGUCCUUGUU-3'[13].
1.2.3 siRNA的转染: 处于对数生长期的SGC-7901细胞常规消化后接种于6孔细胞培养板中, 在0.5 mL无血清, 不含抗生素的DMEM培养基中培养, 转染时细胞密度为70%-90%, 每孔细胞分别用50 μL无血清培养基稀释siRNA和TRANSfection试剂, 温和混匀, 室温放置5 min, 将稀释的siRNA和TRANSfection试剂混合在一起, 室温保温20 min后直接将100 μL混合物加入到每孔, 摇动培养板, 轻轻混匀, 在CO2培养箱中37 ℃培养4-6 h后更换正常含血清培养基培养24 h. 转染中, DNA(μg)与TRANSfection(μL)的比例为1:2.5, siRNA的工作浓度为100 nmol/L[14].
1.2.4 细胞RNA的提取和引物设计: 用TRIzol试剂从细胞中提取总RNA, 样品纯度和浓度经核酸测定仪测定, A260/280 = 1.8-2.0. 分别设计RUNX2和β-actin的上游和下游引物, 各引物序列见表1.
| 名称 | 正义 | 反义 | 产物长度(bp) |
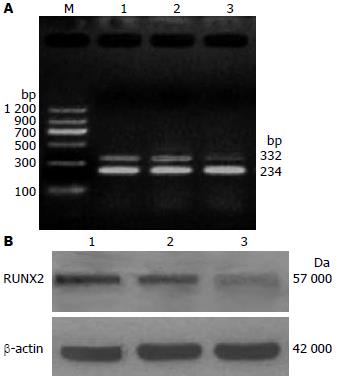
| β-actin | 5'-TGCTGTCCCTCTACGCCTCT-3' | 5'-GCTTCTCCTTGATGTCGC-3' | 234 |
| RUNX2 | 5'-ACCCACGAATGCACTATCCA-3' | 5'-CACAACTGCGACTACCTTG-3' | 332 |
1.2.5 RT-PCR及灰度分析: cDNA合成, 步骤如下: 取MgCl2 2 μL, 10×RT Buffer 1 μL, dNTP混合液(各10 mmol/L)1 μL, RNase inhibitor 0.25 μL, AMV逆转录酶0.5 μL, Oligo(dT)0.5 μL, RNA 1 μL(≤500 ng), RNase Free dH2O 3.75 mL, 总体积10 μL, 离心后混匀, 42 ℃ 30 min, 99 ℃ 5 min, 5 ℃ 5 min. 从上述反应体系中取2 μL, 加5×PCR Buffer 4 μL, TaKaRa Taq酶(5 U/μL)0.1 μL, 50 mmol/L的上游、下游引物各0.5 μL, 灭菌蒸馏水12.9 μL, 总体积为20 μL. 反应条件为: 95 ℃预变性5 min后开始循环, 95 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸45 s, 30个循环, 最后72 ℃延伸5 min, 目的引物与β-actin用一管法进行PCR. PCR产物经1.5%的琼脂糖凝胶电泳检测, RT-PCR结果经凝胶成像软件分析, 检测各组平均吸光度(A)值, 计算目的基因与β-actin的比值, 得到目的基因的相对含量.
1.2.6 Western blot: 转染siRNA后48-72 h, 用预冷的PBS(0.01 mol/L, pH7.4)洗涤细胞2次, 加入100 μL预冷的蛋白裂解液(0.1 mol/L NaCl, 0.01 mol/L Tris-HCl pH7.6, 0.001 mol EDTA pH8.0, 1 mg/L Aprotinin, 100 mg/L PMSF, 1% NP40), 置冰浴中作用60 min, 4 ℃、13 000 r/min×15 min, 取上清20 μL进行蛋白定量, 其余与等体积2×SDS上样缓冲液(Tris 100 mmol/L, DTT 200 mmol/L, SDS 4%, 溴酚蓝0.2%, 甘油20%)混合后煮沸10 min, 缓慢冷却后分装并在-80 ℃冰箱中保存. 制备SDS-PAGE胶, 蛋白浓度80 μg/泳道进行SDS-PAGE电泳; 将蛋白质电转移到NC膜上, 5%脱脂牛奶37 ℃封闭1 h, 加入一抗(1:500, V/V)4 ℃孵育过夜, 加入二抗(1:1 000, V/V), 室温下杂交1 h, 洗膜后用ECL试剂盒暗室显影.
1.2.7 MTT法分析细胞增殖活性: 处于对数生长期的SGC-7901细胞常规消化, 接种于96孔板, 接种量4 000个(200 μL)/孔, 12 h后, 常规转染siRNA, 分别于转染前、转染后24、48、72 h行MTT检测: 将浓度为5 g/L的MTT(Sigma公司)以每孔20 μL加入待测的96孔板内, 37 ℃培养4 h, 弃上清液, 每孔加DMSO 200 μL并振荡15 min, 最后用紫外分光光度计570 nm波长测定各孔的A值, 取每组8孔的均值. 肿瘤细胞增殖率计算方法: 用未转染siRNA的细胞作为对照, 求出实验组肿瘤细胞增殖率. 肿瘤细胞增殖率 = (转染细胞A值/空白对照细胞A值)×100%.
1.2.8 流式细胞术检测细胞增殖凋亡: 转染72 h后, 常规制备单细胞悬液, 用1×Buffer A洗涤细胞1次(离心2 000 r/min, 5 min), 收集并调整细胞浓度为1×106/mL, 加入9倍体积的700 mL/L乙醇, 于-20 ℃固定12 h, 离心收集细胞后, 除去乙醇, 细胞重悬于500 μL Buffer A中, 加入RNaseA使其终浓度为0.25 g/L, 37 ℃, 反应30 min, 加入5 μL PI 室温避光染色30 min, 按Annexin V-FITC PI染色试剂盒说明操作, 以Annexin V阳性/PI阴性判断为早期凋亡, Annexin V阳性/PI阳性判断为晚期凋亡.
统计学处理 用SPSS13.0统计软件进行统计学分析, 所有指标采用ANOVA单因素方差分析, 检验水准α = 0.05.
培养的SGC-7901细胞贴壁生长, 在倒置显微镜下观察细胞呈不规则多边形, 折光性较强, 透明度大. 转染SGC-7901细胞后, 荧光显微镜下转染阳性细胞可看到绿色荧光; 在同一视野相差显微镜下可看到全部细胞. 转染率测定发现转染效率在70%以上, 完全可满足实验的要求.
在胃癌细胞系SGC-7901中, 我们发现在SGC-7901中存在RUNX2的表达. 使用RUNX2 siRNA转染SGC-7901细胞72 h后, 我们发现在转染72 h后, 实验组RUNX2的mRNA表达和空白对照组及空载体组相比明显降低(图1A), 差异有显著统计学意义(0.27±0.068 vs 0.45±0.058, 0.46±0.041, F = 75.6, P<0.01). 说明转染RUNX2 siRNA后, 在U251细胞中, RUNX2 siRNA直接反向作用于RUNX2转录因子, 并显著降低RUNX2的表达; 使用Western blot检测RUNX2蛋白水平表达变化, 结果发现RUNX2蛋白表达也出现了下降(图1B), 且与空白对照组及空载体组相比, 差异有显著统计学意义(F = 123.8, P<0.001).
转染24、48、72 h后, 空白对照组及空载体组细胞的生长未受到明显抑制, 而实验组细胞的生长在转染24 h后即出现明显抑制, 且随转染时间的延长, 抑制程度越明显. 与对照组、空载体组比较, 差异具有显著统计学意义(P<0.001, 表2). 说明降低RUNX2表达后, 可明显降低SGC-7901细胞增殖活性.
| 分组 | 24 h | 48 h | 72 h |
| 空白对照组 | 0.32±0.012 | 0.45±0.074 | 0.72±0.006 |
| 空载体组 | 0.34±0.018 | 0.43±0.015 | 0.71±0.007 |
| 实验组 | 0.23±0.039 | 0.31±0.037 | 0.52±0.021 |
| F值 | 173.744 | 14.012 | 253.145 |
| P值 | <0.001 | <0.001 | <0.001 |
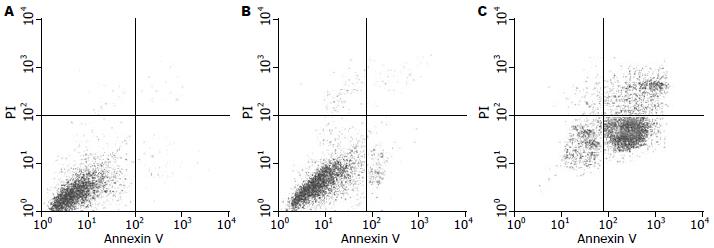
流式细胞术检测用针对RUNX2特异靶点的siRNA转染SGC-7901细胞72 h后, Annexin V/PI标记双染色流式细胞仪检测显示, 空白对照组和空载体组的凋亡率无明显差异(P>0.05), 转染RUNX2 siRNA的SGC-7901细胞出现明显凋亡, 处理72 h后凋亡率达45.65%±0.64%, 与空白对照组(4.46%±0.27%)及空载体组(4.23%±0.33%)相比, 有显著性差异(P<0.01, 图2).
RUNX2是RUNX转录因子家族中的成员之一, 他调控骨组织的分化和形成及在正常发育中T淋巴细胞的分化. RUNX转录因子在肿瘤的生物学行为中所起的作用是不同的, RUNX1表达失调可引起白血病[15], 在小鼠实验中, RUNX2的表达增强可促进淋巴瘤的发生[8], 而RUNX3在胃癌的发生中则起着肿瘤抑制子的作用[16,17]. 作为一个支架蛋白, 他们可以和许多辅助因子及转录因子相互作用形成集合子, 但取决于他们的组成、启动基因的活化或基因阻遏[18].
RUNX2纯合子缺失的小鼠在出生后很快就因呼吸衰竭而死亡, 其主要原因是由于缺乏骨及成骨细胞而导致骨骼畸形[19,20]. 杂合子小鼠显示骨骼异常类似于观察到的人类遗传疾病, 如锁骨、颅骨发育异常, 这类疾病的发生是由于RUNX2基因突变引起[21]. 同在成骨细胞发育早期RUNX2促进其形态形成相反, 在成骨细胞分化的晚期RUNX2对其起抑制作用[22]. RUNX2最初是在胸腺T淋巴细胞的发育过程中出现的, 但他的出现是受限制的, 随后发现, 他在血细胞、骨及上皮的形成中起调控作用[23]. 近来发现, 其在乳腺癌、黑色素瘤及前列腺癌中表达. 目前RUNX2在肿瘤方面的作用逐步被发现, RUNX2在不同的肿瘤中表现不同的作用, 如促进增殖、抑制凋亡、促血管生成、促进侵袭转移等. 在我们的研究中运用RT-PCR检测发现, 在胃癌细胞SGC-7901中存在RUNX2的表达, 但目前未见相关文献报道.
RNA干扰是自然界生物体的一种遗传现象, 可以诱导目的基因沉默, 高效、特异地抑制目的基因的表达. 小分子干扰RNA具有许多传统方法无法比拟的优势, 包括特异性、高效性和放大效应, 因此作为肿瘤术后化疗或放疗基础上的一种附加治疗, 是靶向封闭目的基因的最佳选择, 具有广阔的应用前景[24,25]. 目前国内外针对肝癌、胃癌、宫颈癌等恶性肿瘤特异性靶基因RNA干扰治疗研究已广泛展开[26-28].
本研究应用RNA干扰技术, 将针对RUNX2特异靶点的小干扰RNA转染胃癌细胞株SGC-7901, 我们选用胃癌细胞系SGC-7901, 是1981年建立的一株胃癌淋巴结转移癌细胞系, 该细胞系凋亡率低[29], 是非常符合本研究目的的模型材料. RUNX2 siRNA转染后, RUNX2的表达在mRNA和蛋白水平的表达均下降, 说明转染是成功的, RUNX2 siRNA能够抑制RUNX2的表达. 通过对胃癌细胞增殖活性和凋亡的检测, 发现转染RUNX2 siRNA组细胞增殖能力降低, 细胞生长受到抑制, 细胞凋亡增加. 这说明RUNX2可能参与了胃癌的发生发展, 但具体的发生机制仍有待进一步研究.
总之, RUNX2的表达在胃癌的发生中起重要作用, 利用RNA干扰技术, 将针对RUNX2的siRNA转染胃癌细胞后能明显降低RUNX2蛋白表达, 细胞生长被抑制, 凋亡增加, RUNX2可能成为胃癌靶向基因治疗的新选择.
胃癌在世界范围内仍是引起高死亡率的肿瘤之一, 其发生发展非常复杂, 涉及到多种免疫与分子机制, 与多种基因有关, 包括癌基因激活和抑癌基因失活. 迄今, 有关胃癌的发生与发展的机制尚未取得突破性的进展.
杜雅菊, 主任医师, 哈尔滨医科大学附属第二医院消化内科; 王鲁平, 主任医师, 中国人民解放军北京军区总医院病理科
本文发现在胃癌细胞SGC7901中存在RUNX2的表达, 应用RUNX2-siRNA抑制胃癌细胞SGC7901中RUNX2的表达, 结果发现胃癌细胞的增殖率降低, 细胞凋亡增加, 说明RUNX2参与了胃癌细胞的发生发展.
本文通过分子生物学的方法研究在胃癌细胞SGC7901中RUNX2的表达, 并探讨RUNX2在胃癌发生发展中的作用, 可望为胃癌的个体化治疗提供新的思路和干预靶点.
本文选题恰当, 论据充分, 具有一定的理论及预期临床意义.
编辑: 李薇 电编:何基才
| 1. | NCCN. NCCN Clinical Practice Guidelines in Oncology. NCCN online, 2010, cited 2011. Available from: www.nccn.org. |
| 2. | Corley DA, Buffler PA. Oesophageal and gastric cardia adenocarcinomas: analysis of regional variation using the Cancer Incidence in Five Continents database. Int J Epidemiol. 2001;30:1415-1425. [PubMed] [DOI] |
| 4. | Hoshi T, Sasano H, Kato K, Yabuki N, Ohara S, Konno R, Asaki S, Toyota T, Tateno H, Nagura H. Immunohistochemistry of Caspase3/CPP32 in human stomach and its correlation with cell proliferation and apoptosis. Anticancer Res. 1998;18:4347-4353. [PubMed] |
| 6. | Liu W, Toyosawa S, Furuichi T, Kanatani N, Yoshida C, Liu Y, Himeno M, Narai S, Yamaguchi A, Komori T. Overexpression of Cbfa1 in osteoblasts inhibits osteoblast maturation and causes osteopenia with multiple fractures. J Cell Biol. 2001;155:157-166. [PubMed] [DOI] |
| 7. | Franceschi RT, Xiao G. Regulation of the osteoblast-specific transcription factor, Runx2: responsiveness to multiple signal transduction pathways. J Cell Biochem. 2003;88:446-454. [PubMed] [DOI] |
| 8. | Vaillant F, Blyth K, Andrew L, Neil JC, Cameron ER. Enforced expression of Runx2 perturbs T cell development at a stage coincident with beta-selection. J Immunol. 2002;169:2866-2874. [PubMed] |
| 9. | Stock M, Schäfer H, Stricker S, Gross G, Mundlos S, Otto F. Expression of galectin-3 in skeletal tissues is controlled by Runx2. J Biol Chem. 2003;278:17360-17367. [PubMed] [DOI] |
| 10. | Barnes GL, Javed A, Waller SM, Kamal MH, Hebert KE, Hassan MQ, Bellahcene A, Van Wijnen AJ, Young MF, Lian JB. Osteoblast-related transcription factors Runx2 (Cbfa1/AML3) and MSX2 mediate the expression of bone sialoprotein in human metastatic breast cancer cells. Cancer Res. 2003;63:2631-2637. [PubMed] |
| 11. | Brubaker KD, Vessella RL, Brown LG, Corey E. Prostate cancer expression of runt-domain transcription factor Runx2, a key regulator of osteoblast differentiation and function. Prostate. 2003;56:13-22. [PubMed] [DOI] |
| 12. | Riminucci M, Corsi A, Peris K, Fisher LW, Chimenti S, Bianco P. Coexpression of bone sialoprotein (BSP) and the pivotal transcriptional regulator of osteogenesis, Cbfa1/Runx2, in malignant melanoma. Calcif Tissue Int. 2003;73:281-289. [PubMed] [DOI] |
| 13. | Zhang HY, Jin L, Stilling GA, Ruebel KH, Coonse K, Tanizaki Y, Raz A, Lloyd RV. RUNX1 and RUNX2 upregulate Galectin-3 expression in human pituitary tumors. Endocrine. 2009;35:101-111. [PubMed] [DOI] |
| 14. | Vladimirova V, Waha A, Lückerath K, Pesheva P, Probstmeier R. Runx2 is expressed in human glioma cells and mediates the expression of galectin-3. J Neurosci Res. 2008;86:2450-2461. [PubMed] [DOI] |
| 15. | Ito Y. Oncogenic potential of the RUNX gene family: 'overview'. Oncogene. 2004;23:4198-4208. [PubMed] [DOI] |
| 18. | Durst KL, Hiebert SW. Role of RUNX family members in transcriptional repression and gene silencing. Oncogene. 2004;23:4220-4224. [PubMed] [DOI] |
| 19. | Komori T, Yagi H, Nomura S, Yamaguchi A, Sasaki K, Deguchi K, Shimizu Y, Bronson RT, Gao YH, Inada M. Targeted disruption of Cbfa1 results in a complete lack of bone formation owing to maturational arrest of osteoblasts. Cell. 1997;89:755-764. [PubMed] [DOI] |
| 20. | Otto F, Thornell AP, Crompton T, Denzel A, Gilmour KC, Rosewell IR, Stamp GW, Beddington RS, Mundlos S, Olsen BR. Cbfa1, a candidate gene for cleidocranial dysplasia syndrome, is essential for osteoblast differentiation and bone development. Cell. 1997;89:765-771. [PubMed] [DOI] |
| 21. | Mundlos S, Otto F, Mundlos C, Mulliken JB, Aylsworth AS, Albright S, Lindhout D, Cole WG, Henn W, Knoll JH. Mutations involving the transcription factor CBFA1 cause cleidocranial dysplasia. Cell. 1997;89:773-779. [PubMed] [DOI] |
| 22. | Lin HM, Pestell RG, Raz A, Kim HR. Galectin-3 enhances cyclin D(1) promoter activity through SP1 and a cAMP-responsive element in human breast epithelial cells. Oncogene. 2002;21:8001-8010. [PubMed] [DOI] |
| 23. | Levanon D, Groner Y. Structure and regulated expression of mammalian RUNX genes. Oncogene. 2004;23:4211-4219. [PubMed] [DOI] |
| 24. | Sledz CA, Williams BR. RNA interference in biology and disease. Blood. 2005;106:787-794. [PubMed] [DOI] |
| 25. | Phalon C, Rao DD, Nemunaitis J. Potential use of RNA interference in cancer therapy. Expert Rev Mol Med. 2010;12:e26. [PubMed] [DOI] |
| 26. | Davenport RJ. Gene silencing. A faster way to shut down genes. Science. 2001;292:1469-1471. [PubMed] [DOI] |