修回日期: 2011-10-20
接受日期: 2011-11-04
在线出版日期: 2011-11-28
目的: 探讨中国幽门螺杆菌(Helicobacter pylori, H. pylori)强细胞毒株和弱细胞毒株vacA基因序列差异对其VacA活性的影响.
方法: 从GenBank数据库下载4个强细胞毒株和4个弱细胞毒株vacA基因全长DNA和氨基酸序列, 利用DNAMAN、lasergene 7.0、MEGA 5.0 3个生物信息学软件对其进行分析.
结果: (1)中国H. pylori强细胞毒株、弱细胞毒株和西方强细胞毒株60190株3者之间在vacA基因序列上都存在明显的差异, 这些差异主要集中在vacA基因p55结构域, 表现为疏水性氨基酸与极性氨基酸之间的转换; (2)弱细胞毒株还存在多个插入变异位点; (3)强细胞毒株和弱细胞毒株, 中国和西方分离株在系统发育树中分别聚类为不同的谱系.
结论: vacA基因序列差异和插入突变可能是导致中国不同H. pylori VacA活性差异的重要原因.
引文著录: 杨泽民. 中国幽门螺杆菌强细胞毒株和弱细胞毒株vacA基因全长序列特征与VacA活性的关系. 世界华人消化杂志 2011; 19(33): 3446-3451
Revised: October 20, 2011
Accepted: November 4, 2011
Published online: November 28, 2011
AIM: To evaluate the effect of vacA gene sequence variation on VacA activity by analyzing the full-length sequence of the vacA gene of high- and low-cytotoxic Helicobacter pylori (H. pylori) strains isolated from China.
METHODS: The full-length sequences of the vacA gene of four high- and four low-cytotoxic H. pylori strains were retrieved from GenBank database and analyzed using three bioinformatic programs (DNAMAN, Lasergene 7.0 and MEGA 5.0).
RESULTS: There existed significant sequence variations in the vacA gene among high- and low-cytotoxic H. pylori strains isolated from China and a high-cytotoxic H. pylori 60190 strain isolated from west country. These variations were mainly concentrated on the p55 domain of the vacA gene, resulting in transitions between hydrophobic and polar amino acids. Several insertion variations were detected in low-cytotoxic H. pylori strains compared to the H. pylori 60190 strain. High- and low-cytotoxic strains as well as strains isolated from China and west countrywere clustered as different H. pylori lineages.
CONCLUSION: Sequence and insert variation in the vacA gene might be an important reason resulting in VacA activity difference among H. pylori strains.
- Citation: Yang ZM. Relationship between full-length sequence characteristics of the vacA gene from high-cytotoxic and low-cytotoxic Helicobacter pylori in China and VacA activities. Shijie Huaren Xiaohua Zazhi 2011; 19(33): 3446-3451
- URL: https://www.wjgnet.com/1009-3079/full/v19/i33/3446.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i33.3446
幽门螺杆菌(Helicobacter pylori, H. pylori)是一种人类常见致病菌, 全世界有50%的人终身感染这种细菌. 自从1982年Marshall和Warren发现H. pylori是导致消化性溃疡病的主要致病菌以来, 越来越多的证据表明H. pylori与胃炎、胃溃疡、胃癌、胃黏膜相关的淋巴样组织淋巴瘤(MALT)和胃腺癌密切相关[1,2]. vacA基因编码的空泡毒素A(Vacuolating Cytotoxin, VacA)是H. pylori产生的重要毒力因子, 可导致上皮细胞的空泡变性[3]. VacA是以140 kDa毒素前体的形式被翻译, 在分泌过程中经过N-和C-端的水解产生N-端信号肽(Signal Peptide, SP)(氨基酸残基-33-0)、88 kDa的成熟毒素p88(氨基酸残基1-821)、未知功能的分泌α肽(Secreted Alpha Peptide, SAP)(氨基酸残基822-954)和C-端β-桶型结构域(Beta-Barrel Domain, BBD)(氨基酸残基955-1254). p88含有p33和p55两个结构域, 其中p33(氨基酸残基1-311)与孔的形成有关, 而p55(氨基酸残基312-821)含有一个或多个细胞结合区域. p33和p55结构域共同介导VacA与靶细胞表面的相互作用, 都是VacA发挥毒力作用所必需的[4]. 临床发现所有H. pylori菌株都具有vacA基因, 但是不同的菌株在表达空泡毒性的活性上存在明显的差异[5,6]. 研究发现VacA毒性差异与vacA基因型或序列多态性密切相关, 并且这种序列差异性还表现出明显的地理差异性[7,8]. 根据vacA基因s区(信号序列区)和m区(p55结构域的中间序列区)是否存在标签序列以及两者之间的序列差异, vacA等位基因能够被分成s1/s2、i1/i2、m1/m2不同的家族[8-10]. 通常含有s1、i1和m1的vacA等位基因的菌株更易导致溃疡性疾病或胃癌的发生[8,10].
中国是一个H. pylori感染率高的国家, 也是胃癌的高发区. 目前我国学者对不同省市H. pylori的vacA基因型以及与胃肠疾病的相关性进行了广泛的研究, 其中也有对vacA基因s区或m区部分序列的对比分析[11-13], 但是对vacA基因全长进行深入分析的报道还很少, 尤其是对具有不同细胞毒性活性的H. pylori株的vacA基因全长的对比分析还未见报道. 本研究通过对中国H. pylori强细胞毒株和弱细胞毒株与西方强细胞毒株60190株vacA基因DNA和氨基酸序列进行了深入的对比分析, 探讨中国H. pylori强细胞毒株和弱细胞毒株vacA基因序列的特点及其与西方强细胞毒株的差异, 揭示导致中国H. pylori不同细胞毒株VacA活性差异的原因. 并且利用vacA基因序列构建了中国与西方H. pylori分离株的系统发育树.
本研究数据来源于GenBank数据库, 其相关信息见表1.
| 菌株名称 | 细胞毒性 | 菌株来源 | 基因型 | GenBank登记号 | 文献来源 |
| J128(95-54) | 无 | 意大利 | s1/m2 | U95971; AAC25911 | [14] |
| Tx30a | 无 | 美国 | s2/m2 | U29401; AAA86834 | [8] |
| CHN1811a | 无 | 中国 | s1/m2 | AF050326; AAF26508 | [15,16] |
| CHN5147c | 弱 | 中国 | s1/m2 | AF050320; AAF26502 | [15,16] |
| CHN5114a | 弱 | 中国 | s1/m2 | AF050327; AAF26509 | [15,16] |
| CHN3295b | 弱 | 中国 | s1/m2 | AF050319; AAF26501 | [15,16] |
| CHN5060d | 高 | 中国 | s1/m1 | AF050328; AAF26510 | [15,16] |
| CHN5038c | 高 | 中国 | s1/m1 | AF361702; AAL83505 | [16,17] |
| CHN4611a | 高 | 中国 | s1/m1 | AF361701; AAL83504 | [16,17] |
| CHN3554a | 高 | 中国 | s1/m1 | AF361700; AAL83503 | [16,17] |
| 60190 | 高 | 英国 | s1/m1 | U05676; AAA17657 | [18] |
| NCTC 11637 | 高 | 澳大利亚 | s1/m1 | AF049653; AAD04290 | [7] |
| 185-44 | 高 | 德国 | s1/m1 | Z26883; CAA81528 | [19] |
将强细胞毒性的菌株5060d, 5038c, 4611a和3554a分为一组, 将弱细胞毒性的菌株5147c, 5114a和3295b和无细胞毒性的菌株1811a分为另一组. 以西方强细胞毒株60190株vacA基因作为对照, 利用DNAMAN软件对中国H. pylori强细胞毒株和弱细胞毒株的vacA基因DNA和氨基酸序列进行相似性分析; 利用lasergene 7.0软件的EditSeq功能模块分别对他们的DNA和氨基酸序列进行碱基和氨基酸统计; 利用MEGA 5.0软件采用邻接法(NJ)分别对中国和西方H. pylori分离株vacA基因DNA和氨基酸序列构建系统发育树, 采用Bootstrap的方法重复1 000次进行检验.
中国H. pylori强细胞毒株和弱细胞毒株与60190株vacA基因DNA和氨基酸序列相似性分析结果如表2. 本研究中H. pylori的vacA基因DNA和氨基酸序列相似趋势一致, 并且DNA序列相似性略高于氨基酸序列相似性. 就菌株而言, 组内相似性明显高于组间, 组间相似性以强细胞毒株与60190株为最高. 中国H. pylori分离株与60190株vacA基因DNA序列平均相似性为87.24%, 氨基酸序列平均相似性为85.27%.
| 高细胞毒株 | 弱细胞毒株 | 60190株 | ||||
| 氨基酸序列相似性平均值 | DNA序列相似性平均值 | 氨基酸序列相似性平均值 | DNA序列相似性平均值 | 氨基酸序列相似性平均值 | DNA序列相似性平均值 | |
| 高细胞毒株 | 95.16 | 94.45 | 88.37 | 89.01 | ||
| 弱细胞毒株 | 84.98 | 96.01 | 95.93 | 85.47 | ||
| 60190株 | 88.45 | 82.09 | 100 | 100 | ||
vacA基因碱基和氨基酸统计分析显示, 本研究中不同H. pylori分离株之间碱基数量虽有差异, 但其百分含量没有明显差异(结果未显示). 相对于60190株, 中国H. pylori分离株vacA基因在氨基酸数量和不同类型氨基酸含量都存在明显的差异, 这种差异性表现为酸性、碱性和疏水性氨基酸含量明显增加, 极性中性氨基酸含量明显降低, 其中弱细胞毒株与60190株的差异性更大(表3).
与60190株相比, 中国H. pylori分离株vacA基因氨基酸变异位点有76个(不包括1个插入位点), 变异率为5.91%. 这些差异位点分布在vacA基因不同结构域中, 其中以p55区最多, 变异率最大, 氨基酸变异大部分表现为疏水性氨基酸与极性氨基酸(包括酸、碱和中性氨基酸)之间的转换. 并且弱细胞毒株与60190株的差异性较强细胞毒株的更大. 强细胞毒株与弱细胞毒株之间变异位点有161个(不包括7个插入/缺失位点), 变异率为12.51%, 仅次于弱细胞毒株与60190株之间的变异率(15.07%, 表4). 此外, 弱细胞毒株在vacA基因m区插入了21个氨基酸, 序列完全相同, 但与西方无毒株Tx30a的插入序列有3个氨基酸残基的差异.
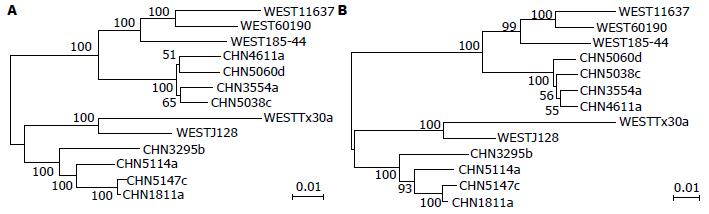
vacA基因DNA和氨基酸序列系统发育树具有非常高的相似性, 其中强细胞毒株和弱细胞毒株各自聚为一类, 在强细胞毒株和弱细胞毒株内部中国分离株和西方分离株又各自聚为一个亚类, 并且每一类或亚类都具有很高的支持率(图1).
vacA基因编码产物是H. pylori的一个重要毒力因子. 虽然所有的H. pylori都存在vacA基因, 但是临床分离的H. pylori菌株仅有50%或者更低的能产生这种毒素. 究其原因, 可能与vacA基因序列变异或多态性密切相关. Atherton等[8]通过对强细胞毒株60190与无细胞毒株Tx30a遗传差异性分析, 发现两者存在vacA基因型差异, 他们认为特定的vacA基因型与其细胞毒性的活性和感染的临床结果密切相关. 目前发现vacA基因主要存在s1/s2, i1/i2和m1/m2 3个等位基因, 这3个等位基因可以组合形成不同的vacA基因型. 此外, vacA基因型的分布有明显的地理差异, 中国各省市vacA基因型虽然存在一定的差异, s1/m2和s1/m1都存在, 但以s1/m2为主[11-13,15]. 本研究中, 强细胞毒株都为s1/m1型, 弱细胞毒株均为s1/m2型(除Tx30a外). 这一定程度上为"含有s1, i1和m1 vacA等位基因的菌株更易导致溃疡性疾病或胃癌的发生"的结果提供了依据.
VacA作为一种蛋白毒素, 特定的空间结构是其发挥功能的必要条件. 然而蛋白空间结构是由蛋白质的一级结构决定, 因此通过对vacA基因DNA和氨基酸序列的分析, 有助于我们揭示导致不同H. pylori菌株存在VacA活性差异的原因. 通过序列相似性分析, 我们发现vacA基因DNA和氨基酸序列相似性顺序为: 中国H. pylori弱细胞毒株组内>强细胞毒株组内>强细胞毒株-60190株>强细胞毒株-弱细胞毒株>弱细胞毒株-60190株. 这一结果说明中国H. pylori分离株vacA基因DNA和氨基酸序列在强细胞毒株和弱细胞毒株组内都非常保守, 强细胞毒株、弱细胞毒株和西方强细胞毒株60190株三者之间相似性明显降低, 存在明显的差异, 并且这种差异性表现为功能上的差异性小于地理上的差异性, 以功能保守为首要的. Gangwer等[4]通过对来自不同国家分离的H. pylori vacA基因进行聚类分析发现m1和m2型菌株各自聚为一类, 其中m1型又分为亚洲分离株和非亚洲分离株两个亚型, m2型菌株虽然亚洲和非亚洲分离株没有完全分开, 但是两者也存在一定程度的分离. 纪徐淮等[15]研究发现不同来源的H. pylori菌株能够分为m1型和m2型两个聚类群, 每一个聚类群又可再分为中国株和西方株两个地理分类亚群. 本研究对中国和西方H. pylori分离株的vacA基因DNA序列和氨基酸序列进行聚类分析, 也发现m1型强细胞毒株和m2型弱细胞毒株各自聚为一类, 在每一大类内部中国分离株和西方分离株又各自聚类为一个亚类. 这进一步说明中国H. pylori强细胞毒株和弱细胞毒株在vacA基因序列上存在明显的差异, 并且同为强细胞毒株的中国和西方H. pylori分离株之间也表现为明显的地理差异.
组成人类蛋白质的常见氨基酸有20种, 共分为疏水性氨基酸、极性中性氨基酸、酸性氨基酸和碱性氨基酸4种, 其中包括酸性、碱性和中性的极性氨基酸常处于蛋白质的外侧, 疏水性氨基酸常处于蛋白质的内侧, 因此蛋白质氨基酸残基极性的改变会直接影响蛋白质空间结构, 进而影响蛋白质的功能发挥. 通过对vacA基因氨基酸序列的深入分析, 我们发现中国H. pylori分离株与60190株相比, vacA基因在氨基酸数量和不同类型氨基酸含量都存在明显的差异, 这种差异性表现为酸性、碱性和疏水性氨基酸含量明显增加, 极性中性氨基酸含量明显降低. 其中弱细胞毒株与60190株的差异性更大. 通过VacA不同结构域的进一步分析, 我们发现这些氨基酸残基的差异位点分布在vacA基因不同结构域中, 其中以p55区最多, 变异率最大, 氨基酸变异同样表现为疏水性氨基酸与极性氨基酸(包括酸、碱和中性氨基酸)之间的转换, 并且弱细胞毒株与60190株的差异性表现更为明显. 这些结果说明组成vacA基因氨基酸残基的极性变异可能是导致VacA结构改变, 进而影响其毒性改变的重要原因. 同时vacA基因p55结构域含有多个细胞结合区域, 他在介导VacA和靶细胞的相互作用方面具有重要作用, 因此, p55区氨基酸残基的大范围变异可能会直接影响VacA与靶细胞的结合, 进而影响VacA活性的发挥. 此外, 与60190株相比, 我们还发现弱细胞毒株在p55结构域中存在多个插入位点, 然而强细胞毒株只存在1个中国H. pylori分离株共有的插入位点. 这一结果说明中国H. pylori弱细胞毒株vacA基因插入变异可能是导致其活性降低的另一原因.
另外, Ito等[7]通过对日本H. pylori有细胞毒株和无细胞毒株vacA基因全长的分析发现, 50%无细胞毒株存在内部复制、大片段删除、1 bp插入等导致VacA翻译提前终止的突变, 然而有细胞毒株不存在这些突变. 在我们的分析中, 弱细胞毒株vacA基因没有发现这些突变, 这可能是我们的样品量较少, 存在这些突变的中国H. pylori菌株没有被采集到用于测序分析. 同时也可能是导致VacA活性差异的原因也像vacA基因型分布一样存在地理差异性.
总之, 通过对vacA基因序列的深入分析, 我们发现中国H. pylori强细胞毒株、弱细胞毒株和西方强细胞毒株三者之间在vacA基因DNA和氨基酸序列上都存在明显的差异, 这些差异性主要集中在vacA基因p55结构域, 表现为疏水性氨基酸与极性氨基酸(包括酸、碱和中性氨基酸)之间的转换. 而且, 弱细胞毒株还存在多个插入变异位点. 所有这些差异可能是导致不同H. pylori VacA活性差异的重要原因. 此外, 中国和西方H. pylori分离株系统发育树分析显示, 强细胞毒株和弱细胞毒株、中国和西方分离株分别聚类为不同谱系.
H. pylori与胃炎、消化性溃疡和胃癌密切相关. vacA基因编码产物是H. pylori的一个重要毒力因子. 临床发现所有H. pylori菌株都具有vacA基因, 但是不同的菌株在表达VacA的活性上存在明显的差异, 究其原因还不清楚.
陈国忠, 副主任医师, 广西中医学院第一附属医院消化内科
研发前沿 基因多态性是导致基因功能变化的遗传基础. VacA氨基酸序列变异会改变其空间结构, 进而影响VacA的毒性, 因此, 通过不同VacA活性的H. pylori菌株的vacA基因序列分析, 可以揭示导致不同H. pylori菌株VacA活性差异的深层原因.
Yoshiyuki等通过对日本H. pylori有细胞毒株和无细胞毒株vacA基因全长的分析发现, 50%无细胞毒株存在内部复制、大片段删除、1 bp插入等导致VacA翻译提前终止的突变, 然而有细胞毒株不存在这些突变.
本研究发现中国H. pylori强细胞毒株、弱细胞毒株和西方强细胞毒株60190株的vacA基因序列存在明显的差异: (1)序列差异主要表现为p55区的疏水性与极性氨基酸之间的转换; (2)弱细胞毒株存在多个插入变异位点; (3)三者在系统发育树中分别聚类为不同的谱系.
vacA基因序列的差异, 为揭示不同H. pylori菌株VacA活性存在差异提供了理论依据. 此外, 通过对vacA基因序列的分析还有利于认识不同vacA基因型与H. pylori感染临床结果之间的关系.
本研究具有一定的创新性, 角度新颖, 充分利用了国际生命信息资源和研究手段, 科学性较好.
编辑: 曹丽鸥 电编:闫晋利
| 1. | Pinto-Santini D, Salama NR. The biology of Helicobacter pylori infection, a major risk factor for gastric adenocarcinoma. Cancer Epidemiol Biomarkers Prev. 2005;14:1853-1858. [PubMed] [DOI] |
| 2. | Santacroce L, Cagiano R, Del Prete R, Bottalico L, Sabatini R, Carlaio RG, Prejbeanu R, Vermesan H, Dragulescu SI, Vermesan D. Helicobacter pylori infection and gastric MALTomas: an up-to-date and therapy highlight. Clin Ter. 2008;159:457-462. [PubMed] |
| 3. | Cover TL, Blaser MJ. Purification and characterization of the vacuolating toxin from Helicobacter pylori. J Biol Chem. 1992;267:10570-10575. [PubMed] |
| 4. | Gangwer KA, Shaffer CL, Suerbaum S, Lacy DB, Cover TL, Bordenstein SR. Molecular evolution of the Helicobacter pylori vacuolating toxin gene vacA. J Bacteriol. 2010;192:6126-6135. [PubMed] [DOI] |
| 5. | Telford JL, Ghiara P, Dell'Orco M, Comanducci M, Burroni D, Bugnoli M, Tecce MF, Censini S, Covacci A, Xiang Z. Gene structure of the Helicobacter pylori cytotoxin and evidence of its key role in gastric disease. J Exp Med. 1994;179:1653-1658. [PubMed] [DOI] |
| 6. | Cover TL, Blanke SR. Helicobacter pylori VacA, a paradigm for toxin multifunctionality. Nat Rev Microbiol. 2005;3:320-332. [PubMed] [DOI] |
| 7. | Ito Y, Azuma T, Ito S, Suto H, Miyaji H, Yamazaki Y, Kohli Y, Kuriyama M. Full-length sequence analysis of the vacA gene from cytotoxic and noncytotoxic Helicobacter pylori. J Infect Dis. 1998;178:1391-1398. [PubMed] [DOI] |
| 8. | Atherton JC, Cao P, Peek RM, Tummuru MK, Blaser MJ, Cover TL. Mosaicism in vacuolating cytotoxin alleles of Helicobacter pylori. Association of specific vacA types with cytotoxin production and peptic ulceration. J Biol Chem. 1995;270:17771-17777. [PubMed] |
| 9. | Atherton JC, Sharp PM, Cover TL, Gonzalez-Valencia G, Peek RM, Thompson SA, Hawkey CJ, Blaser MJ. Vacuolating cytotoxin (vacA) alleles of Helicobacter pylori comprise two geographically widespread types, m1 and m2, and have evolved through limited recombination. Curr Microbiol. 1999;39:211-218. [PubMed] [DOI] |
| 10. | Rhead JL, Letley DP, Mohammadi M, Hussein N, Mohagheghi MA, Eshagh Hosseini M, Atherton JC. A new Helicobacter pylori vacuolating cytotoxin determinant, the intermediate region, is associated with gastric cancer. Gastroenterology. 2007;133:926-936. [PubMed] [DOI] |
| 12. | 陈 学军, 严 杰, 毛 亚飞, 李 立伟. 浙江地区幽门螺杆菌临床菌株vacA优势基因型及其核苷酸序列分析. 浙江大学学报(医学版). 2003;32:24-28. |
| 13. | 刘 改芳, 韩 晓辉, 张 彦普, 吴 婧, 张 玉贞, 赵 丽伟, 徐 华洲, 姚 希肾. 幽门螺杆菌cagA及vacA亚型与胃肠疾病的关系. 中国实用内科杂志. 2006;26:203-205. |
| 14. | Pagliaccia C, de Bernard M, Lupetti P, Ji X, Burroni D, Cover TL, Papini E, Rappuoli R, Telford JL, Reyrat JM. The m2 form of the Helicobacter pylori cytotoxin has cell type-specific vacuolating activity. Proc Natl Acad Sci U S A. 1998;95:10212-10217. [PubMed] [DOI] |
| 15. | 纪徐淮, Daniela Burroni, 许国铭, Cristina Pagliaccia, Jean-Marc Reyrat, 屠振兴, 丁华, 杜奕奇, Rino Rappuoli, John L. Telford. 中国幽门螺杆菌vacA基因的等位变异. 中华消化杂志. 2000;20:151-154. |
| 17. | Ji X, Frati F, Barone S, Pagliaccia C, Burroni D, Xu G, Rappuoli R, Reyrat JM, Telford JL. Evolution of functional polymorphism in the gene coding for the Helicobacter pylori cytotoxin. FEMS Microbiol Lett. 2002;206:253-258. [PubMed] [DOI] |
| 18. | Cover TL, Tummuru MK, Cao P, Thompson SA, Blaser MJ. Divergence of genetic sequences for the vacuolating cytotoxin among Helicobacter pylori strains. J Biol Chem. 1994;269:10566-10573. [PubMed] |
| 19. | Schmitt W, Haas R. Genetic analysis of the Helicobacter pylori vacuolating cytotoxin: structural similarities with the IgA protease type of exported protein. Mol Microbiol. 1994;12:307-319. [PubMed] [DOI] |