修回日期: 2011-07-23
接受日期: 2011-08-05
在线出版日期: 2011-08-18
目的: 探讨野生型p53对人肝癌细胞SMMC-7721细胞增殖和细胞恶性表型影响的一种可能机制.
方法: 设计并构建p53特异性小干扰shRNA绿色荧光真核表达质粒(p53-siRNA)和表达EGFP-p53融合蛋白的p53绿色荧光真核增强表达质粒(pEGFP-p53), 通过脂质体Lipofection-2000介导转染, 将表达pEGFP-p53重组质粒、p53-siRNA及空载体pEGFP-C1转染入SMMC-7721细胞; 经G418筛选, 获得稳定细胞系7721-p53、7721-C1; 7721-p53RNAi、7721-NC. 通过RT-PCR检测转染后p53、POLD1 mRNA表达水平. 通过生长曲线测定, 克隆形成实验, 了解SMMC-7721细胞在p53表达水平改变后细胞癌性的变化.
结果: 与人肝癌细胞SMMC-7721比较, 转染质粒pEGFP-p53的野生型p53高表达组, p53 mRNA表达量增高, POLD1基因的表达量降低; 而转染pGPU6/GFP/neo-p53i-769的p53低表达组, p53 mRNA表达量降低, POLD1 mRNA表达量增高, 其他组较空白对照无明显变化. 对高表达野生型p53的人肝癌细胞系SMMC-7721-pEGFP-C1-p53, 低表达野生型p53的人肝癌细胞系SMMC-7721-pGPU6/GFP/neo-p53i-769, 和普通人肝癌细胞SMMC-7721分别进行MTT和平板克隆形成实验. MTT结果发现: SMMC-7721-pGPU6/GFP/neo-p53i-769的细胞其生长速度比普通肝癌细胞的要快, 而SMMC-7721-pEGFP-p53的细胞较普通肝癌细胞生长速度较慢.克隆形成实验结果显示: pEGFP-C1-p53、pGPU6/GFP/neo-p53i-769组克隆形成率分别为38.1%和72.6%, 与普通肝癌细胞SMMC-7721的克隆形成率52.6%相比, 均有统计学差异(P<0.05).结果显示: 在人肝癌细胞SMMC-7721中, 野生型p53高表达组能够抑制POLD1的基因转录, 并抑制细胞增殖活性; 而低表达组能够促进POLD1的基因转录, 促进细胞增殖.
结论: p53对POLD1的调控作用可能是p53对肝癌细胞增殖和细胞恶性表型影响的一种新的机制.
引文著录: 韦长元, 刘起理, 廖柳凤, 徐恒, 谭晓虹. 野生型p53对肝癌细胞POLD1基因表达及细胞恶性表型的影响. 世界华人消化杂志 2011; 19(23): 2443-2449
Revised: July 23, 2011
Accepted: August 5, 2011
Published online: August 18, 2011
AIM: To investigate the impact of overexpression of wild-type p53 on cell proliferation and malignant phenotype in human hepatocellular carcinoma cell line SMMC-7721 and to explore possible mechanism involved.
METHODS: Enhanced green fluorescence protein gene-containing eukaryotic expression plasmids expressing p53-specific small interfering RNA (shRNA) (p53-siRNA) or wild-type p53 (pEGFP-p53) were constructed and introduced into SMMC-7721 cells by Lipofection-2000-mediated transfection. Meanwhile, the pEGFP-C1 empty vector was also transfected into SMMC-7721 cells. Cell lines stably expressing p53-siRNA, pEGFP-p53 or pEGFP-C1 were screened in medium containing G418. After transfection, the expression of p53 and POLD1 mRNAs was detected by RT-PCR. The changes in malignant cell behavior were determined by cell growth curve determination and colony formation assay.
RESULTS: Compared to control SMMC-7721 cells, p53 mRNA expression was increased and POLD1 gene expression was decreased in SMMC-7721 cells transfected with the plasmid carrying wild-type p53 gene, while p53 mRNA expression was reduced and POLD1 mRNA expression was increased in SMMC-7721 cells transfected with the plasmid carrying p53-siRNA. MTT results showed that cell growth rate was faster in SMMC-7721 cells transfected with the plasmid carrying p53-siRNA than in control SMMC-7721 cells, but was slower in SMMC-7721 cells transfected with the plasmid carrying wild-type p53 gene than in control cells. Colony formation assay showed that colony formation rate was lower in SMMC-7721 cells transfected with the plasmid carrying wild-type p53 gene than in control cells (38.1% vs 52.6%, P < 0.05), but was higher in cells tranfected with the plasmid carrying p53-siRNA than in control cells (72.6% vs 52.6%, P < 0.05). High expression of wild-type p53 inhibited POLD1 transcription and cell proliferation, while low expression of wild-type p53 promoted POLD1 transcription and cell proliferation.
CONCLUSION: Wild-type p53 controls liver cancer cell proliferation and malignant phenotype possibly by regulating POLD1 expression.
- Citation: Wei CY, Liu QL, Liao LF, Xu H, Tan XH. Effect of overexpression of wild-type p53 on POLD1 expression and malignant cell behavior in human hepatocellular carcinoma cell line SMMC-7721. Shijie Huaren Xiaohua Zazhi 2011; 19(23): 2443-2449
- URL: https://www.wjgnet.com/1009-3079/full/v19/i23/2443.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v19.i23.2443
DNA聚合酶δ(DNA polymerase δ, polδ)是真核生物DNA复制的最主要复制酶, 参与到DNA复制、修复、细胞周期调控等过程, 对保持真核生物基因组的结构完整性和遗传稳定性具有重要作用[1,2]. polδ由四个亚基组成, 其催化亚基P125由POLD1基因编码[3]. POLD1基因定位于染色体19q13.3-q13.4, 含有27个外显子和26个内含子[4,5]. 对保持真核生物基因组的结构完整性和遗传稳定性具有重要作用[6-8]. DNA聚合酶δ的聚合酶和外切酶二者活性位于其催化亚基P125. 只有通过P125上的PIP-box和KA-box 2个PCNA相互作用位点, P125与PCNA相互作用才能发挥其功能活性[9,10]. 有研究表明, POLD1基因的表达受到细胞周期的调控, 在POLD1基因启动子中首次鉴定CDE/CHR元件, 并证明此元件与POLD1基因细胞周期调控密切相关[11,12].
野生型p53作为人体内重要的抑癌基因, 存在于几乎所有肿瘤细胞中, p53在DNA复制、修复、重组及细胞周期调控中都起到重要作用. 其对正常细胞生长的维持及肿瘤细胞的抑制作用一直是医学界重要的研究方向[13]. 以往研究[14]发现: 转录因子p53能够与受其调控的基因的启动子或内含子中的保守序列结合, 从而调控受控基因的表达. 另外, POLD1基因的启动子区上可能存在p53及转录因子Sp1的结合位点, p53可能通过与Sp1竞争结合位点从而抑制POLD1启动子活性[15]. 在受p53调控的基因中, 有些基因可能与细胞中POLD1基因的表达调控有关. 如细胞周期抑制因子p21的表达就受p53的正向调控[16,17], 细胞周期抑制因子p21基因启动子区域具有p53结合位点, 野生型p53能与p21结合激活p21基因转录, 而p21又可以通过自身或PCNA、Gadd45、E2F、Myc、Rb等细胞因子的相互作用抑制POLD1启动子的活性, 所以野生型p53通过p21间接抑制了POLD1的活性. 但是, 目前仍不清楚在肝癌细胞中p53对POLD1的调控作用. 本研究通过构建p53的绿色荧光真核增强表达质粒, 以及p53低表达的RNA干扰表达质粒, 并分别导入人肝癌细胞SMMC-7721中, 从正反两方面验证在人肝癌细胞SMMC-7721中p53对POLD1基因表达产生的调控作用. 同时对筛选出来的稳定细胞系进行生长速率检测、平板集落形成率等实验, 检测p53对POLD1基因表达产生调控之后, 对肝癌细胞增殖及细胞恶性表型的影响.
人肝癌细胞SMMC-7721购于中科院上海细胞库; 脂质体Lipofectamine2000和TRIzol试剂购于美国Invitrogen公司; pEGFP-C1空载体由北京师范大学柳惠图教授惠赠; 表达EGFP-p53融合蛋白的pEGFP-p53重组质粒由中国科学院上海生命科学学院吴家睿研究员惠赠; RNA干扰所用的siRNA序列, 及pGPU6/GFP/neo重组真核表达质粒(含有增强型绿色荧光蛋白阅读框, 正确插入目的基因cDNA后, 能够将目的蛋白融合在EGFP的C端)由吉玛公司构建.
1.2.1 siRNA的筛选及p53-siRNA绿色荧光真核表达质粒的构建: 具有互补序列、能够编码短发卡结构RNA(shRNA)的双链寡核苷酸模板DNA转录产物所形成的siRNA的作用靶点为人p53mRNA(GeneBank No, NM_00546.4)第198-1379 bp位核苷酸. 为防止siRNA的off-target, 本研究设计了3对siRNA Oligo, 以下3对siRNA与p53序列无同源性, 亦不与任何人类基因序列同源. 同时以双链寡核苷酸转录产物所形成的siRNA作为阴性对照. 将人肝癌细胞SMMC-7721培养于含有100 mL/L胎牛血清的RPMI1640(Hyclone)培养基中, 并置于37 ℃含50 mL/L CO2的温箱培养. 实验分6组: A: p53i-769组, 转染试剂中加入p53i-769 siRNA Oligo; B: p53i-574组, 转染试剂中加入p53i-574 siRNA Oligo; C: p53i-73组, 转染试剂中加入p53i-73 siRNA Oligo; D: 空白对照组, 即未转染组; E: 脂质体组, 即转染中仅含脂质体, 无siRNA; F: 阴性对照组, 即转染试剂中加入阴性对照siRNA; 以上实验组分别用脂质体Lipofectamine2000介导转染人肝癌细胞SMMC-7721. 分别收集24 h, 48 h, 72 h的未干扰和siRNA干扰的细胞, 用TRIzol法提取细胞总RNA. 用RT-PCR方法检测SMMC-7721细胞中p53 mRNA的表达. PCR反应条件为: 94 ℃预变性4 min, 然后扩增30个循环, 每个循环94 ℃变性45 s、55 ℃退火45 s、72 ℃延伸1 min, 最后72 ℃延伸10 min. p53基因扩增产物为186 bp, 上游引物为5'-GGCCCACTTCACCGTACTAA-3', 下游引物为5'-GTGGTTTCAAGGCCAGATGT-3'. β-actin的扩增产物为661 bp, 上游引物为5'-TGACGGGGTCACCCACACTGTGCCCATCTA-3'; 下游引物为5'-CTAGAAGCATTTGCGGTGGACGATGGAGGG-3'. 以p53与β-actin的条带的灰度比值表示p53 mRNA的相对含量. 经半定量RT-PCR鉴定得到的对p53表达有明显干扰作用的siRNA Oligo送上海吉玛公司构建真核表达载体pGPU6/GFP/neo-p53i-769. 同时由公司用阴性对照siRNA构建阴性对照真核表达pGPU6/GFP/neo-shNC(表1).
| siRNA Oligo | 序列 |
| p53i-769 | 5'-GGAAGACUCCAGUGGUAAUTT-3' |
| 5'-AUUACCACUGGAGUCUUCCAG-3' | |
| p53i-574 | 5'-GCAUCUUAUCCGAGUGGAATT-3' |
| 5'-UUCCACUCGGAUAAGAUGCTG-3' | |
| P53i-73 | 5'-CUACUUCCUGAAAACAACGTT-3' |
| 5'-CGUUGUUUUCAGGAAGUAGTT-3' | |
| 阴性对照 | 5'-UUCUCCGAACGUGUCACGUTT-3' |
| 5'-ACGUGACACGUUCGGAGAATT-3' |
1.2.2 细胞的培养、转染及稳定细胞株的筛选: 将人肝癌细胞SMMC-7721培养于含有100 mL/L胎牛血清的RPMI 1640培养基中, 并置于37 ℃含50 mL/L CO2的温箱培养. 用脂质体Lipofectamine2000分别介导质粒pEGFP-p53、pEGFP-C1、pGPU6/GFP/neo-p53i-769、pGPU6/GFP/neo-shNC转染SMMC-7721细胞, 48 h后, 用荧光显微镜观察绿色荧光蛋白的表达情况. 再用G418(600 mg/L)筛选细胞, 挑取克隆, 16 d后, 各组陆续出现较大的细胞群落, 挑出单克隆扩大培养, 扩大培养后分别得到pEGFP-p53、pEGFP-C1、pGPU6/GFP/neo-p53i-769、pGPU6/GFP/neo-shNC的稳定细胞系. 同时培养SMMC-7721细胞作空白对照组. 转染步骤参照转染剂的操作说明书进行, 筛选方法见参考文献[18].
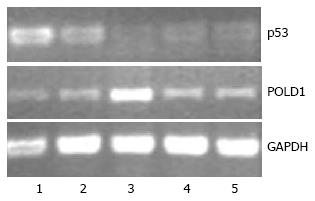
1.2.3 稳定细胞系的鉴定(RT-PCR方法检测p53 mRNA表达): 收集各组稳定细胞系的细胞, 用TRIzol法提取细胞总RNA, 反转录成cDNA, 以基因特异性引物进行PCR扩增. p53的扩增条件同前; 以GAPDH为内参对照. GAPDH基因扩增产物为210 bp, 上游引物为5'-AACGGATTTGGTCGTATTG-3', 下游引物为5'-CTGGAAGATGGTGATGGG-3'. 所有扩增产物经1.5%的琼脂糖凝胶电泳分离后用溴化乙锭染色观察.
1.2.4 RT-PCR分析p53表达变化对POLD1 mRNA表达水平的影响: p53和GAPDH的扩增条件同前; POLD1上游引物为5'-ACCTACTGAGCAACCGCATCG-3', 下游引物为5'-CGGTACTGGCAGAACTCACAC-3'. 扩增条件为: 94 ℃预变性4 min, 然后扩增30个循环; 每个循环94 ℃变性45 s、62 ℃退火45 s、72 ℃延伸1 min, 最后再72 ℃延伸10 min. POLD1基因扩增产物为487 bp, GAPDH作为内参对照, 序列同前. 所有扩增产物经1.5%的琼脂糖凝胶电泳分离后用溴化乙锭染色观察.
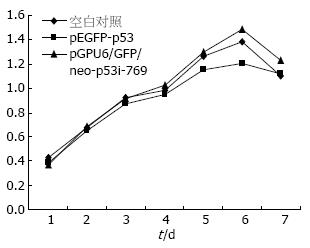
1.2.5 不同细胞系生长速率的比较: 分别取7721、7721-p53、7721- p53i、7721-C1、7721- NC等细胞系处于对数生长期的细胞, 按1×104个细胞/mL的量接种于含100 mL/L胎牛血清及抗生素(青霉素100 kU/L,链霉素100 mg/L)的RPMI1640培养基中, 置于37 ℃、50 mL/L CO2的细胞培养箱培养. 24 h后, 将检测孔的培养基吸尽, 加入100 μL的新培养基和10 μL MTT(5 g/L溶于PBS), 于37 ℃孵育4 h; 再加入150 μL的裂解液DMSO, 震荡10 min后测A490. 以后每个细胞系每隔24 h从孔板中吸取3个孔进行检测, 持续七天. 并绘制曲线图.
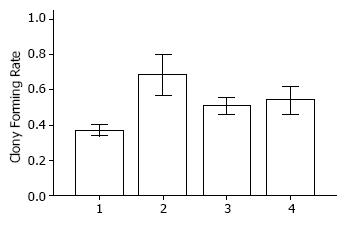
1.2.6 不同细胞系平板集落形成率的比较: 用细胞计数板在显微镜下计数, 以1 000个细胞/孔的量分别吸取SMMC-7721, pGPU6/GFP/neo-shNC、pEGFP-C1-p53、pGPU6/GFP/neo-p53i-769 4组细胞系的细胞, 接种于含有RPMI1640培养基(含100 mL/L胎牛血清、100 kU/L青霉素、100 mg/L链霉素)的6孔板中, 每组细胞系做3个平行孔, 置于37 ℃, 50 mL/L CO2的细胞培养箱中培养. 静置培养10 d后, 观察集落的形态, 并用Giemsa染色法在显微镜下统计50个细胞以上的集落. 计算不同细胞集落形成率. 用SPOT II采集图像.
统计学处理 以上实验数据经SPSS17.0统计处理后, 数据以mean±SD表示. 数据比较采用方差分析, 当P<0.05时则认为差异有显著.
转染p53i-769、p53i-574、p53i-73组的SMMC-7721细胞p53 mRNA表达水平都有所下降(图1). 根据各实验组跑胶结果中p53电泳条带灰度比值比较, 发现相对于空白对照组, p53i-769组的24 h、48 h、72 h的p53表达量分别下调45.64%、60.81%、72.67%; p53i-574组分别下调38.24%、57.34%、64.93%; p53i-73组分别下调32.11%、46.59%、49.31%. 上述实验组抑制作用随处理时间的延长而呈增长趋势, 其余各对照组p53 mRNA表达差异无明显变化.
根据上述结果, 筛选出干扰效果最明显的A组: p53i-769后送上海吉玛生物工程公司分别将p53i-769和阴性对照shNC重组于pGPU6/GFP/neo上, 构建得到绿色荧光真核表达载体: pGPU6/GFP/neo-p53i-769、pGPU6/GFP/neo-shNC.
分别将pEGFP-p53, pEGFP-C1, pGPU6/GFP/neo-p53i-769, pGPU6/GFP/neo-shNC转染SMMC-7721细胞. 48 h后, 将培养的细胞置于荧光显微镜下观察, 镜检发现各组均有一定量的绿色荧光产生, 表明质粒已经成功转染细胞, 并能在细胞内表达.
各组细胞经G418筛选后, 得到稳定的细胞系, 用半定量RT-PCR的方法检测p53 mRNA在不同细胞系中的表达. 结果发现, 与阴性对照组相比, 转染pEGFP-p53的p53高表达组, p53 mRNA表达量增高; 而转染pGPU6/GFP/neo-p53i-769的p53低表达组, p53 mRNA表达量降低(图2). 表明稳定转染的质粒对p53 mRNA表达水平起到了相应的调节作用.
与空白组相比, 转染pEGFP-p53的p53高表达组, 随着p53 mRNA表达量增高, POLD1基因的表达量降低; 而转染pGPU6/GFP/neo-p53i-769的p53低表达组, 随着p53 mRNA表达量降低, POLD1 mRNA表达量增高; 其他组较空白对照无明显变化(图3). 表明p53对POLD1起着负性调节的作用.
3种细胞系的活细胞数目(起始)均随时间的延长而不断增加, 但从第6天转染pEGFP-p53和转染pGPU6/GFP/neo-p53i-769的细胞系其活细胞数处于高峰, 第7天两者的活细胞数都开始下降. 转染pGPU6/GFP/neo-p53i-769的细胞其生长速度比空白对照组的要快, 而转染pEGFP-p53的细胞较之于空白对照组细胞其生长速度较慢(图4).
以SMMC-7721, pGPU6/GFP/neo-shNC细胞为对照, 接种pEGFP-C1-p53、pGPU6/GFP/neo-p53i-769于6孔板中, 每个细胞系做3个平行孔. 经培养后, 发现各个细胞系的克隆集落形成数由少到多排列顺序为: pEGFP-C1-p53<SMMC-7721<pGPU6/GFP/neo-shNC<pGPU6/GFP/neo-p53i-769. 统计分析发现, pEGFP-C1-p53、pGPU6/GFP/neo-p53i-769组克隆形成率分别为38.1%和72.6%, 与对照组SMMC-7721和pGPU6/GFP/neo-shNC的克隆形成率52.6%和49.3%相比, 有统计学差异(P<0.05). 而两个对照组SMMC-7721和pGPU6/GFP/neo-shNC之间的克隆形成率无统计学差异(图5).
肝癌是我国常见的恶性肿瘤之一, 其发病率在我国仍逐年上升. 在我国位列居民癌症死因的第2位[19], 手术切除和肝移植虽已被认为是根治肝癌的最好手段[20], 但转移和复发是影响肝癌术后生存的最大障碍. 探讨肝癌发生发展和转移复发的分子机制, 寻找早期诊断肝癌、预测转移的生物标志和干预治疗的靶点已成为当今肝癌研究的热点[21].
癌细胞的恶性增殖与DNA的复制是密不可分的. 因此, 认识DNA复制调控的机理对了解肝癌细胞恶性增殖至关重要. 癌细胞在大量增殖的过程中需要大量的DNA合成, 作为DNA复制中关键的基因, POLD1基因表达异常调控可能与癌细胞的恶性增殖有关. 已有报道, 在肝癌组织中, POLD1基因和其编码蛋白p125的表达水平均显著高于癌旁组织, 证实了癌的恶性增殖可能与POLD1的表达有关[22]. 而POLD1启动子的结合蛋白又是调控其表达的制约因素. 比如细胞周期素依赖性蛋白激酶(cyclin dependentkinases, CDKs)、细胞分裂周期蛋白(cell division cycle proteins, CDCs)和细胞周期素(Cyclins)、细胞周期抑制因子(cyclin dependent kinas inhibitor, CDKI)如p53、p21、增殖细胞核抗原(proliferating cell nuclear antigen, PCNA)及E2F、Myc、Rb等多种细胞周期调控因子和促进细胞增殖的生长因子参与了此进程的调控[14,23]. 这些调控蛋白和某些未知的上游调控蛋白相互作用形成一个特异的DNA复制调控系统[24-27]. 一旦某个调控环节失常, 会造成DNA复制异常而导致细胞癌变或病变[28,29]. 所以, 认识已知或未知的调控因子, 对POLD1基因异常表达的调控机理, 和细胞恶性增殖的相关性有着至关重要的作用.
Li等[15]研究发现, p53可能通过与转录因子Sp1竞争POLD1基因的启动子区上的结合位点, 从而抑制POLD1启动子活性. 国内专家通过利用染色体免疫共沉淀(ChIP, Chromatin Immunoprecipitation)和荧光素酶报告基因实验证实高表达条件下p53在细胞内(in vivo)能与POLD1启动子直接结合并抑制POLD1启动子活性, 并且这种抑制作用是不依赖于细胞周期的[30]. 除影响p21表达外, 研究还发现, p53可以对Gadd4基因表达进行调控, 而Gadd4表达出的蛋白能够与PCNA, p21及一些细胞周期因子结合而影响DNA复制及细胞周期调控[31,32]. 由此, 我们认为, p53可能通过多条途径直接或间接的影响着POLD1基因的表达, 进而影响DNA的复制, 最终起到抑制肿瘤细胞恶性增殖的作用.
本研究通过构建真核表达载体, 经导入肝癌细胞SMMC-7721后得到可在体内高表达野生型p53的稳定细胞系SMMC-7721-pEGFP-C1-p53. 半定量RT-PCR检测结果发现: 肝癌细胞中p53高表达, 可对POLD1的mRNA表达水平产生明显的抑制作用. 与此同时, 我们还通过RNA干扰技术沉默SMMC-7721肝癌细胞中p53的表达, 结果发现肝癌细胞中p53的低表达后, POLD1基因的mRNA的表达量明显增高. 通过以上正反两方面的对比, 我们认为p53确实具有对POLD1表达的调控作用. 我们通过生长速率检测、平板集落形成率等实验, 发现肝癌细胞中p53表达上调确实能够抑制SMMC-7721细胞的增殖能力和锚定依赖性. 而p53低表达后SMMC-7721细胞的生长速率有所增加, 单细胞锚定依赖性成克隆能力增强. 这意味着p53对POLD1的调控作用可能是p53对细胞增殖和细胞恶性表型影响的一种新的分子通路. 通过从细胞水平和分子水平两个层面, 我们进一步验证了p53对POLD1的抑制作用, 同时也证明了用RNA干扰方法沉默p53可促进POLD1基因的表达. POLD1基因的表达水平与p53呈负相关, 这意味着p53可能参与了POLD1基因的调控, 并且这种作用有可能是p53对细胞增殖抑制及细胞恶性表型变化的调控途径之一. 这也意味着POLD1基因的异常调控和p125功能活性癌性表现与肝癌的发生和发展有着密切的联系.
总之, 野生型p53高表达能够抑制POLD1的基因转录细胞增殖活性, 而低表达能够促进POLD1的基因转录细胞增殖活性. 这对在肝癌细胞中p53影响POLD1基因表达的研究提供了实验依据. 我们将在现有的实验研究基础上, 对p53和其他已知或未知的细胞周期(增殖)调控因子协同调控POLD1基因表达的机理进行深入的研究, 以确定POLD1基因表达在肝癌细胞中的异常调控模式. 通过研究肝癌细胞中对POLD1基因癌性表达和p125功能活性癌性表现的抑制, 寻找肝癌细胞增殖阻断的靶点, 为发展有效的新的抗肝癌或抗肝癌细胞增殖性疾病药物提供基础.
DNA复制是细胞正常分裂、增殖的关键一步, DNA聚合酶δ是真核生物DNA复制的最主要复制酶, DNA聚合酶δ催化亚基基因1(POLD1)基因编码DNA聚合酶δ催化P125. 本文选择对POLD1有调节作用之一的抑癌基因p53, 探讨两者在癌的恶性增殖与DNA的复制之间的相互关系.
葛海燕, 教授, 同济大学附属第十人民医院普通外科; 李淑德, 主任医师, 中国人民解放军第二军医大学长海医院消化内科
研究表明, 在MCF-7细胞中高表达的p53可以抑制POLD1启动子的活性, 并且这种抑制作用具有剂量依赖效应, 但目前仍不清楚, 在肝癌细胞中高表达p53对POLD1的调控作用及改变p53表达水平后, POLD1的相应变化和细胞功能变化.
本文探讨在肝癌细胞SMMC-7721中, 不同p53基因表达水平下, POLD1基因的相应变化, 及由此而引起的细胞功能变化.
对p53和其他已知或未知的细胞周期(增殖)调控因子协同调控POLD1基因表达的机制进行深入的研究, 以确定POLD1基因表达在癌细胞中的异常调控模式. 通过研究癌细胞中对POLD1基因癌性表达, 寻找癌细胞增殖阻断的靶点, 为发展有效的新的抗癌或抗细胞增殖性疾病药物提供基础.
POLD1: 即DNA聚合酶δ催化亚基基因1, DNA聚合酶δ是真核生物DNA复制的最主要复制酶, DNA聚合酶δ催化亚基基因1(POLD1)基因编码DNA聚合酶δ催化亚基P125.
本文探讨野生型p53对肝癌细胞POLD1基因表达及细胞恶性表型的影响, 对探讨肝癌的分子发病机制及进一步指导治疗, 有重要临床意义.
编辑 曹丽鸥 电编 何基才
| 1. | Goldsby RE, Hays LE, Chen X, Olmsted EA, Slayton WB, Spangrude GJ, Preston BD. High incidence of epithelial cancers in mice deficient for DNA polymerase delta proofreading. Proc Natl Acad Sci U S A. 2002;99:15560-15565. [PubMed] [DOI] |
| 2. | Hazane F, Valenti K, Sauvaigo S, Peinnequin A, Mouret C, Favier A, Beani JC. Ageing effects on the expression of cell defence genes after UVA irradiation in human male cutaneous fibroblasts using cDNA arrays. J Photochem Photobiol B. 2005;79:171-190. [PubMed] [DOI] |
| 3. | Chang LS, Zhao L, Zhu L, Chen ML, Lee MY. Structure of the gene for the catalytic subunit of human DNA polymerase delta (POLD1). Genomics. 1995;28:411-419. [PubMed] [DOI] |
| 4. | Chung DW, Zhang JA, Tan CK, Davie EW, So AG, Downey KM. Primary structure of the catalytic subunit of human DNA polymerase delta and chromosomal location of the gene. Proc Natl Acad Sci U S A. 1991;88:11197-11201. [PubMed] [DOI] |
| 5. | Kemper RR, Ahn ER, Zhang P, Lee MY, Rabin M. Human DNA polymerase delta gene maps to region 19q13.3-q13.4 by in situ hybridization. Genomics. 1992;14:205-206. [PubMed] [DOI] |
| 6. | Venkatesan RN, Treuting PM, Fuller ED, Goldsby RE, Norwood TH, Gooley TA, Ladiges WC, Preston BD, Loeb LA. Mutation at the polymerase active site of mouse DNA polymerase delta increases genomic instability and accelerates tumorigenesis. Mol Cell Biol. 2007;27:7669-7682. [PubMed] [DOI] |
| 7. | Meng X, Zhou Y, Zhang S, Lee EY, Frick DN, Lee MY. DNA damage alters DNA polymerase delta to a form that exhibits increased discrimination against modified template bases and mismatched primers. Nucleic Acids Res. 2009;37:647-657. [PubMed] [DOI] |
| 8. | Yabuta T, Shinmura K, Yamane A, Yamaguchi S, Takenoshita S, Yokota J. Effect of exogenous MSH6 and POLD1 expression on the mutation rate of the HPRT locus in a human colon cancer cell line with mutator phenotype, DLD-1. Int J Oncol. 2004;24:697-702. [DOI] |
| 9. | Xu H, Zhang P, Liu L, Lee MY. A novel PCNA-binding motif identified by the panning of a random peptide display library. Biochemistry. 2001;40:4512-4520. [PubMed] [DOI] |
| 10. | Naryzhny SN. Proliferating cell nuclear antigen: a proteomics view. Cell Mol Life Sci. 2008;65:3789-3808. [PubMed] [DOI] |
| 11. | Zeng XR, Hao H, Jiang Y, Lee MY. Regulation of human DNA polymerase delta during the cell cycle. J Biol Chem. 1994;269:24027-24033. [PubMed] |
| 13. | Boehme KA, Blattner C. Regulation of p53--insights into a complex process. Crit Rev Biochem Mol Biol. 2009;44:367-392. [PubMed] [DOI] |
| 14. | Tokino T, Nakamura Y. The role of p53-target genes in human cancer. Crit Rev Oncol Hematol. 2000;33:1-6. [PubMed] [DOI] |
| 15. | Li B, Lee MY. Transcriptional regulation of the human DNA polymerase delta catalytic subunit gene POLD1 by p53 tumor suppressor and Sp1. J Biol Chem. 2001;276:29729-29739. [PubMed] [DOI] |
| 16. | Gartel AL, Tyner AL. Transcriptional regulation of the p21((WAF1/CIP1)) gene. Exp Cell Res. 1999;246:280-289. [PubMed] [DOI] |
| 17. | Liu G, Lozano G. p21 stability: linking chaperones to a cell cycle checkpoint. Cancer Cell. 2005;7:113-114. [PubMed] [DOI] |
| 18. | Spector DL, Goldman RD, Leinwand LA, 黄 培堂, 周 江, 张 映辉. 细胞实验指南(下册). 第1版. 北京: 科学出版社 2001; 783-784. |
| 19. | Parkin DM, Bray F, Ferlay J, Pisani P. Global cancer statistics, 2002. CA Cancer J Clin. 2005;55:74-108. [PubMed] [DOI] |
| 20. | Llovet JM, Burroughs A, Bruix J. Hepatocellular carcinoma. Lancet. 2003;362:1907-1917. [PubMed] [DOI] |
| 23. | Hallstrom TC, Nevins JR. Balancing the decision of cell proliferation and cell fate. Cell Cycle. 2009;8:532-535. [PubMed] [DOI] |
| 24. | Sclafani RA, Holzen TM. Cell cycle regulation of DNA replication. Annu Rev Genet. 2007;41:237-280. [PubMed] [DOI] |
| 25. | Nishitani H, Lygerou Z. Control of DNA replication licensing in a cell cycle. Genes Cells. 2002;7:523-534. [DOI] |
| 26. | Stillman B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 2008;30:259-260. [PubMed] [DOI] |
| 27. | Budzowska M, Kanaar R. Mechanisms of dealing with DNA damage-induced replication problems. Cell Biochem Biophys. 2009;53:17-31. [PubMed] [DOI] |
| 28. | Bandura JL, Calvi BR. Duplication of the genome in normal and cancer cell cycles. Cancer Biol Ther. 2002;1:8-13. [PubMed] [DOI] |
| 29. | Blow JJ, Gillespie PJ. Replication licensing and cancer--a fatal entanglement? Nat Rev Cancer. 2008;8:799-806. [PubMed] [DOI] |
| 30. | 朱 晓宇, 徐 恒, 李 莉, 张 海江, 宋 楠萌, 孙 建华, 石 磊, 桑 建利. p53亚细胞定位变化对POLD1基因启动子活性的影响. 自然科学进展. 2006;16:555-562. |