修回日期: 2010-09-20
接受日期: 2010-09-27
在线出版日期: 2010-11-08
目的: 探讨外周血DNA中肿瘤抑制候选基因4 (tumor suppressor candidate 4, TUSC4)缺失在大肠癌的筛查与早期诊断中的意义.
方法: 采集外周静脉血标本238例, 分为结肠镜正常组(117例), 腺瘤型息肉组(38例)和大肠癌组(83例), 应用PCR法检测TUSC4基因表达及缺失情况. 同时检测各组血浆CEA、CA19-9水平.
结果: 结肠镜正常组TUSC4阴性率14.5%, 腺瘤型息肉组阴性率44.7%, 大肠癌组阴性率77.1%, 3组之间均有统计学差异(P = 0.000). TUSC4阴性在诊断腺瘤型息肉的敏感度为44.7%(95%CI 28.9%-60.5%), 特异度85.5%(95%CI 79.1%-91.9%), PPV为50.0%(95%CI 33.2%-66.8%), NPV为82.6%(95%CI 75.8%-89.4%). TUSC4阴性在诊断大肠癌的敏感度为77.1%(95%CI 68.1%-86.1%), 特异度为85.5%(95%CI 79.1%-91.9%), PPV为79.0%(95%CI 70.1%-87.9%), NPV为84.0%(95%CI 77.4%-90.6%). TUSC4基因缺失与大肠癌发生部位、分化程度及Dukes分期无关. TUSC4阴性在诊断大肠癌时与CEA、CA19-9相比较, 其特异度和PPV无统计学差异(P>0.05), 其敏感度和NPV明显高于CEA和CA19-9(P = 0.000).
结论: 大肠癌患者外周血DNA中TUSC4基因出现明显缺失, 在诊断大肠癌时其敏感度明显高于CEA和CA19-9, 在大肠癌的分子筛查和分子诊断中具有一定应用价值.
引文著录: 裴凤华, 赵晶, 杜雅菊, 程艳秋, 王晓芬, 陈晶, 张会涛, 刘冰熔, 高善玲. 外周血DNA TUSC4基因缺失在大肠癌分子筛查及早期诊断中的应用. 世界华人消化杂志 2010; 18(31): 3320-3326
Revised: September 20, 2010
Accepted: September 27, 2010
Published online: November 8, 2010
AIM: To detect tumor suppressor candidate 4 (TUSC4) gene deletion in peripheral blood DNA and to evaluate its significance for molecular screening and diagnosis of colorectal carcinoma.
METHODS: Polymerase chain reaction was used to detect TUSC4 gene deletion in peripheral blood DNA samples from 238 subjects, including 117 subjects with normal colonoscopic findings, 38 patients with adenoma polyp, and 83 patients with colorectal carcinoma.
RESULTS: TUSC4 gene deletion was detected in peripheral blood DNA samples in 14.5%, 44.7% and 77.1% of normal subjects, adenoma polyp patients and colorectal carcinoma patients, respectively. The percentage of subjects carrying TUSC4 gene deletion differed significantly among the three groups of subjects (all P = 0.000). The sensitivity, specificity, positive predictive value (PPV) and negative predictive value (NPV) of detection of TUSC4 gene deletion in peripheral blood DNA for diagnosis of adenoma polyp were 44.7% (95%CI 28.9%-60.5%), 85.5% (95%CI 79.1%-91.9%), 50.0% (95%CI 33.2%-66.8%) and 82.6% (95%CI 75.8%-89.4%), respectively, while the corresponding values for diagnosis of colorectal carcinoma were 77.1% (95%CI 68.1%-86.1%), 85.5% (95%CI 79.1%-91.9%), 79.0% (95%CI 70.1%-87.9%) and 84.0% (95%CI 77.4%-90.6%). TUSC4 gene deletion in peripheral blood DNA was not significantly related to tumor site, differentiation degree and Dukes stage. Compared with detection of CEA and CA19-9, the specificity and PPV of detection of TUSC4 gene deletion for diagnosis of colorectal carcinoma were not significantly different (all P > 0.05). However, the sensitivity and NPV of detection of TUSC4 gene deletion for diagnosis of colorectal carcinoma were significantly higher than those of detection of CEA and CA19-9 (all P = 0.000).
CONCLUSION: USC4 gene deletion in peripheral blood DNA can be detected in patients with colorectal carcinoma. The sensitivity of detection of TUSC4 gene deletion for diagnosis of colorectal carcinoma is significantly higher than that of detection of CEA and CA19-9. Detection of TUSC4 gene deletion in peripheral blood DNA has significant value for molecular screening and diagnosis of colorectal carcinoma.
- Citation: Pei FH, Zhao J, Du YJ, Cheng YQ, Wang XF, Chen J, Zhang HT, Liu BR, Gao SL. Significance of detection of TUSC4 gene deletion in peripheral blood DNA for molecular screening and diagnosis of colorectal carcinoma. Shijie Huaren Xiaohua Zazhi 2010; 18(31): 3320-3326
- URL: https://www.wjgnet.com/1009-3079/full/v18/i31/3320.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i31.3320
大肠癌的发生、发展是由于一系列癌基因的激活和抑癌基因的失活引起的. 肿瘤抑制候选基因4(tumor suppressor candidate 4, TUSC4, GeneID: 10641)是近年新发现的一个抑癌基因[1], 无论在体内还是体外均能抑制肿瘤的活性, 亦称为NPRL2(nitrogen permease regulator-like 2)基因. TUSC4基因表达于许多正常组织, 在多种上皮性肿瘤组织中的表达明显降低[2], 但在外周血DNA中的缺失情况目前国内外尚无报道. 本文通过研究外周血DNA中TUSC4缺失情况, 探讨该基因对大肠癌的诊断意义, 为寻找新的大肠癌早期筛查方法提供依据.
选取2008-12/2010-04在哈尔滨医科大学附属第二医院消化内镜中心行结肠镜检查者共238例. 根据结肠镜和组织病理结果, 将其分为结肠镜正常组(117例)、腺瘤型息肉组(38例)和大肠癌组(83例). 凡入选患者均签署知情同意书, 并经我院伦理委员会讨论批准. 外周血DNA提取试剂盒购自TIANGEN公司, Premix Taq DNA聚合酶购自TaKaRa公司, PCR引物由上海超世生物科技有限公司设计并合成, 癌胚抗原(carcinoembryonic antigen, CEA)、癌抗原19-9(cancer antigen 19-9, CA19-9)ELISA试剂盒购自Adlitteram Diagnostic Laboratories公司. PCR仪(BIOER XP cycler).
1.2.1 样本处理: 抗凝管采集患者空腹静脉血5-10 mL, 立即离心分离血浆, 将血浆和血细胞成分分别-20 ℃保存备用.
1.2.2 总DNA提取: 按照DNA提取试剂盒说明书进行, 提取出的DNA在紫外分光光度计下测定浓度.
1.2.3 内参β-actin PCR扩增: 扩增体系20 μL: 2×Premix Taq DNA聚合酶10 μL, 上、下游引物各1 μL(10 μmol/L), DNA 2 μL, 去离子水6 μL. 内参β-actin引物序列: F: GGCACCCAGCACAATGAA; R: GGAAGGTGGACAGCGAGG. PCR扩增条件95 ℃预变性5 min, 95 ℃ 30 s, 58 ℃ 30 s, 72 ℃ 30 s, 35个循环, 72 ℃延伸5 min, PCR产物片段长度210 bp.
1.2.4 内参β-actin PCR产物鉴定: 取PCR产物5 μL加样于2%琼脂糖凝胶进行电泳, 电压150 V, 电泳20 min, 用紫外光凝胶成像系统扫描并拍摄, 以肉眼明确判定有目标条带为内参阳性.
1.2.5 TUSC4 PCR扩增: 选取内参阳性的DNA标本, 扩增体系20 μL: 2×Premix Taq DNA聚合酶10 μL, 上、下游引物各1 μL(10 μmol/L), DNA 2 μL, 去离子水6 μL. TUCS4引物序列: F: CACAACAAATCCTGCCCTACA; R: TCTTCATAAGCCCGAACTGG. PCR扩增条件95 ℃预变性5 min, 95 ℃ 30 s, 58 ℃ 30 s, 72 ℃ 30 s, 35个循环, 72 ℃延伸5 min, PCR产物片段长度910 bp.
1.2.6 TUSC4 PCR产物鉴定: 取PCR产物5 μL加样于2%琼脂糖凝胶进行电泳, 电压150 V, 电泳20 min, 用紫外光凝胶成像系统扫描并拍摄, 以肉眼明确判定有目标条带为阳性.
1.2.7 外周血浆CEA、CA19-9检测: 按照ELISA试剂盒说明书进行.
统计学处理 采用统计描述进行分析. 计算敏感度, 特异度, 阳性预测值(positive predictive value, PPV), 阴性预测值(negative predictive value, NPV)及其相应的95%可信区间(95% confidence interval, 95%CI). 计量资料用t检验, 计数资料用χ2检验或Fisher's检验, 数据处理用SPSS13.0软件包, P<0.05为差异有统计学意义.
入选病例共238例, 其中男143例, 女95例. 结肠镜正常117例, 平均年龄47.67岁±11.26岁. 腺瘤型息肉38例, 平均年龄55.89岁±12.97岁. 大肠癌83例, 平均年龄58.51岁±11.66岁, 其中直肠癌38例(45.8%), 结肠癌45例(54.2%), 高分化30例(36.1%), 中分化41例(49.4%), 低分化12例(14.5%). 年龄与大肠癌部位和分化程度无关(表1).
| 分组 | n | 男n(%) | 女n(%) | 平均年龄(岁, mean±SD) | t值 | P值 |
| 结肠镜正常组 | 117 | 64(54.7) | 53(45.3) | 47.67±11.26 | ||
| 腺瘤型息肉组 | 38 | 29(76.3) | 9(23.7) | 55.89±12.79b | -3.784 | 0.000 |
| 大肠癌组 | 83 | 50(60.2) | 33(39.8) | 58.51±11.66b | -6.611 | 0.000 |
| 直肠n(%) | 38(45.8) | 21(55.3) | 17(44.7) | 59.89±11.54 | ||
| 结肠n(%) | 45(54.2) | 29(64.4) | 16(35.6) | 57.33±11.76 | 0.997 | 0.322 |
| 高分化n(%) | 30(36.1) | 20(66.7) | 10(33.3) | 57.30±12.39 | ||
| 中分化n(%) | 41(49.4) | 23(56.1) | 18(43.9) | 59.32±11.63 | -0.702 | 0.485 |
| 低分化n(%) | 12(14.5) | 7(58.3) | 5(41.7) | 58.75±10.52 | -0.357 | 0.723 |
| 合计 | 238 | 143(60.1) | 95(39.9) |
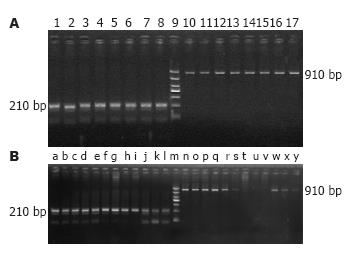
各组内参β-actin PCR产物在210 bp均有清晰条带. TUSC4 PCR产物阳性者在910 bp有清晰条带, TUSC4基因缺失者无条带(图1).
结肠镜正常组、腺瘤型息肉组和大肠癌组TUSC4基因缺失率分别为14.5%、44.7%和77.1%, 3组之间均有统计学差异(P = 0.000). 结肠镜正常组TUSC4缺失者中有结肠癌家族史占41.2%(7/17), 胃癌家族史占5.9%(1/17). 该基因缺失在诊断腺瘤型息肉的敏感度为44.7%(95%CI: 28.9%-60.5%), 特异度85.5%(95%CI: 79.1%-91.9%), PPV为50.0%(95%CI: 33.2%-66.8%), NPV为82.6%(95%CI: 75.8%-89.4%). 在诊断大肠癌的敏感度为77.1%(95%CI: 68.1%-86.1%), 特异度为85.5%(95%CI: 79.1%-91.9%), PPV为79.0%(95%CI: 70.1%-87.9%), NPV为84.0% (95%CI: 77.4%-90.6%, 表2).
直肠癌和结肠癌TUSC4缺失率分别为68.4%和84.4%, 二者无统计学差异(χ2 = 2.997, P = 0.083). 不同分化程度大肠癌的TUSC4缺失率无统计学差异(χ2 = 1.770, P = 0.413). Dukes分期A期和B期与C期和D期相比较, TUSC4缺失率无统计学差异(P = 0.416, 表3).
结肠镜正常组和腺瘤型息肉组CEA和CA19-9均为阴性, CEA诊断大肠癌的敏感度为36.7%, 特异度为100%, PPV为100%, NPV为44.1%; CA19-9敏感度为16.7%, 特异度100%, PPV 100%, NPV 37.5%; 二者联合检测敏感度40.0%, 特异度100%, PPV 100%, NPV 45.5%(表4-6).
| 分组 | n | 阳性数n(%) | 敏感度%(95%Cl) | 特异度%(95%Cl) | PPV%(95%Cl) | NPV%(95%Cl) |
| 结肠镜正常组 | 15 | 0 | ||||
| 腺瘤型息肉组 | 20 | 0 | 0.0 | 100(78.0-100.0) | - | 42.9(26.0-61.0) |
| 大肠癌组 | 30 | 11(36.7) | 36.7(20.0-56.0) | 100(78.0-100.0) | 100(72.0-100.0) | 44.1(27.0-62.0) |
| 分组 | n | 阳性数n(%) | 敏感度%(95%Cl) | 特异度%(95%Cl) | PPV%(95%Cl) | NPV%(95%Cl) |
| 结肠镜正常组 | 15 | 0 | ||||
| 腺瘤型息肉组 | 20 | 0 | 0.0(0.0-17.0) | 100(78.0-100.0) | - | 42.9(26.0-61.0) |
| 大肠癌组 | 30 | 5(16.7) | 16.7(6.0-35.0) | 100(78.0-100.0) | 100(48.0-100.0) | 37.5(23.0-54.0) |
| 分组 | n | 阳性数n(%) | 敏感度%(95%Cl) | 特异度%(95%Cl) | PPV%(95%Cl) | NPV%(95%Cl) |
| 结肠镜正常组 | 15 | 0 | ||||
| 腺瘤型息肉组 | 20 | 0 | 0.0(0.0-17.0) | 100(78.0-100.0) | - | 42.9(26.0-61.0) |
| 大肠癌组 | 30 | 12(40.0) | 40.0(23.0-59.0) | 100(78.0-100.0) | 100(74.0-100.0) | 45.5(28.0-64.0) |
TUSC4阴性与CEA、CA19-9、CEA+CA19-9比较, 其特异度和PPV无统计学差异(P>0.05), 其敏感度和NPV明显高于后者(P = 0.000, 表7).
| 敏感度%(n) | χ2(P)值 | 特异度%(n) | P值1 | PPV%(n) | P值1 | NPV%(n) | χ2(P)值 | |
| TUSC4缺失 | 77.1(64/83) | 85.5(100/117) | 79.0(64/81) | 84.0(100/119) | ||||
| CEA | 36.7(11/30)b | 16.147(0.000) | 100.0(15/15) | 0.216 | 100.0(11/11) | 0.207 | 44.1(15/34)b | 22.570(0.000) |
| CA19-9 | 16.7(5/30)b | 33.857(0.000) | 100.0(15/15) | 0.216 | 100.0(5/5) | 0.578 | 37.5(15/40)b | 32.388(0.000) |
| CEA+CA19-9 | 40.0(12/30)b | 13.779(0.000) | 100.0(15/15) | 0.216 | 100.0(12/12) | 0.114 | 45.5(15/33)b | 20.879(0.000) |
大肠癌是一种常见恶性肿瘤, 随着生活水平的提高, 生活习惯的改变, 大肠癌的发病有逐年增高的趋势, 大肠癌的病死率在美国上升到恶性肿瘤死亡率的第3位, 在中国上升到恶性肿瘤死亡率的第5位. 尽管近些年临床对大肠癌的诊治水平有明显提高, 但手术仍旧是主要的治疗方法, 若早期发现并进行手术治疗, 可以大大提高肿瘤患者的生存率, 因此治疗癌症的关键是早期诊断.
大肠癌常用的筛查方法有: 便潜血检测、血液肿瘤标志物检查、X线钡剂灌肠检查、结肠镜检查等. 较新的大肠癌筛查方法有: PET/CT、外周血或粪便基因检查、胶囊内镜检查等. 其中, 内镜检查或其他影像学检查均具有侵袭性或费用高等缺点, 无法针对普通人群进行大规模的筛查. 便潜血的检测虽然简单, 取材方便, 是大规模筛查的首选方法[3], 但是其影响因素较多, 且需在肿瘤形成后破溃出血的情况下才能检测到阳性结果. 目前, 外周血或粪便的基因检测受到越来越多的关注. 利用基因技术筛查可大大提前早期大肠癌的确诊时间, 甚至在出现典型病理变化之前就可明确诊断.
在结肠癌的发生和发展过程中伴有显著的基因改变. 其中, 原癌基因(如K-ras等)和肿瘤抑制基因(如APC, P53, DCC, DPC1, Bim等)的突变在结肠癌发生中起重要作用. 很多学者在大肠癌患者外周血和粪便中检测到了原癌基因[4,5]的表达和肿瘤抑制基因的失活, 并发现这种诊断方法比结肠镜检查有更高的特异性和敏感性.
TUSC4基因是Lerman等[1]从人3号染色体短臂上克隆得到的一种抑癌基因, 定位于人染色体3p21.3区, 其cDNA全长1 351 bp, 有11个外显子和10个内含子, 编码380个氨基酸的蛋白质, 从低级生物酵母到人类都具有一个高度保守的序列. 人类TUSC4基因表达于许多正常组织(包括心脏、脑、骨骼肌、肝脏、肾脏、胰腺和肺等), 在多种上皮性肿瘤组织中的表达明显降低(如肺癌[6]、肝癌[7]、食管癌[8]、鼻咽癌[9]、乳腺癌、肾细胞癌和卵巢癌等). 研究发现该基因的失活与多种肿瘤的发生、发展关系密切, 但其具体的机制尚不清楚. 该基因可能具有参与DNA错配修饰、调控细胞周期信号转导以及诱导细胞凋亡的生物学功能, 其作用机制可能与TUSC4启动子区纯合子缺失和微卫星不稳定性[10,11]有关, 可能使该基因的表达下调, 并改变了该基因的功能, 这一改变已通过Northern blot获得证实[12]. Li等[2]分析了8种肾癌细胞株, 发现肾癌细胞中TUSC4基因不存在DNA甲基化, 因此推测, TUSC4基因的失活可能与DNA甲基化无关. Kurata等[13]认为TUSC4是一种新的PDK1相关蛋白, 在调节Src/PDK1信号途径和细胞对多种抗癌化疗药的敏感性方面起到重要作用.
本研究应用常规PCR法, 通过检测TUSC4基因在大肠癌和癌前病变中外周血DNA中的缺失情况, 旨在寻找新的大肠癌分子肿瘤标志物, 为大肠癌的筛查与早期诊断提供新的依据. 在检测过程中, 我们应用了DNA的内参β-actin作为参照, 这样就最大限度地避免了其他因素造成的PCR扩增阴性结果对本研究的影响.
本研究发现, 在结肠镜正常组、腺瘤型息肉组和大肠癌组中, 外周血DNA TUSC4缺失率依次上升(分别为14.5%, 44.7%和77.1%), 3组之间互相均有显著性差异(P = 0.000), 说明在大肠癌发生发展过程中TUSC4基因即出现缺失的趋势, 在癌前病变即有近一半的患者出现该基因缺失. 值得高度关注的是, 在正常对照组中, TUSC4基因缺失者有肿瘤家族史者占47.1%(8/17), 其中41.2%(7/17)有大肠癌家族史. 这些患者作为高危人群, 出现TUSC4基因的缺失是否是发生肿瘤的高危信号, 还有待于密切的随访. 同时我们还发现, TUSC4基因缺失与大肠癌发生部位和分化程度无关, 因此推测该基因缺失是肿瘤发生的早期事件, 这更值得我们进一步关注该基因在肿瘤发生中的重要作用. 该基因的缺失还和大肠癌的Dukes分期无关(A、B期与C、D期缺失率无显著差异, P = 0.416), 这表明, 本研究所检测到的基因缺失能代表外周血淋巴细胞DNA(即胚系)所携带的突变, 而不是外周血中肿瘤细胞DNA所携带的突变, 这为进一步研究该基因在肿瘤发生和发展中的作用提供了依据.
外周血肿瘤标志物CEA、CA19-9一直是大肠癌传统的筛查和判断预后的指标[14,15], 本研究将TUSC4基因缺失与CEA、CA19-9在诊断大肠癌的价值做比较, 发现二者特异度和PPV无统计学差异(P>0.05), 而TUSC4基因缺失的敏感度和NPV明显高于CEA和CA19-9(P = 0.000). 鉴于其高敏感性, TUSC4基因缺失可作为一个选择性的基因用于大肠癌的筛查. 但目前尚无其他器官肿瘤外周血DNA中TUSC4基因缺失情况的资料, 推测在其他器官肿瘤中也可能有该基因的缺失, 如果事实如此, 该基因作为大规模筛查肿瘤的目标基因就有更大的应用价值.
大肠癌的防治已成为大肠癌研究的一大热点[16], 寻找与大肠癌发生、发展及预后相关基因和新的干预治疗靶点具有重要意义. TUSC4基因是一个新发现的肿瘤抑制基因, 该基因在多种肿瘤中失活, 表明他是参与肿瘤发生、发展的重要分子. 随着人们对TUSC4基因研究的深入, 其在肿瘤发生、发展过程中的作用将得到更全面、更透彻的阐明, 并能在分子水平上为肿瘤的基因诊断和治疗提供新的思路.
大肠癌是常见恶性肿瘤, 发病率逐年增高. 尽管近些年对大肠癌的诊治水平有明显提高, 但其关键仍是早期诊断. 目前基因检测受到越来越多的关注, 利用基因筛查技术可大大提前大肠癌的确诊时间, 甚至在出现典型病理变化之前就可明确诊断.
徐宁志, 研究员, 中国医学科学院肿瘤研究所细胞生物与分子生物学实验室
TUSC4基因是近年来新发现的肿瘤抑制基因, 在多种上皮性肿瘤组织中表达明显降低, 但在外周血中的表达情况国内外尚无报道.
Schenk等发现, 酵母细胞中的NPR2基因失活后, 该酵母细胞能抵抗顺铂的细胞毒性作用, 而人类NPRL2蛋白与酵母NPR2蛋白的结构十分相似, 可能其也有相同的生物学功能, 恢复耐药肿瘤细胞中NPRL2的正常表达后可以使肿瘤细胞对化疗药物重新敏感.
本研究发现在大肠癌癌变过程中, 外周血TUSC4基因缺失率明显上升, 其敏感性和特异性明显高于传统的肿瘤标志物, 因此在大肠癌的早期筛查中可能具有重要意义.
大肠癌的早期筛查对提高人群生存质量具有重要意义, 外周血TUSC4基因的缺失检测为早期筛查提供了新的分子生物学标记.
本文选题恰当, 科学性一般, 具有一定的临床参考意义.
编辑 李薇 电编 何基才
| 1. | Lerman MI, Minna JD. The 630-kb lung cancer homozygous deletion region on human chromosome 3p21.3: identification and evaluation of the resident candidate tumor suppressor genes. The International Lung Cancer Chromosome 3p21.3 Tumor Suppressor Gene Consortium. Cancer Res. 2000;60:6116-6133. [PubMed] |
| 2. | Li J, Wang F, Haraldson K, Protopopov A, Duh FM, Geil L, Kuzmin I, Minna JD, Stanbridge E, Braga E. Functional characterization of the candidate tumor suppressor gene NPRL2/G21 located in 3p21.3C. Cancer Res. 2004;64:6438-6443. [PubMed] [DOI] |
| 3. | Young GP, St John DJ, Winawer SJ, Rozen P. Choice of fecal occult blood tests for colorectal cancer screening: recommendations based on performance characteristics in population studies: a WHO (World Health Organization) and OMED (World Organization for Digestive Endoscopy) report. Am J Gastroenterol. 2002;97:2499-2507. [PubMed] |
| 4. | Villa E, Dugani A, Rebecchi AM, Vignoli A, Grottola A, Buttafoco P, Losi L, Perini M, Trande P, Merighi A. Identification of subjects at risk for colorectal carcinoma through a test based on K-ras determination in the stool. Gastroenterology. 1996;110:1346-1353. [PubMed] [DOI] |
| 5. | Smith-Ravin J, England J, Talbot IC, Bodmer W. Detection of c-Ki-ras mutations in faecal samples from sporadic colorectal cancer patients. Gut. 1995;36:81-86. [PubMed] |
| 6. | Senchenko VN, Anedchenko EA, Kondratieva TT, Krasnov GS, Dmitriev AA, Zabarovska VI, Pavlova TV, Kashuba VI, Lerman MI, Zabarovsky ER. Simultaneous down-regulation of tumor suppressor genes RBSP3/CTDSPL, NPRL2/G21 and RASSF1A in primary non-small cell lung cancer. BMC Cancer. 2010;10:75. [PubMed] [DOI] |
| 7. | Otani S, Takeda S, Yamada S, Sakakima Y, Sugimoto H, Nomoto S, Kasuya H, Kanazumi N, Nagasaka T, Nakao A. The tumor suppressor NPRL2 in hepatocellular carcinoma plays an important role in progression and can be served as an independent prognostic factor. J Surg Oncol. 2009;100:358-363. [PubMed] [DOI] |
| 8. | Yi Lo PH, Chung Leung AC, Xiong W, Law S, Duh FM, Lerman MI, Stanbridge EJ, Lung ML. Expression of candidate chromosome 3p21.3 tumor suppressor genes and down-regulation of BLU in some esophageal squamous cell carcinomas. Cancer Lett. 2006;234:184-192. [PubMed] [DOI] |
| 9. | Chow LS, Lo KW, Kwong J, To KF, Tsang KS, Lam CW, Dammann R, Huang DP. RASSF1A is a target tumor suppressor from 3p21.3 in nasopharyngeal carcinoma. Int J Cancer. 2004;109:839-847. [PubMed] [DOI] |
| 10. | Economidou F, Tzortzaki EG, Schiza S, Antoniou KM, Neofytou E, Zervou M, Lambiri I, Siafakas NM. Microsatellite DNA analysis does not distinguish malignant from benign pleural effusions. Oncol Rep. 2007;18:1507-1512. [PubMed] |
| 11. | Castagnaro A, Marangio E, Verduri A, Chetta A, D'Ippolito R, Del Donno M, Olivieri D, Di Cola G. Microsatellite analysis of induced sputum DNA in patients with lung cancer in heavy smokers and in healthy subjects. Exp Lung Res. 2007;33:289-301. [PubMed] [DOI] |
| 12. | Takahashi Y, Kondo K, Hirose T, Nakagawa H, Tsuyuguchi M, Hashimoto M, Sano T, Ochiai A, Monden Y. Microsatellite instability and protein expression of the DNA mismatch repair gene, hMLH1, of lung cancer in chromate-exposed workers. Mol Carcinog. 2005;42:150-158. [PubMed] [DOI] |
| 13. | Kurata A, Katayama R, Watanabe T, Tsuruo T, Fujita N. TUSC4/NPRL2, a novel PDK1-interacting protein, inhibits PDK1 tyrosine phosphorylation and its downstream signaling. Cancer Sci. 2008;99:1827-1834. [PubMed] |
| 14. | Kim JY, Kim NK, Sohn SK, Kim YW, Kim KJ, Hur H, Min BS, Cho CH. Prognostic value of postoperative CEA clearance in rectal cancer patients with high preoperative CEA levels. Ann Surg Oncol. 2009;16:2771-2778. [PubMed] [DOI] |
| 15. | Filiz AI, Sucullu I, Kurt Y, Karakas DO, Gulec B, Akin ML. Persistent high postoperative carcinoembryonic antigen in colorectal cancer patients--is it important? Clinics (Sao Paulo). 2009;64:287-294. [PubMed] |
| 16. | Pignone M, Levin B. Recent developments in colorectal cancer screening and prevention. Am Fam Physician. 2002;66:297-302. [PubMed] |