修回日期: 2010-04-10
接受日期: 2010-04-20
在线出版日期: 2010-06-08
目的: 了解hMLH1基因Val384Asp错义突变在家族性胃癌发病中的作用及Val384Asp可能的致病分子机制.
方法: 于江苏省淮安、泰兴及金坛三个肿瘤高发地区行现场调查, 收集到100例2004年新发家族性或疑似家族性胃癌患者、180例健康人的外周血, 应用PCR-DHPLC和DNA序列分析技术检测细胞的DNA, 分析hMLH1基因的第12外显子, 检测Val384Asp错义突变. 生物信息学方法进行蛋白三维模型构建以及结构和功能学分析.
结果: 家族性/疑似家族性胃癌患者Val384Asp检出率显著高于正常对照(OR = 2.84, 95%CI: 1.07-7.81, P<0.05). 分组分析显示, Val384Asp与胃癌相关癌前疾病有协同性(P<0.01), 并在发病年龄≥50岁的患者中显著分布(P<0.05). 此外, 对胃癌家系进行患病风险的评估后, 发现来自高风险家系的患者检出率较高(P<0.05). 蛋白三维模型显示, Val384Asp可以引起氢键的改变, 可能影响hMLH1蛋白局部构象. 功能学分析显示, 降低蛋白功能和影响剪切调控是Val384Asp可能的致病机制.
结论: 中国家族性胃癌家系中的Val384Asp携带者可能有更高的胃癌发病风险.
引文著录: 王德强, 周建农, 宋磊, 李苏平, 丁建华, 李金田, 马国建, 陈森清, 张晓梅. Val384Asp错义突变在家族性胃癌患者中的检出及其致病分子机制. 世界华人消化杂志 2010; 18(16): 1669-1675
Revised: April 10, 2010
Accepted: April 20, 2010
Published online: June 8, 2010
AIM: To investigate the etiological role of the missense mutation, Val384Asp, in the human mutL homolog 1 (hMLH1) gene in familial gastric cancer (FGC) based on a Chinese population in Jiangsu Province.
METHODS: A case-control study was conducted. One hundred newly diagnosed or suspected FGC patients and 180 healthy controls were included in the study. Peripheral white blood cells were obtained from all subjects for DNA extraction. The Val384Asp missense mutation was detected using PCR-based denaturing high-performance liquid chromatography (DHPLC) and verified by DNA sequencing. Bioinformatic software was then used to analyze the etiological mechanism of the Val384Asp missense mutation.
RESULTS: About 5% healthy individuals were Val384Asp carriers. Significant differences were noted for the following comparisons: patients with newly diagnosed or suspected FGC vs healthy controls (OR = 2.84, 95%CI: 1.07-7.81, P < 0.05), patients with an onset age ≥ 50 vs healthy controls (P < 0.05), patients with precancerous disease history vs healthy controls (P < 0.01), and patients having a high-risk family history of GC vs healthy controls (P < 0.05). Bioinformatic analysis showed that the Val384Asp missense mutation might destroy the structure of hMLH1 protein and impair its function. Besides, the conversion of T→A may disrupt pre-mRNA splicing.
CONCLUSION: The Val384Asp missense mutation may be associated with genetic susceptibility to FGC. Detection of the Val384Asp missense mutation may be able to help identify individuals with increased risk of FGC.
- Citation: Wang DQ, Zhou JN, Song L, Li SP, Ding JH, Li JT, Ma GJ, Chen SQ, Zhang XM. Genotype frequencies of the Val384Asp missense mutation in the hMLH1 gene in patients with familial gastric cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(16): 1669-1675
- URL: https://www.wjgnet.com/1009-3079/full/v18/i16/1669.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i16.1669
胃癌是人类常见的疾病, 发病率在全部恶性肿瘤中位居第4位, 致死率高居第2位[1]. 大多数胃癌为散发性, 但约10%的胃癌呈家族聚集性[2]. 病理类型为弥漫型胃癌的家族聚集现象较肠型胃癌更为常见, 弥漫型胃癌患者亲属的患病风险增加达7倍, 远高于肠型的1.4倍[3]. 我国胃癌发病率仍处于上升期, 上述风险性的增加不容忽视, 但目前可用于高危人群筛查的家族性胃癌相关的遗传指标仍十分有限[4]. 在前期研究中, 我们曾在24例有癌症家族史的胃癌患者及其亲属中发现一显著分布的错义突变-错配修复(mismatch repair, MMR)基因hMLH1, Val384Asp[5]. 随后的研究发现, Val384Asp是一个具有东亚人特异性的突变位点, 而西方人群中未见报道[6-9]. 数位研究者在多种肿瘤患者中检出了该突变, 并且证实该突变与其中几种的发病有密切关系[10-12]. 但Val384Asp与家族性肿瘤的关系, 未见进一步的报道. 在此, 我们通过广泛地收集家族性或疑似家族性胃癌样本, 对Val384Asp可能的病因学作用作一研究.
选择分处于江苏省北部、中部和南部的淮安、泰州和金坛三个肿瘤高发地区, 基于当地疾控中心的肿瘤登记制度, 按如下标准筛选研究对象: 家族性胃癌的临床诊断标准[13]: (1)胃癌患者数≥3例, 且为一级亲属者≥1例; (2)至少连续两代发病; (3)发病年龄<50岁者≥1例. 有胃癌或胃癌相关肿瘤家族史但不符合上述标准的为疑似家族性胃癌患者[14]. 7例家族性、93例疑似家族性2004年新发、仍生存且同意采血的胃癌患者入选. 参考大肠癌及HNPCC家系的发病风险预测标准分成两个级别[15]: (1)高风险家系来源45例, 即符合以下任一项: 家系中胃癌或胃癌相关肿瘤患者数≥3例; 一个直系亲属或2级亲属患有≥2种胃癌相关肿瘤; 一个直系亲属的发病年龄<50岁; (2)其余55例不符合上述条件但有明确家族史, 为中等风险家系来源. 此外, 胃溃疡、萎缩性胃炎、残胃炎及胃息肉为公认的胃癌相关癌前疾病[16,17], 本研究有癌前疾病史的患者55例. 正常对照为同一地区的健康人, 无胃癌相关癌前疾病, 各例之间无血缘关系, 其一级亲属中无明确的癌症患者, 男126例, 女54例, 年龄16-70岁.
1.2.1 基因型分析: PCR引物序列: 5'-ACAGACTTTGCTACCAGGACTTG23'(上游), 5'-TGTCTTATCCTCTGTGACAATGG23'(下游). 扩增产物位于hMLH1基因第12外显子上游24位碱基至第12外显子内编码基因的第1 251位碱基, 长度216 bp. 25 μL PCR反应体系, 内含50-100 ng基因组DNA, 0.2 mmol/L dNTP, 上下游引物各0.5 μmol/L, 2 U Taq DNA聚合酶. PT2200梯度扩增循环条件: 94 ℃预变性5 min, 94 ℃ 20 s, 59 ℃ 20 s, 72 ℃ 30 s, 35 个循环, 72 ℃延长5 min, 94 ℃变性4 min, 按0.1 ℃/s的梯度下降温度至25 ℃, 4 ℃保存. 1.2%琼脂糖凝胶电泳检测. PCR产物上DHPLC仪(WAVE系统, Transgenomic)分析. 单次进样5-8 μL, 柱温62 ℃, 流动相为0.1 mol/L N-三乙基乙酰胺(TEAA, 分析纯)和不同浓度梯度的乙腈, 流速0.9 mL/min, 260 nm紫外检测. 对AA纯合变异基因型的筛选: 通过测序得到一部分TT野生型PCR产物, 大量扩增后与DHPLC图谱中未出现异常洗脱峰的PCR产物两两混合(各取5-10 μL), 94 ℃变性4 min, 按0.1 ℃/s的梯度下降温度至25 ℃后得到杂交产物, 原AA型杂交为AT, 原TT型不变, 以上述方式上DHPLC仪分析. 对DHPLC图谱中出现异常洗脱峰的DNA样品作为检测重点, 重新进行PCR扩增并纯化产物后测序. 对于基因分型结果, 再随机挑选10%的样本重复实验.
1.2.2 蛋白模型建立及结构与功能分析: 在Protein Data Bank(PDB)和SWISS-MODEL的模板数据库搜索结构模板, 在SWISS-MODEL中以alignment mode模式进行构建, 并进行质量评价, 方法详见参考文献[18]. 进一步在Modbase[19]中搜索, 与已有的模型进行比对, 在SWISS-MODEL专用软件DeepView中进行优化. 含Val384Asp的模型通过DeepView中的Mutation命令获得, 并通过能量最小化等处理后与正常结构进行对比. 功能学分析使用的软件包括: (1)ClustalW软件[20]对比不同物种间hMLH1同源基因序列, 分析第384位氨基酸是否为一保守位点; (2)SIFT(Sort intolerant from tolerant)及PolyPhen(Polymorphism Phenotype)软件[21,22]预测点突变对蛋白二级结构, 链间反应以及功能位点的影响; (3)ESEfinder3.0软件[23]寻找外显子剪切增强子(exonic splicing enhancers, ESEs)基序, 分析基因变异对剪切调控的影响.
统计学处理 采用Stata9.0软件, Fisher exact χ2检测和风险度分析, P<0.05为有统计学差异.
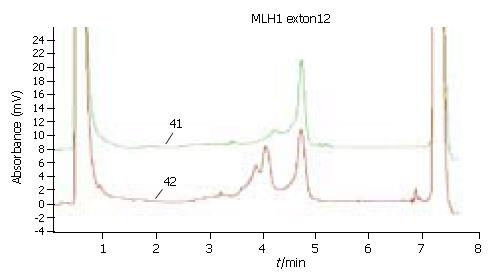
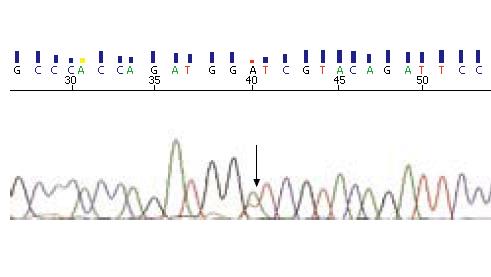
在正常对照中有9例出现一致的DHPLC异常峰型, 相同的峰型见于13例家族性/疑似家族性胃癌患者(图1). DNA序列分析表明这一异常峰型的出现是由于hMLH1基因第1 151位碱基存在遗传性的T→A的颠换(杂合型, 图2), 使得该碱基所处的第384位密码子发生撷氨酸(Val)到天冬氨酸(Asp)的错义突变.
家族性/疑似家族性胃癌患者中Val384Asp检出率高于正常对照, 差异有统计学意义(P<0.05). 风险度分析显示, 携有Val384Asp错义突变的个体患病风险显著升高, 增加值为184%(OR = 2.84, 95%CI: 1.07-7.81, 表1).
| 分组 | Val384Asp检测人数n(%) | P值 | OR(95%CI) | ||
| 携带者 | 非携带者 | 合计 | |||
| 正常对照 | 9(5.0) | 171(95.0) | 180 | 0.021 | 2.84(1.07-7.81) |
| 病例组 | 13(13.0) | 87(87.0) | 100 | ||
组间比较表明, 有癌前疾病史的患者中Val384Asp检出率高于无癌前疾病史的患者, 差异有统计学意义(P<0.05, 表2). 本研究仅检出1例纯合型突变. 我们以等位基因概念比较各组患者中Val384Asp等位基因频率与正常人群的差异, 见表3. 结果表明, Val384Asp在有癌前疾病史的患者中有特别高的检出率(P<0.01); 并在来自高风险家系的患者及发病年龄≥50岁患者中的检出率均显著高于正常人群, 差异有统计学意义(P<0.05, 表3).
| 分组 | Val384Asp检测人数n(%) | P值 | ||
| 携带者 | 非携带者 | 合计 | ||
| 有癌前疾病史 | 11(20.0) | 44(80.0) | 55 | 0.034 |
| 无癌前疾病史 | 2(4.4) | 43(95.6) | 45 | |
| 高风险家系 | 8(17.8) | 37(82.2) | 45 | 0.240 |
| 中等风险家系 | 5(9.1) | 50(90.9) | 55 | |
| <50岁 | 2(9.1) | 20(90.9) | 22 | 0.727 |
| ≥50岁 | 11(14.1) | 67(85.9) | 78 | |
| 分组 | 密码子(%) | P值 | OR(95%CI) | ||
| 384GAT | 384GTT | 合计 | |||
| 正常对照 | 9(2.5) | 351(97.5) | 360 | 1.00 | |
| 有癌前疾病史 | 12(10.9)n | 98(89.1) | 110 | 0.001 | 4.78(1.78-13.18) |
| 无癌前疾病史 | 2(2.2) | 88(97.8) | 90 | 1.000 | 0.89(0.09-4.39) |
| 高风险家系 | 8(8.9) | 82(91.1) | 90 | 0.010 | 3.80(1.23-11.45) |
| 中等风险家系 | 6(5.5)n | 104(94.5) | 110 | 0.129 | 2.25(0.64-7.25) |
| <50岁 | 2(4.5) | 42(95.5) | 44 | 0.341 | 1.86(0.19-9.39) |
| ≥50岁 | 12(7.7)n | 144(92.3) | 156 | 0.013 | 3.25(1.22-8.92) |
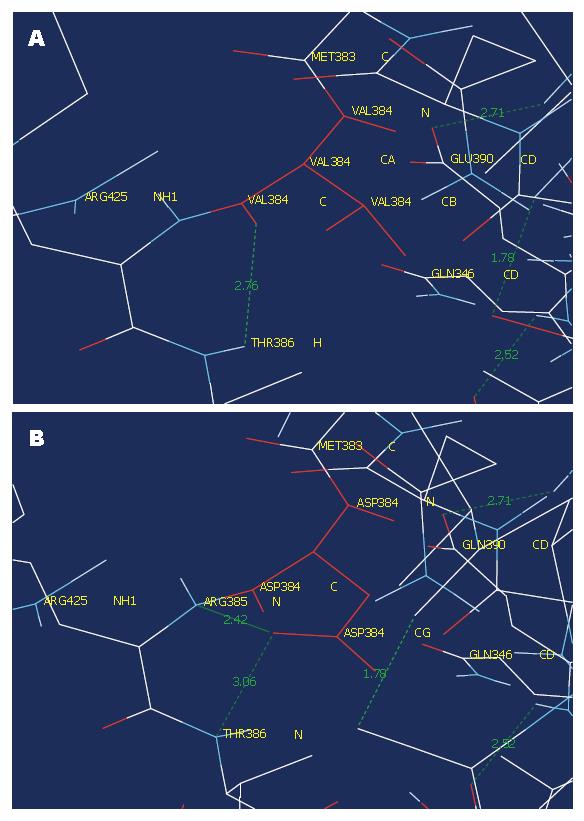
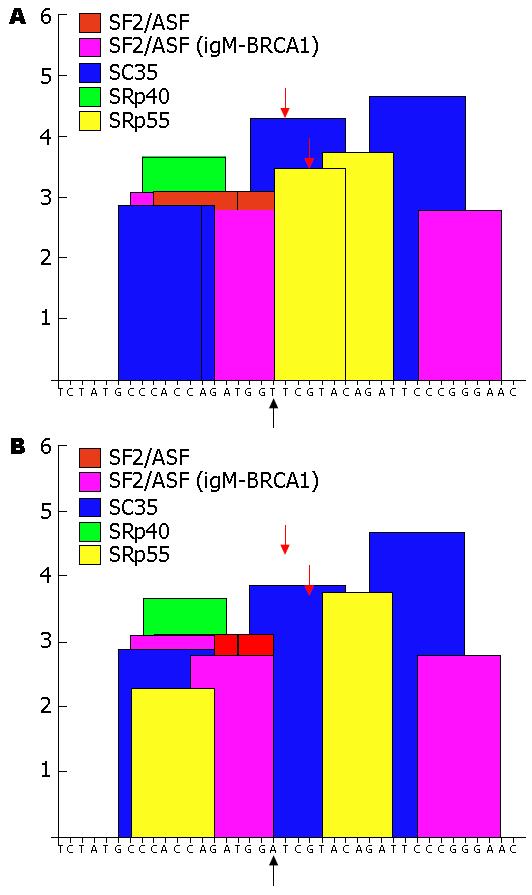
蛋白模型表明, 第384位的Val由Asp所替代, 将使hMLH1蛋白三维结构中的氢键(H-bond)发生改变, 表现为氢键VAL384C...THR386H(2.76Å)改变为ASP384CG...THR386N(3.06Å); 并出现额外的氢键ASP384CG...ARG385N(2.42Å), 见图3. ClustalW软件对比不同物种间hMLH1基因序列发现, 第384位的Val还存在于小鼠或家鼠、果蝇(Drosophila)、斑马鱼(zebrafish)等生物中, 可能是物种进化中的一个保守位点(输出结果未显示); SIFT及PolyPhen均显示该位点中撷氨酸(Val)到天冬氨酸(Asp)的替换可能影响hMLH1蛋白功能, 影响评价分别为"intolerant"、"probably damaging"[21,22](输出结果未显示); ESEfinder3.0软件分析该处碱基T→A的颠换对剪切调控的影响, 发现突变碱基A可能改变作为剪切调控元件的外显子剪切增强子的活性(图4).
家族性胃癌发病的遗传学基础因地区和人种不同而存在差异, 表现为欧洲家系以钙黏附素基因(E-cadherin gene, CDH1)突变常见, 但日本和韩国等东亚家系较少发现相关CDH1突变, 而在MET, TP-53等基因中检出了大量胚系突变[24-34]. 中国的家族性胃癌还存在大量MMR基因胚系突变或多态位点[14,35].
MMR基因介导的错配修复是机体内DNA修复的一种重要机制, 对维护基因组稳定起着关键作用[36]. MMR基因异常与肿瘤的相关性已得到广泛证实, 但其与家族性胃癌的关系仍存争议[13]. 有人认为家族性胃癌中MMR基因的异常主要表现为启动子序列的高甲基化, 而基因变异较少见[37]. 但是, 中国家族性胃癌相关的MMR基因变异可能是一经常性事件, 因为中国以MMR基因变异为主要病因的遗传性非息肉病性结直肠癌(hereditary nonpolyposis colorectal cancer, HNPCC)家系中, 胃癌的发生率远高于西方, 是中国HNPCC最常见的肠外肿瘤, 常形成胃癌的家族聚集现象[38,39], 提示MMR基因与中国家族性胃癌的密切关系.
Val384Asp位于MMR基因之一的hMLH1第12外显子上, 该错义突变具有种族特异性, 在中国、日本和韩国人群中均有分布, 突变碱基A的频率在3%-6%间, 但在德国及美国人群中尚无发现[6-9]. Val384Asp与肿瘤的关系已得到广泛证实. Wang等首先于胃癌、大肠癌中发现了该突变, 并证实其为二者中低龄散发患者的遗传易感因素[5,10]. 随后的研究发现, Val384Asp与非小细胞肺癌的发病也存在密切关系, 但却可能是前列腺癌的保护因素, 而与食管癌及乳腺癌的发病无显著关系[10-12]. 但这些研究均局限于散发性肿瘤患者, 而Val384Asp在遗传影响度更高的家族性患者中是否有类似作用, 尚未见报道.
本研究显示, 在家族性及疑似家族性胃癌患者中, Val384Asp仍存在显著分布(P<0.05), 使携带者的患病风险大为增加. 但通过对年龄的分层分析后发现, 与散发性胃癌中该多态主要分布于低龄患者的现象相反[5,10], 其仅在高龄家族性(疑似)胃癌患者中较对照有显著差异, 差异的具体原因尚不清楚, 但不能排除源于小样本相关偏倚的可能.
分析还显示Val384Asp与癌前疾病有协同作用. 胃癌的癌前疾病是指与胃癌相关的胃良性疾病, 有发生胃癌的危险性, 常是幽门螺杆菌感染相关的[40]. 研究表明, 幽门螺杆菌对hMLH1, hMSH2等错配修复基因正常表达的抑制可能是其致癌的重要机制之一[41]. Val384Asp则可能进一步加重这一状况. 因为既往研究认为, Val384Asp可能削弱hMLH1蛋白的功能, 导致DNA错配修复能力下降, 产生基因组的不稳定性. 这可能使癌前疾病中已有癌变倾向的上皮细胞对环境因素更易感, 基因突变频率增加.
另外, 流行病学研究证明, 影响肿瘤家族聚集性最重要的临床因素是一等亲中患病的人数和发病年龄. 以大肠癌为例, 一等亲中如有一人患大肠癌, 则罹患该病的危险比普通人群增加2-4倍; 如一等亲中有2人患大肠癌, 则罹患该病的危险比普通人群增加3-6倍; 如果某一等亲在<50岁时被诊断为大肠癌, 则与前述情况具有相似的危险; 而二等亲或三等亲患大肠癌, 罹患该病的危险比普通人群仅增加30%-50%[42-44]. 与之类似, 胃癌家系之间因一等亲中患病的人数和发病年龄的差异, 其家系成员也应有不同的患病风险. 因此, 我们借鉴了大肠癌及HNPCC家系的发病风险预测标准对本研究的患者进行评估, 发现高风险家系来源的患者中有更显著的Val384Asp分布, 提示Val384Asp可协助家系危险性的评价.
Val384Asp的结构和功能学研究尚缺如, 本研究用生物信息学方法对此进行了分析. 生物信息学是一门新兴的交叉学科, 包含了大量权威机构精心设计的分析软件及各种数据库汇聚所得的巨量实用信息, 已成为生物学研究的得力辅助工具. 我们发现Val384可能是一个保守位点, 研究表明MMR蛋白保守区域中能够使氨基酸残基发生改变的变异常是致病性的[45,46]. 三维结构模型显示Val384Asp将引起氢键的改变, 而氢键是蛋白结构特别是二级结构的重要组成, 提示相关改变可能影响蛋白功能. 其余软件的分析也进一步表明Val384Asp可能削弱hMLH1蛋白的功能, 并且MMR基因中类似变异对相应蛋白功能的下调已得到证实[47]. 此外, 我们还发现Val384Asp的另一可能的致病机制, 即突变碱基对剪切调控的影响. 事实上, UniProt蛋白数据库将Val384Asp定义为"不能分类的变异"(unclassified variants, Uvs), 而Uvs的致病机制正与剪切调控有关[48,49]. 这种影响在一定条件下可能使相应外显子在转录后剪切修饰过程中部分或全部失落(skipping), 导致编码蛋白的功能严重缺陷[50]. 上述发现有利于对Val384Asp病因学的解释, 但仍需体内外研究的确认.
总之, Val384Asp与我国家族性/疑似家族性胃癌患者的发病有一定的关联, 现有证据表明其可能是一低度外显、条件性致病的遗传风险因素, 是对我国家族性胃癌遗传背景研究的有效补充. 进一步扩大样本量并在各家系中展开纵向研究以及体内外的功能学研究是必要的.
目前可用于高危人群筛查的家族性胃癌相关的遗传指标仍十分有限. 在前期研究中, 本课题组曾在24例有癌症家族史的胃癌患者及其亲属中发现一显著分布的错义突变-错配修复(MMR)基因hMLH1, Val384Asp. 随后的研究发现, Val384Asp是一个具有东亚人特异性的突变位点, 而西方人群中未见报道. 数位研究者在多种肿瘤患者中检出了该突变, 并且证实该突变与其中几种的发病有密切关系. 但Val384Asp与家族性肿瘤的关系, 未见进一步的报道.
刘改芳, 主任医师, 河北医科大学第三医院消化内科
MMR基因与中国家族性胃癌可能存在密切关系, 但目前相关研究甚少. MMR基因变异位点的功能学研究是目前的热点.
本研究了Val384Asp与中国家族性/疑似家族性胃癌患者发病的关系, 并通过生物信息学方法初步探索了Val384Asp可能的致病机制.
本研究是对中国家族性胃癌遗传背景研究的有效补充, 发现了Val384Asp可作为中国家族性/疑似家族性胃癌高危人群的候选筛查指标. 进一步扩大样本量并在各家系中展开纵向研究以及体内外的功能学研究是必要的.
本研究选题新颖, 层次和表达清晰, 资料齐全, 具有一定的流行病学和临床价值.
编辑: 李军亮 电编: 何基才
| 1. | Parkin DM, Pisani P, Ferlay J. Estimates of the worldwide incidence of 25 major cancers in 1990. Int J Cancer. 1999;80:827-841. [PubMed] [DOI] |
| 2. | Oliveira C, Seruca R, Carneiro F. Genetics, pathology, and clinics of familial gastric cancer. Int J Surg Pathol. 2006;14:21-33. [PubMed] [DOI] |
| 3. | Lehtola J. Family study of gastric carcinoma; With special reference to histological types. Scand J Gastroenterol Suppl. 1978;50:3-54. [PubMed] |
| 5. | 王 亚平, 周 建农, 李 忠佑, 王 建东, 李 金田, 高 长明, 高 萍. 中国人消化道肿瘤发病的遗传背景因素-错配修复基因hMLH1错义突变Va1384Asp. 中华医学遗传学杂志. 2000;17:82-86. |
| 6. | Wang Y, Friedl W, Lamberti C, Nöthen MM, Kruse R, Propping P. A novel missense mutation in the DNA mismatch repair gene hMLH1 present among East Asians but not among Europeans. Hum Hered. 1998;48:87-91. [PubMed] [DOI] |
| 7. | Mei Q, Yan HL, Ding FX, Xue G, Huang JJ, Wang YZ, Sun SH. Single-nucleotide polymorphisms of mismatch repair genes in healthy Chinese individuals and sporadic colorectal cancer patients. Cancer Genet Cytogenet. 2006;171:17-23. [PubMed] [DOI] |
| 8. | Kim JC, Roh SA, Koo KH, Ka IH, Kim HC, Yu CS, Lee KH, Kim JS, Lee HI, Bodmer WF. Genotyping possible polymorphic variants of human mismatch repair genes in healthy Korean individuals and sporadic colorectal cancer patients. Fam Cancer. 2004;3:129-137. [PubMed] [DOI] |
| 9. | Moslein G, Tester DJ, Lindor NM, Honchel R, Cunningham JM, French AJ, Halling KC, Schwab M, Goretzki P, Thibodeau SN. Microsatellite instability and mutation analysis of hMSH2 and hMLH1 in patients with sporadic, familial and hereditary colorectal cancer. Hum Mol Genet. 1996;5:1245-1252. [PubMed] [DOI] |
| 10. | 张 晓梅, 李 金田, 朱 明, 吴 晓, 柳 高萍, 周 平, 王 亚平. 错配修复基因hMLH1错义突变Val384Asp与四种肿瘤遗传易感性的研究. 中华流行病学志. 2004;25:978-981. |
| 11. | Tanaka Y, Zaman MS, Majid S, Liu J, Kawakami K, Shiina H, Tokizane T, Dahiya AV, Sen S, Nakajima K. Polymorphisms of MLH1 in benign prostatic hyperplasia and sporadic prostate cancer. Biochem Biophys Res Commun. 2009;383:440-444. [PubMed] [DOI] |
| 12. | Shi X, Xu G, Zhao C, Ma J, Zhang Y, Lv S, Yang Q. A single-strand conformation polymorphism method by capillary electrophoresis with laser-induced fluorescence for detection of the T1151A mutation in hMLH1 gene. Electrophoresis. 2003;24:2316-2321. [PubMed] [DOI] |
| 13. | Barber M, Fitzgerald RC, Caldas C. Familial gastric cancer - aetiology and pathogenesis. Best Pract Res Clin Gastroenterol. 2006;20:721-734. [PubMed] [DOI] |
| 14. | Zhang Y, Liu X, Fan Y, Ding J, Xu A, Zhou X, Hu X, Zhu M, Zhang X, Li S. Germline mutations and polymorphic variants in MMR, E-cadherin and MYH genes associated with familial gastric cancer in Jiangsu of China. Int J Cancer. 2006;119:2592-2596. [PubMed] [DOI] |
| 15. | Hampel H, Sweet K, Westman JA, Offit K, Eng C. Referral for cancer genetics consultation: a review and compilation of risk assessment criteria. J Med Genet. 2004;41:81-91. [PubMed] [DOI] |
| 16. | Hatakeyama M. Helicobacter pylori and gastric carcinogenesis. J Gastroenterol. 2009;44:239-248. [PubMed] [DOI] |
| 17. | El-Zimaity H. Gastritis and gastric atrophy. Curr Opin Gastroenterol. 2008;24:682-686. [PubMed] [DOI] |
| 18. | Arnold K, Bordoli L, Kopp J, Schwede T. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling. Bioinformatics. 2006;22:195-201. [PubMed] [DOI] |
| 19. | Pieper U, Eswar N, Webb BM, Eramian D, Kelly L, Barkan DT, Carter H, Mankoo P, Karchin R, Marti-Renom MA. MODBASE, a database of annotated comparative protein structure models and associated resources. Nucleic Acids Res. 2009;37:D347-D354. [PubMed] [DOI] |
| 20. | Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R. Clustal W and Clustal X version 2.0. Bioinformatics. 2007;23:2947-2948. [PubMed] [DOI] |
| 21. | Ng PC, Henikoff S. Predicting deleterious amino acid substitutions. Genome Res. 2001;11:863-874. [PubMed] [DOI] |
| 22. | Ramensky V, Bork P, Sunyaev S. Human non-synonymous SNPs: server and survey. Nucleic Acids Res. 2002;30:3894-3900. [PubMed] [DOI] |
| 23. | Cartegni L, Wang J, Zhu Z, Zhang MQ, Krainer AR. ESEfinder: A web resource to identify exonic splicing enhancers. Nucleic Acids Res. 2003;31:3568-3571. [PubMed] [DOI] |
| 24. | Guilford P, Hopkins J, Harraway J, McLeod M, McLeod N, Harawira P, Taite H, Scoular R, Miller A, Reeve AE. E-cadherin germline mutations in familial gastric cancer. Nature. 1998;392:402-405. [PubMed] [DOI] |
| 25. | Brooks-Wilson AR, Kaurah P, Suriano G, Leach S, Senz J, Grehan N, Butterfield YS, Jeyes J, Schinas J, Bacani J. Germline E-cadherin mutations in hereditary diffuse gastric cancer: assessment of 42 new families and review of genetic screening criteria. J Med Genet. 2004;41:508-517. [PubMed] [DOI] |
| 26. | Kim HC, Wheeler JM, Kim JC, Ilyas M, Beck NE, Kim BS, Park KC, Bodmer WF. The E-cadherin gene (CDH1) variants T340A and L599V in gastric and colorectal cancer patients in Korea. Gut. 2000;47:262-267. [PubMed] [DOI] |
| 27. | Keller G, Vogelsang H, Becker I, Plaschke S, Ott K, Suriano G, Mateus AR, Seruca R, Biedermann K, Huntsman D. Germline mutations of the E-cadherin(CDH1) and TP53 genes, rather than of RUNX3 and HPP1, contribute to genetic predisposition in German gastric cancer patients. J Med Genet. 2004;41:e89. [PubMed] [DOI] |
| 28. | Richards FM, McKee SA, Rajpar MH, Cole TR, Evans DG, Jankowski JA, McKeown C, Sanders DS, Maher ER. Germline E-cadherin gene (CDH1) mutations predispose to familial gastric cancer and colorectal cancer. Hum Mol Genet. 1999;8:607-610. [PubMed] [DOI] |
| 29. | Yabuta T, Shinmura K, Tani M, Yamaguchi S, Yoshimura K, Katai H, Nakajima T, Mochiki E, Tsujinaka T, Takami M. E-cadherin gene variants in gastric cancer families whose probands are diagnosed with diffuse gastric cancer. Int J Cancer. 2002;101:434-441. [PubMed] [DOI] |
| 30. | Gayther SA, Gorringe KL, Ramus SJ, Huntsman D, Roviello F, Grehan N, Machado JC, Pinto E, Seruca R, Halling K. Identification of germ-line E-cadherin mutations in gastric cancer families of European origin. Cancer Res. 1998;58:4086-4089. [PubMed] |
| 31. | Pharoah PD, Guilford P, Caldas C. Incidence of gastric cancer and breast cancer in CDH1 (E-cadherin) mutation carriers from hereditary diffuse gastric cancer families. Gastroenterology. 2001;121:1348-1353. [PubMed] [DOI] |
| 32. | Yamada H, Shinmura K, Okudela K, Goto M, Suzuki M, Kuriki K, Tsuneyoshi T, Sugimura H. Identification and characterization of a novel germ line p53 mutation in familial gastric cancer in the Japanese population. Carcinogenesis. 2007;28:2013-2018. [PubMed] [DOI] |
| 33. | Kim IJ, Park JH, Kang HC, Shin Y, Lim SB, Ku JL, Yang HK, Lee KU, Park JG. A novel germline mutation in the MET extracellular domain in a Korean patient with the diffuse type of familial gastric cancer. J Med Genet. 2003;40:e97. [PubMed] [DOI] |
| 34. | Shinmura K, Tani M, Isogaki J, Wang Y, Sugimura H, Yokota J. RER phenotype and its associated mutations in familial gastric cancer. Carcinogenesis. 1998;19:247-251. [PubMed] [DOI] |
| 37. | Yanagisawa Y, Akiyama Y, Iida S, Ito E, Nomizu T, Sugihara K, Yuasa Y, Maruyama K. Methylation of the hMLH1 promoter in familial gastric cancer with microsatellite instability. Int J Cancer. 2000;85:50-53. [PubMed] [DOI] |
| 38. | Yuan Y, Ye J, Zheng S. Clinical and genetic features of International Collaborative Group-hereditary nonpolyposis colorectal cancer families and suspected hereditary nonpolyposis colorectal cancer families. Chin Med J (Engl). 2004;117:748-752. [PubMed] |
| 40. | Correa P, Houghton J. Carcinogenesis of Helicobacter pylori. Gastroenterology. 2007;133:659-672. [PubMed] [DOI] |
| 41. | 范 凯, 马 坚妹, 胡 少为, 王 朝晖, 刘 敏, 吕 申, 刘 丽娜, 许 国旺. 胃黏膜细胞hMSH2, hMLH1和p53基因表达与幽门螺杆菌感染的关系. 世界华人消化杂志. 2004;12:2904-2907. [DOI] |
| 42. | Fuchs CS, Giovannucci EL, Colditz GA, Hunter DJ, Speizer FE, Willett WC. A prospective study of family history and the risk of colorectal cancer. N Engl J Med. 1994;331:1669-1674. [PubMed] [DOI] |
| 43. | Slattery ML, Kerber RA. Family history of cancer and colon cancer risk: the Utah Population Database. J Natl Cancer Inst. 1994;86:1618-1626. [PubMed] [DOI] |
| 44. | Ahsan H, Neugut AI, Garbowski GC, Jacobson JS, Forde KA, Treat MR, Waye JD. Family history of colorectal adenomatous polyps and increased risk for colorectal cancer. Ann Intern Med. 1998;128:900-905. [PubMed] [DOI] |
| 45. | Nyström-Lahti M, Wu Y, Moisio AL, Hofstra RM, Osinga J, Mecklin JP, Järvinen HJ, Leisti J, Buys CH, de la Chapelle A. DNA mismatch repair gene mutations in 55 kindreds with verified or putative hereditary non-polyposis colorectal cancer. Hum Mol Genet. 1996;5:763-769. [PubMed] [DOI] |
| 46. | Baudi F, Fersini G, Lavecchia A, Terracciano R, Leone F, Quaresima B, Faniello MC, De Paola L, Doldo P, Cuda G. A novel missense germline mutation in exon 2 of the hMSH2 gene in a HNPCC family from Southern Italy. Cancer Lett. 2005;223:285-291. [PubMed] [DOI] |
| 47. | Barnetson RA, Cartwright N, van Vliet A, Haq N, Drew K, Farrington S, Williams N, Warner J, Campbell H, Porteous ME. Classification of ambiguous mutations in DNA mismatch repair genes identified in a population-based study of colorectal cancer. Hum Mutat. 2008;29:367-374. [PubMed] [DOI] |
| 48. | Auclair J, Busine MP, Navarro C, Ruano E, Montmain G, Desseigne F, Saurin JC, Lasset C, Bonadona V, Giraud S. Systematic mRNA analysis for the effect of MLH1 and MSH2 missense and silent mutations on aberrant splicing. Hum Mutat. 2006;27:145-154. [PubMed] [DOI] |