修回日期: 2010-03-10
接受日期: 2010-03-23
在线出版日期: 2010-03-18
目的: 分析中国北方地区胃癌家系人群及胃癌散发患者hMLH1和hMSH2基因的突变.
方法: 收集胃癌家族史的胃癌患者16例及5个家系健康人114例, 无胃癌家族史的胃癌患者56例, 正常人群对照100例. 采取外周血, 用小样本血液DNA提取试剂盒提取DNA. 分别扩增hMLH1的外显子3、8、12、13和外显子16以及hMSH2的外显子5和外显子7, 热变性后, 用毛细管电泳进行单链构象多态性的分析, 对可疑样本进行测序.
结果: 突变出现在hMLH1基因第8、第12和第16外显子, 而外显子3和外显子13没有检测到突变, hMSH2基因的外显子5和外显子7也没有检测到突变. 在有家族史的胃癌患者的16例外周血标本中, 有6例出现突变, 突变率37%; 无胃癌家族史的56例患者, 总计6例出现突变, 突变率11%; 胃癌家系健康人群的114例样本中, 31例出现突变, 突变率27%; 在100例对照样本中, 5例出现突变, 突变率5%. 第8外显子的突变点位于219位密码子的第一个碱基(ATC→GTC); 第12外显子的突变点位于第384位密码子的第二个碱基(GTT→GAT); 第16外显子的突变点位于第553位密码子的第二个碱基(AGT→AGG), 这三个突变都是碱基置换. 但有家族史的胃癌患者和胃癌家系健康成员的突变率明显高于对照组(P = 0.001, 0.000), 无家族史的胃癌患者突变率虽然略高于对照组, 但差异不显著(P = 0.204). 第16外显子的突变是目前尚未见报道的新的突变.
结论: 胃癌家系人群体细胞存在与遗传性非息肉性结肠癌(hereditary nonpolyposis colorectal cancer, HNPCC)相似的基因突变.
引文著录: 李建华, 吕申, 石先哲, 刘敏, 王彦. hMLH1和hMSH2基因在胃癌易感人群中的突变. 世界华人消化杂志 2010; 18(11): 1121-1126
Revised: March 10, 2010
Accepted: March 23, 2010
Published online: March 18, 2010
AIM: To analyze human mutL homolog 1 (hMLH1) and hMSH2 gene mutations in Chinese familial gastric cancer kindreds and sporadic gastric cancer patients.
METHODS: Blood samples from 16 gastric cancer patients and 114 healthy members with familial clustering of gastric cancer and 56 sporadic gastric cancer patients were collected. After DNA extraction, the fragments of exons 3, 8, 12, 13 and 16 of the hMLH1 gene and exons 5 and 7 of the hMSH2 gene were amplified by polymerase chain reaction (PCR), followed by single-stranded conformation polymorphism/capillary electrophoresis (SSCP-CE) genotyping assay. The fragments suspected to harbor mutations were sequenced.
RESULTS: Mutations were detected in exon 8, 12 and 16 of the hMLH1 gene, whereas no mutation was detected in exons 5 and 7 of the hMSH2 gene. In subjects with familial clustering of gastric cancer, the mutation frequency was 37% (6/16) in gastric cancer patients and 27% (31/114) in healthy members, both significantly higher than that in controls (5%; P = 0.001 and 0.000, respectively). The mutation frequency was 11% in gastric patients without familial clustering, significantly lower than that in subjects with familial clustering. The mutation at codon 219 in exon 8 of the hMLH1 gene (A→G) results in a conversion of Ile→Val (ATC→GTC), whereas the mutation at codon 384 in exon 12 of the hMLH1 gene (T→A) results in a conversion of Asp→Val (GTT→GAT). The mutation at codon 553 in exon 16 of the hMLH1 gene (T→G) results in a conversion of Ser→Arg (AGT→AGG), which represents a novel mutation of the hMLH1 gene that has not been reported before.
CONCLUSION: Familial gastric cancer kindreds may have similar hMLH1 gene mutations to those observed in hereditary nonpolyposis colorectal carcinoma.
- Citation: Li JH, Lv S, Shi XZ, Liu M, Wang Y. hMLH1 and hMSH2 gene mutations in a population susceptible to gastric cancer. Shijie Huaren Xiaohua Zazhi 2010; 18(11): 1121-1126
- URL: https://www.wjgnet.com/1009-3079/full/v18/i11/1121.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i11.1121
胃癌在我国是发病率较高的恶性肿瘤, 其发病与结肠癌相似, 具有一定的家族积聚现象, 这两种肿瘤的发生除了与环境因素有关, 遗传因素也相当重要. 已有大量的研究证实, 结肠癌的发生与错配修复基因的突变有关[1-3], 错配修复基因是DNA复制时保证细胞基因组稳定的卫士, 错配修复基因突变或功能丧失后, 其下游的癌基因和抑癌基因等多种基因的突变因得不到修复而逐渐积累, 使细胞发生恶变的危险性增高[4-6]. 除了遗传性非息肉性结肠癌(hereditary nonpolyposis colorectal cancer, HNPCC), 在散发性结肠癌也检测到了错配修复基因的突变, 尤其是作为错配修复起始因子的hMLH1和hMSH2基因, 突变更为常见[7,8], 然而在胃癌有关这两种基因突变的报道却很少. 虽然有学者曾经按照HNPCC的标准定义过遗传性胃癌, 但对胃癌基因背景的研究并不多. 至于胃癌的发生为什么有家族积聚性, 更很少有研究对这一现象进行解释. 通过临床及流行病学调查, 我们掌握了5个胃癌家系, 这些家系的特点是: 每个家系均有3例以上、连续2代发病, 其中至少2例为直系亲属, 这些特点与HNPCC相似, 并且近些年来, 这些家系的人群中结肠癌的发病率有增高的趋势. 另外, 在HNPCC也常有胃癌的发生[9,10]. 既然胃癌和结肠癌的组织结构和临床发病特点如此相似, 我们推测他们基因遗传背景可能有相同之处. 本研究中, 我们在胃癌易感人群体细胞检测HNPCC常出现的突变位点, 并与HNPCC相比较, 以获取我国胃癌高发人群的基因背景资料.
无胃癌家族史的胃癌患者的56份外周血采自我国东北地区的胃癌患者, 具有胃癌家族史的胃癌患者的16份外周血和胃癌家系健康成员的114份外周血样采自中国辽宁省南部地区, 这5个胃癌家系均满足以下条件: 每个家系有3例以上、连续2代发病, 其中2例为一级亲属. 男64例, 年龄8-75(中位年龄38)岁, 女50例, 年龄14-68(中位年龄40)岁. 正常对照的100份血样采自大连地区的志愿者, 其直系亲属无胃癌患者. 无家族史胃癌患者是指一、二级亲属中未发现胃癌或结肠癌, 有家族史胃癌患者是指一级亲属中有胃癌患者, 胃癌家系健康者是指在进行调查和采血时未发现患有肿瘤. Perkin-Elemer Model 2700 PCR扩增仪(美国ABI公司); P/ACE MDQ型毛细管电泳(美国Beckman公司); ABI Prism 310基因分析仪(美国ABI公司). 血液DNA提取纯化试剂盒(上海华舜生物工程有限公司)、丙烯酰胺、四甲基乙二胺(TEMED)、过硫酸铵(APS)、γ-甲基丙烯酰基-三(甲氧基)硅烷、TBE电泳缓冲液(Sigma公司, 美国), PCR反应试剂、引物(宝生物工程公司, 中国), 其他试剂均为国产分析纯.
1.2.1 血液DNA的提取: 用试剂盒提取血液有核细胞DNA.
1.2.2 聚合酶链反应: PCR引物设计见表1. 以提取的基因组DNA为模板, 扩增 hMLH1和hMSH2基因的外显子. PCR反应条件相同, PCR反应体积25 μL, 其中模板DNA 100 ng, 4×dNTPs终浓度各0.2 mmol/L, MgCl2浓度1.5 mmol/L, Taq酶1.5 U, 上下游引物各0.15 μmol/L. 循环条件: 94 ℃预变性5 min后, 94 ℃ 40 s, 55 ℃ 40 s, 72 ℃ 60 s, 35个循环周期, 最后72 ℃延长10 min.
| 基因 | 位点 | 引物序列 | 温度(℃) | 长度(bp) |
| hMSH2 | 外显子5 | 5'-FAM-ACTGTCTGCGGTAATCAAGT | 58 | 131 |
| 5'-FAM-AAAGGTTAAGGGCTCTGACT | ||||
| 外显子7 | 5'-FAM-CAAGCAGCAAAGTTAGAAGA | 56 | 176 | |
| 5'-FAM-ACCACCACCAACTTTATGAG | ||||
| hMLH1 | 外显子3 | 5'-FAM-AGAGATTTGGAAAATGAGTAAC | 50 | 208 |
| 5'-FAM-ACAATGTCATCACAGGAGG | ||||
| 外显子8 | 5'-FAM-CTCAGCCATGAGACAATAAATCC | 50 | 217 | |
| 5'-FAM-GGTTCCCAAATAATGTGATGG | ||||
| 外显子12 | 5'-FAM-ACAGACTTTGCTACCAGGACTTG | 55 | 216 | |
| 5'-FAM-TGTCTTATCCTCTGTGACAATGG | ||||
| 外显子13 | 5'-FAM-TGCAACCCACAAAATTTGGC | 55 | 292 | |
| 5'-FAM-CTTTCTCCATTTCCAAAACC | ||||
| 外显子16 | 5'-FAM-CATTTGGATGCTCCGTTAAAGC | 55 | 276 | |
| 5'-FAM-CACCCGGCTGGAAATTTTATTTG | ||||
| 5'-FAM-CTATTGTTGGATCATATTCGTCC |
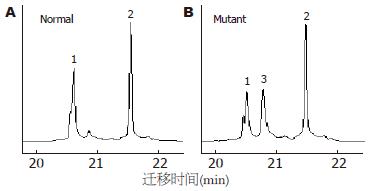
1.2.3 SSCP-CE分析: 取2 μL PCR产物用纯水稀释20倍, 95℃变性5 min后迅速冰浴冷却, 待测. 电泳时由毛细管电泳仪压力系统自动填充聚合物溶液到毛细管(总长40 cm, 有效长度30 cm, 内径75 μm)中, 采用电动进样和反极性恒压电泳模式分离. 激光诱导荧光检测器检测DNA片段, 激发波长488 nm, 发射波长520 nm. MDQ数据处理软件采集数据. 与正常对照相比, 出现异常峰为可疑突变.
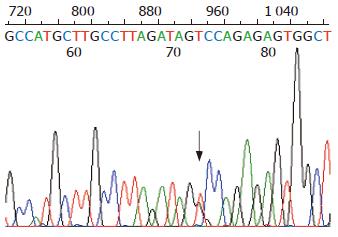
1.2.4 DNA序列分析: SSCP-CE分析出现异常的PCR产物, 按照ABI PRISM 310基因分析手册中的方法用乙酸钠和乙醇进行纯化. 取纯化的双链DNA2ng作为模板, 相应的非荧光标记的PCR引物为测序引物(3 pmol), 用ABI公司的Big Dye terminator cycle sequencing ready reaction kit, 在ABI PRISM 310型全自动DNA测序分析仪上进行PCR产物的直接测序.
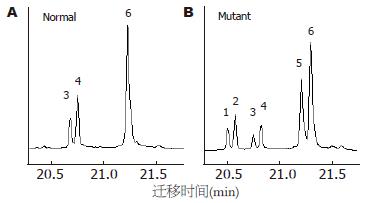
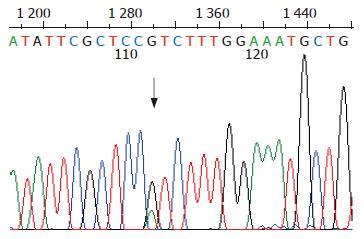
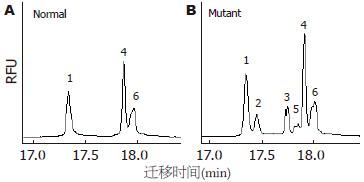
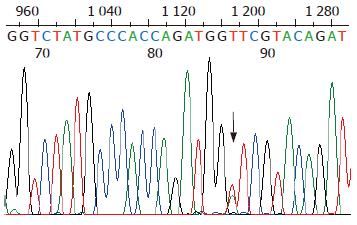
突变位点出现在hMLH1基因的第8、第12和第16外显子, 外显子3和外显子13没有检测到突变, hMSH2基因的外显子5和外显子7也没有检测到突变. 有胃癌家族史的16例患者, 总计6例出现突变, 突变率37%, hMLH1基因第8、第12和第16外显子突变分别有2例、2例和3例, 其中1例出现了第8和第12外显子的同时突变. 无胃癌家族史的56例患者, 总计6例出现突变, 突变率11%, hMLH1基因第8、第12和第16外显子突变分别有2例、2例和2例. 家系114例健康者样本中, 总计31例出现突变, 突变率27%, hMLH1基因第8、第12和第16外显子突变分别有12例、7例和13例, 其中1例出现第8和第16外显子的同时突变. 在100例对照样本中, 总计5例出现突变, 突变率5%, 第8、第12和第16外显子突变分别有2例、2例和1例. 由于每个家系单个外显子的突变例数较少, 因此, 合计各外显子进行统计见表2. 第8外显子的突变点位于219位密码子的第一个碱基(ATC→GTC, Ile→Val)(图1, 2); 第12外显子的突变点位于第384位密码子的第二个碱基(GTT→GAT, Val→Asp)(图3, 4); 第16外显子的突变点位于第553位密码子的第三个碱基(GTC→GGC, Ser→Arg)(图5, 6).
错配修复基因的主要功能是修复DNA复制时出现的错配, 维持基因组的稳定性[11,12]. 当错配修复基因本身发生了改变或错配修复功能降低时, DNA复制时出现的错配得不到及时纠正, 使基因组中的突变增多[13,14]. 癌基因和抑癌基因突变时, 细胞发生恶性转化的危险性随之增高[15,16]. 欧美诸国多见的HNPCC发病的主要原因之一就是错配修复基因的突变[17,18]. 到目前为止, 已经检测到400多种错配修复基因的突变, MLH1约占50%, MSH2约占40%[19,20]. 突变形式多为引起短缩蛋白的移码突变以及错义突变. 据统计, 有37.4%的HNPCC患者发病是由hMLH1基因突变引起的[21,22]. 错配修复基因突变是否也与中国北方发病率很高且有一定遗传倾向的胃癌发病相关, 迄今为止尚未见报道.
我们对胃癌患者和5个胃癌家系非肿瘤人群的体细胞hMLH1和hMSH2基因突变进行了检测, 结果在hMLH1的第8和第12外显子发现了与HNPCC患者相同的外显子突变位点: 第8外显子的突变点位于219位密码子的第1个碱基(ATC→GTC), 第12外显子的突变点位于第384位密码子的第2个碱基(GTT→GAT), 虽然这两种突变在散发性大肠癌和胃癌中也见有报道, 但却是偶发事件. 第12外显子的突变(GTT→GAT, Val384Asp)曾被认为是非功能性突变[23], 因为Val384Asp不处于hMLH1基因的高度保守区内, 其存在不会导致hMLH1基因功能的完全丧失. 然而第384位氨基酸残基是hMLH1基因相对可变区内的一个保守位点, 在电中性的缬氨酸被带负电荷的天冬氨酸替代时, 可能会使基因产物的结构发生某种变化. 这种杂合性突变可能会提高宿主罹患消化系肿瘤的危险性, 在环境因素给予二次打击时, 易导致肿瘤发生. 第16外显子的突变点位于第553位密码子的第3个碱基(AGT→AGG), 该突变到目前为止尚未见报道. 该突变是否能引起蛋白质功能性变化, 还有待于探讨. 胃癌患者和胃癌家系健康者的体细胞hMLH1基因出现的突变位点相同, 但有家族史的胃癌患者和胃癌家系健康成员的突变率明显高于对照组, 无家族史的胃癌患者突变率虽然略高于对照组, 但差异不显著. 胃癌家系非肿瘤人群体细胞基因组也出现了HNPCC常有的外显子的突变, 且每个家系的基因突变率均明显高于对照人群. 这一现象提示: 有胃癌家族史的人群可能有着与HNPCC相似的遗传背景, 即错配修复基因突变. 我国北方地区人们的饮食习惯与欧美国家有很大的差异, 比如常食用酸菜, 饮食中粗纤维含量高和高温、高盐饮食等. 在相同的基因遗传背景条件下, 肿瘤好发部位的不同是否是由于饮食习惯等流行病学原因所引起, 还有待于进一步的分子流行病学的研究.
无家族史的胃癌患者体内hMLH1基因的突变率与正常对照相似, 明显低于家系人群. 尽管该56例胃癌组织中, 我们曾经在14例(25%)检测到MSI-H, 但体细胞检测到MMR突变的只有2例(14%)[24], 基因突变可能不是这些胃癌错配修复功能的缺陷的主要原因, 家族性胃癌和散发性胃癌的发病机制和途径可能不同.
错配修复基因是DNA复制时保证细胞基因组稳定的卫士, 错配修复基因突变或功能丧失后, 其下游的癌基因和抑癌基因等多种基因的突变因得不到修复而逐渐积累, 使细胞发生恶变的危险性增高. 欧美诸国多见的HNPCC发病的主要原因之一就是错配修复基因的突变. 已经检测到的400多种错配修复基因的突变中, MLH1约占50%, MSH2约占40%.
倪润洲, 教授, 南通大学附属医院消化内科.
错配修复基因突变是否也与中国北方发病率很高且有一定遗传倾向的胃癌发病相关, 迄今为止尚未见报道.
本研究通过对胃癌家系人群、胃癌患者及正常对照人群错配修复基因MLH1和 MSH2的突变位点的分析, 在胃癌家系人群检测到较高频率的突变, 并发现了新的突变位点.
对于胃癌家系人群中错配修复基因的携带者进行预警、干预, 防治肿瘤的发生, 或者早发现, 早治疗, 提高治愈率.
hMSH2和hMLH1基因: 为保证DNA复制的真实性, 维持遗传的稳定, 无论原核生物还是真核生物, 都有一套完整的修复受损或突变DNA的修复系统, 其中修复碱基错配的系统称为错配修复系统, 其功能是修复复制过程中发生的碱基错配, 避免复制错误的积累. 参与组成这一系统的主要基因称为错配修复基因, hMSH2和hMLH1基因是错配修复系统中最为重要的两个.
本文通过对中国北方地区胃癌家系人群及胃癌散发患者hMLH1和hMSH2基因突变的分析, 来探索hMLH1和hMSH2基因突变与胃癌的关联, 是一篇质量较高的研究型论文.
编辑: 李军亮 电编:吴鹏朕
| 1. | Park JG, Vasen HF, Park KJ, Peltomaki P, Ponz de Leon M, Rodriguez-Bigas MA, Lubinski J, Beck NE, Bisgaard ML, Miyaki M. Suspected hereditary nonpolyposis colorectal cancer: International Collaborative Group on Hereditary Non-Polyposis Colorectal Cancer (ICG-HNPCC) criteria and results of genetic diagnosis. Dis Colon Rectum. 1999;42:710-715; discussion 715-716. [PubMed] |
| 2. | Lynch HT, Watson P, Shaw TG, Lynch JF, Harty AE, Franklin BA, Kapler CR, Tinley ST, Liu B, Lerman C. Clinical impact of molecular genetic diagnosis, genetic counseling, and management of hereditary cancer. Part II: Hereditary nonpolyposis colorectal carcinoma as a model. Cancer. 1999;86:2457-2463. [PubMed] |
| 3. | Dieumegard B, Grandjouan S, Sabourin JC, Le Bihan ML, Lefrère I, Bellefqih , Pignon JP, Rougier P, Lasser P, Bénard J. Extensive molecular screening for hereditary non-polyposis colorectal cancer. Br J Cancer. 2000;82:871-880. [PubMed] |
| 4. | Wang Q, Lasset C, Desseigne F, Saurin JC, Maugard C, Navarro C, Ruano E, Descos L, Trillet-Lenoir V, Bosset JF. Prevalence of germline mutations of hMLH1, hMSH2, hPMS1, hPMS2, and hMSH6 genes in 75 French kindreds with nonpolyposis colorectal cancer. Hum Genet. 1999;105:79-85. [PubMed] |
| 5. | Peltomäki P, Vasen HF. Mutations predisposing to hereditary nonpolyposis colorectal cancer: database and results of a collaborative study. The International Collaborative Group on Hereditary Nonpolyposis Colorectal Cancer. Gastroenterology. 1997;113:1146-1158. [PubMed] |
| 6. | Peltomäki P. Deficient DNA mismatch repair: a common etiologic factor for colon cancer. Hum Mol Genet. 2001;10:735-740. [PubMed] |
| 7. | Koopman M, Kortman GA, Mekenkamp L, Ligtenberg MJ, Hoogerbrugge N, Antonini NF, Punt CJ, van Krieken JH. Deficient mismatch repair system in patients with sporadic advanced colorectal cancer. Br J Cancer. 2009;100:266-273. [PubMed] |
| 8. | Poulogiannis G, Frayling IM, Arends MJ. DNA mismatch repair deficiency in sporadic colorectal cancer and Lynch syndrome. Histopathology. 2010;56:167-179. [PubMed] |
| 9. | Gylling A, Abdel-Rahman WM, Juhola M, Nuorva K, Hautala E, Järvinen HJ, Mecklin JP, Aarnio M, Peltomäki P. Is gastric cancer part of the tumour spectrum of hereditary non-polyposis colorectal cancer? A molecular genetic study. Gut. 2007;56:926-933. [PubMed] |
| 10. | La Vecchia C, Negri E, Franceschi S, Gentile A. Family history and the risk of stomach and colorectal cancer. Cancer. 1992;70:50-55. [PubMed] |
| 11. | Duval A, Hamelin R. Genetic instability in human mismatch repair deficient cancers. Ann Genet. 2002;45:71-75. [PubMed] |
| 12. | Jacob S, Praz F. DNA mismatch repair defects: role in colorectal carcinogenesis. Biochimie. 2002;84:27-47. [PubMed] |
| 13. | Ling XL, Fang DC, Wang RQ, Yang SM, Fang L. Mitochondrial microsatellite instability in gastric cancer and its precancerous lesions. World J Gastroenterol. 2004;10:800-803. [PubMed] |
| 14. | Kobayashi K, Matsushima M, Koi S, Saito H, Sagae S, Kudo R, Nakamura Y. Mutational analysis of mismatch repair genes, hMLH1 and hMSH2, in sporadic endometrial carcinomas with microsatellite instability. Jpn J Cancer Res. 1996;87:141-145. [PubMed] |
| 15. | Stoffel E, Mukherjee B, Raymond VM, Tayob N, Kastrinos F, Sparr J, Wang F, Bandipalliam P, Syngal S, Gruber SB. Calculation of risk of colorectal and endometrial cancer among patients with Lynch syndrome. Gastroenterology. 2009;137:1621-1627. [PubMed] |
| 16. | Oliveira C, Velho S, Domingo E, Preto A, Hofstra RM, Hamelin R, Yamamoto H, Seruca R, Schwartz S Jr. Concomitant RASSF1A hypermethylation and KRAS/BRAF mutations occur preferentially in MSI sporadic colorectal cancer. Oncogene. 2005;24:7630-7634. [PubMed] |
| 17. | Han HJ, Maruyama M, Baba S, Park JG, Nakamura Y. Genomic structure of human mismatch repair gene, hMLH1, and its mutation analysis in patients with hereditary non-polyposis colorectal cancer (HNPCC). Hum Mol Genet. 1995;4:237-242. [PubMed] |
| 18. | Mueller J, Gazzoli I, Bandipalliam P, Garber JE, Syngal S, Kolodner RD. Comprehensive molecular analysis of mismatch repair gene defects in suspected Lynch syndrome (hereditary nonpolyposis colorectal cancer) cases. Cancer Res. 2009;69:7053-7061. [PubMed] |
| 19. | Kim JC, Kim HC, Roh SA, Koo KH, Lee DH, Yu CS, Lee JH, Kim TW, Lee HL, Beck NE. hMLH1 and hMSH2 mutations in families with familial clustering of gastric cancer and hereditary non-polyposis colorectal cancer. Cancer Detect Prev. 2001;25:503-510. [PubMed] |
| 20. | Bellizzi AM, Frankel WL. Colorectal cancer due to deficiency in DNA mismatch repair function: a review. Adv Anat Pathol. 2009;16:405-417. [PubMed] |
| 21. | Bonis PA, Trikalinos TA, Chung M, Chew P, Ip S, DeVine DA, Lau J. Hereditary nonpolyposis colorectal cancer: diagnostic strategies and their implications. Evid Rep Technol Assess (Full Rep). 2007;1-180. [PubMed] |
| 22. | Lagerstedt Robinson K, Liu T, Vandrovcova J, Halvarsson B, Clendenning M, Frebourg T, Papadopoulos N, Kinzler KW, Vogelstein B, Peltomäki P. Lynch syndrome (hereditary nonpolyposis colorectal cancer) diagnostics. J Natl Cancer Inst. 2007;99:291-299. [PubMed] |