修回日期: 2009-01-30
接受日期: 2009-02-09
在线出版日期: 2009-03-08
目的: 设计并构建ASGPR1靶向的microRNA表达载体, 观察其对ASGPR1基因的抑制作用及其在HBV感染基因治疗中的应用价值.
方法: 以ASGPR1为靶基因, 设计并构建3个针对ASGPR1不同位点的microRNA表达载体, 通过脂质体方法转染HepG2.2.15细胞, RT-PCR和Western blot检测其对ASGPR1 mRNA和蛋白的抑制作用, 乙肝五项定量和HBV DNA检测其对HBV的抑制作用.
结果: ASGPR1 mRNA和蛋白的平均水平分别下降了57.3%和49.8%(P<0.01); 在病毒水平3种amiRNA均能明显抑制相应细胞株中HBsAg和HBeAg的分泌, 其中以amiRNA-ASGPR1-610抑制作用最强, 对培养72 h的细胞上清中的HBsAg和HBeAg抑制率分别为31.3%和33.6%(P<0.01), HBV DNA的抑制率为29.7%(P<0.01).
结论: 靶向ASGPR1的外源性microRNA能显著抑制靶基因的表达, 进而抑制HBV的复制和表达. ASGPR1可以作为慢性HBV感染基因治疗的候选靶点.
引文著录: 郜玉峰, 余莉, 李家斌, 魏少峰, 李旭, 沈继龙. 靶向ASGPR1的外源性microRNA对HBV表达和复制的抑制作用. 世界华人消化杂志 2009; 17(7): 699-704
Revised: January 30, 2009
Accepted: February 9, 2009
Published online: March 8, 2009
AIM: To investigate the inhibitory effects on hepatitis B virus (HBV) replication and expression by transfecting artificial microRNA targeted ASGPR1 into HepG2.2.15 cells.
METHODS: Three amiRNA-HBV plasmids were constructed and transfected into HepG2.2.15 cells via LipofectamineTM 2000 reagent. The level of ASGPR1 mRNA was measured by semi-quantitative RT-PCR. The level of ASGPR1 protein was measured by western blot. HBV antigen secretion was detected in the cells with transient and stable transfection by time-resolved fluoroimmunoassays (TRFIA). HBV DNA replication was examined by fluorescence quantitative PCR.
RESULTS: Three amiRNA significantly reduced ASGPR1 mRNA and protein expression, and the greatest reduction was seen in amiRNA-ASGPR1-610 transfected group. Expressions of ASGPR1 mRNA and protein were down-regulated by 57.3% and 49.8% at 72 h(P < 0.01). At the virus level, three amiRNA-ASGPR1 plasmids obviously inhibited the secretion of HBsAg and HBeAg with the greatest reduction seen in amiRNA-ASGPR1-610 transfected group. Expression levels of HBsAg and HBeAg were down-regulated by 31.3% and 33.6% after 72 h (P < 0.01) and HBV DNA level was down-regulated by 29.7% at 72 h (P < 0.01).
CONCLUSION: In HepG2.2.15 cells, HBV replication and expression could be inhibited by artificial microRNA targeted ASGPR1. Artificial microRNA targeted ASGPR1 could be a promising therapeutic approach for chronic HBV infection.
- Citation: Gao YF, Yu L, Li JB, Wei SF, Li X, Shen JL. Inhibition of hepatitis B virus gene expression and replication by artificial microRNA targeted ASGPR1. Shijie Huaren Xiaohua Zazhi 2009; 17(7): 699-704
- URL: https://www.wjgnet.com/1009-3079/full/v17/i7/699.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i7.699
去唾液酸糖蛋白1(asialoglycoprotein receptor 1, ASGPR1)是在肝细胞膜表面特异性表达的转膜分子, 可介导去唾液酸糖蛋白黏附并进入肝细胞[1]. 研究表明, ASGPR1可能是介导HBV进入肝细胞的主要受体之一, 应用ASGPR1的配基、抗体等抑制ASGPR1的水平可阻断HBV进入肝细胞, 进而抑制HBV的复制[2-3]. 进一步研究发现, 在已感染HBV的肝细胞中, 阻断ASGPR1表达后, HBV的表达和复制水平下降[4]. 因此, 推测阻断ASGPR1的表达可抑制HBV的复制, ASGPR1可成为抗HBV的潜在作用靶点.
MicroRNA(miRNA)是一种大小约22 nt的非编码小分子单链RNA, 可以通过对靶基因mRNA的剪切或抑制靶基因mRNA的翻译调控靶基因的表达[5]. miRNA作用方式很大程度上和siRNA介导的RNAi途径重叠[6]. 与siRNA相比, miRNA仅仅需要和靶基因部分的结合即可以发挥作用, 因而可以在很大程度上避免因为病毒变异导致的RNAi治疗失败, 这将对容易发生变异的病毒感染(如HCV, HBV, HIV)的治疗更有优势[7-8]. miRNA功能的研究可能有助于提出治疗病毒感染性疾病的新方法, 但尚未见microRNA介导的RNAi用于HBV基因治疗研究的报道. 为探讨ASGPR1在HBV RNA干扰基因治疗中的应用价值, 本研究设计并合成靶向ASGPR1的microRNA序列, 构建外源性microRNA的表达载体, 转染HepG2.2.15细胞, 观察其对ASGPR1的抑制作用, 并进一步观察ASGPR1抑制后对HBV表达和复制的影响.
pcDNA6.2-G/W-EmGFP-miRNA表达质粒购自美国Invitrogen公司, 稳定表达HBV的HepG2.2.15细胞系由本室保存, DMEM为Gibco公司产品, T4 DNA连接酶为TaKaRa公司产品, TRIzol、脂质体Lipofectamine 2000为invitrogen公司产品, A3500逆转录试剂盒、质粒纯化试剂盒和G418为Promega公司产品, 山羊抗人ASGPR1多克隆抗体和鼠抗人actin抗体购自Santa Crusz公司. 乙肝五项定量检测试剂盒为苏州宏波生物有限公司产品, 其他试剂均为国产或进口分析纯.
1.2.1 microRNA表达载体的构建、质粒纯化: 根据ASGPR1基因序列(NM_001671), 应用Invitrogen公司的在线RNAi设计软件设计针对ASGPR1基因的microRNA, 利用BLAST对选择的靶序列进行同源分析, 排除非特异性microRNA. miRNA相应的单链DNA序列由上海英俊公司合成. 退火合成双链, 利用T4连接酶与载体链接, 将连接后的载体转化入TOP10细菌, 质粒抗性筛选, 提取质粒测序证实插入序列的正确性.
1.2.2 HepG2.2.15细胞的转染: 将HepG2.2.15细胞按每孔1×105的数量接种于6孔培养板, DMEM培养液(含100 g/L小牛血清, 100 kU/L青霉素, 100 kU/L链霉素)37℃、50 mL/L CO2培养, 待细胞生长融合至80%左右, 更换无抗生素、无血清的DMEM培养液继续培养4 h后, 用脂质体2000进行转染. 按其说明书方法进行, 每孔细胞分别加入2 μg质粒和5 μL脂质体, 转染6 h更换含100 g/L小牛血清的无抗生素的DMEM培养液. 转染后24、48、72 h分别收集细胞和培养上清液待检.
1.2.3 RT-PCR检测转染后HepG2.2.15细胞ASGPR1 mRNA的表达: 细胞转染后72 h, 在每孔细胞中加入0.5 mL TRIzol, 按照说明书方法提取总RNA, 各取1 μg总RNA, 按照逆转录试剂盒说明进行cDNA第一链的合成. 用于扩增ASGPR1基因上游引物为P1: 5'-TGCTGCTTGTGGTTGTCTGTG-3'; 下游引物为P2: 5'-AGCCCGTCTCGTAGTCCGTCCC-3', 预期扩增片段长535 bp. 用于扩增内参照GAPDH基因的上游引物为P3: 5'-CCACTCCTCCACCTTTGACGC-3'; 下游引物为P4: 5'-AGTTATTTCATGGGACACGAGTT-3'; 预期扩增片段长337 bp. PCR 反应条件为: 94℃预变性5 min; 94℃ 30 s, 58℃ 30 s, 72℃ 30 s, 循环30次; 最后72℃延伸10 min. 扩增结束后取PCR产物5 μL琼脂糖凝胶电泳, 观察并记录结果.
1.2.4 Western blot检测HepG2.2.15细胞转染后ASGPR1蛋白的表达: 细胞转染72 h后, 在每孔细胞中加入RIPA裂解液裂解细胞, 获得蛋白, 裂解上清用BCA试剂盒定量蛋白浓度. 各组分别取蛋白30 μg, 100 g/L SDS-PAGE后200mA转膜2 h, 用含50 g/L牛奶的PBST室温封闭1 h, 洗膜3次, 加入稀释的山羊抗人ASGPR1多克隆抗体(1∶4000)或鼠抗人actin(1∶5000), 室温轻摇1 h. PBST洗膜3次, 每次10 min. 分别加入稀释的辣根过氧化物酶标记的羊抗鼠IgG、兔抗羊IgG酶标二抗, 稀释比例为1∶5000-10000, 室温轻摇1 h. 用PBST洗膜3次, 每次10 min, 暗室下ECL发光显色, 曝光, 观察结果.
1.2.5 乙肝五项定量和HBV DNA荧光定量PCR检测: 乙肝五项定量采用双抗体夹心时间分辨免疫荧光分析法进行检测, HBV DNA应用ABI GeneAmp PE7500荧光定量PCR仪检测, 正向引物: 5'-GGAGTATGG ATTCGCACTCCTC-3'; 反向引物: 5'-TTGTTGTTGTAGGGGACCTGCCT-3', 荧光探针: 5'-ACTTCCGGAAACTACTGTTAGACGA-3'.
统计学处理 数据以mean±SD表示, 由SPSS13.0统计软件处理, 采用t检验和单因素方差分析得出P值, P<0.05为差异有统计学意义.
将3对合成的64 nt的寡核苷酸序列进行合成双链, 合成双链后经3%琼脂糖凝胶电泳进行确认. 然后进行载体的连接, 经测序引物进行PCR扩增插入片段后送Invitrogen公司测序证实插入序列的正确性, 均排除碱基的突变, 分别命名为amiRNA-ASGPR1-188, 610, 772.
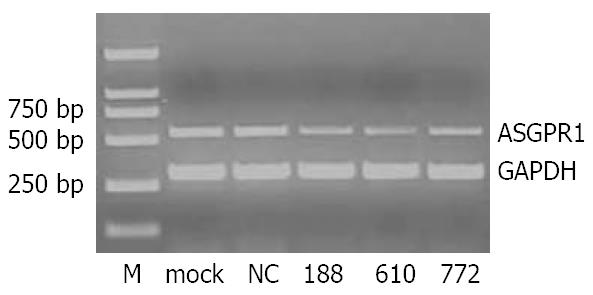
靶向ASGPR1 mRNA的3个质粒转染HepG2.2.15细胞72 h后, 提取细胞总RNA并逆转录, 以看家基因GAPDH为内参照, 进行PCR反应, 扩增产物经10 g/L琼脂糖凝胶电泳分析, 然后对电泳条带应用半定量分析, 以对照组细胞ASGPR1 mRNA与GAPDH mRNA的比值为100%, 计算治疗组的相对百分比, 结果显示: 转染72 h后, amiRNA-ASGPR1-188、amiRNA-ASGPR1-610和amiRNA-ASGPR1-772组ASGPR1 mRNA水平较空白转染组和阴性质粒转染组均显著降低(P<0.05), 而空白组和阴性质粒转染组相比无显著性差异(P>0.05, 图1). 在这三组质粒中, amiRNA-ASGPR1-610组的ASGPR1 mRNA水平下降最为显著, 抑制率为57.3%, amiRNA- ASGPR1-188和amiRNA-ASGPR1-772组的抑制率分别为44.5%和35.6%.
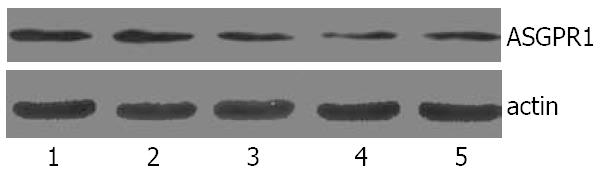
Western blot印迹结果显示转染后72 h ASGPR1蛋白的表达量与空白转染组和阴性质粒转染组相比均有显著降低(P<0.05), amiRNA-La-610组的ASGPR1蛋白表达减少最明显, 为49.8%, amiRNA-ASGPR1-188组为46.3%, amiRNA-ASGPR1-772组为39.3%, 而阴性质粒组组表达量无明显改变(图2), P>0.05, 与mRNA检测结果基本一致.
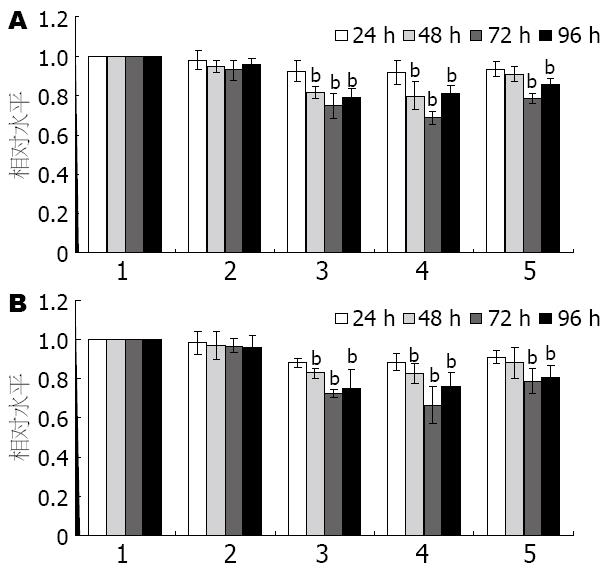
转染后的第2天细胞上清中的HBsAg和HBeAg即开始受到抑制, 但无明显显著性差别. 在转染后48 h抑制效果较明显, 72 h病毒抑制达到高峰, 96 h后抑制作用开始渐减弱. 3个质粒对HBV的表达均有抑制作用, 其中, 以amiRNA-ASGPR1-610作用最为明显, 而阴性对照组对蛋白表达的影响不明显. amiRNA-ASGPR1-610在转染后的48、72和96 h三个时间点对HBsAg的抑制率分别为20.2%, 31.3%和19.1%(图3A), 对HBeAg的抑制率分别为17.6%, 33.6%和24.3%(图3B), 其他2个质粒的抑制率相对较低. 考虑到瞬时转染质粒的转染效率仅能达到50%-60%左右, 所以我们判断amiRNA-ASGPR1-610的实际抗原分泌抑制率最高能达到50-60%以上, 说明在提高转染效率的前提下, amiRNA-ASGPR1介导的RNA干扰能达到较好的治疗效果, 可以作为抗HBV的基因治疗靶位.
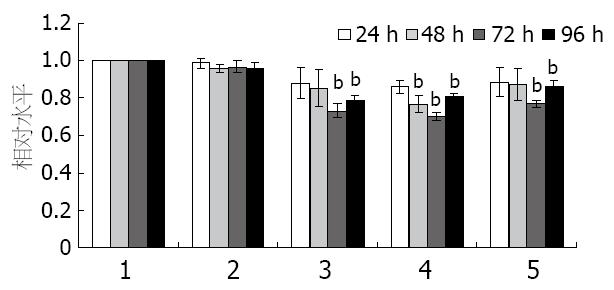
为明确amiRNA-ASGPR1转染后对HBV复制的影响, 对瞬时转染各个时间点培养上清中的HBV DNA水平采用实时荧光定量PCR进行了检测, 结果表明, 特异性靶向ASGPR1的amiRNA能降低细胞培养上清中的拷贝数. 与HBsAg和HBeAg抑制的作用相似, 3个质粒对培养上清中的HBV DNA也均有不同程度的抑制作用, 而阴性对照组载体对HBV DNA几乎没有作用(图4). 其中amiRNA-ASGPR1-610的抑制效果最为明显. amiRNA-ASGPR1-610在转染后48、72和96 h对HBV DNA的抑制率为23.2%, 29.7%和19.0%. 因为载体的转染效率较低只有50%-60%, 同样可以认为, amiRNA-HBV实际的病毒复制抑制率可以达到50%以上.
质粒转染后, 每日在倒置显微镜下观察细胞形态, 治疗质粒转染组和对照组形态无明显变化, 在开始的24 h细胞生长较空白细胞生长的速度稍慢, 其后基本和空白对照生长速度一致. 在转染细胞后72 h, 加入MTT, 继续孵育4 h后, 490 nm波长检测各细胞培养孔中的吸光度, 以细胞对照组的A值为1, 计算各个质粒转染组的相对A值, 结果显示: 各质粒转染组的A值与空白细胞相比无显著性差异, P>0.05.
RNA干扰(RNA interference, RNAi)是一项新兴的基因沉默技术, 主要通过siRNA和microRNA(miRNA)两种小RNA分子来发挥作用[9]. 大量体内外实验研究已经证明, 使用siRNA控制HBV表达和复制具有高效性和可行性[10-13]. 但越来越多的研究发现, 许多病毒在治疗选择压力下可以通过靶基因序列的突变来逃避siRNA介导的RNAi, 针对病毒的RNAi治疗效果可能会因为病毒的变异而降低疗效, 甚至失去治疗作用[14-17]. microRNA通过两种作用方式发挥其生物功能:当他们与靶mRNA完全或几乎完全配对时, 可以引起靶mRNA的剪切反应即RNA干扰作用; 在不完全配对的时候, 可以引起靶mRNA的翻译抑制[7-8], 这是其较之siRNA优越之处.
外源性人工合成的microRNA(artificial microRNA, amiRNA)技术是指利用内源性miRNA前体分子产生小RNA去介导动物或植物中的靶基因沉默[18-20]. 最近, 几个基于miRNAs表达框架的pol Ⅱ启动子驱动的RNAi载体被构建, 这种RNAi载体能表达外源性miRNA通过RNA干扰途径指导RNA的清除或转录抑制[8,21-22]. 利用microRNA表达框架产生外源性miRNA特异性降解靶基因的体内外实验已经展示了极好的应用前景. 我们采用的以microRNA介导的RNAi研究显示: 靶向ASGPR1的外源性miRNA表达质粒能特异性的显著降低ASGPR1 mRNA和蛋白水平的表达.
另一种有效的病毒RNAi治疗途径将是针对病毒感染和复制所需要的宿主细胞因素进行RNA干扰研究. 以宿主基因为靶位RNAi治疗的优点是他不依赖于病毒的基因组, 因而能避免病毒通过突变逃避RNAi的缺点. Xue et al针对HCV复制所需的几个宿主基因进行了RNA干扰治疗研究. 结果显示针对宿主基因hVAP-A、La和PTB的siRNA能显著降低宿主基因的表达, 同时发现HCV的复制水平受到了显著的抑制[23-24]. 另外, 这些治疗对细胞的活性并没有明显的影响. 提示与病毒复制相关的宿主基因可能作为抑制病毒复制的一个靶点. ASGPR1不仅是HBV感染肝细胞的候选受体, 而且可以促进HBV在体内的复制. Yang et al[4]通过反义寡核苷酸技术研究显示: 靶向ASGPR1的反义寡核苷酸可以特异性和剂量依赖性地抑制HepG2.2.15细胞中的ASGPR1表达, ASGPR1表达下调后, HepG2.2.15细胞培养液中的HBV DNA, HBsAg和HBeAg的水平也显著降低, 证实阻断ASGPR1的表达可阻断HBV的复制. 我们采用的以microRNA介导的RNAi研究显示: 靶向ASGPR1的外源性amiRNA表达质粒能特异性的阻断ASGPR1 mRNA和蛋白水平的表达. 随后的病毒抑制水平检测显示: 阻断ASGPR1表达后, 细胞培养上清中的HBsAg、HBeAg和HBV DNA水平有明显的下降, 其中amiRNA-ASGPR1-610治疗组对HBV DNA的抑制率可以达到32.4%, 如果考虑到转染效率的影响, 我们判断实际的抑制率可以达到50%-60%左右, 与Yang et al的研究结果一致, 也和我们以前的稳转实验研究抑制效率相符[25]. 说明ASGPR1的确在HBV的感染和复制中起到重要作用, ASGPR1可以作为HBV抗病毒治疗的一个候选靶点. 目前尚无HBV易感的肝细胞株, 如果利用HBV易感肝细胞株进行可能会取得更理想的疗效. 另外, 为了证实靶向ASGPR1的外源性amiRNA对HBV的复制不是由质粒和脂质体的细胞毒性引起, 我们检测了治疗组转染细胞的细胞增殖情况. 实验结果显示, 在转染后72 h, 转染组细胞的形态无明显变化, 转染组细胞的A值与空白细胞对照组相比没有显著的差异, 因此, 可以判断对HBV复制的抑制并不是由细胞毒性引起的.
外源性amiRNA介导的RNA干扰可以高效阻断ASGPR1宿主靶基因的表达. ASGPR1的表达水平与HBV的复制和表达有密切关系, 进一步证实其在HBV的感染和复制中起着关键作用. 与HBV感染和复制相关的宿主基因可以作为HBV基因治疗的一个候选靶位. 在提高转染效率的情况下, 外源性amiRNA能够作为一种新的抗HBV的基因治疗手段. microRNA介导的RNAi作为一种新型的HBV基因治疗技术, 展示了良好的应用前景, 但RNAi要应用于临床, 其组织靶向性和表达效率、药代动力学及如何选择最有效的靶序列等问题仍有待解决, 比如本研究中所采用的质粒载体, 若具有肝器官靶向性, 则可能提高其效果.
大量研究发现, 许多病毒在治疗选择压力下可以通过靶基因序列的突变来逃避RNAi, 针对病毒的RNAi治疗可能会因为病毒变异而降低疗效. 针对病毒复制所需要宿主基因进行RNAi可能是避免病毒抗性突变株出现的一种解决办法.
范学工, 教授, 中南大学湘雅医院感染病科
RNAi抗HBV基因治疗有着广泛的应用前景, 已有对HCV、HIV病毒感染宿主基因治疗的报道, 均取得了较好的疗效. 包括宿主基因在内的多靶位的RNAi已成为抗病毒基因治疗的热点.
Yang et al通过反义寡核苷酸技术研究显示, 靶向ASGP R1的反义寡核苷酸可以特异性和剂量依赖性地阻断ASGPR1的表达, 并进一步抑制HBV的复制.
siRNA通过序列特异性的方式发挥作用, 而microRNA可以通过与靶mRNA完全或部分配对两种作用方式发挥其生物功能. 本研究首次采用microRNA介导的RNAi技术干扰宿主基因用于HBV基因治疗研究.
本文为HBV基因治疗的研究提供了新的思路, 并为慢性HBV感染临床基因治疗提供新的基因靶点.
MicroRNA (miRNA): 一种大小约22 nt的非编码小分子单链RNA, 他可以通过对靶基因mRNA的剪切或抑制靶基因mRNA的翻译调控靶基因的表达.
本文对靶向ASGPR1的外源性microRNA抑制HBV表达和复制进行了初步观察, 其结果有较好的科学意义和潜在的应用价值.
编辑: 李军亮 电编:吴鹏朕
| 1. | Meier M, Bider MD, Malashkevich VN, Spiess M, Burkhard P. Crystal structure of the carbohydrate recognition domain of the H1 subunit of the asialoglycoprotein receptor. J Mol Biol. 2000;300:857-865. [PubMed] [DOI] |
| 2. | Treichel U, Meyer zum Buschenfelde KH, Dienes HP, Gerken G. Receptor-mediated entry of hepatitis B virus particles into liver cells. Arch Virol. 1997;142:493-498. [PubMed] [DOI] |
| 3. | Treichel U, Meyer zum Buschenfelde KH, Stockert RJ, Poralla T, Gerken G. The asialoglycoprotein receptor mediates hepatic binding and uptake of natural hepatitis B virus particles derived from viraemic carriers. J Gen Virol. 1994;75:3021-3029. [PubMed] [DOI] |
| 4. | Yang J, Bo XC, Ding XR, Dai JM, Zhang ML, Wang XH, Wang SQ. Antisense oligonucleotides targeted against asialoglycoprotein receptor 1 block human hepatitis B virus replication. J Viral Hepat. 2006;13:158-165. [PubMed] [DOI] |
| 5. | Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 2004;116:281-297. [PubMed] [DOI] |
| 6. | Cullen BR. Derivation and function of small interfering RNAs and microRNAs. Virus Res. 2004;102:3-9. [PubMed] [DOI] |
| 7. | Ossowski S, Schwab R, Weigel D. Gene silencing in plants using artificial microRNAs and other small RNAs. Plant J. 2008;53:674-690. [PubMed] [DOI] |
| 8. | Stegmeier F, Hu G, Rickles RJ, Hannon GJ, Elledge SJ. A lentiviral microRNA-based system for single-copy polymerase II-regulated RNA interference in mammalian cells. Proc Natl Acad Sci U S A. 2005;102:13212-13217. [PubMed] [DOI] |
| 9. | Agrawal N, Dasaradhi PV, Mohmmed A, Malhotra P, Bhatnagar RK, Mukherjee SK. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 2003;67:657-685. [PubMed] [DOI] |
| 10. | Arbuthnot P, Longshaw V, Naidoo T, Weinberg MS. Opportunities for treating chronic hepatitis B and C virus infection using RNA interference. J Viral Hepat. 2007;14:447-459. [PubMed] [DOI] |
| 11. | Ying RS, Zhu C, Fan XG, Li N, Tian XF, Liu HB, Zhang BX. Hepatitis B virus is inhibited by RNA interference in cell culture and in mice. Antiviral Res. 2007;73:24-30. [PubMed] [DOI] |
| 12. | Li GQ, Gu HX, Li D, Xu WZ. Inhibition of Hepatitis B virus cccDNA replication by siRNA. Biochem Biophys Res Commun. 2007;355:404-408. [PubMed] [DOI] |
| 13. | Xuan B, Qian Z, Tan C, Min T, Shen S, Huang W. esiRNAs purified with chromatography suppress homologous gene expression with high efficiency and specificity. Mol Biotechnol. 2005;31:203-209. [PubMed] [DOI] |
| 14. | Zheng ZM, Tang S, Tao M. Development of resistance to RNAi in mammalian cells. Ann N Y Acad Sci. 2005;1058:105-118. [PubMed] [DOI] |
| 15. | Das AT, Brummelkamp TR, Westerhout EM, Vink M, Madiredjo M, Bernards R, Berkhout B. Human immunodeficiency virus type 1 escapes from RNA interference-mediated inhibition. J Virol. 2004;78:2601-2605. [PubMed] [DOI] |
| 16. | Konishi M, Wu CH, Kaito M, Hayashi K, Watanabe S, Adachi Y, Wu GY. siRNA-resistance in treated HCV replicon cells is correlated with the development of specific HCV mutations. J Viral Hepat. 2006;13:756-761. [PubMed] [DOI] |
| 17. | Wu HL, Huang LR, Huang CC, Lai HL, Liu CJ, Huang YT, Hsu YW, Lu CY, Chen DS, Chen PJ. RNA interference-mediated control of hepatitis B virus and emergence of resistant mutant. Gastroenterology. 2005;128:708-716. [PubMed] [DOI] |
| 18. | Niu QW, Lin SS, Reyes JL, Chen KC, Wu HW, Yeh SD, Chua NH. Expression of artificial microRNAs in transgenic Arabidopsis thaliana confers virus resistance. Nat Biotechnol. 2006;24:1420-1428. [PubMed] [DOI] |
| 19. | Zeng Y, Wagner EJ, Cullen BR. Both natural and designed micro RNAs can inhibit the expression of cognate mRNAs when expressed in human cells. Mol Cell. 2002;9:1327-1333. [PubMed] [DOI] |
| 20. | Schwab R, Ossowski S, Riester M, Warthmann N, Weigel D. Highly specific gene silencing by artificial microRNAs in Arabidopsis. Plant Cell. 2006;18:1121-1133. [PubMed] [DOI] |
| 21. | Chung KH, Hart CC, Al-Bassam S, Avery A, Taylor J, Patel PD, Vojtek AB, Turner DL. Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155. Nucleic Acids Res. 2006;34:e53. [PubMed] [DOI] |
| 22. | Du G, Yonekubo J, Zeng Y, Osisami M, Frohman MA. Design of expression vectors for RNA interference based on miRNAs and RNA splicing. FEBS J. 2006;273:5421-5427. [PubMed] [DOI] |
| 23. | Xue Q, Ding H, Liu M, Zhao P, Gao J, Ren H, Liu Y, Qi ZT. Inhibition of hepatitis C virus replication and expression by small interfering RNA targeting host cellular genes. Arch Virol. 2007;152:955-962. [PubMed] [DOI] |
| 24. | Zhang J, Yamada O, Sakamoto T, Yoshida H, Iwai T, Matsushita Y, Shimamura H, Araki H, Shimotohno K. Down-regulation of viral replication by adenoviral-mediated expression of siRNA against cellular cofactors for hepatitis C virus. Virology. 2004;320:135-143. [PubMed] [DOI] |
| 25. | Gao YF, Yu L, Wei W, Li JB, Luo QL, Shen JL. Inhibition of hepatitis B virus gene expression and replication by artificial microRNA. World J Gastroenterol. 2008;14:4684-4689. [PubMed] [DOI] |