修回日期: 2009-09-24
接受日期: 2009-10-12
在线出版日期: 2009-12-18
目的: 观察溃疡性结肠炎(UC)和肠易激综合征(IBS)之间基因表达上的差异, 从基因水平探讨UC的病理机制及其与IBS的区别.
方法: 从GEO下载UC和IBS基因芯片资料, 用BRB-ArrayTools 3.6软件对该数据进行挖掘, 并对差异表达基因进行生物信息学分析.
结果: 两者具有131条差异基因, 涉及炎症反应、免疫、信号传导、细胞外分泌、基因转录调节、细胞结构、细胞死亡等. 与IBS 相比, UC上调基因124条, 下调基因7条. 聚类分析发现, 两类样品可根据基因表达进行区分.
结论: 与IBS相比, UC肠黏膜在炎症、分泌、细胞生长和增殖等的相关基因表达增高; 利用基因芯片能够对两种疾病进行区别.
引文著录: 陈玉龙, 苗艳艳, 吕翠田. 溃疡性结肠炎与肠应激综合征间差异基因表达. 世界华人消化杂志 2009; 17(35): 3601-3605
Revised: September 24, 2009
Accepted: October 12, 2009
Published online: December 18, 2009
AIM: To identify differentially expressed genes between ulcerative colitis (UC) and irritable bowel syndrome (IBS) and analyze the difference in the pathogenesis between UC and IBS at the genetic level.
METHODS: The microarray data for UC and IBS were downloaded from the Gene Express Omnibus (GEO) and mined using the BRB-Array Tools software package to obtain a set of differential genes. Differentially expressed genes were then analyzed using bioinformatic tools.
RESULTS: A total of 131 differential genes were identified between UC and IBS. These genes are involved in inflammatory response, immunity, signal transduction, cell secretion, transcriptional regulation, cell structure, cell death, etc. Compared to IBS, 124 genes were up-regulated and 7 down-regulated in UC. Cluster analysis showed that the two kinds of samples could be distinguished by specific gene expression profiles.
CONCLUSION: Compared to IBS, many genes involved in inflammatory response, and cell growth, proliferation and secretion are upregulated in UC. Microarray analysis can successfully distinguish UC and IBS samples.
- Citation: Chen YL, Miao YY, Lv CT. Identification of differentially expressed genes between ulcerative colitis and irritable bowel syndrome. Shijie Huaren Xiaohua Zazhi 2009; 17(35): 3601-3605
- URL: https://www.wjgnet.com/1009-3079/full/v17/i35/3601.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i35.3601
溃疡性结肠炎(ulcerative colitis, UC)是一种慢性非特异性肠道炎症性疾病, 主要表现为腹痛、腹泻、黏液便或便血等症状, 该病与遗传易感性、免疫应答、环境等多种因素有关[1]. 已有很多利用基因芯片技术研究该病与克隆恩氏病及其他炎性肠病间差异基因、UC相关肿瘤(UC-associated colorectal cancer, UC-CRC)基因、不同造模方法基因表达差异等的报道[2-3], 从分子生物学角度探讨了该病的发病机制及与其他疾病的区别. 肠易激综合征(irritable bowel syndrome, IBS)为功能性肠功能紊乱, 主要以排便伴有腹部不适或腹痛, 或有不规则排便习惯的改变为症状[4]. 其发病机制与遗传、心理、胃肠运动失常、腹部高敏、感染和炎症等多种因素有关[5]. 该病与溃疡性结肠在症状上有一定重叠[6], 两者有时相互误诊, 目前应用基因芯片研究UC和IBS区别的报道尚少. 本研究从基因表达资料库(gene express omnibus, GEO)下载两者相互比较的基因芯片资料, 进行数据挖掘, 探讨两者在基因表达上的差异.
1.1.1 GSE2461数据集: 从GEO下载GSE2461数据集(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE2461), 进行归类整理. 该数据集是基于GPL96芯片平台, GSE2461是由丹麦学者Jorgen Olsen于2005-04公布在GEO, 是关于IBS和UC基因芯片数据集, 集中有8例样品数据由GPL96平台得来, 他们为: GSM46539, 男性IBS; GSM 46541, 女性IBS; GSM 46542, 女性IBS; GSM 46543, 女性IBS; GSM 46544, 女性IBS; GSM 46540, 男性UC; GSM46545, 男性UC; GSM46546, 女性UC.
1.1.2 GPL96芯片平台: GPL96(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL96)又称人类基因组HG-U133A芯片集(affymetrix), 包含1 000 000寡核苷酸片断, 覆盖39 000个转录变异体, 代表33 000条人全长基因. 实际研究中应用的GPL96平台, 涉及18 400个转录本和400条表达序列标签共22 000条已明确注释的基因. 在公开发表的报道中, 目前已有15 914份样品是用该平台进行研究的.
采用数据挖掘软件BRB-ArrayTools 3.6版(BRB)对该数据库进行挖掘, 筛选差异表达基因, 然后对差异表达基因进行生物信息学分析. 资料缺失50%, 或少于20%的表达资料大于或等于1.5倍单方向基因中位数值被过滤, 重复基因点取平均值, 应用HG-U133第7张芯片中的看家基因中位数进行均一化. 应用随机变量模型双样品T检验筛选差异基因, 对样品和基因进行层级聚类.
22 283条基因片段中1 633条通过过滤条件. 对两组进行随机变量模型双样品T检验, UC/IBS比率(ratio)ratio<1为UC下调基因, ratio>1为UC上调基因. 设定两个统计差异基因条件: (1)P<0.01; (2)P<0.01, ratio<0.5或ratio>2.0. 结果发现1 633条中, 符合条件(1)的差异基因片段141条(Gb), 涉及基因131条(Hs). UC基因下调7条, 其余124条都是上调. 符合条件(2)的差异基因片段33条, 涉及基因28条, 下调3条, 为免疫球蛋白κ链(IGKC///IGKV1-5///IGKV2-24)和免疫球蛋白重链α1(IGHA1///IGHA2), 肿瘤坏死因子受体脱氨基肽激酶调节因子1(tumor necrosis factor-receptor kinase regulator de-aminopeptidase1, ARTS-1); 上调基因25条, 主要为一氧化氮合酶2、黏液蛋白MUC3B、囊泡关联膜蛋白2、jun D原癌基因、蛋白磷酸激酶调节亚基15等(表1).
| 检验参数P-value值 | IBS组基因密度值几何均数 | UC组基因密度值几何均数 | 基因均数ratio值 | 探针号 | 基因名称 |
| 0.000244 | 205.9285517 | 417.7994053 | 0.4928886 | 214676_x_at | MUC3B |
| 0.0019582 | 90.8868736 | 184.8229232 | 0.4917511 | 202014_at | PPP1R15A |
| 5.43E-05 | 72.2036604 | 149.7764105 | 0.4820763 | 214792_x_at | VAMP2 |
| 0.0062763 | 133.4356297 | 296.1121829 | 0.4506253 | 214326_x_at | JUND |
| 0.0006259 | 53.8173706 | 120.5370706 | 0.4464798 | 34478_at | RAB11B |
| 0.009928 | 41.9325934 | 94.7902392 | 0.4423725 | 210037_s_at | NOS2A |
| 5.55E-05 | 119.2627791 | 294.06674 | 0.4055636 | 204907_s_at | BCL3 |
| 0.0044962 | 2736.27977 | 831.7465272 | 3.2898001 | 217022_s_at | IGHA1/IGHA2 |
| 0.0001021 | 228.8090336 | 98.3600146 | 2.3262403 | 209788_s_at | ARTS-1 |
| 0.009715 | 3350.126035 | 1527.193161 | 2.1936492 | 221651_x_at | IGKC/IGKV1-5/2-24 |
141条基因片段通过滤过标准, 检验了250个GO分类. 显著水平设定为默认值P<0.005. 有51条基因片段经过LS或KS参数检验, 归为结构分子活性7条, 调节RNA polymeraseⅡ启动子转录12条、细胞死亡16条.
2.3.1 KEGG通路: 131条基因中具有KEGG通路的30条, 有两条下调基因, 分别为钙连接蛋白和免疫球蛋白κ, 主要参与了抗原呈递过程. 其余为上调基因, 参与了MAPK信号通路5条基因; 精氨酸和脯氨酸代谢2条基因; 钙离子信号通路3条基因; 细胞通讯2条基因; 胰岛素信号通路2条基因, SANRE与囊泡相互作用1条基因、细胞因子相互作用1条基因等(表2).
| Kegg通路 | 基因名称 | Biocarta通路 | 基因名称 |
| SANRE与囊泡相互作用 | VAMP2 | 磷酸肌醇及下游通路 | GSK3A RD |
| 细胞通讯 | NES | IL-2受体Beta链对T细胞激活 | CISH |
| 钙离子信号通路 | TNFRSF6B | MAPK信号通路 | DUSP8 |
| 精氨酸和脯氨酸代谢 | NOS1 | 细胞的生存 | JUND |
| MAPK信号通路 | JUND | MAPK信号通路 | MAP2K2 |
| 胰岛素信号通路 | IRS2 | 由PPARα(alpha)产生的基因调控 | NOS2A |
| 精氨酸和脯氨酸代谢 | NOS2A | ER降解通路 | CANX |
| 抗原呈递 | CANX | 凋亡Apoptotic DNA片断组织稳定 | HMGB1 |
| 抗原呈递 | IGKC/IGKV1-5 | mRNA多腺苷化 | PAPOLA |
2.3.2 BioCarta通路: 131条基因中具有BioCarta 通路信息的18条基因, 3条下调基因, 分别为多聚腺苷酸聚合酶α、高迁移率组框和钙连接蛋白, 主要参与了mRNA多腺苷化和凋亡Apoptotic DNA片断组织稳定、ER降解通路. 其余为上调基因, 主要参与的细胞的生存、由PPARα(alpha)产生的基因调控、MAPK信号通路、IL-2受体 Beta链对T细胞的激活过程、磷酸肌醇及下游通路等(表2).
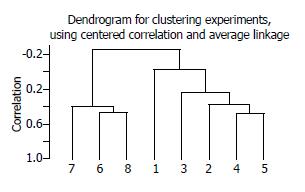
2.4.1 样品聚类: 根据各样品基因表达情况, 判别样品之间的差距离, 差距最近的样品聚为一类, 然后不断层积, 8个样品可聚为两大类, 溃疡性结肠炎样品为一类, 其中6号样品和8号样品差距最小; IBS样品为一类. 4号样品和5号样品差距最小(图1).
2.4.2 基因聚类: 根据差异基因在各样品表达情况, 判别基因之间的差距离, 差距最近的基因聚为一类, 然后不断层积, 基因也可聚为两大类, 一类为UC/IBS ratio >1.0, 一类为UC/IBS ratio<1, 每个大类包含差距离不断缩小的不同类别.图2所示为UC/IBS ratio<1的一类. 图中两个免疫球蛋白基因之间差距离最小, 聚为一类, 然后和热休克蛋白差距离较近; 肿瘤坏死因子脱氨基肽激酶调节因子1(ARTS-1)和poly(A)多聚酶(PAPOLA)差距离最小, 聚为一类; 如此不断层积, UC/IBSratio<1的基因聚为1大类. 由此结果看出, 差距离最小的1类基因在功能上或其转录调节可能非常相近, 如结果中2个免疫球蛋白分子; 两者为1类, 也可能就目前知识水平两者的功能或转录调节关系不明, 但提示在所检测的疾病中两者关系密切, 需要进一步研究, 如结果中的ARTS-1和PAPOLA.
UC和IBS是两种常见的胃肠道疾病, 都会出现腹痛、腹泻、黏液便等症状, 虽然结肠镜下两者能很好地进行鉴别, 但由于IBS急性期时也常常有轻微炎症改变, 因此也常常相互误诊. 同时, 他们的发病机制都和环境、遗传、炎症等因素密切相关, 在具体环节上有很多重叠, 但两者肯定又有很多不同. 基因芯片技术能够高通量、敏感、便捷地比较不同疾病状态下上万个基因差异, 是研究疾病之间分子差异的有力工具[7]. 近年来已有可观的利用基因芯片研究疾病病理机制和进行疾病分类比较的数据积累, 为了方便进行交流和资源共享, 已建立了一批网络数据库, GEO就是其中之一. 鉴于利用基因芯片研究UC和IBS之间基因表达差异的报道尚少, 而他们病理机制及其之间的异同未明, 我们利用GEO资源, 下载两者相互比较的基因芯片数据资料, 进行比较分析.
本研究发现了131条差异基因, 涉及炎症反应、免疫、信号传导、细胞分泌、基因转录调节、细胞结构、细胞死亡等. 与IBS相比, UC上调基因124条, 下调基因7条. 基于UC在机体病理状态表现比IBS更为严重的缘故, 很多介导细胞生存和死亡、炎症和抗炎等相互对立的相关基因表达升高, 这与慢性UC相对亢进的损伤和修复机制的病理状态一致, 从结果来看, UC表现7条下调基因中, 有2条是免疫球蛋白, 而且UC/IBS ratio<0.5, 免疫球蛋白κ链是所有免疫球蛋白轻链, 免疫球蛋白重链α1/α2是组成IgA1/IgA2的重链, 两者在芯片中表达一致, 层级聚类显示为差距离最小的一类, 说明两者实质上是代表免疫球蛋白IgA. IgA主要由黏膜相关淋巴样组织产生, 其中大部分是由胃肠淋巴样组织所合成, 是黏膜局部免疫的最重要因素. 结肠黏膜中IgA合成和分泌减少, 说明黏膜屏障功能降低. 由此可知, UC与IBS相比其黏膜免疫屏障功能降低更为严重. IgA以分泌性为主, 在UC与IBS之间表达有如此大差异, 可以通过粪便进行检测, 有望成为便捷地鉴别两种疾病的指标.
肿瘤坏死因子是重要的炎症因子, 与UC发生和发展关系密切. 研究发现, UC患者结肠黏膜中TNF高表达, 且其基因多态性与UC易感性明显相关. TNF-α抗体infliximab已成为临床治疗UC药物, 具有较好的治疗效果[8-10]. 本研究发现肿瘤因子受体超家族6(tumor necrosis factor receptor superfamily, member 6b, RTEL1///TNFRSF6B)在UC中高表达, 而对肿瘤坏死因子受体介导的信号传导具有负调节作用的ARTS-1[11]低表达, 也说明与IBS相比, TNF受体介导的信号导致的炎症反应在UC中起更重要作用.
差异基因中调节炎症反应的基因除TNF相关的两条基因外, 还有一氧化氮合酶(nitric oxide synthase2A/1, NOS2A/NOS1)、B-cell CLL/lymphoma 3(BCL3)、基质金属蛋白酶15(metalloproteinase15, MMP15)、磷脂酶A2(phospholipase A2 G4C, PLA2G4C)、过氧化物同系物(peroxidasin homolog (Drosophila), PXDN)、干扰素(alpha, beta and omega)受体2(interferon receptor, IFNAR2)、细胞因子诱导的SH2包含蛋白(cytokine inducible SH2-containing protein, CISH)、S100钙离子结合蛋白A8(S100 calcium-binding protein A8, S100A8)等. 这些基因在结肠黏膜表达UC比IBS高, 说明UC炎症严重程度和黏膜损伤要比IBS高.
本研究发现, 在UC中很多与细胞生长和增殖相关基因高表达, 如jun D原癌基因、丝裂活性蛋白激酶、Amphiregulin、RAS原癌基因家族RAB11B等. 其中有6条基因参与了MAPK信号通路, 该通路主要调节细胞的生长、增殖和炎症反应. 推测这些基因高表达原因: (1)促进炎症细胞的增殖分裂, 在UC结肠黏膜中具有更多炎症细胞聚集和更重的炎症表现; (2)肠黏膜损伤修复机制的启动, 严重的UC结肠黏膜损伤促使应激修复和抗损伤机制表现得也更为强烈, 黏膜细胞增殖也越明显.
黏蛋白(mucin, MUC)是肠黏膜化学屏障的重要组成部分, 对损伤的结肠黏膜具有重要保护和修复功能. 有研究表明, UC结肠黏膜上MUC-2、MUC12表达下调, MUC-3、MUC-4表达没有差异, MUC-5, MUC-6没有检出, 而MUC -1表达上调, 特别是肠隐窝脓肿位置[12]. 另外, 用基因芯片研究发现, 与正常人相比UC肠黏膜中MUC1、MUC2、MUC4、MUC5B、MUC12、MUC13、MUC17、MUC20表达降低[13]. 本研究发现与IBS相比, MUC5AC、MUC3B在UC结肠黏膜上高表达, 这可能是因为所比较的对象不同. 与正常人相比, 在IBS黏膜中他们的表达也许更低.
胞吐是细胞进行分泌的形式, 包括酶、激素、细胞因子的分泌, 其中有很多细胞器和多种分子参与[14]. 在本研究中发现多种胞吐相关的分子基因在UC-结肠黏膜中高表达, 包括vesicle-associated membrane protein 2(VAMP2)、solute carrier family 22 member 14(SLC15A1)、solute carrier organic anion transporter family, member 2A1(SLCO2A1)、WW domain binding protein 2 (WBP2)等. 说明UC结肠黏膜中有许多细胞分泌旺盛, 也可能是因为炎症细胞聚集分泌各种炎症因子的缘故.
总之, 我们利用GEO基因芯片资源对IBS和UC进行了比较分析, 发现了许多差异基因, 部分差异基因的表达与他人的研究相一致. 与IBS相比, UC肠黏膜炎症相关基因表达增强, 说明其黏膜炎症较重, 基本上反映了两种疾病的病理差异. 免疫球蛋白IgA在两者之间的差异, 是本研究的重要发现, 由于其以分泌型为主, 有望成为无创伤鉴别两者的分子标志物, 但需要进一步研究证实. 对于其他差异基因, 我们将深入研究分析.
基因芯片技术已广泛应用于疾病的诊断和分型, 并从差异表达基因角度研究不同疾病的发病机制及其异同, 包括UC和其他炎性肠病间比较. 但是, 应用该技术研究UC和IBS关系的报道尚少.
杜群, 副研究员, 广州中医药大学脾胃研究所药理室.
UC和IBS在症状和发病机制上有一定重叠, 特别是UC缓解期与IBS急性期, 两者有时相互误诊, 有必要深入研究两者的差异. 基因芯片技术是研究疾病之间分子差异的有力工具, 本文利用该技术资料探讨两者在基因表达上的差异.
Wu et al应用基因芯片技术比较了UC和Crohn's基因表达差异, 探讨了两者之间病理机制的不同; Watanabe et al应用该技术对UC和UC相关结肠癌进行了比较研究, 探讨了UC相关结肠癌发生机制及基因表达标志.
本文应用GEO资源, 从差异基因、本体功能、信号通路、聚类分析等方面进行了数据挖掘, 从差异基因角度比较了UC和IBS病理机制异同, 其中两者之间免疫球蛋白IgA的差异, 是本研究的重要发现.
本研究比较了UC和IBS间基因表达的差异, 为两者之间的鉴别提供了参考, 特别是两者之间免疫球蛋白IgA的差异, 有望成为便捷地鉴别两种疾病的标志.
本研究选题尚可, 方法新颖, 结果可靠, 具有一定的科学意义.
编辑: 李瑞敏 电编:吴鹏朕
| 1. | Hanauer SB. Inflammatory bowel disease: epidemiology, pathogenesis, and therapeutic opportunities. Inflamm Bowel Dis. 2006;12 Suppl 1:S3-S9. [PubMed] [DOI] |
| 2. | Wu F, Dassopoulos T, Cope L, Maitra A, Brant SR, Harris ML, Bayless TM, Parmigiani G, Chakravarti S. Genome-wide gene expression differences in Crohn's disease and ulcerative colitis from endoscopic pinch biopsies: insights into distinctive pathogenesis. Inflamm Bowel Dis. 2007;13:807-821. [PubMed] [DOI] |
| 3. | Watanabe T, Kobunai T, Toda E, Kanazawa T, Kazama Y, Tanaka J, Tanaka T, Yamamoto Y, Hata K, Kojima T. Gene expression signature and the prediction of ulcerative colitis-associated colorectal cancer by DNA microarray. Clin Cancer Res. 2007;13:415-420. [PubMed] [DOI] |
| 4. | Longstreth GF, Thompson WG, Chey WD, Houghton LA, Mearin F, Spiller RC. Functional bowel disorders. Gastroenterology. 2006;130:1480-1491. [PubMed] [DOI] |
| 5. | Ohman L, Simrén M. New insights into the pathogenesis and pathophysiology of irritable bowel syndrome. Dig Liver Dis. 2007;39:201-215. [PubMed] [DOI] |
| 6. | Quigley EM. Irritable bowel syndrome and inflammatory bowel disease: interrelated diseases? Chin J Dig Dis. 2005;6:122-132. [PubMed] [DOI] |
| 7. | Lee NH, Saeed AI. Microarrays: an overview. Methods Mol Biol. 2007;353:265-300. [PubMed] |
| 9. | Parkes M, Barmada MM, Satsangi J, Weeks DE, Jewell DP, Duerr RH. The IBD2 locus shows linkage heterogeneity between ulcerative colitis and Crohn disease. Am J Hum Genet. 2000;67:1605-1610. [PubMed] [DOI] |
| 10. | Wilhelm SM, McKenney KA, Rivait KN, Kale-Pradhan PB. A review of infliximab use in ulcerative colitis. Clin Ther. 2008;30:223-230. [PubMed] [DOI] |
| 11. | Cui X, Hawari F, Alsaaty S, Lawrence M, Combs CA, Geng W, Rouhani FN, Miskinis D, Levine SJ. Identification of ARTS-1 as a novel TNFR1-binding protein that promotes TNFR1 ectodomain shedding. J Clin Invest. 2002;110:515-526. [PubMed] |
| 12. | Longman RJ, Poulsom R, Corfield AP, Warren BF, Wright NA, Thomas MG. Alterations in the composition of the supramucosal defense barrier in relation to disease severity of ulcerative colitis. J Histochem Cytochem. 2006;54:1335-1348. [PubMed] [DOI] |
| 13. | Moehle C, Ackermann N, Langmann T, Aslanidis C, Kel A, Kel-Margoulis O, Schmitz-Madry A, Zahn A, Stremmel W, Schmitz G. Aberrant intestinal expression and allelic variants of mucin genes associated with inflammatory bowel disease. J Mol Med. 2006;84:1055-1066. [PubMed] [DOI] |
| 14. | Stow JL, Manderson AP, Murray RZ. SNAREing immunity: the role of SNAREs in the immune system. Nat Rev Immunol. 2006;6:919-929. [PubMed] [DOI] |