修回日期: 2009-09-26
接受日期: 2009-09-28
在线出版日期: 2009-10-18
目的: 系统分析基因甲基化在肝癌发生中涉及的分子途径, 从中选取代表性基因进一步分析其甲基化在细胞癌变过程中的生物学及临床意义.
方法: 对10年间关于肝癌基因甲基化相关文献进行数据发掘与功能分类, 在其中2个与肝癌发生相关的生物途径中选取APC和CDKN2A基因, 采用实时定量甲基化特异性PCR技术对46例成对肝癌与癌旁组织中基因的甲基化水平进行定量分析并评估其诊断价值.
结果: 生物信息学分析显示肝癌中发生甲基化的基因主要涉及Wnt/β-catenin、p16及p53 3个主要分子途径; 甲基化分析表明CDKN2A基因在肝癌组织中甲基化水平极显著高于癌旁组织(P<0.0001), 并且癌组织中甲基化程度在高龄患者中较高(P = 0.0027); ROC曲线分析表明通过CDKN2A甲基化定量分析能够高效区分癌与非癌组织(AUC = 0.8526). APC基因在癌与癌旁间总体甲基化水平无显著性差异(P = 0.0656).
结论: CDKN2A基因甲基化水平的升高在肝细胞癌变过程中可能具有重要作用, 其甲基化水平的升高可作为一种鉴别癌与非癌组织的分子标志物. CDKN2A甲基化模式的特异性使其在肝癌早期筛查和诊断中具有一定的临床应用价值.
引文著录: 白同, 杨斌, 娄诚, 张晔, 高英堂, 王毅军, 杜智, 宋文芹. APC和CDKN2A基因甲基化定量分析对肝细胞癌的诊断价值. 世界华人消化杂志 2009; 17(29): 3001-3007
Revised: September 26, 2009
Accepted: September 28, 2009
Published online: October 18, 2009
AIM: To investigate methylation-related molecular pathways associated with hepatocellular carcinogenesis and determine the clinical implications of representative genes involved in these pathways.
METHODS: A PubMed search was performed to retrieve and analyze relevant publications during the past 10 years via literature mining. Related methylated genes were classified according to Gene Ontology criteria. The methylation of adenomatous polyposis coli (APC) and cyclin-dependent kinase inhibitor 2A (CDKN2A) genes was quantitatively determined in 46 paired tumor and adjacent non-tumor tissues by MethyLight assay. The methylation levels of the two genes were evaluated to determine their implications for diagnosis of hepatocellular carcinoma (HCC).
RESULTS: Bioinformatic analysis revealed that methylated genes were mainly involved in the Wnt/-catenin, p16 and p53 molecular pathways. The methylation level of CDKN2A was significantly higher in tumor tissues than in adjacent non-tumor tissues (P < 0.0001). Moreover, the methylation level of CDKN2A was significantly higher in older patients than in younger ones (P = 0.0027). Receiver operating characteristic (ROC) analysis demonstrated that CDKN2A methylation level could be used to distinguish neoplastic lesions from non-malignant tissues.
CONCLUSION: Elevation of CDKN2A methylation level may play an important role in hepatocarcinogenesis. CDKN2A methylation level can serve as a biomarker for distinguishing tumor tissues from non-tumor tissues and be used for screening or diagnosis of HCC.
- Citation: Bai T, Yang B, Lou C, Zhang Y, Gao YT, Wang YJ, Du Z, Song WQ. Clinical implications of quantitative methylation analysis of the adenomatous polyposis coli and cyclin-dependent kinase inhibitor 2A genes in hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2009; 17(29): 3001-3007
- URL: https://www.wjgnet.com/1009-3079/full/v17/i29/3001.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i29.3001
基因组DNA甲基化模式的异常改变是癌细胞的一个重要特征, 在1996年Herman et al建立了针对特定基因区域进行甲基化检测技术之后[1], 近十多年来抑癌基因启动子区甲基化在癌症发生与发展中的机制得到了深入研究, 同时也积累了大量相关的文献报道. 对这些文献进行系统分析与数据发掘, 不仅有助于全面理解DNA甲基化这一表观遗传学方式在肿瘤形成过程的详细作用, 同时也可为用于癌症筛查、诊断或预后等各类甲基化生物标志物的开发提供选择依据. 本研究在文献发掘基础上, 从肝癌发生的相关分子途径中选取APC和CDKN2A 2个典型基因, 运用定量甲基化分析技术对成对肝癌与癌旁组织间甲基化水平进行比较分析, 探讨了其甲基化在肝癌发生过程中的生物学意义; 同时也依照生物标志物的发现、筛选、验证与鉴定等技术流程对这两个基因甲基化在肝癌中的诊断价值进行了评估.
46例肝细胞癌组织取自天津市第三中心医院2001-2005年手术切除标本并经病理确诊, 标本在术后立即置于-70℃低温冰箱冷冻保存备用. 标本取材获得患者书面知情同意, 并经本院医学伦理委员会批准.
1.2.1 文献发掘及基因分类的生物信息学分析: 首先以"hepatocellular carcinoma"和"methylation"为关键词, 在NCBI生物医学文献数据库中检索1997-2007年肝癌中基因甲基化的文献, 经过鉴别和筛选后运用文献发掘工具BiblioSphere(V7.12, Genomatix Software)对与肝癌发生或临床表现密切相关的研究论文及其中涉及的基因[2]进行相关性(gene connection)分析与分类(gene ontology, GO)(http://www.geneontology.org).
1.2.2 APC与CDKN2A基因启动子区序列分析: 在Ensemble基因组浏览器中获取2个基因的结构和序列后, 用EBI的CpG Plot工具扫描鉴定基因启动子区域的CpG岛(CG>50%, Obs/Esp>0.6), 以此作为甲基化检测与分析的目的片段.
1.2.3 MethyLight定量PCR方法及相关引物和探针: MethyLight是一种建立在TaqMan探针法实时荧光PCR技术上的甲基化定量分析技术, 他通过一对甲基化特异性扩增引物和一条覆盖若干CpG二核苷酸的水解探针来对目的基因进行定量[3]; 在本实验中通过ACTB(β-actin)参比基因来校正样品间在模板量上的差异. 引物和探针由上海超世生物科技有限公司合成, 序列见表1.
| 基因 | PCR引物及TaqMan探针序列(5'-3') |
| APC (98 bp) | F: TATTGCGGAGTGCGGGTC |
| R: TCGACGAACTCCCGACGA | |
| P: FAM-ACGCCCTAATCCGCATCCAACGA-BHQ | |
| CDKN2A (83 bp) | F: TCGAATAGTTACGGTCGGAGGTC |
| R: GTCCTCCAAAATCGCCCG | |
| P: FAM-CATCCCCTACTCCCGCTACAAACCCTC-BHQ | |
| ACTB (133 bp) | F: TGGTGATGGAGGAGGTTTAGTAAGT |
| R: AACCAATAAAACCTACTCCTCCCTTAA | |
| P: FAM-ACCACCACCCAACACACAATAACAAACACA-BHQ |
1.2.4 MethyLight定量PCR体系与扩增条件: 标本组织基因组DNA的提取、模板的亚硫酸氢化修饰、甲基化特异性PCR(Methylation-Specific PCR, MSP)检测、扩增片段的序列验证、重组质粒的制备、定量标准曲线的构建等具体操作方法参照前文所述[4-5]. 定量PCR使用ABI Prism® 7000型荧光定量PCR仪(Applied Biosystems)在96孔板上进行扩增, 25 μL反应体系包含1×Real time PCR Master Mix(Toyobo Co., Ltd)、400 nmol/L引物、200 nmol/L探针以及修饰后的DNA模板; PCR使用两步法进行扩增: 95℃预变性2 min后, 95℃变性15 s, 60℃退火/延伸45 s, APC基因扩增40个循环, CDKN2A基因扩增45个循环. 每次检测通过质粒标准品制作定量标准曲线, 并同时设置阴性对照(健康人白细胞基因组DNA)、阳性对照(SssI甲基转移酶体外修饰基因组DNA)和空白对照. 定量PCR实时数据的采集和分析用ABI公司的Sequence Detection Systems(V1.1, Applied Biosystems)软件完成.
1.2.5 定量PCR的数据处理: 在本研究中使用甲基化百分比参数(percentage of methylated reference, PMR)来描述基因在组织中的甲基化水平[6]; 其计算过程是首先通过ACTB参比基因来对目的基因进行相对定量, 然后再用甲基化酶体外修饰DNA的相对定量值对实验样品值进一步作归一化处理. 计算公式如下: PMR = (GENEsample/REFsample)/(GENEIVD /REFIVD)×100%(GENE: 目的基因; REF: 参比基因; Sample: 样品; IVD: 体外修饰DNA).
统计学处理 肝癌与癌旁组织中总体甲基化水平以"PMR中位值(IQR, 25%-75%)"表示; 二者间甲基化水平的比较用Mann-Whitney U检验; 病例个体中癌与癌旁甲基化水平的比较用Wilcoxon配对秩和检验; 通过ROC曲线分析来根据甲基化水平鉴别肝癌的灵敏度与特异性; 基因甲基化程度与临床病理特征间的联系通过Fisher精确检验分析; 不同甲基化水平患者术后总体生存期、无瘤生存期的比较通过Kaplan-Meier生存曲线进行分析. 以上检验均为双侧检验, P<0.05为有显著性差异, 分析使用GraphPad Prism(V5.0, GraphPad Software)进行.
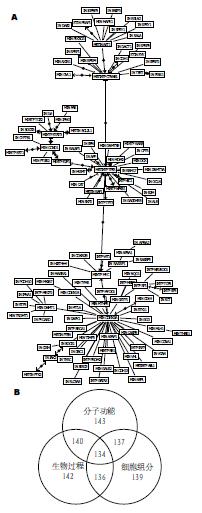
通过对文献数据库检索, 得到10年间共330篇关于肝癌中基因甲基化的文献; 对其中91篇研究性论文中涉及的153个基因进行基于文本的相关性分析与GO分类(图1). 如图1A所示, 153个基因分别以CTNNB1、TP53和CDKN2A 3个节点(node)为中心形成3个基因簇(cluster); 此外, 153个基因中145个具有GO注释, 分类结果表明这些基因广泛地参与分子功能(molecular function, 143个)、生物过程(biology process, 142个)及细胞组成(cell component, 139个)3类生物学活动(图1B).
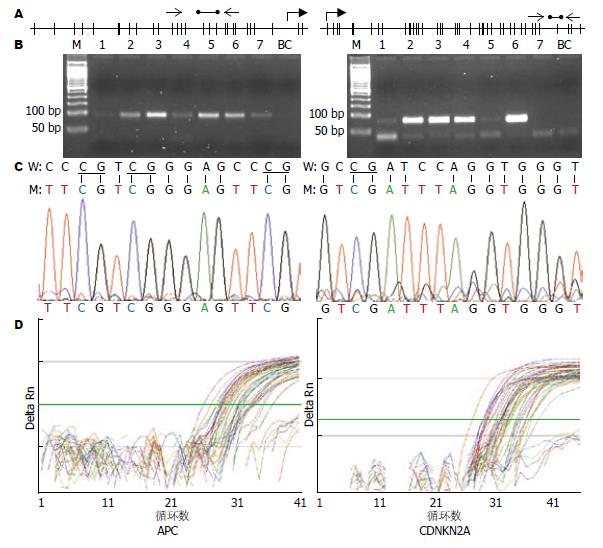
根据对肝癌相关基因生物信息学分析的结果, 分别选取上述基因簇1和3中的代表性基因APC和CDKN2A(P16, INK4A)进行分析. 经基因结构分析和CpG岛扫描后, 选择位于转录起始位点(transcription start site, TSS)附近的区段作为检测的目的片段(图2A). 为了对所用引物和扩增条件进行验证, 首先对10例肝癌组织标本进行MSP扩增, 其中7例的电泳分析如图2B所示, 检测结果表明对2个目的基因扩增片段大小正确; MSP阳性片段的测序结果进一步显示, 序列中CG二核苷酸中的C保持不变, 而CG二核苷酸之外的C均已转变成为T(图2C). 因此, MSP定性检测和测序结果证实了引物扩增及模板修饰的正确性.
图2D所示为APC和CDKN2A两个基因在46例癌组织中的实时荧光PCR扩增曲线, 所测标本约在25个循环后相继进入指数扩增期, 35个循环后到达平台期; 这一阶段曲线呈陡峭坡度上升, 且在样本间具有较好的平行性; 同时, 实验中阴性对照、空白对照以及阴性标本在扩增终点的ΔRn值均保持在较低水平, 为检测阈值的设定提供了较宽的选择范围. 这些结果反映出PCR扩增的高效性以及水解探针与目的片段结合的高特异性, 这也是保证定量数据准确性的前提条件.
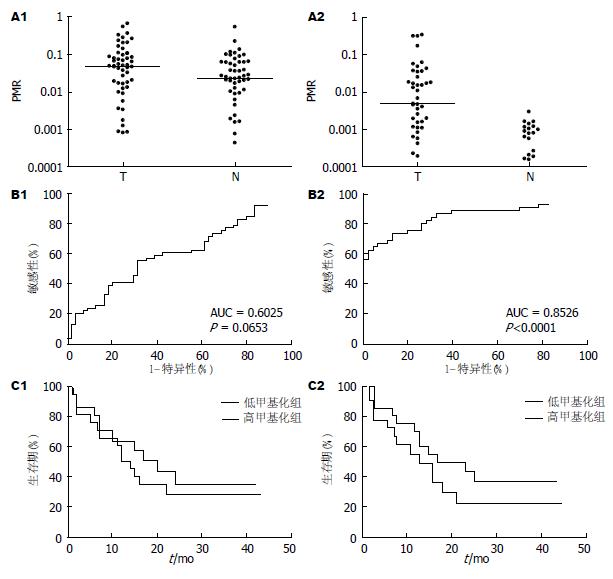
2个基因在肝癌与癌旁组织中甲基化水平的比较如图3A所示: 46例标本中, APC在癌组织中PMR的中位值为0.0472(IQR, 0.0098-0.1025), 在癌旁组织中为0.0230(IQR, 0.0040-0.0637); CDKN2A在癌组织中PMR的中位值为0.0048(IQR, 0.0008-0.0293), 在癌旁组织中为0(IQR, 0-0.0006). 其中APC基因在肝癌与癌旁组织中总体甲基化水平无显著性差异(P = 0.0656), 但对个体而言, 癌组织中甲基化程度略高于癌旁组织(P = 0.0329); 而CDKN2A基因无论是在总体还是个体水平, 癌组织中甲基化程度均极显著地高于癌旁组织(P<0.0001).
通过ROC曲线来分析基因甲基化的定量值能否以较高的效率(灵敏度和特异性)将癌组织与非癌组织进行区分. 如图3B所示, 以癌组织中的PMR值作为病例组, 癌旁组织中的PMR值作为对照组, APC基因的AUC值(0.6025)仅略高于0.5, 统计学比较表明该基因在癌与癌旁甲基化的定量分析不足以将二者区分(P = 0.0653); 而CDKN2A基因甲基化水平的差异则可以使其高效地从非癌组织中鉴别出癌变组织(AUC = 0.8526, P<0.0001).
首先以各自基因PMR值的中位值作为阈值, 通过二分法将连续型的PMR值转换为离散型数据, 即PMR值高于中位值的组织为高甲基化, 低于中位值的组织为低甲基化. 将分层后的甲基化数据与临床病理资料进行统计学分析, 结果表明CDKN2A在高年龄(>55岁)组中的甲基化水平显著高于低年龄(≤55岁)组患者(P = 0.0027)(表2). 此外, Kaplan-Meier分析显示癌组织APC及CDKN2A基因高甲基化组与低甲基化组的患者的术后总体生存期与无瘤生存期(图3C)均无显著差异.
| APC | CDKN2A | |||||
| 高甲基化 | 低甲基化 | P值 | 高甲基化 | 低甲基化 | P值 | |
| 性别 | ||||||
| 男 | 19 | 19 | 17 | 21 | ||
| 女 | 4 | 4 | 1.000 | 6 | 2 | 0.2427 |
| 年龄(岁) | ||||||
| ≤55 | 13 | 12 | 7 | 18 | ||
| >55 | 10 | 11 | 1.000 | 16 | 5 | 0.0027 |
| 病毒感染 | ||||||
| 阳性1 | 20 | 20 | 21 | 19 | ||
| 阴性 | 3 | 3 | 1.000 | 2 | 4 | 0.6652 |
| AFP水平(mg/L) | ||||||
| ≤400 | 14 | 13 | 16 | 11 | ||
| >400 | 9 | 10 | 1.000 | 7 | 12 | 0.2307 |
| 肿瘤直径(cm) | ||||||
| ≤5.0 | 8 | 9 | 7 | 10 | ||
| >5.0 | 15 | 14 | 1.000 | 16 | 13 | 0.5420 |
| 肿瘤数目 | ||||||
| 单发 | 14 | 14 | 13 | 15 | ||
| 多发 | 9 | 9 | 1.000 | 10 | 8 | 0.7631 |
| 血管侵犯 | ||||||
| 有 | 9 | 8 | 8 | 9 | ||
| 无 | 14 | 15 | 1.000 | 15 | 14 | 1.0000 |
全基因组范围内的CpG岛芯片扫描技术和基于文献发掘的生物信息学分析都是肿瘤表观遗传学研究的重要手段[7], 同时也是筛选各类甲基化生物标志物的主要方法, 如Zindy et al就将这两方法相结合, 鉴定出一个在肝纤维化与肝癌发生过程起重要作用的表达上调基因MEN1. 本研究通过基于文献的数据发掘得到3个与肝癌发生相关的分子途径, 即以β-catenin(CTNNB1基因产物)为核心的Wnt途径、以p16(CDKN2A基因产物)为核心的细胞周期途径、以及以p53(TP53基因产物)为核心的细胞凋亡等多功能分子途径, 这一生物信息学分析结果与多篇文献综述中总结的肝癌发生的分子途径非常一致[8-9]. 这表明由甲基化导致的抑癌基因失调在肝细胞癌变过程中具有普遍意义; 同时, 文献发掘的分析结果也为我们探索肝癌相关基因甲基化的临床意义或应用提供了线索和依据.
在前期研究中, 我们用甲基化特异性PCR技术检测了包括APC和CDKN2A基因在内的8个肝癌相关基因在肝癌、癌旁、肝硬化、肝炎以及正常组织中的甲基化发生频率[10]; 本研究的重点在于通过分析成对癌与癌旁组织间基因甲基化水平的变化程度, 探讨甲基化的积累在细胞癌变这一特定阶段中的作用和意义. APC基因的突变可以通过激活Wnt/β-catenin途径导致肝癌形成[11], 然而这种情况在实际的肝癌病例中却仅有个案报道[12]; 而APC在肝癌组织中发生甲基化的现象却非常普遍, 但同时在肝硬化、肝炎甚至正常肝组织中也观察到较高比例的甲基化[13]. 本研究结果表明, APC基因的甲基化在肝细胞癌变之前就已积累到较高水平, 因此, 即使在癌变过程中仍略有持续升高, 但此时这种高水平的甲基化现象显然不是导致癌变的一个主要因素; 同时, 由于该基因在癌与非癌组织在甲基化模式上相差不大, 因此其作为诊断标志物的价值有限. 这一结论可以得到欧洲和亚洲其他地区相关研究结果的支持[14-15].
CDKN2A基因的产物p16在细胞周期的调控中处于核心地位, 因此该基因在肝癌中甲基化也得到了广泛关注和研究. 然而, 由于肝癌在不同地区或人种间具有不同的病因学, 各研究小组所用检测方法也不尽相同, 因此对于CDKN2A在肝癌中的甲基化模式及其临床相关性, 很多文献报道尚存在差异, 尤其是对于甲基化与病毒、年龄等因素在肝癌发生过程中的时序与因果关系都还有待深入探讨[15-16]. 本研究所观察到CDKN2A基因在癌与癌旁组织间甲基化水平的显著差异, 反映了该基因甲基化在肝癌发生中的阶段特异性; 或者说CDKN2A基因甲基化水平的急剧升高是肝细胞癌变这一特定阶段的重要事件, 随后由甲基化导致基因失活及表达缺失进一步促进了细胞转化. 虽然在高龄组肝癌患者癌组织中甲基化水平显著高于低龄组, 然而无论高龄组还低龄组, 其癌旁组织中的甲基化水平都仅维持在极低的水平, 这表明高龄患者癌组织中的高甲基化并非是简单地通过时间的积累而造成的, 这一现象可能是细胞在衰老过程中, CDKN2A等衰老相关基因与致癌因素交互作用下更容易发生甲基化的结果[17].
CDKN2A甲基化在肝癌早期筛查和诊断中具有一定的临床应用前景, 在本研究中我们着重评估了该基因甲基化水平在肝癌诊断上的可行性. 由于DNA甲基化的异常改变要早于mRNA或蛋白质表达等癌变过程中的分子事件, 更要早于细胞形态上的改变, 因此, 对于CDKN2A这类具有肿瘤特异性和阶段性的抑癌基因, 其甲基化水平极有潜力作为一种分子诊断的手段. 基因甲基化作为一种诊断标志物的关键在于他应当能够高效地将癌变的组织从非癌组织中区分开. 本研究中ROC曲线分析表明, 尽管在癌旁组织中由于环境因素而存在相当比例的CDKN2A甲基化, 但通过甲基化定量分析则可以高效地将癌组织从这些低甲基化水平的"噪音"中区分出来. 目前, 在肝炎或肝硬化等肝癌高危人群中, 通过超声手段发现的可疑病灶往往需要穿刺活检进行确诊, 考虑到基因甲基化标志物在肝癌诊断中的角色是在血清学、影像学和病理学等多种传统方法诊断基础上提供分子生物学上的证据, 因此在诊断阈值的选择上优先考虑特异性(95%), 而此时仍可保证较高的诊断灵敏度(65%). 这些结果表明CDKN2A的甲基化水平可以作为一种肝癌诊断的候选标志物.
总之, 本研究通过对APC和CDKN2A两个基因在癌变过程中甲基化水平的定量分析表明, CDKN2A基因甲基化的积累在肝细胞癌变过程中可能具有重要作用, 而且这种具有组织和阶段特异性的甲基化模式也使CDKN2A基因甲基化可以作为一种具有临床诊断价值的分子标志物; 同时, 本研究中所建立的候选生物标志物的发现、筛选、验证与评价流程也可以为肝癌其他甲基化标志物的研发提供参考.
肝细胞癌是临床上常见且病死率较高的恶性肿瘤, 早期诊断及早期治疗对提高患者的生存率具有极其重要的意义. 随着表观遗传学的深入研究, 基因的甲基化检测有望应用于肝细胞癌的早期诊断.
陈洪, 副教授, 东南大学附属中大医院消化科; 肖恩华, 教授, 中南大学湘雅二医院放射教研室.
肝细胞癌的表观遗传学方向的研究前景比较乐观, 但是目前尚处于起步阶段, 表观遗传改变的一个重要特点在于其改变是可逆的, 即在药物作用下可以发生逆转, 肿瘤的分子靶向药物治疗可能成为继分子诊断的又一热点与重点.
Yang et al通过对肝细胞癌的癌组织及手术切缘的CDKN2A基因在26个CpG二核苷酸位点具体甲基化状态分析得出, 肝细胞癌中CDKN2A基因在体内甲基化的延伸过程是一种"渐进式"的方式, 而不是"全或无"的方式.
本研究对10年间关于肝癌基因甲基化相关文献进行数据发掘与功能分类, 选取APC和CDKN2A基因, 采用实时定量甲基化特异性PCR技术检测基因的甲基化水平对肝细胞癌的诊断价值. 为肝癌其他甲基化标志物的研发提供参考.
本研究发现CDKN2A甲基化在肝癌早期筛查和诊断中具有一定的临床应用价值, 具有一定特色.
编辑: 李军亮 电编:吴鹏朕
| 1. | Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci U S A. 1996;93:9821-9826. [PubMed] [DOI] |
| 2. | Zindy PJ, L'Helgoualc'h A, Bonnier D, Le Béchec A, Bourd-Boitin K, Zhang CX, Orlando Musso O, Glaise D, Bérangère Troadec M, Loréal O. Upregulation of the tumor suppressor gene menin in hepatocellular carcinomas and its significance in fibrogenesis. Hepatology. 2006;44:1296-1307. [PubMed] [DOI] |
| 4. | Yang B, Gao YT, Du Z, Zhao L, Song WQ. Methylation-based molecular margin analysis in hepatocellular carcinoma. Biochem Biophys Res Commun. 2005;338:1353-1358. [PubMed] [DOI] |
| 5. | Yang B, Lou C, Gao Y, Du Z, Song W. [Detection of methylation in hepatocellular carcinoma using SYBR Green fluorescent quantitative PCR]. Zhonghua Yixue Yichuanxue Zazhi. 2008;25:534-537. [PubMed] |
| 6. | Eads CA, Lord RV, Wickramasinghe K, Long TI, Kurumboor SK, Bernstein L, Peters JH, DeMeester SR, DeMeester TR, Skinner KA. Epigenetic patterns in the progression of esophageal adenocarcinoma. Cancer Res. 2001;61:3410-3418. [PubMed] |
| 7. | Zheng D, Liu BB, Liu YK, Kang XN, Sun L, Guo K, Sun RX, Chen J, Zhao Y. [Screening for differential methylation status by CpG island microarray in the hepatocellular carcinoma cell lines]. Zhonghua Zhongliu Zazhi. 2008;30:891-896. [PubMed] |
| 8. | Laurent-Puig P, Zucman-Rossi J. Genetics of hepatocellular tumors. Oncogene. 2006;25:3778-3786. [PubMed] [DOI] |
| 9. | Farazi PA, DePinho RA. Hepatocellular carcinoma pathogenesis: from genes to environment. Nat Rev Cancer. 2006;6:674-687. [PubMed] [DOI] |
| 10. | Lou C, Yang B, Gao YT, Wang YJ, Nie FH, Yuan Q, Zhang CL, Du Z. [Aberrant methylation of multiple genes and its clinical implication in hepatocellular carcinoma]. Zhonghua Zhongliu Zazhi. 2008;30:831-836. [PubMed] |
| 11. | Colnot S, Decaens T, Niwa-Kawakita M, Godard C, Hamard G, Kahn A, Giovannini M, Perret C. Liver-targeted disruption of Apc in mice activates beta-catenin signaling and leads to hepatocellular carcinomas. Proc Natl Acad Sci U S A. 2004;101:17216-17221. [PubMed] [DOI] |
| 12. | Chan DW, Chan CY, Yam JW, Ching YP, Ng IO. Prickle-1 negatively regulates Wnt/beta-catenin pathway by promoting Dishevelled ubiquitination/degradation in liver cancer. Gastroenterology. 2006;131:1218-1227. [PubMed] [DOI] |
| 13. | Harder J, Opitz OG, Brabender J, Olschewski M, Blum HE, Nomoto S, Usadel H. Quantitative promoter methylation analysis of hepatocellular carcinoma, cirrhotic and normal liver. Int J Cancer. 2008;122:2800-2804. [PubMed] [DOI] |
| 14. | Nishida N, Nagasaka T, Nishimura T, Ikai I, Boland CR, Goel A. Aberrant methylation of multiple tumor suppressor genes in aging liver, chronic hepatitis, and hepatocellular carcinoma. Hepatology. 2008;47:908-918. [PubMed] [DOI] |
| 15. | Su PF, Lee TC, Lin PJ, Lee PH, Jeng YM, Chen CH, Liang JD, Chiou LL, Huang GT, Lee HS. Differential DNA methylation associated with hepatitis B virus infection in hepatocellular carcinoma. Int J Cancer. 2007;121:1257-1264. [PubMed] [DOI] |
| 16. | Jicai Z, Zongtao Y, Jun L, Haiping L, Jianmin W, Lihua H. Persistent infection of hepatitis B virus is involved in high rate of p16 methylation in hepatocellular carcinoma. Mol Carcinog. 2006;45:530-536. [PubMed] [DOI] |
| 17. | Ahuja N, Issa JP. Aging, methylation and cancer. Histol Histopathol. 2000;15:835-842. [PubMed] |