修回日期: 2009-06-16
接受日期: 2009-06-23
在线出版日期: 2009-07-18
目的: 构建TROP2特异性短发夹环RNA(shRNA)真核表达载体, 抑制人胃癌BGC-823细胞TROP2基因的表达.
方法: 构建TROP2短发夹环RNA, 产生重组质粒转染胃癌BGC-823细胞, 转染24 h后用G418 (浓度400 mg/L)筛选, 待细胞稳定后收集, 分别命名为W组(未处理组), HK组(随机阴性对照质粒组), KB组(空质粒组), T1组, T2组, T3组. 并运用实时荧光定量PCR和Western blot检测TROP2的表达.
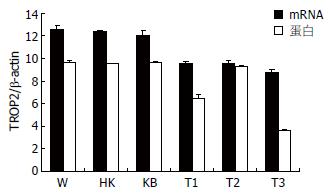
结果: TROP2特异性shRNA片段被成功克隆进pGensil1.1质粒中, 重组质粒shRNA编码序列与设计片断的序列完全一致. 与未转染细胞组、随机阴性对照组、空质粒组相比, 转染shRNA重组质粒的人胃癌BGC-823细胞TROP2表达在mRNA和蛋白水平都受到抑制. 与T1、T2组相比, T3组对TROP2 mRNA和蛋白抑制作用最明显, 差异具统计学意义(8.79±0.23 vs 9.54±0.20, 9.57±0.23; 3.66±0.11 vs 6.46±0.36, 9.31±0.11, 均P<0.05).
结论: 成功构建了针对TROP2的特异性shRNA真核表达载体并抑制了TROP2的表达, 为进一步研究其基因功能打下了基础.
引文著录: 戴东方, 陈德玉, 邵世和. TROP2 shRNA真核表达载体转染胃癌BGC-823细胞的沉默作用. 世界华人消化杂志 2009; 17(20): 2021-2025
Revised: June 16, 2009
Accepted: June 23, 2009
Published online: July 18, 2009
AIM: To construct eukaryotic expression vectors of short hairpin RNA (shRNA) specific for TROP2 gene and observe its silencing effect on TROP2 gene in BGC-823 cells.
METHODS: The eukaryotic expression vectors of short hairpin RNA (shRNA) specific for TROP2 were constructed by ligating pGensil1.1 plasmid (containing human U6 shRNA promoter) to TROP2-specific shRNA target sequences. After the resulting recombinant plasmids were transfected into BGC-823 cells, the cells were screened in medium containing G418 (at a concentration of 400 mg/L) for 24 h to obtain stably transfected cell strain. These stably transfected cells were then divided into W group (untransfected), HK group (transfected with random negative control plasmid), KB group (transfected with empty plasmid), T1 group, T2 group and T3 group. The expression of TROP2 gene in stably transfected cells was determined by real-time PCR and Western blot.
RESULTS: TROP2-specific shRNA target sequences were successfully inserted into eukaryotic expression vector pGensil1.1. The sequences of shRNAs inserted into the recombinant plasmids were identical to those expected. The expression levels of TROP2 mRNA and protein in cells transfected with recombinant plasmids were lower than those in untransfected cells or cells transfected with random negative control plasmid or empty plasmid. Compared with the T1 and T2 groups, TROP2-specific shRNA in the T3 group had the more strong inhibitory effect on the expression of TROP mRNA (8.79 ± 0.23 vs 9.54 ± 0.20, 9.57 ± 0.23) and protein (3.66 ± 0.11 vs 6.46 ± 0.36, 9.31 ± 0.11), and the differences were significant (all P < 0.05).
CONCLUSION: Eukaryotic expression vectors of shRNA specific for TROP2 gene are constructed successfully. The expression of TROP2 mRNA and protein is down-regulated in cells transfected with recombinant plasmids. Our study provides a new approach to the study of the function of TROP2 gene.
- Citation: Dai DF, Chen DY, Shao SH. Short hairpin RNA-mediated silencing of TROP2 gene in gastric cancer BGC-823 cells. Shijie Huaren Xiaohua Zazhi 2009; 17(20): 2021-2025
- URL: https://www.wjgnet.com/1009-3079/full/v17/i20/2021.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v17.i20.2021
RNA干扰是通过核酸酶将双链RNA切割成21-25 nt的干扰RNA(siRNA), 按碱基配对原则特异性识别并切割同源性靶mRNA分子而实现的[1]. TROP2在人类的角膜、胰腺、前列腺等器官中均有表达, 并在正常的滋养层细胞、多层上皮细胞及上皮癌中高度表达[2-5], 他的表达和肿瘤侵袭行为有关[6-9]. 他的生理功能还不清楚, 可能为一种钙信号换能器[10-11]. 目前TROP2与胃癌的关系国内尚未见文献报道. 本研究拟用RNA干扰技术, 构建针对TROP2基因的特异性短发夹环RNA(shRNA)真核表达载体, 获得稳定转染该载体的BGC-823细胞, 实现对TROP2表达的抑制, 为研究TROP2基因功能以及探索基因治疗打下一定的基础.
胃癌BGC-823细胞, 大肠埃希菌DH5α株和含绿色荧光蛋白、耐卡那霉素、抗新霉素Rneo基因及人U6 启动子的质粒pGensil1.1由江苏大学基础医学与医学技术学院病原生物学实验室保存, DMEM培养基为Gibco公司产品, 新生牛血清为杭州四季青产品, pMD18T-vector和Real Time PCR扩增试剂盒购自TaKaRa公司, T4 DNA连接酶、各种限制性内切酶均为晶美公司产品, 质粒提取试剂盒购自Promega公司, LipofectamineTM 2000和TRIzol购自美国Invitrogen公司, 逆转录试剂盒为Toyobo产品, TROP2基因RNAi寡核苷酸, TROP2基因和β-actin基因PCR引物均由上海生物工程公司合成, 鼠抗人TROP2及GAPDHAb和酶标羊抗鼠IgG为Santa Cruze公司产品.
1.2.1 shRNA设计与合成: 针对TROP2基因设计3条特异性的shRNA. 从GenBank中获取TROP2 mRNA的完整序列, 基因序列号为NM_002353, 从目标基因开放阅读框起始密码(ATG)下游75至100碱基位置开始, 寻找"AA"二连序列后的19个碱基序列. 设计shRNA时不针对5'和3'端的非编码区, 分析获得的序列, 选择GC比为40%-55%的靶基因序列作为潜在优选. 使用BLAST将选定的序列和相应的人基因组数据库进行比较. pGensil1.1质粒当中在U6启动子后连接针对TROP2基因的干扰片段, 反义片段以后接TTTTTT以终止转录. 为了便于克隆与鉴定, shRNA 5'端和3'端分别带有Eco31Ⅰ酶切位点, 终止信号后带有SacⅠ的酶切位点, 形成CACC+Sense+Loop+Antisense+终止信号+SacⅠ的结构, 基因的shRNA序列分别选择TROP2外显子448-466位点、1030-1048位点、1102-1120位点(表1). 同时合成目标基因序列为GACTTCATAAGGCGCATGC的随机阴性对照序列.
| shRNA序列 | 碱基序列 |
| pGensil1.1/TROP21 | 正义 5'-CACCGCCACCAACAAGATGACCGTTTCAA |
| GACGACGGTCATCTTGTTGGTGGTTTTTTG-3' | |
| 反义 5'-AGCTCAAAAAACCACCAACAAGATGACCG | |
| TCGTCTTGAAACGGTCATCTTGTTGGTGGC-3' | |
| pGensil1.1/TROP22 | 正义 5'-CACCAGGGCGAGTCTCTATTCCATTCAAG |
| ACGTGGAATAGAGACTCGCCCTTTTTTTG-3' | |
| 反义 5'-AGCTCAAAAAAAGGGCGAGTCTCTATTCC | |
| ACGTCTTGAATGGAATAGAGACTCGCCCT-3' | |
| pGensil1.1/TROP23 | 正义 5'-CACCGCACGCTCATCTATTACCTTTCAAG |
| ACGAGGTAATAGATGAGCGTGCTTTTTTG-3' | |
| 反义 5'-AGCTCAAAAAAGCACGCTCATCTATTACCT | |
| CGTCTTGAAAGGTAATAGATGAGCGTGC-3' |
1.2.2 载体质粒的构建与鉴定: 将pGensil1.1质粒和合成的shRNA用足量的Eco31Ⅰ酶切消化, 低融点10 g/L琼脂糖凝胶电泳后回收大片段; 将上述2个回收片段在T4连接酶作用下, 22℃连接反应过夜; 各取5 μL连接产物转化DH5α, 涂布于含Kana抗性的LB平皿上, 37℃恒温过夜, 从平皿中各挑选3个单克隆菌落于3 mL含Kana抗性的LB培养液中, 37℃, 250 r/min摇床培养过夜; 用质粒纯化试剂盒提取质粒, 并做SacⅠ酶切鉴定; 挑选鉴定正确的克隆进行DNA测序分析.
1.2.3 细胞转染: 在预实验中, 荧光定量PCR法检测人食管癌EC/CUHK1细胞, 肝癌HepG2细胞, 胃癌BGC-823细胞3株细胞系TROP2 mRNA的表达情况, 发现在这3株消化系肿瘤细胞中均有表达, 且以肝癌HepG2细胞表达最低, 胃癌BGC-823细胞表达最高. 选择胃癌BGC-823细胞作为研究对象, 收集同批未转染的胃癌BGC-823细胞作为阴性对照, 同时取随机阴性对照质粒, 空质粒及pGensil1.1/TROP21, pGensil1.1/TROP22及pGensil1.1/TROP23重组体分别转染对数生长期BGC-823细胞, 转染方法参照LipofectamineTM 2000转染试剂操作说明进行, 转染前1 d将2.5×105个细胞接种于6孔培养板中, 次日达到80%汇合, 质粒与脂质体按1:4形成DNA与脂质体混合物逐滴加入并混匀, 添加无血清、无抗生素DMEM培养基至总体积为2 mL, 培养5 h后换DMEM完全培养液继续培养. 采用盲法随机计数显微镜下10个视野的阳性细胞数与总细胞数, 发绿色荧光的细胞数与总细胞数之比即为转染效率. 根据预实验的结果, 转染24 h后用G418(浓度400 mg/L)筛选, 待细胞稳定后收集, 分别命名为W组(未处理组), HK组(随机阴性对照质粒组), KB组(空质粒组), T1组, T2组, T3组.
1.2.4 Real Time PCR分析: 利用Primer 5.0软件设计TROP2基因引物, 由上海生物工程公司合成. 上游引物序列为: 5'-CCT CAT CGC CGT CAT CGT-3'; 下游引物序列为: 5'-CGG TTC CTT TCT CAA CTC CC-3', 扩增片段长度136 bp. β-actin上游引物序列为: 5'-CAC GAA ACT ACC TTC AAC TCC-3'; 下游引物序列为: 5'-CAT ACT CCT GCT TGC TGA TC-3', 扩增片段长度262 bp. 提取1 μg总RNA逆转录后行PCR反应. 将以上2种扩增片段分别与pMD-18T载体连接, 筛选后得到重组标准品质粒进行酶切、PCR和测序鉴定, 质粒抽提得到的TROP2和β-actin质粒用作实时PCR标准品. 采用双标准曲线法, 用Stratagene Mx3000P荧光定量PCR分析仪进行PCR反应, 扩增条件为: 95℃ 10 s; 95℃ 5 s; 60℃20 s, 40个循环. 每个循环后采集荧光生成扩增曲线, 并在反应后生成熔解曲线, 采用Mx3000P软件进行数据分析, 依据TROP2拷贝数/β-actin拷贝数比值提示mRNA表达水平, 进行统计学分析.
1.2.5 Western blot检测TROP2蛋白的表达: 培养细胞蛋白质样品的制备: 收集各组细胞, 用细胞裂解液和蛋白保护液裂解细胞, 经涡旋离心后取上清. 制备SDS-PAGE凝胶, 并做预电泳; 每孔加样品24 μL, Marker加10 μL, 电泳; 染色和脱色, 切胶, 电转; 封闭, 加一抗孵育, TBST洗后加酶标二抗; TBST洗后, 显影, 定影; 用GAPDH作内参照验证蛋白的含量.
统计学处理 实验所得数据使用SPSS13.0统计分析软件进行方差分析, 比较各组间的差异, P<0.05为有统计学意义.
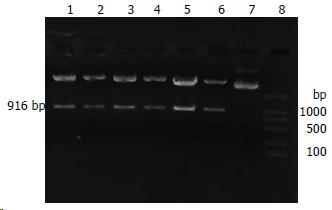
完成构建的载体质粒经SacⅠ酶切, 在插入的目的基因片段里, 我们分别设计了一个SacⅠ的酶切位点, 而质粒pGenesil1.1本来就有一个SacⅠ的酶切位点, 如若插入正确, 质粒就能被SacⅠ酶切出1条约916 bp的DNA小带. 经电泳分析, 构建质粒均被切出1条916 bp大小的DNA带(图1), 说明重组质粒即为克隆正确的质粒.
经酶切后, 挑取鉴定正确的克隆进行测序分析, 结果显示, 3个重组质粒shRNA编码序列与设计的片段完全一致, 说明重组质粒构建成功.
对TROP2、β-actin质粒稀释浓度的对数和相应的拷贝数进行直线回归分析. 模板的融解曲线显示为单峰, 且峰值单一, 说明产物特异. T1组、T2组、T3组较W组、HK组、KB组都统计学意义(P<0.05), 但以T3组差异最显著, T1、T2组之间无统计学意义(表2).
T1组、T2组和T3组与随机阴性对照组、空质粒组, 对照组相比蛋白条带减弱. T1组和T3组与对照组比较有统计学意义(P<0.05), 以T3组蛋白表达下降最明显(表2).
荧光实时定量PCR和Western blot检测显示, T3组干扰质粒对TROP2基因mRNA和蛋白表达水平抑制作用最明显. 与未转染细胞对照组相比, T3组mRNA表达抑制率为29.9%, 蛋白表达抑制率为62.0%, 差异具统计学意义(P<0.05, 图2).
TROP2亦称为TACSTD2、M1S1、GA733-1、EGP-1, 定位在1号染色体短臂D1S2890和D1S2801之间一个长约2.6 cm的区域上[12]. 他是GA733基因家族的其中一员, 是无内含子的基因, 所编码的产物为含有323个氨基酸、35.7 kDa的蛋白质, 被认为是一种癌症相关抗原. 他包含一个上皮生长因子样的重复区, 一个甲状腺球蛋白样重复区, 其分子羟基端含有丝氨酸和酪氨酸位点及PIP-2结合区域, 其丝氨酸残端为激酶C所磷酸化. 早期人们发现TROP2基因突变与胶样滴状角膜营养不良(gelatinous drop-like corneal dystrophy, GDLD)有关, GDLD是一种罕见的常染色体隐性遗传病, 可导致视力严重障碍甚至失明[13-15]. 2008年Wang et al[16]用RNA干扰技术, 向我们展示了TROP2的表达与结肠癌细胞的发生及侵袭密切相关, 干扰后这两种特性均受到抑制, 同时针对TROP2胞外域的抗体能降低肿瘤细胞的侵袭性. 也有研究认为TROP2是独立的肠型胃癌复发预测因子, 因为他分布广泛有可能成为新的治疗靶位[17]. 以上提示我们利用抑制TROP2基因表达的方法治疗结肠癌和胃癌可能具有较好的应用前景.
胃癌是最常见的恶性肿瘤之一, 其发生发展是以癌基因的激活与抑癌基因的失活为基础的多步骤、多阶段过程, 是遗传和环境因素相互作用的结果. 因此寻找阻断癌基因激活的siRNA有可能抑制胃癌的发生. 由于RNAi技术可以利用siRNA或shRNA表达载体快速、经济、简便的以序列特异方式剔除目的基因表达, 所以现在已经成为探索基因功能的重要研究手段, 并在肿瘤基因治疗方面显示出了广阔的应用背景[18]. siRNA在体内的半衰期短, 对基因的抑制作用较短暂[19], 而shRNA表达载体可以在体内持续产生siRNA, 并且用含有hU6启动子的载体对靶基因的沉默效果较好, 沉默时间较久[20], 同时以9个核苷酸序列环为发夹的shRNA产生的基因干扰效果最明显[21], 因此我们采用构建shRNA表达质粒来沉默TROP2基因. 将siRNA转入细胞是RNA干扰关键技术之一, 常规方法有: 脂质体转染、电穿孔法、微注射法[22-24]. 本实验用脂质体LipfectamineTM 2000转染试剂介导的转染, 操作简单, 不需要复杂的设备, 转染效率较高. 另T3组抑制效果较T1、T2组更为明显, 提示RNA干扰技术抑制基因表达的效果与靶位点的选择密切相关.
总之, 本实验中我们针对TROP2基因分别设计了3条shRNA, 从中筛选出最有效的短链片段, 获得稳定转染该有效片段的BGC-823细胞, 其mRNA和蛋白表达抑制率为29.9%和62.0%, 将为进一步的基础和临床研究提供更多的依据.
胃癌是最常见的恶性肿瘤之一, 5年生存率仍在30%左右, 寻找新的治疗方法已成为目前胃癌研究的重要问题. RNA干扰被广泛应用于各种肿瘤研究, 能高效、特异地阻断细胞外源或内源性靶基因的表达, 其在胃癌治疗方面的研究具有重要的现实意义.
王晓艳, 副教授, 湖南长沙中南大学湘雅三医院消化内科
通过抑制TROP2基因表达以达到治疗胃癌的目的是胃癌基因治疗领域的新思路.
国外已有学者对TROP2在结肠癌、食管癌、乳腺癌等恶性肿瘤中的作用进行了研究, 但关于其在胃癌发生发展中发挥的作用研究尚少.
本研究利用RNA干扰的方法, 通过构建shRNA表达质粒下调BGC-823细胞的TROP2 mRNA和蛋白水平.
本研究构建并筛选出靶向TROP2基因的shRNA重组质粒, 经检测证实对TROP2基因有明显的干扰作用, 为进一步观察TROP2基因在胃癌中作用的研究打下基础.
本研究选题有一定的科研价值, 实验方法先进, 具有一定的参考价值.
编辑: 李军亮 电编:何基才
| 1. | Yu JY, DeRuiter SL, Turner DL. RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells. Proc Natl Acad Sci U S A. 2002;99:6047-6052. [PubMed] [DOI] |
| 2. | Birkenkamp-Demtroder K, Olesen SH, Sørensen FB, Laurberg S, Laiho P, Aaltonen LA, Orntoft TF. Differential gene expression in colon cancer of the caecum versus the sigmoid and rectosigmoid. Gut. 2005;54:374-384. [PubMed] [DOI] |
| 3. | Nakashima K, Shimada H, Ochiai T, Kuboshima M, Kuroiwa N, Okazumi S, Matsubara H, Nomura F, Takiguchi M, Hiwasa T. Serological identification of TROP2 by recombinant cDNA expression cloning using sera of patients with esophageal squamous cell carcinoma. Int J Cancer. 2004;112:1029-1035. [PubMed] [DOI] |
| 4. | Santin AD, Zhan F, Bellone S, Palmieri M, Cane S, Bignotti E, Anfossi S, Gokden M, Dunn D, Roman JJ. Gene expression profiles in primary ovarian serous papillary tumors and normal ovarian epithelium: identification of candidate molecular markers for ovarian cancer diagnosis and therapy. Int J Cancer. 2004;112:14-25. [PubMed] [DOI] |
| 5. | Iacobuzio-Donahue CA, Maitra A, Shen-Ong GL, van Heek T, Ashfaq R, Meyer R, Walter K, Berg K, Hollingsworth MA, Cameron JL. Discovery of novel tumor markers of pancreatic cancer using global gene expression technology. Am J Pathol. 2002;160:1239-1249. [PubMed] |
| 6. | Huang E, Cheng SH, Dressman H, Pittman J, Tsou MH, Horng CF, Bild A, Iversen ES, Liao M, Chen CM. Gene expression predictors of breast cancer outcomes. Lancet. 2003;361:1590-1596. [PubMed] [DOI] |
| 7. | Coldren CD, Helfrich BA, Witta SE, Sugita M, Lapadat R, Zeng C, Barón A, Franklin WA, Hirsch FR, Geraci MW. Baseline gene expression predicts sensitivity to gefitinib in non-small cell lung cancer cell lines. Mol Cancer Res. 2006;4:521-528. [PubMed] [DOI] |
| 8. | Ohmachi T, Tanaka F, Mimori K, Inoue H, Yanaga K, Mori M. Clinical significance of TROP2 expression in colorectal cancer. Clin Cancer Res. 2006;12:3057-3063. [PubMed] [DOI] |
| 9. | Shimada A, Kano J, Ishiyama T, Okubo C, Iijima T, Morishita Y, Minami Y, Inadome Y, Shu Y, Sugita S. Establishment of an immortalized cell line from a precancerous lesion of lung adenocarcinoma, and genes highly expressed in the early stages of lung adenocarcinoma development. Cancer Sci. 2005;96:668-675. [PubMed] [DOI] |
| 10. | Ripani E, Sacchetti A, Corda D, Alberti S. Human Trop-2 is a tumor-associated calcium signal transducer. Int J Cancer. 1998;76:671-676. [PubMed] [DOI] |
| 11. | Kinoshita S, Adachi W, Sotozono C, Nishida K, Yokoi N, Quantock AJ, Okubo K. Characteristics of the human ocular surface epithelium. Prog Retin Eye Res. 2001;20:639-673. [PubMed] [DOI] |
| 12. | Tsujikawa M, Kurahashi H, Tanaka T, Nishida K, Shimomura Y, Tano Y, Nakamura Y. Identification of the gene responsible for gelatinous drop-like corneal dystrophy. Nat Genet. 1999;21:420-423. [PubMed] [DOI] |
| 13. | Zhang B, Yao YF, Zhou P. Two novel mutations identified in two Chinese gelatinous drop-like corneal dystrophy families. Mol Vis. 2007;13:988-992. [PubMed] |
| 14. | Markoff A, Bogdanova N, Uhlig CE, Groppe M, Horst J, Kennerknecht I. A novel TACSTD2 gene mutation in a Turkish family with a gelatinous drop-like corneal dystrophy. Mol Vis. 2006;12:1473-1476. [PubMed] |
| 15. | Alavi A, Elahi E, Tehrani MH, Amoli FA, Javadi MA, Rafati N, Chiani M, Banihosseini SS, Bayat B, Kalhor R. Four mutations (three novel, one founder) in TACSTD2 among Iranian GDLD patients. Invest Ophthalmol Vis Sci. 2007;48:4490-4497. [PubMed] [DOI] |
| 16. | Wang J, Day R, Dong Y, Weintraub SJ, Michel L. Identification of Trop-2 as an oncogene and an attractive therapeutic target in colon cancers. Mol Cancer Ther. 2008;7:280-285. [PubMed] [DOI] |
| 17. | Mühlmann G, Spizzo G, Gostner J, Zitt M, Maier H, Moser P, Gastl G, Zitt M, Müller HM, Margreiter R. TROP2 expression as prognostic marker for gastric carcinoma. J Clin Pathol. 2009;62:152-158. [PubMed] [DOI] |
| 18. | Shankar P, Manjunath N, Lieberman J. The prospect of silencing disease using RNA interference. JAMA. 2005;293:1367-1373. [PubMed] [DOI] |
| 19. | Holen T, Amarzguioui M, Wiiger MT, Babaie E, Prydz H. Positional effects of short interfering RNAs targeting the human coagulation trigger Tissue Factor. Nucleic Acids Res. 2002;30:1757-1766. [PubMed] [DOI] |
| 20. | Mäkinen PI, Koponen JK, Kärkkäinen AM, Malm TM, Pulkkinen KH, Koistinaho J, Turunen MP, Ylä-Herttuala S. Stable RNA interference: comparison of U6 and H1 promoters in endothelial cells and in mouse brain. J Gene Med. 2006;8:433-441. [PubMed] [DOI] |
| 21. | Brummelkamp TR, Bernards R, Agami R. A system for stable expression of short interfering RNAs in mammalian cells. Science. 2002;296:550-553. [PubMed] [DOI] |
| 22. | Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes Dev. 2001;15:188-200. [PubMed] [DOI] |