修回日期: 2008-03-19
接受日期: 2008-05-10
在线出版日期: 2008-05-18
目的: 通过基因芯片技术检测亚硝胺诱导食管癌变过程中, 食管组织基因表达的差异, 分析不同阶段基因表达的特点以及基因差异表达与DNA甲基化的相关性.
方法: 小鼠随机分为2组, A组(对照组)40只, 灌喂蒸馏水; B组(实验组)70只, 灌喂诱导混合物(20 g/L亚硝酸钠+200 g/L N, N-二甲基苄胺). 在诱导时间4、8、20 wk时, 提取食管组织RNA, 反转录合成cDNA, 并与芯片杂交, 对芯片结果进行分析, 比较诱导进程中不同组别的表达差异.
结果: 亚硝胺诱导早期病变进程中原癌基因表达的变化呈现整体逐步上升的趋势, 且阶段表达特性显著, 但多数抑癌基因未表现出明显变化. 甲基化酶基因在4 wk无明显改变, 但在8、20 wk稍有升高, 去甲基化酶基因中Mbd2b从8 wk开始上调, 但Gadd45a从4 wk就已诱导上调, 8、20 wk依然明显上调. 未发现明显改变的组蛋白乙酰化酶基因.
结论: 在亚硝胺诱导食管癌变过程中, 大量基因的上调表达可能与早期基因组的低甲基化密切相关, Gadd45a可能参与了诱导早期的脱甲基作用.
引文著录: 吴强强, 魏亚宁, 张素珍, 舒青. 亚硝胺诱导食管癌变早期基因差异表达与表观遗传修饰的关系. 世界华人消化杂志 2008; 16(14): 1487-1492
Revised: March 19, 2008
Accepted: May 10, 2008
Published online: May 18, 2008
AIM: To detect the differentially expressed genes in the process of esophageal carcinoma induced by nitrosamine, and analyze the property of gene expression at different stages, especially the relationship between differentially expressed genes and DNA methylation.
METHODS: One hundred and ten mice were randomly divided into group A (n = 40) and B (n = 70). The mice in group A were fed with distilled water, while those in group B were fed with carcinoma-inducing mixture containing 20 g/L sodium nitrite and 200 g/L N, N-dimethyl benzyl amine. At the 4, 8, and 20 wk of induction stages, the total RNA from esophageal tissues was isolated and reversely transcripted, and then hybridization with gene-chip was performed. The differentially expressed genes of the two groups were analyzed.
RESULTS: During the process of induction, the number of up-regulated oncogenes increased in a step-by-step fashion in group B as compared with that in group A, and the phase-specificity of oncogenes was observed. But most antioncogenes did not change remarkably. Most genes of DNA methylation did not change at 4 wk, but were up-regulated at 8 and 20 wk. The DNA demethylation gene Mdb2b was up-regulated from the 8 wk, but Gadd45a was up-regulated from the 4 wk and maintained a high level at 8 and 20 wk. Genes of histone acetylation, which were changed obviously, were not found.
CONCLUSION: During the process of nitrosamine-induced esophageal carcinoma, up-regulation of numerous genes may associate with the demethylation of genomic DNA at the early stage. Gadd45a may be involved in the demethylation during the early weeks.
- Citation: Wu QQ, Wei YN, Zhang SZ, Shu Q. Relationship between differentially expressed genes and epigenetic modification during the early stage of esophageal carcinoma induced by nitrosamine. Shijie Huaren Xiaohua Zazhi 2008; 16(14): 1487-1492
- URL: https://www.wjgnet.com/1009-3079/full/v16/i14/1487.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v16.i14.1487
食管癌(esophageal carcinoma, EC) 是常见的消化道肿瘤之一, 在癌症致死率中位居第6位[1]. 2007年全球新增食管癌病例529 000例, 有442 000死亡[2]. 食管癌的诱因有多种, 其中化学致癌物质亚硝胺的诱导作用已经被证实, 通常认为内源性和外源性的亚硝胺经机体代谢产生的中间代谢物能够引起基因组DNA的损伤[3-5], 损伤的积累最终导致了细胞遗传的不稳定性, 从而引起癌变或肿瘤的形成. 基因芯片技术可快速高效地检测大量基因的表达情况, 是一种检测基因差异的重要方法, 已被广泛地应用于肿瘤发生的分子生物学研究领域. 本研究目的通过使用亚硝胺诱导小鼠食道癌变发生来模拟人类食管癌的发生过程, 通过基因芯片技术检测该进程中食管组织基因表达的变化趋势, 分析早期不同阶段癌基因的表达特点, 特别是表观遗传修饰相关的基因, 试图为研究亚硝胺诱导食管癌变早期进程中的机制提供新的线索.
♂BALB/c二级健康小鼠110只, 20-25 g, 由中国人民解放军第四军医大学实验动物中心提供; 亚硝酸钠(NaNO2)、N, N-二甲基苄胺(N, N-Dimethylbenzylamine), 购自中国人民解放军第四军医大学试剂中心.
将小鼠随机分为2组, A组(对照组)40只, 灌喂蒸馏水; B组(实验组)70只, 灌喂诱导混合物(20 g/L亚硝酸钠+200 g/L N, N-二甲基苄胺), 剂量为50 μL/只, 每日1次. 在第4、8、20周时, 分别取两组小鼠各5只, 解剖小鼠后取等量食管组织样本用作芯片分析. 将待检样本匀浆后用TRIzol法提取RNA, 检测后合成cDNA, 并进行芯片检测分析. 检测芯片为Agilent小鼠全基因组芯片(G4112A), 由陕西北美基因公司提供, 包含44290个基因位点. 芯片杂交过程及信号扫描处理由北美基因公司检测中心完成, 芯片结果数据为5例样本的均值. 基因芯片结果经归一化处理后, 平均信号强度之比(实验组/对照组)大于2.0倍的基因定义为上调基因, 小于0.5倍的基因定义为下调基因, 0.5-2.0为未发生显著变化.
在所检测的诱导时间点4、8、20 wk, 与对照组相比诱导表达上调的基因数目分别是3556、15 573和23 889, 而诱导表达下调的基因数目分别是602、1275和618.
在诱导4、8、20 wk共同上调的基因数目为1582, 共同下调的基因数目仅为160. 从基因芯片的结果中筛选出细胞癌基因136个, 其中4 wk上调表达的26个, 8 wk上调表达的97个, 20 wk上调表达的130个, 可见癌基因上调表达的特点是随着诱导时间的延长, 基因上调数目明显增多; 另一个特点是基因表达的时段差异明显, 即有些基因仅在某一特定时间表达上调, 如Mup1、Alb1仅在4 wk检测出表达上调, 在8 wk和20 wk无变化; Rras、Ski等仅在8 wk检测出高表达, 而在4 wk和20 wk却无变化; Mos、Kit等仅在20 wk高表达, 而在4和8 wk无变化. 还有一些基因从4 wk开始上调后, 在8、20 wk时依然维持一个较高的水平, 如Crk、Jun、Myc等; Maf(Maff、Mafk、Mafg)家族和Ras(Kras、Mras、Nras)家族成员多是从8 wk开始上调, 20 wk时持续上调. 也有的癌基因在检测的3个时间点未发现变化, 如Mpl(表1). 有的基因在4、8、20 wk都表现出了明显的变化, 在不同时段差异甚至可达50倍以上, 如钙调蛋白家族的Calm2、Calm4, 铁蛋白Fth, 钙结合蛋白Cab39, Mafb等(表2). 从基因芯片的结果中筛选出相关的抑癌基因, 发现多数抑癌基因在4、8 wk未见变化而少数仅在20 wk开始出现高表达如, 如Apc、Dcc等; 一些抑癌基因在3个时间点都未检测出差异, 如Wtap、Ptch2(表3).
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Akt3 | NM_011785 | - | - | - |
| Met | NM_008591 | - | - | - |
| Otc | NM_008769 | - | - | - |
| Alb1 | NM_009654 | 9.77 | - | - |
| Mup1 | NM_031188 | 21.88 | - | - |
| Apoh | NM_013475 | 6.59 | - | - |
| Rras | NM_009101 | - | 2.72 | - |
| Ski | NM_011385 | - | 4.86 | - |
| Frat2 | NM_177603 | - | 2.59 | - |
| Ets1 | NM_011808 | - | - | 7.54 |
| Fgr | A130081E08 | - | - | 2.55 |
| Kit | NM_021099 | - | - | 2.71 |
| Mafa | NM_194350 | - | - | 2.8 |
| Mos | NM_020021 | - | - | 4.62 |
| Mybl2 | NM_008652 | - | - | 2.39 |
| Rel | NM_009044 | - | - | 5.8 |
| Bcl2 | 6030439K11 | - | - | 12.46 |
| Braf | AK036798 | - | - | 5.46 |
| Cdk2 | NM_183417 | - | - | 3.00 |
| Dntt | NM_009345 | - | - | 29.08 |
| Plg | NM_008877 | - | - | 6.57 |
| Sod1 | AK080908 | - | - | 109.38 |
| Crkl | NM_007764 | - | 2.31 | 3.80 |
| Fos | NM_010234 | - | 15.84 | 10.52 |
| Maff | NM_010755 | - | 32.53 | 22.96 |
| Mafk | NM_010757 | - | 14.24 | 11.20 |
| Mafg | NM_010756 | - | 2.17 | 3.11 |
| Myb | NM_033597 | - | 9.42 | 18.64 |
| Araf | 1200013E08 | - | 8.40 | 14.80 |
| Kras2 | NM_021284 | - | 13.77 | 55.81 |
| Mras | NM_008624 | - | 16.00 | 16.46 |
| Nras | NM_010937 | - | 9.83 | 14.42 |
| Src | NM_009271 | - | 2.40 | 3.46 |
| Yes | NM_009535 | - | 2.70 | 10.42 |
| Abl1 | NM_009594 | - | 19.13 | 12.46 |
| Crk | NM_133656 | 2.10 | 38.40 | 57.81 |
| Junb | NM_008416 | 3.56 | 16.49 | 83.45 |
| Jun | NM_010591 | 6.27 | 169.95 | 94.38 |
| Myc | NM_010849 | 2.34 | 55.93 | 37.98 |
| Raf1 | NM_029780 | 2.36 | 85.86 | 74.91 |
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Rab5a | NM_025887 | 2.59 | 56.35 | 102.30 |
| Rab11a | NM_017382 | 2.32 | 55.23 | 72.80 |
| Raf1 | NM_029780 | 2.36 | 85.86 | 74.91 |
| Hras1 | NM_008284 | 4.53 | 89.70 | 87.69 |
| Rab6 | NM_024287 | 4.25 | 72.04 | 101.41 |
| Rab14 | NM_026697 | 11.70 | 346.95 | 248.19 |
| Rab7 | NM_009005 | 4.56 | 193.14 | 258.92 |
| Mafb | NM_010658 | 0.00 | 0.00 | 990.00 |
| Rab3d | 6720486C03 | 0.00 | 20.76 | 14.86 |
| Rab2 | NM_021518 | 0.00 | 40.28 | 58.38 |
| Vav3 | NM_146139 | 0.00 | 28.00 | 42.46 |
| Rab34 | 6430710G15 | 0.00 | 30.48 | 32.74 |
| Rab10 | NM_016676 | 0.00 | 31.33 | 40.31 |
| Rab8a | NM_023126 | 0.00 | 36.00 | 39.76 |
| Rab11b | NM_008997 | 0.00 | 59.47 | 33.87 |
| Akt1 | NM_009652 | 0.00 | 65.78 | 45.00 |
| Rala | NM_019491 | 2.18 | 70.56 | 100.05 |
| Hras1 | NM_008284 | 4.53 | 89.70 | 87.69 |
| Calm4 | NM_020036 | 3.49 | 548.92 | 784.20 |
| Calm2 | NM_007589 | 11.36 | 472.14 | 847.66 |
| Cycs | NM_007808 | 17.10 | 169.20 | 336.39 |
| Mt4 | NM_008631 | 7.69 | 87.61 | 77.89 |
| Snx1 | NM_019727 | 4.48 | 76.10 | 139.85 |
| Ccnh | 1810059M17 | 6.32 | 67.67 | 102.66 |
| Psmc6 | NM_025959 | 22.00 | 274.50 | 1134.35 |
| Eif2s2 | 2700038J22 | 24.80 | 393.75 | 1761.23 |
| Creg | 1600000E17 | 22.00 | 1457.10 | 1430.31 |
| Pts | 0610012K15 | 0.00 | 392.40 | 558.00 |
| Fth | NM_010239 | 14.24 | 807.34 | 319.01 |
| Cab39 | NM_133781 | 2.89 | 245.9 0 | 115.46 |
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Brca1 | NM_009764 | 0.00 | 0.00 | 3.31 |
| Brca2 | NM_009765 | 0.00 | 6.64 | 15.92 |
| Pten | NM_008960 | 0.00 | 36.38 | 38.08 |
| Apc | NM_007462 | 0.00 | 0.00 | 3.05 |
| Ptch1 | NM_008957 | 0.00 | 0.00 | 2.85 |
| Dcc | NM_007831 | 0.00 | 0.00 | 8.11 |
分析了DNA甲基化相关基因的表达变化情况, 发现DNA甲基化酶Dnmt1、Dnmt2基因在4 wk无明显改变, 但在8、20 wk稍有升高. Dnmt3a、Dnmt3b基因在诱导的3个时段均无明显变化(表4).
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Dnmt1 | NM_010066 | 0.00 | 5.65 | 6.80 |
| Dnmt2 | NM_010067 | 0.00 | 2.11 | 4.15 |
| Dnmt3a | G431002P10 | 0.00 | 0.00 | 0.00 |
| Dnmt3b | NM_010068 | 0.00 | 0.00 | 0.00 |
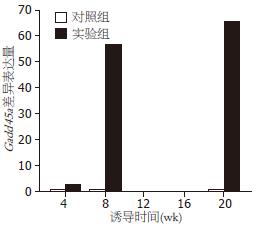
另一方面也分析了DNA脱甲基相关蛋白基因的表达变化情况, 由于目前在哺乳动物细胞中发现的DNA脱甲基酶有限, 其中Mbd2(Mbd2b)是一种具有甲基-CpG结合结构域(methyl CpG-binding domain, MBD)的蛋白分子, 具有DNA脱甲基作用[6]. 芯片分析结果显示Mbd2基因在4 wk无明显改变, 但在8、20 wk稍有升高, 分别为对照组的2.29和4.00倍. Gadd45a蛋白是最近证实的具有主动DNA脱甲基活性的蛋白分子[7], 其基因表达改变在诱导4 wk就已经出现上调表达, 与对照相比升高2.91倍, 在8、20 wk Gadd45a依然持续表达上调, 分别上升56.70和65.47倍(表5, 图1).
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Mbd2 | NM_010773 | 0.00 | 2.29 | 4.00 |
| Gadd45a | NM_007836 | 2.91 | 56.70 | 65.47 |
芯片分析结果并未筛选出明显改变的组蛋白乙酰化酶(histone acetyltransferases, HAT)相关基因, 而组蛋白脱乙酰酶(histone deacetylase, HDAC)相关基因在诱导4 wk时基本无变化, 多数从8 wk开始变化, 在20 wk时大多数呈现上调趋势, 其中只有Hdac11在3个时段都无变化(表6).
| 基因 | GenBank ID | Signal Log Ratio | ||
| 4 wk | 8 wk | 20 wk | ||
| Hdac2 | NM_008229 | 0.00 | 0.00 | 15.23 |
| Hdac10 | NM_199198 | 0.00 | 20.14 | 25.79 |
| Hdac7a | NM_019572 | 0.00 | 0.00 | 3.76 |
| Hdac5 | NM_010412 | 0.00 | 40.74 | 21.21 |
| Hdac3 | NM_010411 | 0.00 | 5.82 | 6.65 |
| Hdac1 | NM_008228 | 0.00 | 4.32 | 5.17 |
| Hdac11 | NM_144919 | 0.00 | 0.00 | 0.00 |
| Hdac6 | NM_010413 | 0.00 | 0.00 | 2.58 |
通过前期魏亚宁 et al[8]的研究结果发现亚硝胺诱导食管病变进程中早期基因组DNA甲基化水平呈现逐渐下降趋势, 到第4周降到最低, 随后直到第48周处于稳定状态. 而芯片分析结果显示出随着诱导时间的延长基因表达呈现整体上升的趋势, 实验研究证实DNA甲基化与基因转录活性相关[9-12], 通常DNA甲基化与基因沉默密切相关, 但甲基化只是基因沉默的结果并非原因. 而低甲基化往往与基因表达活跃相关. 因此推测这种癌基因随着诱导时间整体水平的表达上调有可能与诱导早期的基因组DNA甲基化水平逐渐下降密切相关.
通过对Mbd2(Mbd2b)和Gadd45a这两种目前发现的DNA脱甲基相关蛋白的表达分析发现Mbd2基因表达改变滞后于基因组DNA甲基化水平的下降, 所以推测早期DNA低甲基化可能不是由Mbd2介导的脱甲基作用实现的. 而Gadd45a表达的改变在诱导早期4 wk就已经出现, 并且改变的时段发生于基因组DNA甲基化水平下降之前. 8、20 wk Gadd45a持续表达上调, 这很可能与基因组DNA甲基化水平维持一个较低的水平有关[8].
Gadd45a是一个生长阻滞和DNA损伤基因, 是p53下游基因, 可以通过p53依赖及非依赖两条途径被诱导表达增高, 参与细胞周期监测点、细胞凋亡、DNA损伤修复以及信号传导等重要细胞生命活动的调节, 在维持基因组稳定性中发挥重要的功能[13]. 人们对其在维持基因组稳定性中的功能进行了大量的研究. 发现多种应激因素例如紫外辐射、电离辐射、甲磺酸盐、亚砷酸盐等[14-18], 可以通过不同的传导通路显著诱导Gadd45a的表达上调. 而诱导表达增加的Gadd45a通过多种方式参与对细胞基因组稳定性的调节[19], 而近来发现的可通过修复介导的脱甲基作用, 说明Gadd45a在表观遗传修饰方面参与细胞功能调节, 但具体的调节机制还知之甚少, 特别是在亚硝胺的早期诱导过程中Gadd45a是通过直接还是间接途径与基因组DNA的低甲基化发生关系以及这一过程中涉及的分子机制仍然需要更多的试验证据证实.
芯片分析显示8、20 wk时甲基化酶Dnmt1、Dnmt2表达上调, 但基因组DNA甲基化水平却在一个较低水平, Dnmt1主要功能是维持DNA的CpG甲基化, 其活性的高低是影响DNA甲基化水平的重要因素, Dnmt3a、Dnmt3b基因在诱导的三个时段均无明显变化, 而DNMT3主要参与DNA的创建性甲基化[20-21]. 这一矛盾说明这种基因组DNA甲基化水平逐渐下降现象可能不是通过下调或抑制DNA甲基化酶基因的作用实现的(表4). 而Dnmt1、Dnmt2的上调提示可能他们参与诱导抑癌基因失活.
芯片分析未发现表达改变的组蛋白乙酰化酶基因, 组蛋白乙酰化可使染色质结构重塑而激活基因转录[22-23], 说明亚硝胺诱导的基因表达升高可能不是通过组蛋白乙酰化实现的. 芯片分析还发现脱乙酰酶却在不同阶段表达上调, 组蛋白去乙酰化可导致相应基因的转录抑制[24-26], 推测这种表达升高可能与后期癌变过程中一些抑癌基因的失活相关.
肿瘤或者癌变的发生发展是一个多因素、多步骤的过程, 这一过程涉及了众多基因表达的改变. 如原癌基因的激活以及抑癌基因的失活, 凋亡基因与增殖基因的失衡等. 亚硝胺诱导食管癌变进程中基因差异表达的情况通过基因芯片分析使我们有了一个初步的认识, 这一进程中基因表达的变化呈现上升的趋势这一结果可能是由于Gadd45a介导的脱甲基作用引起的基因转录活化. 随着诱导时间的延长, 基因表达呈现的时段特点说明了不同的阶段存在不同的基因在诱导癌变过程中起作用, 大量基因的异常表达可能在后期引起细胞发生表型改变.
有越来越多的试验证据证明肿瘤或癌变过程涉及了表观遗传修饰的变化[27-29], 如Ebert et al[30]通过对胃癌APC基因分析发现基因没有发生突变, 用甲基化特异性聚合酶链反应(MS-PCR)对胃癌标本和细胞株进行分析, 发现82%的原发性胃癌和98%胃癌患者的癌旁胃黏膜和10个胃癌细胞株APC启动子1A呈高甲基化, 而启动子1B却未发生甲基化. p16基因高甲基化与散发性大肠癌中有相关性, 而对50例结肠癌研究则发现了存在CpG岛异常甲基化状态[31]. Issa et al[32]证实了CpG岛甲基化表型(CpG island methylator phenotype, CIMP)的存在, 其特征是同时存在的多种基因具有肿瘤特异性的CpG岛甲基化. 研究还发现在胃、膀胱、非白血性白血病、大肠、肺等各种组织类型的肿瘤中发现了甲基化酶DNMT活性水平的提高.
我们利用基因芯片分析发现了亚硝胺诱导过程基因差异表达现象与基因表观遗传修饰的变化相关, 推测表观修饰的改变导致了细胞基因组的不稳定性产生, 基因的异常表达在后期引起细胞发生表型改变, 并逐渐向恶变细胞过渡, 但具体的分子机制及脱甲基酶Gadd45a的作用途径还有待更深入的研究.
根据美国癌症协会的统计2007年全球新增食管癌病例529 000例, 死亡442 000例, 其中85%的病例发生于发展中国家. 食管癌的诱因很多, 包括生活习惯, 饮食卫生以及环境中的致癌物. 通常癌症的发生涉及大量基因表达的改变, 如癌基因的激活与抑癌基因的抑制, 而基因表达改变又与DNA甲基化等表观遗传修饰的变化相关, 因此对癌症形成过程中表观遗传水平改变的研究可以为阐明癌症的发病机制提供线索.
刘平, 教授, 上海中医药大学
亚硝胺诱导进程中, 食管组织基因差异表达明显. 这可能与甲基化基因失活或脱甲基化酶相关基因的活化有关, 也有可能是两者共同作用的结果.
魏亚宁 et al发现亚硝胺诱导的食管癌变过程存在基因组DNA甲基化水平逐渐降低的现象.
本研究利用基因芯片技术分析了亚硝胺诱导食管癌变进程中基因表达的差异情况, 并着重对表观遗传修饰相关基因的变化进行分析, 以期发现真正在表观遗传水平起作用的分子.
通过对食管癌形成过程中基因表达差异的分析和表观遗传修饰对差异表达基因的作用, 可以为食管癌的形成机制和早期诊断提供依据.
表观遗传学(Epi- genetics): 是研究表观遗传变异的遗传学学科. 表观遗传修饰是指并非由DNA序列改变导致的可遗传的基因表达水平的变化, 不符合孟德尔遗传规律的核内遗传, 是环境因素与细胞的遗传物质相互作用的结果. 表观遗传调控主要是通过DNA甲基化和组蛋白修饰控制细胞在适当的时间、空间位置表达适当的基因, 从而控制细胞的增殖和分化, 维持机体的稳定, 表观遗传调控属于转录前水平以染色质为基础的基因表达调控. 广义上, DNA甲基化、基因沉默、基因组印记、染色质重塑、组蛋白共价修饰、X染色体失活等均可归为表观遗传的范畴. 例如现在认为基因完全相同的同卵孪生出现个体差异的现象被认为可能和表观遗传修饰有关.
本研究选题较为新颖, 设计合理, 研究结果可信; 写作条理清晰, 语言较为流畅, 反映了当前国内外食管癌的研究水平, 具有发表的价值.
编辑: 程剑侠 电编:何基才
| 1. | Russo A, Franceschi S. The epidemiology of esophageal cancer. Ann Ist Super Sanita. 1996;32:65-72. [PubMed] |
| 2. | Garcia M, Jemal A, Ward EM, Center MM, Hao Y, Siegel RL, Thun MJ. Global Cancer Facts & Figures 2007. Atlanta, GA: American Cancer Society 2007; 25-26. |
| 3. | Magin MN, O'Connor PJ, Craig AW, Margison GP. The effect of hormone induced stress upon the extent of alkylation of rat liver nucleic acids by N-methyl-N-nitrosourea. Z Krebsforsch Klin Onkol Cancer Res Clin Oncol. 1975;84:217-222. [PubMed] [DOI] |
| 4. | Montesano R. Alkylation of DNA and tissue specificity in nitrosamine carcinogenesis. J Supramol Struct Cell Biochem. 1981;17:259-273. [PubMed] [DOI] |
| 5. | Drabløs F, Feyzi E, Aas PA, Vaagbø CB, Kavli B, Bratlie MS, Peña-Diaz J, Otterlei M, Slupphaug G, Krokan HE. Alkylation damage in DNA and RNA--repair mechanisms and medical significance. DNA Repair (Amst). 2004;3:1389-1407. [PubMed] [DOI] |
| 6. | Cervoni N, Bhattacharya S, Szyf M. DNA demethylase is a processive enzyme. J Biol Chem. 1999;274:8363-8366. [PubMed] [DOI] |
| 7. | Barreto G, Schäfer A, Marhold J, Stach D, Swaminathan SK, Handa V, Döderlein G, Maltry N, Wu W, Lyko F. Gadd45a promotes epigenetic gene activation by repair-mediated DNA demethylation. Nature. 2007;445:671-675. [PubMed] [DOI] |
| 9. | Aggerholm A, Holm MS, Guldberg P, Olesen LH, Hokland P. Promoter hypermethylation of p15INK4B, HIC1, CDH1, and ER is frequent in myelodysplastic syndrome and predicts poor prognosis in early-stage patients. Eur J Haematol. 2006;76:23-32. [PubMed] [DOI] |
| 10. | Wolffe AP, Matzke MA. Epigenetics: regulation through repression. Science. 1999;286:481-486. [PubMed] [DOI] |
| 11. | Wee G, Shim JJ, Koo DB, Chae JI, Lee KK, Han YM. Epigenetic alteration of the donor cells does not recapitulate the reprogramming of DNA methylation in cloned embryos. Reproduction. 2007;134:781-787. [PubMed] [DOI] |
| 12. | Lee WJ, Kim HJ. Inhibition of DNA methylation is involved in transdifferentiation of myoblasts into smooth muscle cells. Mol Cells. 2007;24:441-444. [PubMed] |
| 13. | 姬 峻芳, 吴 旻, 詹 启敏. Gadd45a在抑制细胞转化和肿瘤恶性进展中的作用. 生物化学与生物物理进展. 2006;33:1146-1153. |
| 14. | Zhang D, Song L, Li J, Wu K, Huang C. Coordination of JNK1 and JNK2 is critical for GADD45alpha induction and its mediated cell apoptosis in arsenite responses. J Biol Chem. 2006;281:34113-34123. [PubMed] [DOI] |
| 15. | Zhan Q, Fan S, Smith ML, Bae I, Yu K, Alamo I Jr, O'Connor PM, Fornace AJ Jr. Abrogation of p53 function affects gadd gene responses to DNA base-damaging agents and starvation. DNA Cell Biol. 1996;15:805-815. [PubMed] |
| 16. | Zhan Q, Chen IT, Antinore MJ, Fornace AJ Jr. Tumor suppressor p53 can participate in transcriptional induction of the GADD45 promoter in the absence of direct DNA binding. Mol Cell Biol. 1998;18:2768-2778. [PubMed] |
| 17. | Schmidt-Kastner R, Zhao W, Truettner J, Belayev L, Busto R, Ginsberg MD. Pixel-based image analysis of HSP70, GADD45 and MAP2 mRNA expression after focal cerebral ischemia: hemodynamic and histological correlates. Brain Res Mol Brain Res. 1998;63:79-97. [PubMed] [DOI] |
| 18. | Liebermann DA, Hoffman B. Gadd45 in the response of hematopoietic cells to genotoxic stress. Blood Cells Mol Dis. 2007;39:329-335. [PubMed] [DOI] |
| 19. | Chang Q, Bhatia D, Zhang Y, Meighan T, Castranova V, Shi X, Chen F. Incorporation of an internal ribosome entry site-dependent mechanism in arsenic-induced GADD45 alpha expression. Cancer Res. 2007;67:6146-6154. [PubMed] [DOI] |
| 20. | Oka M, Meacham AM, Hamazaki T, Rodić N, Chang LJ, Terada N. De novo DNA methyltransferases Dnmt3a and Dnmt3b primarily mediate the cytotoxic effect of 5-aza-2'-deoxycytidine. Oncogene. 2005;24:3091-3099. [PubMed] [DOI] |
| 21. | Kaneda M, Okano M, Hata K, Sado T, Tsujimoto N, Li E, Sasaki H. Essential role for de novo DNA methyltransferase Dnmt3a in paternal and maternal imprinting. Nature. 2004;429:900-903. [PubMed] [DOI] |
| 22. | Claus R, Fliegauf M, Stock M, Duque JA, Kolanczyk M, Lübbert M. Inhibitors of DNA methylation and histone deacetylation independently relieve AML1/ETO-mediated lysozyme repression. J Leukoc Biol. 2006;80:1462-1472. [PubMed] [DOI] |
| 24. | Causevic A, Gentil MV, Delaunay A, El-Soud WA, Garcia Z, Pannetier C, Brignolas F, Hagège D, Maury S. Relationship between DNA methylation and histone acetylation levels, cell redox and cell differentiation states in sugarbeet lines. Planta. 2006;224:812-827. [PubMed] [DOI] |
| 25. | Allen ML, Antoniou M. Correlation of DNA methylation with histone modifications across the HNRPA2B1-CBX3 ubiquitously-acting chromatin open element (UCOE). Epigenetics. 2007;2:227-236. [PubMed] |
| 26. | Walton TJ, Li G, Seth R, McArdle SE, Bishop MC, Rees RC. DNA demethylation and histone deacetylation inhibition co-operate to re-express estrogen receptor beta and induce apoptosis in prostate cancer cell-lines. Prostate. 2008;68:210-222. [PubMed] [DOI] |
| 28. | 府 伟灵, 黄 庆. 肿瘤表观遗传学. 国外医学•临床生物化学与检验学分册. 2004;25:289-290. |
| 30. | Ebert MP, Yu J, Hoffmann J, Rocco A, Röcken C, Kahmann S, Müller O, Korc M, Sung JJ, Malfertheiner P. Loss of beta-catenin expression in metastatic gastric cancer. J Clin Oncol. 2003;21:1708-1714. [PubMed] [DOI] |
| 31. | Yi J, Wang ZW, Cang H, Chen YY, Zhao R, Yu BM, Tang XM. p16 gene methylation in colorectal cancers associated with Duke's staging. World J Gastroenterol. 2001;7:722-725. [PubMed] |
| 32. | Issa JP. CpG island methylator phenotype in cancer. Nat Rev Cancer. 2004;4:988-993. [PubMed] [DOI] |