修回日期: 2007-11-28
接受日期: 2007-11-28
在线出版日期: 2007-12-08
目的: 使用RNA干扰技术阻断人结肠癌细胞cyclin D1的表达, 检测其对细胞生长增殖的影响和机制.
方法: 将人结肠癌细胞株HT-29分成3组, antisense组(转染反义链组)、sense组(转染正义链组)和对照组. 免疫沉淀和蛋白质印迹法观察cyclin D1 siRNA对细胞cyclin D1蛋白质表达的影响, 并对蛋白质电泳图像进行分析, MTT比色法绘制细胞生长曲线, 3H-TdR技术和流式细胞技术观测细胞周期变化.
结果: 反义siRNA有效抑制了cyclin D1蛋白质表达, MTT实验显示, 转染反义siRNA的细胞, 生长代谢减慢, 与对照组细胞差异显著(P<0.01). 而antisense组细胞24 h的3H-TdR渗入量, G0/G1期和S期细胞较对照组和sense组明显减少(3H-TdR: 1181.8±117.97 vs 1798.4±55.36, 1851.4±83.46; P<0.01; G0/G1期: 79.31% vs 60.87%, 59.14%; S期: 13.67% vs 26.42%, 27.93%; P<0.01).
结论: RNA干扰技术能有效减弱cyclin D1蛋白质表达, 使细胞周期受阻, 并抑制结肠癌细胞的生长增殖.
引文著录: 丁健, 李丹, 王承党, 吴婷, 王小众, 于皆平. RNA干扰阻断cyclin D1表达抑制结肠癌细胞增殖. 世界华人消化杂志 2007; 15(34): 3572-3576
Revised: November 28, 2007
Accepted: November 28, 2007
Published online: December 8, 2007
AIM: To inhibit the expression of cyclin D1 in a human colorectal cancer cell line using RNA interference, and to examine its effect on the growth and proliferation of colorectal cancer cells.
METHODS: Human colorectal cancer HT-29 cells were divided into three groups: antisense group transfected with antisense siRNA against cyclin D1, sense group transfected with sense siRNA against cyclin D1, and control group. The inhibitory effect of antisense siRNA on cyclin D1 protein expression was detected by immunoprecipitation and Western blotting. MTT assay was used to detect the effect of antisense siRNA on the growth and metabolism of HT-29 cells. 3H-TdR incorporation assay and flow cytometry were used to test the metabolism and cell cycle changes in HT-29 cells transfected with antisense siRNA against cyclin D1.
RESULTS: The expression level of cyclin D1 was clearly decreased in HT-29 cells in the antisense group. MTT assay showed a significant decrease in the metabolism of HT-29 cells in the antisense group, between 12 h and 72 h, compared with that in the control and sense groups (P < 0.01). 3H-TdR incorporation was lower in the antisense group than that in the control and sense groups (3H-TdR: 1181.8 ± 117.97 vs 1798.4 ± 55.36, 1851.4 ± 83.46; P < 0.01; G0/G1: 79.31% vs 60.87%, 59.14%; S: 13.67% vs 26.42%, 27.93%; P < 0.01).
CONCLUSION: RNA interference can effectively inhibit the expression of cyclin D1, and inhibition of cyclin D1 expression causes a decrease in the growth, metabolism and proliferation of colorectal cancer cells, due to a delay in the progression of the cell cycle from G1 to S phase.
- Citation: Ding J, Li D, Wang CD, Wu T, Wang XZ, Yu JP. RNA interference inhibits the proliferation of human colorectal cancer cells by blocking the expression of cyclin D1. Shijie Huaren Xiaohua Zazhi 2007; 15(34): 3572-3576
- URL: https://www.wjgnet.com/1009-3079/full/v15/i34/3572.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i34.3572
哺乳动物细胞的生长增殖受到细胞周期蛋白的调控. 其中cyclin D1在细胞从G1期进入S期的过程中发挥关键作用, 决定细胞是否继续生长和增殖[1-6]. 肿瘤细胞的生长同样受到cyclin D1调控, 在许多肿瘤细胞内发现cyclin D1的过表达, 比如肝癌[7-10]、食管癌[11-14]、胰腺癌[15]、乳腺癌[16]、喉癌[17-18]、膀胱癌[19]、纤维肉瘤[20]和肺癌[21]. 使用细胞转录因子Cdx1可以降低细胞内cyclin D1的含量, 达到抑制结肠癌增殖的目的[22]. 因此, 通过降低cyclin D1表达来抑制肿瘤的发生和转移成为肿瘤治疗的一个新思路. RNA干扰(RNA interference, RNAi)是由双链RNA分子介导的序列特异性转录后的基因静默过程, 是一种高效的基因阻断技术[23]. 本研究选择cyclin D1作为靶基因, 使用RNA干扰技术, 合成小干扰RNA(small interfering RNA, siRNA), 并将其转导人结肠癌细胞株HT-29细胞, 观察siRNA对cycling D1的阻断效果, 检测随之而来结肠癌细胞增殖和细胞周期的变化, 对cyclin D1的生物学作用进行补充, 同时为结肠癌的治疗探求一条新的途径.
人结肠癌细胞株HT-29购自上海生物细胞研究所, Lipofectamine2000是Invitrogen公司产品, 兔抗人cyclin D1抗体购自Santa Cruz公司, 电化学发光法检测试剂盒(包括二抗)购自武汉博士德公司, RPMI 1640、小牛血清试剂为GIBCO-BRL公司产品, 蛋白A、噻唑蓝(MTT)和二甲基亚枫均为Sigma公司产品, 3H-TdR为美国Ameshan公司产品, 液体闪烁技术仪BeckmanL-9800型为美国贝克曼公司, 流式细胞仪FACSCalibur型是美国BD公司产品.
1.2.1 细胞培养: 人结肠癌细胞株HT-29使用含100 mL/L小牛血清的1640培养液, 置于37℃、100%湿度和50 mL/L CO2的培养箱中培养.
1.2.2 siRNA的设计和合成: 根据GenBank检索cyclin D1的mRNA序列, 使用美国Ambion公司siRNA设计软件设计, 由大连TaKaRa公司合成双链小RNA. 正义链: UGACCCCGCACGAUUUCAUtt, 反义链: AUGAAAUCGUGCGGGGUCAtt. G/C含量: 47.6%, 在GenBank中使用同源序列查询, 确定与人类基因外显子无同源性.
1.2.3 siRNA的转染和细胞分组: 人结肠癌细胞株HT-29细胞生长至80%融合时, 使用Lipofectamine2000将siRNA转染入细胞, 操作过程按Lipofectamine2000的说明书进行. 根据转染siRNA的不同和对照组一起, 建立3个细胞组: 转染: antisense组、转染: sense组和对照组即正常HT-29细胞组. 转染结束后, 即进入下一步实验.
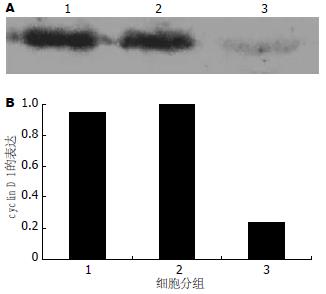
1.2.4 免疫沉淀和蛋白质印迹法检测cyclin: D1蛋白质表达: 转染48 h后, 取各细胞组细胞, 裂解, 离心后上清液蛋白定量. 三组细胞各取等量蛋白加兔抗人cyclin D1抗体和琼脂糖交联的蛋白A沉淀蛋白质, 振荡过夜后, 离心取沉淀. 行SDS-PAGE电泳, 之后以电转印法将蛋白转印到硝酸纤维素膜上, 封闭后加兔抗人cyclin D1抗体, 使用电化学发光法试剂盒进行显影. 使用图像分析软件Image-Pro Plus对蛋白质电泳图像进行分析. 为方便直观比较, 将蛋白质表达的最大值定义为100%.
1.2.5 MTT比色法检测细胞增殖实验: 使用胰蛋白酶消化antisense组、sense组和对照组细胞. 以每孔104个细胞的密度将细胞接种于96孔板中, 每组接种5孔. 置于37℃、50 mL/L CO2细胞培养箱中, 分别于12、24、36、48、60、72 h每孔加入5 g/L的MTT溶液20 μL, 继续培养4 h, 吸去上清液, 每孔加入二甲基亚枫150 μL, 振荡10 min. 在酶联免疫监测仪上, 使用595的波长进行检测, 记录吸光值. 以时间为横坐标, 吸光值为纵坐标绘制细胞生长曲线.
1.2.6 3H-TdR掺入法实验检测细胞周期的变化: 转染24 h后, 使用胰蛋白酶消化antisense组、sense组和对照组细胞. 以每孔104个细胞的密度将细胞接种于96孔板中, 每组5孔, 每孔加入2.0 μL的3H-TdR(3.7×1013 Bp/L)继续培养24 h. 使用胰蛋白酶消化细胞, 收集细胞. 破膜后使用液体闪烁计数仪测定3H-TdR渗入量的CPM值.
1.2.7 流式细胞技术检测细胞周期的改变: 细胞转染后48 h后, 使用胰蛋白酶消化并收获antisense组、sense组和对照组细胞. 用4℃预冷的700 mL/L乙醇固定1 h, 然后使用PBS洗涤细胞3次. 将细胞重悬于50 mg/L的PI染液(含20 g/L RNase)中, 室温孵育1 h, 使用流式细胞仪对细胞周期进行分析.
统计学处理 其他数据与最大值的比率定义为表达量. 使用成组t检验对实验数值进行分析.
转染反义siRNA的antisense组细胞cyclin D1表达明显低于sense组和对照组. Image-Pro Plus图像分析显示: antisense组细胞cyclin D1的表达只有对照组的24.77%, 而转染正义siRNA的sense组和对照组cyclin D1表达相似(图1A-B).
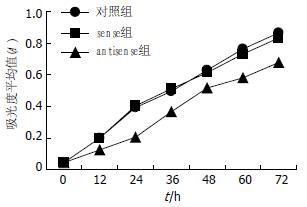
开始检测时, antisense组、对照组和sense组细胞吸光度无差别(P>0.05). 从12 h开始, antisense组细胞的吸光度显著低于对照组和sense组(P<0.01). 在此后的每一个检测时间, 即24、36、48和72 h, antisense组细胞的吸光度均显著低于对照组和sense组(P<0.01)(表1, 图2).
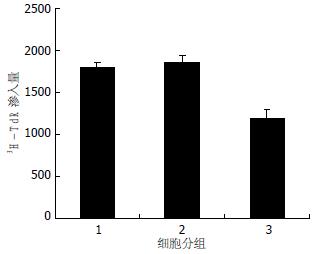
对照组和sense组24 h的3H-TdR渗入量分别为1798.4±55.36和1851.4±83.46, 而antisense组细胞24 h的3H-TdR渗入量为1181.8±117.97, 较对照组和sense组明显减少(P<0.01). 对照组和sense组24 h的3H-TdR渗入量无差别(P>0.05)(图3).
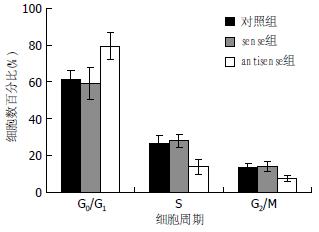
正常细胞组即对照组细胞有60.87%处于G0/G1期, 26.42%处于S期, sense组细胞有59.14%处于G0/G1期, 27.93%处于S期, 而转染反义siRNA的antisense组细胞有79.31%处于G0/G1期, 13.67%处于S期. 与对照组和sense组相比, 处于G0/G1期的细胞和处于S期的细胞, 均有显著性差异(P<0.01, 表2, 图4).
在细胞周期中, G1到S期的转变非常重要. 细胞一旦从G1期跨入S期就可以不再依靠外来信息的刺激而自动完成分裂过程. 与G1期相关的细胞周期素主要是cyclin D, 他包括三个亚型: D1、D2和D3, 其中D1和肿瘤细胞的关系最为密切. cyclin D1基因位于人染色体11q13上, 全长15 kb, 共5个外显子. cDNA全长4.2 kb, 编码295个氨基酸残基组成的蛋白质, 分子量为36 kDa[24]. cyclin D1于G1期最先合成, 形成cyclin D1-CDK4或cyclin D1-CDK6复合物, 激活CDK磷酸化底物pRB, 使之释放出结合的转录因子E2F, 促进DNA转录, 使细胞从G1期进入S期, 从而促进细胞生长和增殖[25]. 目前已在多种肿瘤细胞内发现cyclin D1表达增多, 提示cyclin D1在肿瘤的发生过程中发挥重要作用. 通过抑制其基因的表达达到抑制肿瘤生长的目的已经引起人们重视[26].
在现有抑制基因表达技术中, RNA干扰技术是被公认为最有效果的手段. 干扰RNA合成的方法有化学合成、体外转录、长片段双链RNA消化和体内表达等方法[27-31]. 其中化学合成法具有快捷, 方便, 使用剂量容易控制, 转染效果稳定, 可重复性强等优点.
本实验选择化学合成cyclin D1的siRNA对cyclin D1基因表达进行封闭, 观察cyclin D1蛋白质表达被部分封闭后, 结肠癌HT-29细胞生物学功能变化. 转染cyclin D1的反义siRNA后, 使用免疫沉淀和蛋白质印迹法证实cyclin D1蛋白质表达明显下降, 只有对照组的24.77%. 蛋白质是基因的最终表达产物, 此结果说明反义siRNA有效封闭了cyclin D1基因, 造成cyclin D1蛋白质量的减少. cyclin D1-CDK4或cyclin D1-CDK6复合物可以促使细胞从G1期进入S期, 完成分裂增殖. 本实验试图观察cyclin D1表达被有效降低后, 细胞的生长增殖情况. MTT实验和3H-TdR渗入实验证实了cyclin D1降低会导致细胞活性和增殖能力的下降, 同时3H-TdR渗入实验还说明了生长代谢减慢与细胞从G1进入S期受阻有关. 本实验使用了流式细胞技术, 进一步明确转染siRNA细胞出现活性减低和增殖能力下降是因为在G1期受阻. 此结果和cyclin D1作用特点相符. 总之, 本实验成功设计合成cyclin D1 siRNA, 转染后有效阻断cyclin D1表达并减弱结肠癌细胞的生长增殖明显, 出现细胞周期停滞. 一方面进一步验证cyclin D1是细胞周期进展中的关键信号分子, 发挥重要生物学功能. 另一方面证实, 封闭cyclin D1表达可以抑制结肠癌增殖, 为肿瘤的基因治疗开辟一条新思路.
肿瘤细胞不同于正常细胞的重要特点在于其生长和增殖的失控. 细胞的生长增殖受细胞周期蛋白的调节, 现有资料证实多种肿瘤表达异常增多的细胞周期蛋白, 其中报道最多是cyclin D1, cyclin D1已被证实是调节肿瘤细胞生长增殖的关键信号分子.
本文使用RNA干扰技术, 封闭cyclin D1表达, 观察随之而来的细胞生物学功能的变化, 旨在进一步了解cyclin D1在人结肠癌发生发展过程中的作用, 并试图通过阻断细胞生长和增殖过程中的信号传导, 达到治疗结肠癌的目的.
研究证实许多抑制肿瘤细胞生长和增殖的转录因子, 其作用是通过降低cyclin D1表达来实现的. 研究者发现Cdx1通过干扰cyclin D1转录, 减少结肠癌细胞中cyclin D1的蛋白质水平, 使细胞生长停滞于G0/G1期; 而不表达Cdx1的变异株则无此作用.
本文寻找到了高效阻断cyclin D1表达的基因序列和随之而来的系统化研究是对现有相关资料的补充.
本文针对肿瘤细胞生长周期进行抑制, 达到治疗肿瘤生长增殖的目的, 为类似的研究打下基础, 提供了新思路. 高效的siRNA序列将来可以用来进行动物实验和临床前期实验, 并可以试验用于其他肿瘤细胞.
本文图文并茂, 层次清楚, 有一定的可读性和参考价值.
编辑: 程剑侠 电编:郭海丽
| 1. | Fu M, Wang C, Li Z, Sakamaki T, Pestell RG. Minireview: Cyclin D1: normal and abnormal functions. Endocrinology. 2004;145:5439-5447. [PubMed] |
| 3. | Baker GL, Landis MW, Hinds PW. Multiple functions of D-type cyclins can antagonize pRb-mediated suppression of proliferation. Cell Cycle. 2005;4:330-338. [PubMed] |
| 4. | Gladden AB, Diehl JA. Location, location, location: the role of cyclin D1 nuclear localization in cancer. J Cell Biochem. 2005;96:906-913. [PubMed] |
| 5. | Rampalli S, Pavithra L, Bhatt A, Kundu TK, Chattopadhyay S. Tumor suppressor SMAR1 mediates cyclin D1 repression by recruitment of the SIN3/histone deacetylase 1 complex. Mol Cell Biol. 2005;25:8415-8429. [PubMed] |
| 6. | Zhang ZK, Davies KP, Allen J, Zhu L, Pestell RG, Zagzag D, Kalpana GV. Cell cycle arrest and repression of cyclin D1 transcription by INI1/hSNF5. Mol Cell Biol. 2002;22:5975-5988. [PubMed] |
| 7. | Zhang YJ, Jiang W, Chen CJ, Lee CS, Kahn SM, Santella RM, Weinstein IB. Amplification and overexpression of cyclin D1 in human hepatocellular carcinoma. Biochem Biophys Res Commun. 1993;196:1010-1016. [PubMed] |
| 8. | Zhang YJ, Chen SY, Chen CJ, Santella RM. Polymorphisms in cyclin D1 gene and hepatocellular carcinoma. Mol Carcinog. 2002;33:125-129. [PubMed] |
| 9. | Xu JM, Wen JM, Zhang M, Lu GL, Wu LZ, Wang WS. A study of gene amplification and expression of cyclin D1 in hepatocellular carcinoma. Zhonghua Bing Li Xue Za Zhi. 2004;33:26-30. [PubMed] |
| 10. | Nishida N, Fukuda Y, Komeda T, Kita R, Sando T, Furukawa M, Amenomori M, Shibagaki I, Nakao K, Ikenaga M. Amplification and overexpression of the cyclin D1 gene in aggressive human hepatocellular carcinoma. Cancer Res. 1994;54:3107-3110. [PubMed] |
| 11. | Jiang W, Zhang YJ, Kahn SM, Hollstein MC, Santella RM, Lu SH, Harris CC, Montesano R, Weinstein IB. Altered expression of the cyclin D1 and retinoblastoma genes in human esophageal cancer. Proc Natl Acad Sci U S A. 1993;90:9026-9030. [PubMed] |
| 12. | Nakagawa H, Zukerberg L, Togawa K, Meltzer SJ, Nishihara T, Rustgi AK. Human cyclin D1 oncogene and esophageal squamous cell carcinoma. Cancer. 1995;76:541-549. [PubMed] |
| 13. | Jiang W, Kahn SM, Tomita N, Zhang YJ, Lu SH, Weinstein IB. Amplification and expression of the human cyclin D gene in esophageal cancer. Cancer Res. 1992;52:2980-2983. [PubMed] |
| 14. | Gramlich TL, Fritsch CR, Maurer D, Eberle M, Gansler TS. Differential polymerase chain reaction assay of cyclin D1 gene amplification in esophageal carcinoma. Diagn Mol Pathol. 1994;3:255-259. [PubMed] |
| 15. | Huang L, Lang D, Geradts J, Obara T, Klein-Szanto AJ, Lynch HT, Ruggeri BA. Molecular and immunochemical analyses of RB1 and cyclin D1 in human ductal pancreatic carcinomas and cell lines. Mol Carcinog. 1996;15:85-95. [PubMed] |
| 16. | Gillett C, Fantl V, Smith R, Fisher C, Bartek J, Dickson C, Barnes D, Peters G. Amplification and overexpression of cyclin D1 in breast cancer detected by immunohistochemical staining. Cancer Res. 1994;54:1812-1817. [PubMed] |
| 17. | Jares P, Fernandez PL, Campo E, Nadal A, Bosch F, Aiza G, Nayach I, Traserra J, Cardesa A. PRAD-1/cyclin D1 gene amplification correlates with messenger RNA overexpression and tumor progression in human laryngeal carcinomas. Cancer Res. 1994;54:4813-4817. [PubMed] |
| 18. | Bellacosa A, Almadori G, Cavallo S, Cadoni G, Galli J, Ferrandina G, Scambia G, Neri G. Cyclin D1 gene amplification in human laryngeal squamous cell carcinomas: prognostic significance and clinical implications. Clin Cancer Res. 1996;2:175-180. [PubMed] |
| 19. | Bringuier PP, Tamimi Y, Schuuring E, Schalken J. Expression of cyclin D1 and EMS1 in bladder tumours; relationship with chromosome 11q13 amplification. Oncogene. 1996;12:1747-1753. [PubMed] |
| 20. | Hochhauser D, Schnieders B, Ercikan-Abali E, Gorlick R, Muise-Helmericks R, Li WW, Fan J, Banerjee D, Bertino JR. Effect of cyclin D1 overexpression on drug sensitivity in a human fibrosarcoma cell line. J Natl Cancer Inst. 1996;88:1269-1275. [PubMed] |
| 21. | Reissmann PT, Koga H, Figlin RA, Holmes EC, Slamon DJ. Amplification and overexpression of the cyclin D1 and epidermal growth factor receptor genes in non-small-cell lung cancer. Lung Cancer Study Group. J CancerRes Clin On. col. 1999;125:61-70. [PubMed] |
| 22. | Lynch J, Keller M, Guo RJ, Yang D, Traber P. Cdx1 inhibits the proliferation of human colon cancer cells by reducing cyclin D1 gene expression. Oncogene. 2003;22:6395-6407. [PubMed] |
| 23. | Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 1998;391:806-811. [PubMed] |
| 26. | Petty WJ, Dragnev KH, Dmitrovsky E. Cyclin D1 as a target for chemoprevention. Lung Cancer. 2003;41 Suppl 1:S155-S161. [PubMed] |
| 27. | Paul CP, Good PD, Winer I, Engelke DR. Effective expression of small interfering RNA in human cells. Nat Biotechnol. 2002;20:505-508. [PubMed] |
| 28. | Lewis DL, Hagstrom JE, Loomis AG, Wolff JA, Herweijer H. Efficient delivery of siRNA for inhibition of gene expression in postnatal mice. Nat Genet. 2002;32:107-108. [PubMed] |
| 29. | Castanotto D, Li H, Rossi JJ. Functional siRNA expression from transfected PCR products. RNA. 2002;8:1454-1460. [PubMed] |
| 30. | Kawakami S, Hashida M. Targeted delivery systems of small interfering RNA by systemic administration. Drug Metab Ph. armacokinet. 2007;22:142-151. [PubMed] |
| 31. | Natt F. siRNAs in drug discovery: target validation and beyond. Curr Opin Mol Ther. 2007;9:242-247. [PubMed] |