修回日期: 2007-10-30
接受日期: 2007-10-28
在线出版日期: 2007-11-28
目的: 研研究西藏拉萨地区乙型肝炎病毒基因型的分布与特点.
方法: 采集92份西藏拉萨地区乙型肝炎患者的血清, 参照GenBank中HBV DNA序列设计寡核苷酸探针并制备HBV基因分型芯片, 利用套式PCR扩增HBV S基因部分片段, 结合基因芯片、DNA测序和BioEdit软件进行基因分型检测, 并对其与乙肝标志物、DNA含量、性别和民族之间的关系进行分析.
结果: 在92例血清标本中, 套式PCR检测73例HBV DNA阳性可进行基因分型检测, 其中B型13例(17.8%), C型18例(24.7%), D型39例(53.4%)和B/D混合基因型3例(4.1%). 统计学分析3种基因型分布在不同乙肝标志物阳性、不同DNA含量和不同性别之间无差异, 但与民族存在统计学差异(χ2 = 7.179, P<0.05). B型以汉族为主(9/13), 而C、D型以藏族为主(12/18、28/39). 将基因芯片分型的B、C、D型和B/D混合型进行DNA序列分析, 表明两种分型方法的结果完全一致.
结论: PCR结合基因芯片技术可用于HBV基因分型. 西藏拉萨地区HBV基因型包括B、C、D和B/D混合型, 其中以D型为主.
引文著录: 曹占良, 高英堂, 刘霜, 景丽, 吉宗, 刘彤, 刘持佳, 杜智. 基因芯片技术检测西藏拉萨地区的乙型肝炎病毒基因型. 世界华人消化杂志 2007; 15(33): 3500-3506
Revised: October 30, 2007
Accepted: October 28, 2007
Published online: November 28, 2007
AIM: To investigate the distribution and characteristics of HBV genotypes in Lhasa.
METHODS: A total of 92 serum samples were collected from chronic hepatitis patients in Lhasa infected with HBV. Oligonucleotide probes were designed according to the DNA sequences from GenBank and used to prepare the genotyping chip. The fragments of HBV S gene were amplified using nested PCR. The genotypes were identified by gene chip and then confirmed by sequencing. The phylogeny was analyzed with BioEdit software. The relationship with hepatitis markers, DNA load, gender and race was also analyzed.
RESULTS: Among the 73 patients positive for HBV DNA, 13 (17.8%) were genotype B, 18 (24.7%) genotype C, 39 (53.4%) genotype D, and 3 (4.1%) mixed genotype B/D. The distribution of the genotypes was not associated with serum HBV markers, HBV DNA load or gender, but was significantly associated with nationality (χ2 = 7.179, P < 0.05). Genotype B (9/13) was mostly distributed in the Han region, and genotypes C (12/18) and D (28/39) in Tibet. In addition, we selected 7 cases of genotype B, 6 of genotype C, 16 of genotype D, and 3 of mixed genotype B/D, identified by HBV typing chip which were further confirmed by sequencing, and the results of these two methods were consistent.
CONCLUSION: Gene chip technology can be used for HBV genotyping. HBV genotypes distributed in Lhasa include B, C, D and B/D, of which genotype D is the predominant strain.
- Citation: Cao ZL, Gao YT, Liu S, Jing L, Ji Z, Liu T, Liu CJ, Du Z. Detection of HBV genotypes in Lhasa by gene chip. Shijie Huaren Xiaohua Zazhi 2007; 15(33): 3500-3506
- URL: https://www.wjgnet.com/1009-3079/full/v15/i33/3500.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i33.3500
自1988年Okamoto et al[1]将18株不同血清亚型的HBV DNA全序列分为A、B、C和D 4个基因型之后, 相继又有E、F、G和H型4个基因型被确定[2-4]. 在我国乙型肝炎患者中, 南方以B型为主, 北方以C型为主. D型仅见于少数民族地区, 如西藏、新疆等地区, A和F型偶有发现, 尚无E、G和H型的报道[5-12]. 西藏地处青藏高原, 地域相对封闭, 而又靠近中亚地区, 因此, 其乙肝病毒基因型的分布特点非常值得研究. 本文应用基因芯片技术和DNA序列分析西藏拉萨地区92例乙肝患者的基因型, 进一步了解拉萨地区HBV基因型分布情况.
1.1.1 血清标本: 92例血清标本均采自武警西藏总队医院门诊及住院的HBV患者, 患者均来自西藏拉萨地区, 年龄4-67岁. 32例为HBsAg、HBeAg和抗-HBcAb阳性, 46例为HBsAg、抗-HBeAb和抗-HBcAb阳性, 14例为HBsAg和抗-HBcAb阳性. 汉族男性21例, 汉族女性17例, 藏族男性26例, 藏族女性28例. 肘静脉采血5 mL, 分离血清于-80℃保存, 运输采用保温冰盒的特快专递.
1.1.2 主要试剂和仪器: HBV DNA定量检测试剂盒购自中山医科大学达安基因诊断中心, PCR仪为美国ABI公司的ABI Prism 7000实时荧光定量PCR仪, 芯片检测所需的尼龙膜、DIG-11-dUTP、过氧化物酶标记的地高辛抗体、封阻试剂等均购自美国罗氏公司, Taq酶、UNG酶、dNTP、PCR产物克隆试剂等购自大连宝生物工程公司, 其他生化试剂均为国产分析纯. PCR引物和芯片探针由上海生工生物工程公司合成, 芯片制作采用中科院电工所的DY2003生物芯片点样仪, 探针固定采用美国UVP公司的CL-1000M紫外交联仪, 杂交使用国产多功能杂交反应仪, DNA测序由北京三博远志公司完成.
1.2.1 HBVDNA定量检测: 采用荧光定量PCR法, HBV DNA提取、PCR扩增和数据分析均按达安基因诊断中心的试剂盒操作说明进行.
1.2.2 HBV: DNA基因分型芯片的制备与检测: (1)基因分型芯片的制备, 根据GenBank中HBV 7个基因型105株序列设计HBV S基因区的PCR引物和寡核苷酸探针, 探针经一定修饰后点到尼龙膜上并交联固定, 即成为HBV基因分型芯片. 引物和探针序列见表1, 探针在芯片上的排列位置见图1. (2) 地高辛标记的套式PCR, 第一次PCR反应体系: 2 µL血清裂解液, 10×PCR缓冲液2.5 µL, 2 mmol/L dNTP(dTTP: Dig-dUTP为10:1)2 µL, UNG酶0.5 µL, Taq酶1 µL, 上下游外引物1 µL, 超纯水16 µL, 总计25 µL. PCR扩增条件: 50℃ 5 min, 94℃ 2 min预变性, 94℃ 1 min, 55℃ 45 s, 72℃ 45 s, 35个循环, 然后72℃延伸5 min. 对于强阳性标本(HBV DNA定量大于5×108拷贝/L)取5 µL一次PCR标记产物即可用于芯片杂交, 而其他标本需进行第二次PCR标记反应. 第二次PCR反应参照一次PCR进行, 区别在于使用一对内引物, 取5 µL二次PCR产物电泳, 依据电泳带的强弱取5-20 µL用于芯片杂交. 用灭菌水做套式PCR的阴性对照, 每6个标本设置1个对照. (3) 预杂交、杂交和显色, 参照Roche公司地高辛标记检测试剂盒并加以修改, 杂交温度为46℃. 显色采用华美公司的DAB试剂, 显色5-10 min, 用1×PBS终止反应. (4) 芯片杂交信号的判读, 芯片杂交结果可直观判断, 整张芯片背景不着色, 特异杂交点和非特异杂交点之间差别明显. 进一步将芯片通过扫描仪输入到计算机中, 利用实验室研发的分析软件(Chip3.0)进行灰度值分析, 从而判断患者的基因型. 判断标准: 特异杂交点的灰度值不小于阳性对照点(102稀释)的50%, 特异杂交点和非特异杂交点之间的探针灰度值比值不小于3. 对出现混合基因型的标本从处理血清步骤开始重复实验确证.
| 引物/探针 | 位置 | 序列组成 | |
| 引物 | 上游外引物 | 182-202 nt | 5′TAG GAC CCC TGC TCG TGT TAC |
| 下游外引物 | 805-824 nt | 5′CA(A/G) AGA CAA AAG AAA ATT GG | |
| 上游内引物 | 246-266 nt | 5′GAG TCT AGA CTC GTG GTG GAC | |
| 下游内引物 | 777-797 nt | 5′GG(T/C) A(T/A)A AAG GGA CTC A(A/C)G ATG | |
| 探针 | 通用探针A-G1 | 377-393 nt | 5′GG ATG TGT CTG CGG CGT |
| 通用探针A-G2 | 393-377 nt | 5′ACG CCG CAG ACA CAT CC | |
| A1 | 483-502 nt | 5′A ATT CCA GGA TCA ACA ACA A | |
| A2 | 502-483 nt | 5′T TGT TGT TGA TCC TGG AAT T | |
| BE1 | 483-502 nt | 5′A ATT CCA GGA TCA TCA AC(C/A) A | |
| BE2 | 502-483 nt | 5′T (G/T)GT TGA TGA TCC TGG AAT T | |
| C1 | 483-502 nt | 5′A CTT CCA GGA ACA TCA ACT A | |
| C2 | 502-483 nt | 5′T AGT TGA TGT TCC TGG AAG T | |
| ABDFG1 | 596-613 nt | 5′GCA CCT GTA TTC CCA TCC | |
| ABDFG2 | 613-596 nt | 5′GGA TGG GAA TAC AGG TGC | |
| CEF1 | 596-613 nt | 5′GCA CTT GTA TTC CCA TCC | |
| CEF2 | 613-596 nt | 5′GGA TGG GAA TAC AAG TGC | |
| A3 | 483-502 nt | 5′A ATT CCA GGA TCC ACA ACA A | |
| C3 | 524-540 nt | 5′CC TGC ACG ATT CCT GCT | |
| C4 | 540-524 nt | 5′AGC AGG AAT CGT GCA GG | |
| D1 | 549-566 nt | 5′C TCT ATG TAT CCC TCC TG | |
| D2 | 284-300 nt | 5′GG GGA ACC ACC GTG TGT | |
| D3 | 284-300 nt | 5′GG GGA ACT ACC GTG TGT | |
| E1 | 613-630 nt | 5′CCA TCA TCA TGG GCT TTC | |
| E2 | 700-716 nt | 5′TGG TTC GCC GGG CTT TC | |
| F1 | 680-699 nt | 5′TA CTA GTG CAA TTT GTT CAG | |
| F2 | 699-680 nt | 5′CTG AAC AAA TTG CAC TAG TA | |
| G1 | 483-500 nt | 5′G ATT CCA GGA TCC TCG AC |
1.2.3 PCR产物的测序: 根据芯片杂交结果, 选取7例B基因型、6例C基因型、16例D基因型和3例B/D混合基因型样品进行PCR产物序列分析, 即使用PCR内引物扩增100 µL产物直接测序. 为进一步确定混合基因型的准确性, 对1例B/D混合基因型的PCR产物进行A/T克隆, 采用大连宝生物工程公司的PMD18T载体及其连接、转化试剂, 均按其说明书进行操作. 挑选10个白色克隆进行PCR鉴定证明含有PCR产物的插入子, 随后将此10个克隆进行测序.
1.2.4 核苷酸序列相似性和系统发生树分析: 从GenBank中选取HBV 7个基因型的参照株序列, A-G型的基因号依次为M57663、D00329、M12906、X02496、X75657、X69798和AF160501. 利用BioEdit7.0.0软件包进行序列比对分析, 其中使用Clustal W 1.4软件作多重序列比较, 使用Sequence Identity Matrix程序进行核苷酸相似性比较, 系统发生树分析采用Neighbor phylogenetic tree方法.
统计学处理 应用SPSS13.0统计软件中的χ2检验分析数据.
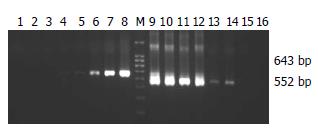
荧光定量PCR检测乙肝标志物HBsAg、HBeAg和抗-HBcAb阳性患者的HBV DNA含量均不低于1.0×103拷贝/mL, 而HBsAg、抗-HBeAb和抗-HBcAb阳性和HBsAg和抗-HBcAb阳性的患者只有11例和9例大于1.0×103拷贝/mL. 对于用于基因分型的地高辛标记套式PCR, DNA含量不低于1.0×103拷贝/mL的52例标本均阳性, 而定量小于1.0×103拷贝/mL的小三阳和一、五阳的患者中又有19例和2例套式二次PCR阳性, 其中17例的DNA含量介于102-103拷贝/mL之间(表2), 部分标本的套式PCR电泳结果见图2. 为保证这些低拷贝标本扩增的准确性, PCR时严格设置空白对照并且不与高拷贝标本同时扩增, 并且重复PCR扩增仍获得相同结果, 表明这些低拷贝标本的阳性扩增带是可信的. 因此, 可以用于芯片杂交的标本总计73例.
| 分组 | n | ≥1.0X103/阳性 | <1.0X103/阳性 |
| HBsAg、HBeAg和抗-HBcAb阳性 | 32 | 32(7.33±1.12)/32 | 0/0 |
| HBsAg、抗-HBeAb和抗-HBcAb阳性 | 46 | 11(5.06±1.18)/11 | 35(1.11±0.98)/19 |
| HBsAg和抗-HBcAb阳性 | 14 | 9(5.72±1.17)/9 | 5(1.40±1.14)/2 |
| 合计 | 92 | 52/52 | 40/21 |
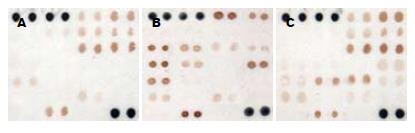
2.2.1 芯片特异性验证: 为了确定芯片杂交结果的准确性, 我们从3个方面进行了试验检测. (1) 芯片上探针点样量的检测, 用点样针将HBV DNA的PCR产物点于膜上, 再用标记的PCR产物杂交, 观察各点之间点样量的误差. 变异系数小于20%, 满足芯片定性检测的需要. (2)使用人工合成的互补探针检测芯片的特异性, 均可获得对应探针的强杂交信号. (3)对32份PCR产物直接测序表明基因芯片的判读结果与测序结果一致(下文2.3详述). 通过以上试验表明, 此芯片可初步用于该项研究.
2.2.2 HBV: DNA基因型分布: 在73例基因分型的标本中, B型13例(17.8%), C型18例(24.7%), D型39例(53.4%)和B/D混合基因型3例(4.1%), 表明D型可能是拉萨地区最常见的HBV基因型, 芯片杂交图见图3, 基因型的详细分布见表3, 由于B/D混合型仅3例, 未进行统计分析. 由表3可知, 3种基因型的分布在不同乙肝标志物阳性、不同DNA含量和不同性别之间无统计学差异, 但与民族存在统计学差异(χ2 = 7.179, P<0.05). B型更多的存在于汉族中(9/13), 而C、D型更多的存在于藏族中(12/18、28/39).
| 分组 | n | B | C | D | B/D | P值 |
| 乙肝标志物 | ||||||
| HBsAg、HBeAg和抗-HBcAb阳性 | 32 | 7 | 6 | 19 | 0 | |
| HBsAg、抗-HBeAb和抗-HBcAb阳性 | 30 | 4 | 9 | 14 | 3 | |
| HBsAg和抗-HBcAb阳性 | 11 | 2 | 3 | 6 | 0 | 0.778 |
| 荧光定量PCR | ||||||
| ≥1.0×103拷贝/mL | 52 | 11 | 10 | 28 | 3 | |
| <1.0×103拷贝/mL | 21 | 2 | 8 | 11 | 0 | 0.205 |
| 性别 | ||||||
| 男性 | 38 | 6 | 8 | 21 | 3 | |
| 女性 | 35 | 7 | 10 | 18 | 0 | 0.767 |
| 民族 | ||||||
| 汉族 | 28 | 9 | 6 | 11 | 2 | |
| 藏族 | 45 | 4 | 12 | 28 | 1 | 0.028 |
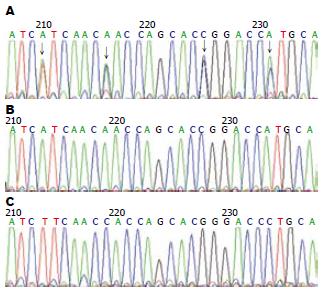
选取基因芯片分型的7例B基因型、6例C基因型、16例D基因型和3例B/D混合基因型的PCR产物直接测序. 测序使用上游内引物, 获得有效序列长度480 bp, 利用BioEdit7.0.0软件包的Sequence Identity Matrix程序结合7个基因型的7株参照株进行序列相似性比较. 结果表明, 基因芯片分型的7例B基因型标本与B型参照株(D00329)的相似性大于98.2%,基因芯片分型的6例C基因型标本与C型参照株(M12906)的相似性大于98.5%, 基因芯片分型的16例D基因型标本与D型参照株(X02496)的相似性大于98.5%. 29例单一基因型的标本其两种分型方法完全一致, 而3例混合型按测序公司提供的序列文件分析显示D基因型. 进一步分析其提供的测序峰图文件, 发现3例B/D混合基因型的测序峰图在B、D基因型差异位点存在2个碱基的双峰(图4). 为确定混合型的真实性, 再次重新裂解血清、PCR扩增、芯片杂交和PCR产物直接测序, 杂交和测序结果均表明3例B/D混合基因型的真实性. 同时, 我们将1例B/D混合基因型的PCR产物进行A/T克隆, 挑选10个白色克隆进行PCR鉴定, 证明7个克隆为B型, 3个克隆为D型. 再次证明PCR产物直接测序观察到的双峰是由于B/D基因型混合造成的.
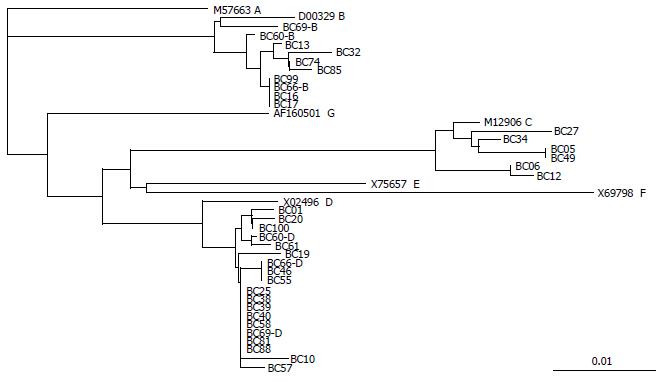
在上述测序分析基础上, 构建7株参照株序列和35株测序序列(将3株B/D混合型序列分解成B、D各3个序列)的系统发生树(图5). 从系统发生树中清楚看出, 35株测序序列包括10个B基因型(其中3株B/D混合型的B序列)、6个C基因型和19个D基因型(其中3株B/D混合型的D序列), 与芯片杂交完全吻合.
将35株DNA测序序列翻译成氨基酸序列, 根据血清型的定义发现, 10个B基因型的血清型均为adw2, 6个C基因型的血清型均为adrq+, 19个D基因型的血清型均为ayw2.
HBV基因分型已被广泛应用于HBV的分子流行病学、病因学和临床诊治研究, 因此, 建立一种准确、简便快速的分型方法就显得尤为重要. HBV基因型是根据其基因组核苷酸全序列异质性≥8%划分的[1], 因而HBV全基因组核苷酸序列分析是确定基因分型最可靠的经典方法, 是许多其他分型方法的参考标准, 并且在证实新基因(亚)型时具有举足轻重的作用. 但由于设备技术条件要求高、成本贵、操作繁琐等原因导致其难以在临床上推广应用. 为建立一种临床可行的分型方法, 多位学者通过对GenBank中众多HBV全基因组序列的比对分析和建立分子系统发生树, 发现基因组中前S/S基因或前C/C基因的差异与整个病毒核苷酸序列的异质性是一致的, 因此只要通过PCR扩增具代表性的部分基因片段, 再对PCR扩增产物直接进行测序并与参照株序列比对即可得出分型结果[1-3]. 在此基础上, 相继建立了PCR-限制性片段长度多态性分型法(PCR-RFLP)[13-14]、型特异引物PCR
法[15]、荧光探针PCR[16-17]、基因芯片法(型特异探针反向杂交法)[18-20]、特异性表位mAb的ELISA[21]等多种方法. 这些方法虽然可用于HBV基因分型, 但各有利弊, 均需进一步的改进、优化, 尚无一种公认的标准分型方法. 本文在比对GenBank中HBV 7个基因型105株序列的基础上, 设计型特异的寡核苷酸探针制成HBV基因分型芯片. 该芯片的特点是: (1)芯片基质为尼龙膜, 显色采用DAB. 尼龙膜与玻璃片等基质相比, 膜更易于吸附固定DNA分子, 可以保证更稳定和准确的信号强度. 膜的制备工艺和杂交条件等更成熟, 结果相对更稳定. 各种荧光的干扰因素对尼龙膜的杂交和DAB显色无影响, 并且不需昂贵的荧光扫描仪等设备, 操作简便, 凡能开展PCR检测的实验室都可应用. (2)同一基因型采用多个型特异探针鉴别, 通过序列比对发现, 即使是最保守区域的型特异探针, 在同一基因型内也有少数序列存在差异, 同时其他型的个别序列又与此一致, 这样用单一探针检测时即可能出现漏检也可能出现假阳性. 因此, 针对我国流行的B、C和D 3种基因型, 我们均设计2个以上的探针进行检测. (3)该芯片成本低廉、试验条件简便、易于推广. 利用此芯片对西藏拉萨地区92例血清标本的检测和其中32份PCR产物测序验证, 表明该基因芯片具有较好的检测效果, 可以广泛用于各项HBV基因分型研究.
本文依据HBV S基因核苷酸序列对西藏拉萨地区73例乙肝患者进行的基因型分析表明, 优势基因型为D型, 占53.4%, 其次为C型(24.7%)和B型(17.8%), 以及4.1%的B/D混合基因型. 对比以往文献, Cui et al[6]对26例西藏藏族乙肝携带者的S基因核苷酸序列分析表明, 1例为C型, 其余25例均为D型. 姜双应 et al[22]对9例青海同德地区藏族乙肝感染者的S基因核苷酸序列分析表明, 7例为D型, 2例为C型. 边中启 et al[23]报道了1例云南少数民族地区的藏族慢性乙肝患者为B型. 因此, 在西藏地区即存在D、C等优势基因型, 同时也存在B、B/D等少数基因型.
由表3可知, 3种基因型的分布在不同DNA含量和不同性别之间无统计学差异, 这与最近的两篇报道相一致. 赵鸿 et al[7]对新疆等8个地区884例乙肝感染者分型表明, B、C型最为常见, 占20.77%和78.22%, 病毒载量在两基因型之间无差别. 高俊薇 et al[8]对11个内地城市1214例乙肝感染者分型表明, B、C型也是最常见的基因型, 其在不同性别中的分布无统计学差异.
众多文献报道国内南方以B型为主, 北方以C型为主, 其他型只有零星报道[5-12]. 而与西藏相邻的中东地区流行株为D型[24]. 因此, 西藏独特的地理位置和人口迁移等因素可能会影响该地区HBV基因型演化和分布规律. 拉萨地区三种基因型的分布在不同民族之间存在统计学差异, 不能简单地认为某种民族更容易感染某种基因型, 而应更深层次地分析基因型的演化历史. D、C型是优势基因型, 各占53.4%和24.7%, 并且更多的存在于藏族中(28/39、12/18), 表明这两种基因型可能是拉萨地区早期的基因型. B型仅占17.8%, 且多存在于汉族中(9/13), 推测其可能是随着汉族人在西藏的定居而引入的; 另一方面, 11例汉族D基因型、4例藏族B基因型、2例汉族B/D基因型和1例藏族B/D基因型的存在, 也说明即使在国内其他省市几乎无D基因存在的情况下, 当汉族人定居西藏时可以感染上D基因型, 同样, 藏族人也可以感染上B基因型. 对HBV基因型在更多地区和不同民族之间分布特点的分析, 将有助于阐明其演化历史.
HBV基因分型已广泛应用于HBV 的分子流行病学、病因学和临床诊治研究, 因此, 建立一种准确、简便快速的分型方法就显得尤为重要. 目前已有核苷酸测序、PCR-RFLP、型特异引物、基因芯片等多种方法用于HBV分型.
HBV基因型是目前乙型肝炎病毒的研究热点之一, 众多文献表明HBV基因型呈明确的地域性分布. HBV基因型与其他临床资料之间的关系虽呈一定的相关性, 但各文献报道结果尚不一致. 因此, HBV基因型与疾病的关系及其相关机制需要深入研究.
尽管国内多个地区的HBV基因型分布已被报道, 但对于地处青藏高原的西藏, 其乙肝病毒基因型的分布特点及其与临床的相关性研究目前报道较少. 本文采用基因芯片技术对西藏拉萨地区的HBV感染者进行基因分型测定, 并探讨其与患者的乙肝标志物、DNA含量、性别和民族之间的关系.
通过基因芯片和DNA测序结果的比较, 表明PCR结合基因芯片具有较好的检测效果, 可以广泛用于各项HBV基因分型研究. 西藏拉萨地区HBV基因型包括B、C、D和B/D混合型, 其中以D型为主. 统计学分析3种基因型分布在不同乙肝标志物阳性、不同DNA含量和不同性别之间无差异, 但与民族存在统计学差异.
本文研究方法先进, 数据可靠, 分析客观, 有一定的学术价值.
编辑: 李军亮 电编:马文华
| 1. | Okamoto H, Tsuda F, Sakugawa H, Sastrosoewignjo RI, Imai M, Miyakawa Y, Mayumi M. Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes. J Gen Virol. 1988;69:2575-2583. [PubMed] |
| 2. | Norder H, Courouce AM, Magnius LO. Complete genomes, phylogenetic relatedness, and structural proteins of six strains of the hepatitis B virus, four of which represent two new genotypes. Virology. 1994;198:489-503. [PubMed] |
| 3. | Stuyver L, De Gendt S, Van Geyt C, Zoulim F, Fried M, Schinazi RF, Rossau R. A new genotype of hepatitis B virus: complete genome and phylogenetic relatedness. J Gen Virol. 2000;81:67-74. [PubMed] |
| 4. | Arauz-Ruiz P, Norder H, Robertson BH, Magnius LO. Genotype H: a new Amerindian genotype of hepatitis B virus revealed in Central America. J Gen Virol. 2002;83:2059-2073. [PubMed] |
| 5. | Zeng G, Wang Z, Wen S, Jiang J, Wang L, Cheng J, Tan D, Xiao F, Ma S, Li W. Geographic distribution, virologic and clinical characteristics of hepatitis B virus genotypes in China. J Viral Hepat. 2005;12:609-617. [PubMed] |
| 6. | Cui C, Shi J, Hui L, Xi H, Zhuoma , Quni , Tsedan , Hu G. The dominant hepatitis B virus genotype identified in Tibet is a C/D hybrid. J Gen Virol. 2002;83:2773-2777. [PubMed] |
| 8. | 高 俊薇, 李 雅娟, 庄 辉, 李 杰, 王 佳, 董 庆鸣, 陈 雅洁, 牛 俊奇, 马 为民, 赵 伟. 中国11城市乙型肝炎病毒慢性感染者中乙型肝炎病毒基因型分布. 中华流行病学杂志. 2007;28:315-318. |
| 9. | 庄 林, 游 晶, 陈 红英, 俞 岚, 孔 蕾, 唐 宝璋, 黄 俊华, 袁 绍明, Hutcha S, Virasakdi C. 云南地区乙型肝炎病毒基因型分布与临床的相关性. 世界华人消化杂志. 2007;15:2120-2127. [DOI] |
| 10. | 郭 瑜, 刘 晓燕, 胡 惠梅, 陈 斯勇, 伊 瑶, 陈 向伟, 毕 胜利. 山西省乙型肝炎病毒基因型别的初步研究. 中华实验和临床病毒学杂志. 2006;20:361-363. |
| 11. | 马 英, 窦 晓光, 李 智伟, 黄 芬, 乔 光彦, 闻 颖, 张 明香, 于 蛟, 赵 贺群. 沈阳地区乙型肝炎病毒基因型分子流行病学研究. 中华实验和临床病毒学杂志. 2006;20:349-351. |
| 13. | Bartholomeusz A, Schaefer S. Hepatitis B virus genotypes: comparison of genotyping methods. Rev Med Virol. 2004;14:3-16. [PubMed] |
| 14. | Zeng GB, Wen SJ, Wang ZH, Yan L, Sun J, Hou JL. A novel hepatitis B virus genotyping system by using restriction fragment length polymorphism patterns of S gene amplicons. World J Gastroenterol. 2004;10:3132-3136. [PubMed] |
| 15. | Naito H, Hayashi S, Abe K. Rapid and specific genotyping system for hepatitis B virus corresponding to six major genotypes by PCR using type-specific primers. J Clin Microbiol. 2001;39:362-364. [PubMed] |
| 16. | Yeh SH, Tsai CY, Kao JH, Liu CJ, Kuo TJ, Lin MW, Huang WL, Lu SF, Jih J, Chen DS. Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting curve analysis. J Hepatol. 2004;41:659-666. [PubMed] |
| 17. | Liu Y, Hussain M, Wong S, Fung SK, Yim HJ, Lok AS. A genotype-independent real-time PCR assay for quantification of hepatitis B virus DNA. J Clin Microbiol. 2007;45:553-558. [PubMed] |
| 18. | Osiowy C, Giles E. Evaluation of the INNO-LiPA HBV genotyping assay for determination of hepatitis B virus genotype. J Clin Microbiol. 2003;41:5473-5477. [PubMed] |
| 19. | Hussain M, Chu CJ, Sablon E, Lok AS. Rapid and sensitive assays for determination of hepatitis B virus (HBV) genotypes and detection of HBV precore and core promoter variants. J Clin Microbiol. 2003;41:3699-3705. [PubMed] |
| 20. | Tang XR, Zhang JS, Zhao H, Gong YH, Wang YZ, Zhao JL. Detection of hepatitis B virus genotypes using oligonucleotide chip among hepatitis B virus carriers in Eastern China. World J Gastroenterol. 2007;13:1975-1979. [PubMed] |
| 21. | Usuda S, Okamoto H, Tanaka T, Kidd-Ljunggren K, Holland PV, Miyakawa Y, Mayumi M. Differentiation of hepatitis B virus genotypes D and E by ELISA using monoclonal antibodies to epitopes on the preS2-region product. J Virol Methods. 2000;87:81-89. [PubMed] |
| 24. | Alavian SM, Keyvani H, Rezai M, Ashayeri N, Sadeghi HM. Preliminary report of hepatitis B virus genotype prevalence in Iran. World J Gastroenterol. 2006;12:5211-5213. [PubMed] |