修回日期: 2007-08-08
接受日期: 2007-08-28
在线出版日期: 2007-08-18
目的: 构建肝癌抗原基因cDNA噬菌体表达文库.
方法: 从肝癌细胞提取总RNA, 纯化mRNA, 用逆转录酶链式反应(RT-PCR)反转录合成cDNA第1链, LD-PCR合成cDNA第2链, 除去<500 bp小片段, 与λTripLEx2噬菌体载体连接并体外包装, 转化大肠感菌E.coli XL1-blue, 测定文库的库容和重组率, PCR鉴定插入cDNA片段大小.
结果: 构建的肝癌抗原基因cDNA噬菌体表达文库, 原始库容为3.4×106 pfu/mL, 重组率为98.2%; 文库扩增后库容为9.6×108 pfu/mL, 重组率为97.2%. 重组子插入cDNA片段大小0.6-2(平均1.4) kb.
结论: 成功构建高质量的肝癌抗原基因cDNA噬菌体表达文库, 为肝癌特异性抗原的筛选和鉴定奠定了基础.
引文著录: 丁世华, 杨冬华, 汤绍辉, 叶刚. SMART技术构建人肝癌抗原基因cDNA噬菌体表达文库. 世界华人消化杂志 2007; 15(23): 2530-2533
Revised: August 8, 2007
Accepted: August 28, 2007
Published online: August 18, 2007
AIM: To construct a cDNA phage expression library of hepatocellular carcinoma antigens.
METHODS: Total RNA was extracted from hepatocellular carcinoma cells and mRNA was purified. Single- and double-stranded cDNA were synthesized through reverse transcription polymerase chain reaction (RT-PCR) and long distance polymerase chain reaction (LD-PCR). cDNA fragments, after removing those smaller than 500 bp, were recombined to a λTripLEx2 phage vector. The recombined cDNA was packaged in vitro with Gigapack Ш Gold packing extract. A small portion of package phage was then used to infect Escherichia coli XL1-Blue for titration and determination of the percentage of recombinant clones. PCR was used to identify the size of the inserted cDNA.
RESULTS: The constructed cDNA phage expression library of hepatocellular carcinoma antigens consisted of 3.4×106 pfu/mL independent clones with a recombination rate of 98.2%. After amplification, the constructed cDNA phage expression library of hepatocellular carcinoma antigens consisted of 9.6×108 pfu/mL independent clones with a recombination rate of 97.2%. The lengths of the inserted cDNA fragments ranged from 0.6 to 2 kb, with an average of 1.4 kb.
CONCLUSION: A high-quality cDNA phage expression library of hepatocellular carcinoma antigens was successfully constructed. This is an essential step for screening and identifying tumor-specific antigens of hepatocellular carcinoma.
- Citation: Ding SH, Yang DH, Tang SH, Ye G. Switch mechanism at 5' end of mRNA template technology used to construct a cDNA phage expression library of hepatocellular carcinoma antigens. Shijie Huaren Xiaohua Zazhi 2007; 15(23): 2530-2533
- URL: https://www.wjgnet.com/1009-3079/full/v15/i23/2530.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i23.2530
我们利用噬菌体抗体库技术成功制备了3株高亲和力的特异性抗肝癌单链抗体[1-2], 筛选和鉴定这3株单链抗体识别的特异性肝癌抗原, 可以为肝癌的免疫治疗提供新的靶点. 筛选和鉴定特异性肝癌抗原的前提和关键是构建高质量的肝癌抗原基因cDNA噬菌体表达文库. 传统的cDNA文库构建方法很难包括5'端全长基因, 所建的文库代表性差, 文库筛选复杂、耗时、工作量大. 以3株人肝癌细胞为材料, 利用SMART(switch mechanism at 5' end of mRNA template)技术构建了高质量的肝癌抗原基因cDNA噬菌体表达文库, 为肝癌特异性肿瘤抗原筛选和鉴定提供了研究基础.
人肝癌细胞株HepG2、BeL-7402及SMMC-7721为本室保存; TRIzoL试剂, RPMI 1640培养液购自美国Gibco公司; 优等胎牛血清为TBD产品, 购自广州展晨生物技术公司; mRNA分离纯化试剂盒购自Premega公司; SmartTM cDNA Library Construction kit购自美国Clontech公司; 噬菌体包装蛋白Gigapack ⅢGold packing extract kit购自Stratagene公司.
培养肝癌细胞HepG2、BeL-7402及SMMC-7721至旺盛生长期, 倒去培养液后加入TRIzoL试剂, 裂解细胞并抽提总RNA, 用紫外分光光度计测定其浓度及纯度. 用Premega的mRNA试剂盒分离纯化mRNA, 10 g/L琼脂糖凝胶电泳观察. 取mRNA样品0.5 μg RT-PCR合成第1链, 加SmartⅣTM oligonucleotide随机引物5'-AAGCAGTGGTATCAACGCAG-AGTGGCCATTACGGCCGGG-3' 1 μL, CDSⅢ/3' PCR随机引物5-ATTCTAG-AGGCCGAGGCGGCCGACATG-d(T)30N-1N-3' (N = A、G、C or T; N-1 = A、G or C)1 μL, 灭菌去离子水2.55 μL, 总体积5 μL, 72℃温育2 min, 冰浴2 min. 依次加入5×First strand buffer 2.0 μL, 20 mmoL/L DTT 1.0μL, 10 mmoL/L dNTP mix 1.0 μL, 逆转录酶SupercriptⅡ 1.0 μL, 总体积5.0 μL, 42℃ 1 h, 冰浴终止反应. 用LD-PCR合成第2链, 取第1链2.0 μL, 加入灭菌去离子水80.0 μL, 10×Advantage 2 PCR buffer 10.0 μL, dNTPmix 2.0 μL, 5' PCR引物(5'-AAGCAGTGG-TATCAACGCAGAGT-3')2.0 μL, CDSⅢ/3' PCR随机引物2.0 μL, 50×Advantage 2 polymerase mix 2.0 μL, 总体积100 μL. 反应条件: 在95℃ 1 min后, 95℃ 15 s, 68℃ 6 min, 20个循环, 10 g/L琼脂糖凝胶电泳观察. 取已被蛋白酶K消化的cDNA 79 μL, 10×Sfi buffer 10 μL, SfiⅠ酶10 μL, 100×BSA 1 μL, 总体积100 μL, 50℃温育酶切2 h. 加入2 μL 10 g/L的二甲苯青蓝混匀, 短暂离心. 在经Chroma Spin-400柱子分离小于500 bp的小片段后, 用灭菌去离子水7 μL溶解沉淀. 取cDNA1.0 μL, 500 g/L已去磷酸化的λTripLEx2噬菌体载体1.0 μL, 10×Ligation buffer 0.5 μL, 10 mmoL/L ATP 0.5 μL, T4 DNA连接酶0.5 μL, 灭菌去离子水1.5 μL, 总体积5.0 μL, 16℃连接16 h. 加入包装蛋白25 μL, 22℃孵育2 h, 加入1×Lambda dilution buffer 500 μL, 加入氯仿20 μL混匀, 短暂离心. 即已构建成人肝癌抗原cDNA噬菌体表达文库.
1.2.1 文库的滴度和重组率滴定: 取原始文库1 μL, 以1×Lambda dilution buffer作1:5, 1:10, 1:20稀释, 分别取不同倍数的原始文库1 μL加入E.coli XL1-blue过夜培养物200 μL中, 37℃孵育15 min, 往混合物中加入3 mL熔化的LB/MgSO4软琼脂, 铺在LB/MgSO4 90 mm平板上, 37℃孵育15 h. 文库的滴度pfu/mL = (噬斑数×稀释倍数×103 mL/L)/铺平板用稀释库的μL数. 用IPTG和X-GaL滴定重组率, 重组率 = 白色噬斑数/(蓝色噬斑数+白色噬斑数)×100%. 文库扩增后加二甲亚砜终浓度至70 g/L, -80℃保存.
1.2.2 重组噬菌体PCR鉴定: 随机挑取13个白色噬斑分别加入至装有噬菌体稀释液30 μL中, 取噬菌体稀释液5 μL, 在反应体系50 μL中加入载体自带的随机引物各1 μL, 进行PCR扩增, 反应参数: 95℃预变性5 min, 95℃变性30 s, 68℃延伸4 min, 循环35次后, 68℃保温8 min, 10 g/L琼脂糖凝胶鉴定插入片段的大小.
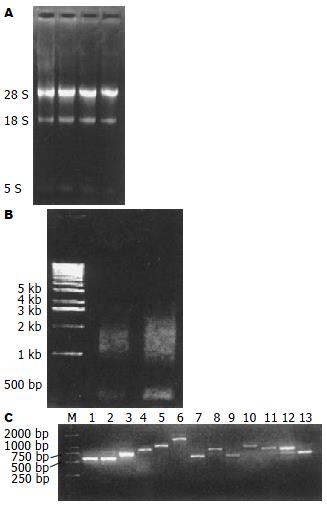
总RNA的A260/A280为1.86, 电泳可见28 S, 18 S, 5 S三条带, 28 S和18 S亮度比约为2:1, 符合建库要求(图1A). 电泳结果显示双链cDNA分布在0.5-4 kb之间, 呈长瀑布条带, 符合哺乳动物细胞DNA的分布规律(图1B). 原始文库的库容为3.4×106 pfu/mL, 重组率为98.2%; 文库扩增后库容为9.6×108 pfu/mL, 重组率为97.2%. 随机挑选13个重组噬菌体, 经PCR鉴定, 插入cDNA片段大小0.6-2(平均1.4)kb(图1C).
利用抗体寻找抗原最有效的方法是构建肝癌抗原基因cDNA噬菌体表达文库, 继而进行免疫学筛选, 获取特异性的肝癌肿瘤抗原, 而构建高质量的cDNA文库是前提和关键. 国内已开展肝癌cDNA文库构建和肝癌相关肿瘤抗原筛选方面的研究[3-6]; 但是, 传统的cDNA文库构建方法很难包括5'端全长基因, 所建的文库代表性差, 文库筛选复杂、耗时、工作量大, 很难获得特异性的肝癌肿瘤抗原. SMART方法构建cDNA文库是一种新颖而实用技术, 所构建的克隆为含有完整的5'端非翻译区的全长型cDNA, 而将此技术用于肝癌抗原基因cDNA噬菌体表达文库的构建还鲜见于报道. SMART cDNA文库构建技术, 利用一种经修饰的Oligo(dT)(CDSⅢ/3' PCR primer)引导第一链合成, 并加入SMARTⅣOligo在mRNA 5'末端作为短的延伸模板. 当逆转录到5'末端时, 逆转录酶的末端转移酶活性在第1链cDNA的3'末端加上一小段脱氧胞苷. 这些脱氧胞苷与SMARTⅣ Oligo 3'端含有的寡核苷酸(G)序列配对, 形成延伸模板, 逆转录酶转换模板并将SMART ⅣOligo链转录完全. 所合成的单链cDNA包含了完整的mRNA 5'末端, 并且附加了SMART ⅣOligo的互补链[7]. 只有那些5'末端含有SMART 锚定序列的单链cDNA才能在随后的LD-PCR过程中进行指数扩增, 不完整的cDNA和无polyA的cDNA因缺乏SMART锚定引物而无法被扩增, 可消除基因组DNA和无polyA的RNA, 并且允许用ng级的总RNA或mRNA构建高库容量、高比例的全长cDNA文库[8-10]. 新合成的cDNA 5'和3'末端分别含有非均一的sfiⅠ限制性酶切位点, 由于sfiⅠ限制性酶识别cDNA 的5'和3'末端碱基序列并不完全相同, cDNA和λTripIEx2载体经sfiⅠ酶消化后, 可以直接进行克隆连接, 并且消除了接头的自连接, 提高了文库的重组效率. 而且sfiⅠ的酶切位点在哺乳动物中罕见, cDNA经过sfiⅠ酶消化后仍可保持其完整性.
cDNA文库的质量主要反映在文库的代表性和重组cDNA片段序列完整性两个方面. 一个具有完好代表性的cDNA文库理论上应有99%的概率保证文库中包含细胞表达的任何一种mRNA序列信息, 库容量至少应具有106以上. 重组cDNA片段的完整性要求文库中重组cDNA片段为全长的cDNA, 尽可能完整地反映天然基因的结构, 插入子的片段大小平均要在1 kb以上. 我们选取3株人肝癌细胞建库, 有着相对单一的转录本库, 比新鲜肿瘤标本更具有代表性; 利用SMART方法构建的克隆含有完整的5'端非翻译区, 能够反映基因的天然结构. 我们构建的肝癌抗原基因cDNA噬菌体表达文库, 原始库容为3.4×106 pfu/mL, 重组率为98.2%; 文库扩增后库容为9.6×108 pfu/mL, 重组率为97.2%; 重组子经PCR鉴定, 插入cDNA片段大小从0.6-2(平均1.4)kb; 到达高质量文库的要求, 为肝癌特异性或相关性肿瘤抗原基因的筛选和鉴定奠定了基础.
原发性肝癌发病率高, 恶性程度 高, 预后差, 传统手术、化疗、放射治疗效果差. 肝癌特异性抗体介导的靶向治疗是肝癌治疗的新策略, 本文利用噬菌体抗体库技术成功制备了高亲和力的特异性抗肝癌单链抗体, 筛选和鉴定与其结合的抗原可为肝癌的免疫治疗提供新的靶点.
肝癌特异性肿瘤抗原的筛选和鉴定是肝癌研究的热点和难点, 高质量cDN A文库的构建是其前提和关键.
SMART方法构建的克隆含有完整的5 ' 端非翻译区的全长型cDNA,能够达到高质量cDNA文库的要求, 适合用于肝癌特异性抗原基因的筛选.
肝癌抗原基因 cDNA噬菌体表达文库的成功构建为肝癌特异性肿瘤抗原的筛选和鉴定奠定了基础.
SMART技术:利用Powerscript TMRT反转录酶的反转录、末端转移酶活性和内切酶sfiⅠ的特性,快速、简单地构建全长cDNA文库的方法.
本文为肝癌特异性抗原的筛选和鉴定成功的构建了高质量的肝癌抗原基因cDN A噬菌体表达文库, 设计科学, 方法先进, 结果明确, 有很强的科学性和可读性.
编辑: 何燕 电编:何基才
| 2. | Lu XH, Yang DH, Zhou M, Tang SH. Affinity maturation of a single-chain antibody against hepatocellular carcinoma. Zhonghua Gan Zang Bing Za Zhi. 2006;14:192-195. [PubMed] |
| 3. | Uemura M, Nouso K, Kobayashi Y, Tanaka H, Nakamura S, Higashi T, Ono T, Nakayama E, Hanafusa T, Shiratori Y. Identification of the antigens predominantly reacted with serum from patients with hepatocellular carcinoma. Cancer. 2003;97:2474-2479. [PubMed] |
| 4. | Shi YY, Wang HC, Yin YH, Sun WS, Li Y, Zhang CQ, Wang Y, Wang S, Chen WF. Identification and analysis of tumour-associated antigens in hepatocellular carcinoma. Br J Cancer. 2005;92:929-934. [PubMed] |
| 5. | Zhong X, Ran YL, Lou JN, Hu D, Yu L, Zhang YS, Zhou Z, Yang ZH. Construction of human liver cancer vascular endothelium cDNA expression library and screening of the endothelium-associated antigen genes. World J Gastroenterol. 2004;10:1402-1408. [PubMed] |
| 6. | Cui XN, Tang JW, Hou L, Song B, Ban LY. Identification of differentially expressed genes in mouse hepatocarcinoma ascites cell line with low potential of lymphogenous metastasis. World J Gastroenterol. 2006;12:6893-6897. [PubMed] |
| 7. | Wellenreuther R, Schupp I, Poustka A, Wiemann S. SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones. BMC Genomics. 2004;5:36. [PubMed] |
| 8. | Fu GK, Stuve LL. Improved method for the construction of full-length enriched cDNA libraries. Biotechniques. 2003;34:954-957. [PubMed] |
| 9. | Oh JH, Kim YS, Kim NS. An improved method for constructing a full-length enriched cDNA library using small amounts of total RNA as a starting material. Exp Mol Med. 2003;35:586-590. [PubMed] |
| 10. | Suzuki Y, Sugano S. Construction of a full-length enriched and a 5'-end enriched cDNA library using the oligo-capping method. Methods Mol Biol. 2003;221:73-91. [PubMed] |