修回日期: 2007-07-21
接受日期: 2007-07-28
在线出版日期: 2007-08-08
目的: 应用表面增强激光解吸附电离飞行时间质谱技术(SELDI-TOF-MS)对肝细胞癌组织的蛋白表达谱进行分析, 从中筛选出标记蛋白.
方法: 采用CM10芯片及SELDI-TOF-MS技术对肝细胞癌组织26例和肝硬化组织18例进行蛋白指纹图谱检测分析; 比较高、中、低分化肝细胞癌组织, 不同TNM分期肿瘤以及AFP阴、阳性肝细胞癌组织的蛋白表达差异, 所得结果均采用Biomarker Wizard软件进行分析; 通过查询蛋白库, 对特定分子质量所对应的标记蛋白进行初步确定.
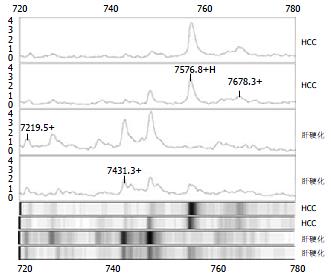
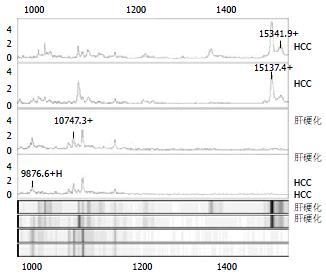
结果: 肝细胞癌和肝硬化组织蛋白表达图谱之间存在16个稳定的标志蛋白, 7个蛋白在肝细胞癌组织中表达上调, 9个蛋白在肝细胞癌组织中表达下调, 其中4.7 kDa, 7.2 kDa和9.8 kDa蛋白峰差异性最明显; 中分化和高分化肝细胞癌之间存在11个标志蛋白; 通过查询蛋白库ExPasy并进行筛选, 初步确定特定分子质量所对应的蛋白.
结论: 采用SELDI-TOF-MS技术, 证实在肝细胞癌组织中存在多种高度特异性低分子蛋白.
引文著录: 李冬, 张建中, 郑燕华, 纪小龙, 尤红, 舒清明, 范丽娜, 李享春. 蛋白芯片技术对肝细胞癌组织蛋白谱的建立及标志蛋白的筛选. 世界华人消化杂志 2007; 15(22): 2424-2430
Revised: July 21, 2007
Accepted: July 28, 2007
Published online: August 8, 2007
AIM: To use the surface-enhanced laser desorption and ionization time-of-flight mass spectrometry (SELDI-TOF-MS) technique to screen for tissue biomarkers in patients with hepatocellular carcinoma (HCC), but with different clinicopathological features.
METHODS: Proteomic spectra were examined and analyzed by mass spectroscopy in 44 cases, including 26 specimens of HCC tissue that had been pathologically confirmed in patients aged 34-68 years, and 18 specimens of liver cirrhosis tissue in patients aged 38-70 years. The spectra obtained were analyzed using the biomarker wizard system, and the biomarkers were defined by searching www.ExPasy.org.
RESULTS: A total of 16 distinguished proteomic biomarkers, 7 up-regulated and 9 down-regulated, were detected from screening HCC tissue, in contrast with liver cirrhosis tissue. There were significant differences in the protein peaks of different molecular masses of 4.7, 7.2 and 9.8 kDa between HCC and liver cirrhosis tissues. Eleven distinguished proteomic biomarkers were screened when comparing cases of moderately and highly differentiated HCC tissue. All proteins were confirmed by searches of www.ExPasy.org.
CONCLUSION: The SELDI-TOF-MS technique offers a unique platform for proteomic detection in HCC. It is also a non-invasive method for studying proteomic changes in the development and progression of HCC.
- Citation: Li D, Zhang JZ, Zheng YH, Ji XL, You H, Shu QM, Fan LN, Li XC. Using protein chip technology to screen for tissue proteomic profiles and tumor markers in hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi 2007; 15(22): 2424-2430
- URL: https://www.wjgnet.com/1009-3079/full/v15/i22/2424.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i22.2424
在全世界范围内, 肝细胞癌(HCC)在所有恶性肿瘤中的发病率居于第5位, 致死率在所有恶性肿瘤中位居第3[1], 每年大约造成1 000 000人死亡[2]. HCC通常伴发其他慢性肝病, 主要为肝硬化[3]. HCC的致病因素包括慢性病毒性肝炎、酒精肝、代谢性疾病或其他环境因素[4], 其中HBV感染是我国肝硬化和HCC的主要病因. 由于HCC对化疗不敏感, 手术, 包括病灶切除或者肝移植, 是目前唯一可能治愈HCC的疗法. 然而, 由于HCC常常晚期才表现出明显的临床症状, 因此难于早期诊断. 实际上, 仅有约15%的患者能够及时接受手术. 总体而言, HCC的5 年存活率始终低于5%[5]. 在HCC的临床治疗取得突破性进展之前, 研究重点是发现新的治疗靶分子以及早期诊断分子标记物. 最近的研究已经证实, 与甲胎蛋白相比, 还存在其他有诊断价值的分子标记物[6-7], 然而, 到目前为止, 还没有哪种标记物具有足够的特异性; 同时, 大多数相关研究都是关联性或回顾性的. 因此, 有必要采用新的蛋白质组学方法寻找具有HCC诊断、预后和治疗意义的蛋白分子. 最直接的方法是采用蛋白质谱对HCC组织和血清进行分析, 从中寻找差异蛋白, 因为恶性肿瘤的起始和演进都源于蛋白表达水平改变及其累积效应.
传统的双向凝胶电泳(2D-PAGE)是分离复杂蛋白复合物时最常用的方法[8], 并且在HCC蛋白组学研究中应用最为广泛. 2D-PAGE可以同时观察到上千个蛋白位点, 允许对蛋白进行分离, 可重复性好, 并且具有足够的敏感度. 但2D-PAGE并不是一种可以进行大规模样本分析的技术, 也无法观察到所有种类的蛋白(例如膜蛋白, 低丰度蛋白). Ciphergen公司的蛋白芯片技术通过将固相色谱和质谱测量结合[9-11], 可以对组织液、细胞裂解液等生物样本进行分析. 这种所谓的表面增强激光解吸附和离子化飞行时间(SELDI-TOF)方法由Yip et al[12]首次描述, 他利用芯片表面的具有特殊亲和力的分子和样本中的蛋白结合, 在激光照射下使其解吸附, 通过特定电场, 并根据其带电量的多少和通过电场的时间来推算出分子质量, 可以俘获fmoL级的小分子(<2500 kDa)、低丰度蛋白质, 大大提高了实验的精确性和特异性.
蛋白芯片技术目前已经广泛应用于各种癌症的血清学分析[13-14], 但少见于对新鲜组织进行的分析[15-16]. 我们通过对肝细胞癌和肝硬化组织蛋白指纹图谱的初步观察, 筛选HCC特异的标志蛋白, 并分析不同病理学特征肿瘤组织蛋白表达的差异, 旨在发现并鉴定参与HCC肿瘤发生有关的各种蛋白, 并探讨其在临床诊断、预后以及治疗上的意义.
尿素、乙腈、三氟乙酸、Tris-HCl pH9.0、CHAPS、DTT、NaAc、HPLC H2O、Hepes购自Sigma公司, 丝氨酸蛋白酶抑制剂(PMSF)购自Solarbio公司. 收集解放军306医院武警总医院及北京友谊医院手术时切除的新鲜HCC组织26例, 男25例, 女1例, 平均年龄55.5(34-68)岁, 低分化3例, 中分化13例, 高分化10例, 癌结节直径大于2 cm 15例, 小于2 cm 11例, 根据免疫组化染色甲胎蛋白(AFP)阳性15例, 阴性11例; 收集武警总医院肝移植手术中切除的肝硬化组织18例, 平均年龄52(30-70)岁, 男15例, 女3例. 所有新鲜肝癌组织均仔细切割, 保证未带有癌旁组织, 手术切除后的组织被立刻投入液氮中. 材料被分为肿瘤和非肿瘤2组. 蛋白飞行时间质谱(protein chip biology system): 美国Ciphergen公司生产, 型号PBSⅡ-C. 蛋白质芯片: 采用CM10(弱阳离子交换芯片), 表面结合弱阴型离子羧基, 可以和被分析物表面的正电荷基团相互作用而俘获蛋白, 用于检测高等电点的蛋白质和生物标记物.
融解冰冻组织, 每个样品取100 mg, 放入匀浆器中加U9缓冲液1 mL和少量PMSF在冰上将组织匀浆. 匀浆后液体取出置于1.5 mL离心管中以8500 g 4℃离心10 min. 每个样品取750 μL置于2 mL专用离心管, 以26000 g 15℃离心2 h. 分离上清液并以50 μL分装冻存于-80℃. 实验前采用考马斯亮蓝法确定裂解液中蛋白含量在2-10 g/L. 将已加样处理的CM10芯片装入Bioprocessor中, 每孔加入 200 μL结合缓冲液, 室温振荡洗涤2次, 每次5 min, 甩干. 每孔加入样品混合液100 μL, 振荡孵育1 h, 甩去样品, 用洗脱缓冲液(100 mmol/L, NaAc, pH4.0) 200 μL室温振荡洗涤2次, 每次5 min, 甩干; 再用HPLC H2O洗涤1次, 立即甩干. 拆开Bioprocessor, 取出芯片, 晾干后, 每点加2次SPA 0.5 μL, 晾干后上机测试. 采用蛋白飞行质谱仪对结合在弱阳离子CM10芯片上的血清蛋白进行读取分析, 设定最高检测分子质量为200 kDa, 优化范围为2-20 kDa, 激光强度185, 检测敏感度为9. 考虑到基质峰的存在, 将1 kDa以下的峰滤去, 以免基质峰对结果造成影响. 仪器每天采用塞弗吉公司提供的ALL-IN-ONE蛋白质标准分子校正, 系统的质量偏差≤0.1%. 采用Ciphergen Proteinchip3.1版本的分析软件自动采集数据.
统计学处理 采用Ciphergen Proteinchip 3.1软件对数据进行统计学处理, 确定两组间蛋白峰比较时, P<0.01时具有统计学意义, 以Biomarker patterns system软件分析数据建模.
对HCC和非HCC组蛋白表达谱进行对比分析后, 发现两组间存在16个稳定的差异表达蛋白, 其中7个表达上调, 9个表达下调(表1, 图1-2). 通过检索ExPasy蛋白数据库(http: //http://www. expasy.org), 初步确定特定分子质量所对应的候选蛋白, 检索时分子质量设定为质/核比±0.1%, PI值9.0±5.0. 检索结果见表2. 分析检索结果表明, 一些蛋白或肽分子只有一个候选结果, 可初步判定检索结果即为该分子质量标记蛋白, 其他多候选结果者, 根据其组织来源、生物学功能, 初步进行筛选(表3). 根据AFP免疫组化染色结果, 将HCC组织病例分为AFP(-)和AFP(+)组, 对其蛋白表达谱进行分析, 结果显示二者之间并没有统计学差异.
| 分子质量(Da) | 蛋白表达 | P |
| 2273 | ↓ | 0.0077157109 |
| 2641 | ↓ | 0.0025471256 |
| 4524 | ↓ | 0.0065551667 |
| 4674 | ↓ | 0.0001131722 |
| 4974 | ↑ | 0.0071142201 |
| 6758 | ↓ | 0.0083623824 |
| 7219 | ↓ | 0.000175627 |
| 7431 | ↓ | 0.0077157109 |
| 7576 | ↑ | 0.0055540603 |
| 7678 | ↑ | 0.002125878 |
| 9846 | ↓ | 0.0000063936 |
| 10747 | ↓ | 0.0002695077 |
| 15137 | ↑ | 0.0046930365 |
| 15341 | ↑ | 0.0051071705 |
| 31037 | ↑ | 0.0036265565 |
| 31748 | ↑ | 0.0016126005 |
| 分子量(Da) | 检索结果 |
| Anaphase-promoting complex subunit 11 | |
| (Q9NYG5) | |
| Cyclin-dependent kinases regulatory sub- | |
| unit 2 (P33552) | |
| Pro-MCH-like protein 2 (Q9BQD1) | |
| Protein transport protein Sec61 subunit | |
| beta (P60468) | |
| 9846.428 | Caspase-9 subunit p10 (P55211) |
| 4674.091 | Bax protein, cytoplasmic isoform gamma |
| (Q07815) | |
| 7219.294 | Platelet basic protein precursor (P02775) |
| Cholecystokinins precursor (P06307) | |
| NADH-ubiquinone oxidoreductase chain 4L | |
| (P03901) | |
| 10747.82 | Protein S100-A5 (P33763) |
| 31748.55 | Nit protein 2 (Q86X76) |
| S-adenosylmethionine decarboxylase | |
| proenzyme (P17707) | |
| 7678.362 | Platelet factor 4 variant precursor (P10720) |
| 2641.05 | Endothelin-3 precursor (P14138) |
| 31037.76 | enoyl-coenzyme A hydratase 1, short- |
| chain (Q58EZ5) | |
| vascular endothelial growth factor (VEGF) | |
| isoform | |
| 15137.76 | Growth/differentiation factor 9 precursor |
| (O60383) | |
| Beta-glucuronidase-like protein 1 | |
| (Q9NQQ0) | |
| Interleukin-2 precursor (P09105) | |
| 15341.82 | Interleukin-2 precursor (P60568) |
| 7576.037 | Dipeptidyl-peptidase 1 precursor (P53634) |
| 4524.928 | Orexigenic neuropeptide QRFP precursor |
| (P83859) | |
| Beta-defensin 107A precursor (Q8IZN7) | |
| 4974.046 | Gastric inhibitory polypeptide precursor |
| (P09681) | |
| 2273.905 | Prolactin-releasing peptide precursor |
| (P81277) | |
| Guanine nucleotide-binding protein G(I)/ | |
| G(S)/G(O) gamma-T2 subunit precursor | |
| (O14610) | |
| 7431.591 | Growth-regulated protein alpha precursor |
| (P09341) | |
| Cytochrome c oxidase polypeptide VIIa- | |
| heart, mitochondrial precursor (P24310) | |
| 6758.022 | WAP four-disulfide core domain protein |
| 10A precursor (Q9H1F0) |
| 分子量(Da) | 确定的HCC蛋白(肽分子) |
| 9846.428 | Caspase-9亚单位p10 |
| 4674.091 | BAX蛋白, 胞质异构体gamma |
| 7219.294 | 血小板碱性蛋白前体 |
| 10747.82 | 胆囊收缩素前体 |
| 31748.55 | Nit蛋白2 |
| 7678.362 | 血小板因子Ⅳ变异体前体 |
| 2641.05 | 内皮素3前体 |
| 31037.76 | 烯酰辅酶A水解酶1, 短链 |
| 15137.76 | 血管内皮生长因子(VEGF)异构体 |
| 15341.82 | β-葡糖苷酸酶样蛋白1 |
| 7576.037 | 二肽基肽酶1前体 |
| 4524.928 | 促食肽QRFP前体 |
| 4974.046 | β-防卫素107A前体 |
| 2273.905 | 催乳素释放肽前体 |
| 7431.591 | 生长调节蛋白α前体 |
| 6758.022 | WAP 4-二硫化物核心区蛋白10A前体 |
通过对3例低分化, 13例中分化和10例高分化HCC的蛋白表达谱进行对比分析, 寻找具有统计学意义的标记蛋白. 低分化组和中分化组、高分化组蛋白表达谱之间都没有统计学意义的差异. 中分化组和高分化组蛋白表达谱之间存在11个具有统计意义的标记蛋白, 其中3个在中分化HCC中低表达, 8个在中分化HCC中高表达. 通过检索ExPasy蛋白数据库, 初步确定特定分子质量所对应的候选蛋白, 检索时分子质量设定为质/核比±0.1%, PI值9.0±5.0. 根据组织来源和生物学功能, 对所得候选蛋白(肽段)进行筛选(表4).
| 分子量(Da) | 蛋白表达 | P | 对应HCC蛋白(肽段) |
| 10945.197 | ↓ | 0.00919 | 60S 核糖体蛋白 L37(P61927) |
| 11675.955 | ↓ | 0.000349 | 糖蛋白激素α-2前体(Q96T91) |
| 11893.256 | ↓ | 0.003095 | Caspase-2前体(P42575) |
| 2553.324 | ↑ | 0.005411 | 内皮素-2前体(P20800) |
| 2911.205 | ↑ | 0.007077 | Orexin前体(O43612) |
| 3099.609 | ↑ | 0.00919 | 神经肽B前体(Q8NG41) |
| 3627.127 | ↑ | 0.00929 | Apelin前体(Q9ULZ1) |
| 6186.219 | ↑ | 0.005411 | 60S核糖体蛋白L40(P62987) |
| 6554.271 | ↑ | 0.004107 | 表面活性相关蛋白G(Q6UW10) |
| 9627.260 | ↑ | 0.005411 | 细胞色素C氧化酶多肽VIa-liver, 线粒体前体(P12074) |
| 7155.589 | ↑ | 0.00919 | FXYD区粒子 迁 移调节子3前 体(Q14802) |
目前, HCC发生的分子机制还不是十分清楚, 但经常观察到非整倍体和多基因改变, 如P53, β-catenin, c-myc以及cyclin-D1[17-19], 同时杂合子丢失(LOH)也可伴有肿瘤抑制基因的失活[20-22]. 此外, 一些生长因子也证明参与HCC的发生, 其中AFP已被证实与HCC的侵袭性相关. 然而, 这些发现并不能整合成一个清晰的HCC蛋白谱, 同时, 这些生物标记物不具有足够的生物特异性, 因为HCC是一种异质性肿瘤[23], 在分子和亚细胞水平可能存在肿瘤发生发展过程中的多重改变. 为了解决这一问题, 需要可以在蛋白和RNA/DNA水平上进行复杂分析的自动化和高通量技术. 蛋白组学继基因组学之后, 更彻底的改变我们对肿瘤生物学的理解. 通过比较正常组织和病变组织或体液(血清、尿液等)中的蛋白改变, 可以鉴定出与肿瘤进展侵袭和转移相关的蛋白, 进一步推断出肿瘤复发和转移的可能性. 此外还可以发展出更具有特异性的个性化肿瘤治疗. 然而, 相关的新标记物仅在个别肿瘤中得到初步建立[24]. SELDI技术是发现肿瘤标记物最有希望的工具之一[25-26], 被广泛应用于体液以及组织蛋白的分析, 但不论是血清还是组织, 个体之间的蛋白表达谱都存在差异. 性别、激素水平、炎症都可以明显改变蛋白表达谱, 因此, 具有特异性的蛋白必须有很高的表达水平, 以便在这些差异分析中被观察到. SELDI技术可以得出蛋白或肽段的近似分子质量, 但却无法鉴定是何种蛋白, 较简便的解决方法是采用查询蛋白库, 如ExPasy蛋白文库, 可以初步确定某分子质量代表何种蛋白. 但是, 这种办法也有其局限性, 许多蛋白在生物体内是经过修饰, 如糖基化或甲基化, 分子质量已发生变化, 因此, 检索时可能出现误差. 就目前而言, 最准确的方法仍需要将蛋白纯化后再进行鉴定.
根据此前进行的预实验[27], 本实验在蛋白检测时采用CM10(弱阳离子)芯片, 而未采Chiphergen公司的强阴离子交换芯片、疏水芯片和固定金属亲和表面芯片, 以便能够俘获更多的差异蛋白, 但我们并不排除其他芯片也可能获得少量蛋白峰. HCC和肝硬化组之间共有16个具有统计意义的标记蛋白, 其中差异最明显的蛋白峰出现4674, 7219和9846 Da, 通过蛋白库检索, 可以初步确定其代表Bax蛋白细胞质异构体γ, 血小板基本蛋白前体和细胞周期蛋白激酶调节亚基, Bax是一种细胞凋亡的促进因子, 其促进凋亡的作用可能通过诱导Caspase的释放, 激活来自线粒体的细胞色素C实现. 目前认为, Bax是一种具有抑癌作用的蛋白分子[28]. 与之相对应, 9846 Da对应的应为Caspase-9亚单位P10. Caspase是半胱氨酸蛋白的一种酶, 在细胞凋亡中发挥关键性作用. Caspase家族正常情况下均以无活性的酶原形式存在, 当细胞接受死亡信号时, Caspase酶原被蛋白酶裂解而活化, 在目前研究已知的14种Caspase中, Caspase-3, -8, -9和细胞凋亡的关系最为密切. 考虑到9846 Da蛋白在HCC组织和肝硬化组织中表达的高特异性, 该蛋白的低表达很可能在肝硬化向HCC转化过程中发挥了重要作用, 并且有可能成为新的HCC诊断和治疗靶分子.
通过结合检索结果和Melle et al[29]以前的研究, 有理由认为15.13 kDa代表一个糖基化的血管内皮生长因子异构体(VEGF110+ N-Glyc, 分子质量15128 Da). VEGF可以促进微血管形成, 促进肿瘤侵袭和转移, 是目前肿瘤病理学和免疫治疗的一个热点. 31.74 kDa和31.03 kDa分别代表Nit蛋白2(Nit 2)和乙酰辅酶A裂解酶1(enoyl -coenzyme A hydratase 1)的短链, 这也和先前的研究相符[30], Nit2和乙酰辅酶A裂解酶均参与线粒体的能量代谢, 他们在HCC中的高表达可能与肿瘤细胞旺盛的能量消耗有关.
在中分化、高分化HCC组织蛋白中存在11个标记蛋白, 其中11893 Da在中分化HCC中低表达, 经检索发现其应为Caspase-2前体. Caspase2作为细胞凋亡级联反应的早期因子, 可通过调控Caspase-8, -9参与细胞凋亡, 目前认为通常定位于细胞核, 但对于其在肝脏中的表达情况尚缺乏相关研究. 2553 Da对应的蛋白经分析应为内皮素(endothelin)前体, 内皮素是一组缩血管活性很强的多肽物质, 其作用十分广泛且与恶性肿瘤生长有关. 已有研究证明, HCC患者血清的内皮素水平要明显高于正常人群[31], 但本实验首次证实其在HCC组织中的存在. 同时, 分析发现中分化与低分化HCC、高分化和低分化HCC之间并不存在具有明显差异的标记蛋白, 分析其原因, 应当是低分化HCC的病例太少(3例), 不具有统计学意义. 根据AFP表达情况将HCC分为阳性(15例)和阴性(11例)对照分析后发现, 两组肿瘤之间并不存在明显差异. 分析其原因, 一是因为AFP属于大分子蛋白(70 kDa), 超出SELDI的检测范围, 二是因为不同病理医生在诊断时的尺度把握有所差异, 以及免疫组化染色存在差异, 造成AFP阴性、阳性没有绝对意义. 如果以患者血清AFP水平为分组标准, 则可能发现某些有统计意义的差异蛋白.
比较本实验与此前进行的HCC血清标记蛋白的研究, 并未发现二者的蛋白谱之间存在明显相关性, 但由于缺乏HCC组织对应的血清样本, 无法进行相应分析. Scarlett et al[32]采用SELDI-TOF-MS技术对胆管癌(CC)患者的血清和组织蛋白进行分析后, 曾发现一个重合的稳定高表达的蛋白峰. 考虑到血清学检测是HCC早期诊断最有效的手段, 因此, 在以后的实验重点应采用SELDI-TOF-MS技术对同一病例组织和血清的蛋白谱进行分析比较, 进一步完善具有诊断和预后意义的HCC蛋白表达谱系.
总的来说, 此研究显示蛋白芯片技术可以绘制较完整的HCC低分子质量差异蛋白表达谱, 并用来找到新的肿瘤蛋白标记. 然而, 鉴于SELDI蛋白质谱技术的局限性, 还需要进一步的研究, 如分析这些蛋白在血清中的表达是否有所差异、分离纯化并鉴定这些蛋白, 这将会有助于更深层次的理解HCC基因学、蛋白组学改变以及其对肿瘤发生机制的认识.
肝细胞癌作为最常见和恶性程度较高的恶性肿瘤之一, 难于早期诊断, 仅有少数患者能够及时接受手术, 因此在HCC临床治疗没有取得突破性进展之前,研究重点是建立完整的HCC差异蛋白表达谱, 并从中发现新的治疗靶分子以及早期诊断的分子标记物.
目前发现除甲胎蛋白以外, 还存在其他具有HCC诊断价值的分子标记物. 采用新的蛋白质组学方法, 可以直接对HCC组织和血清进行分析, 具有灵敏度 高、检测样本量大的优点. 其中, SELDI蛋白指纹技术作为一种较新的质谱技术, 擅长于检测小分子低丰度蛋白分子. 目前其在美国已应用于卵巢癌和前列腺癌的诊断, 其特异性和敏感性远远高于传统方法.
目前利用SELDI技术已发现HCC患者和正常人的血清蛋白质指纹图谱之间有17个稳定的标志蛋白,并筛选出两个标志蛋白建立起一个HCC的诊断模型, 对HCC的诊断特异性为97.06%,灵敏度为91.18%.
我国几乎所有 HCC均起源于肝炎后肝硬化, 与国外HCC发病情况相比, 病因学有很大不同, 本文为建立适用于我国人群的肝细胞癌差异蛋白库提供了实验依据, 在寻找特异诊断分子和临床治疗靶分子方面具有重要的意义.
本文采用蛋白芯片技术建立HCC组织蛋白谱, 并筛选出一些标志蛋白, 为下一步进行分离纯化鉴定这些蛋白提供了依据, 有助于更深层次的理解HCC基因组学和蛋白组学改变.
表面增强激光解吸电离飞行时间质谱(SELDI-TOF-MS): 该技术主要是在飞行质谱的检测系统中, 芯片特异性地与被测标本中蛋白结合, 再通过选择性清洗, 获得高分辨率的保留蛋白质谱. 当加入能量吸收分子后, 芯片上保留的蛋白形成晶体. 在激光照射下, 晶体发生解离作用, 带电分子在通过电场时加速, 检测仪记录飞行时间的长短, 质荷比(M/Z)越小,飞行时间越短, 就会被最先检测到. 被测定蛋白质以一系列峰值的形式呈现.
本文采用蛋白芯片技术对肝细胞癌组织中的标志蛋白进行了筛选, 采用的技术先进, 设计合理, 得出的结果和结论较可信, 有一定的参考价值.
编辑: 何燕 电编:郭海丽
| 1. | Lee SG, Rho HM. Transcriptional repression of the human p53 gene by hepatitis B viral X protein. Oncogene. 2000;19:468-471. [PubMed] |
| 2. | El-Serag HB, Rudolph KL. Hepatocellular carcinoma: epidemiology and molecular carcinogenesis. Gastroenterology. 2007;132:2557-2576. [PubMed] |
| 3. | del Olmo JA, Serra MA, Rodriguez F, Escudero A, Gilabert S, Rodrigo JM. Incidence and risk factors for hepatocellular carcinoma in 967 patients with cirrhosis. J Cancer Res Clin Oncol. 1998;124:560-564. [PubMed] |
| 4. | Tiribelli C, Melato M, Croce LS, Giarelli L, Okuda K, Ohnishi K. Prevalence of hepatocellular carcinoma and relation to cirrhosis: comparison of two different cities of the world--Trieste, Italy, and Chiba, Japan. Hepatology. 1989;10:998-1002. [PubMed] |
| 5. | El-Shanawani FM, Abdel-Hadi AA, Abu Zikri NB, Ismail A, El-Ansary M, El-Raai A. Clinical significance of aflatoxin, mutant P53 gene and sIL-2 receptor in liver cirrhosis and hepatocellular carcinoma. J Egypt Soc Parasitol. 2006;36:221-239. [PubMed] |
| 6. | Qin LX, Tang ZY. The prognostic significance of clinical and pathological features in hepatocellular carcinoma. World J Gastroenterol. 2002;8:193-199. [PubMed] |
| 7. | Nakatsura T, Yoshitake Y, Senju S, Monji M, Komori H, Motomura Y, Hosaka S, Beppu T, Ishiko T, Kamohara H. Glypican-3, overexpressed specifically in human hepatocellular carcinoma, is a novel tumor marker. Biochem Biophys Res Commun. 2003;306:16-25. [PubMed] |
| 8. | Klose J, Kobalz U. Two-dimensional electrophoresis of proteins: an updated protocol and implications for a functional analysis of the genome. Electrophoresis. 1995;16:1034-1059. [PubMed] |
| 9. | Davies H, Lomas L, Austen B. Profiling of amyloid beta peptide variants using SELDI Protein Chip arrays. Biotechniques. 1999;27:1258-1261. [PubMed] |
| 10. | Jr GW, Cazares LH, Leung SM, Nasim S, Adam BL, Yip TT, Schellhammer PF, Gong L, Vlahou A. Proteinchip(R) surface enhanced laser desorption/ionization (SELDI) mass spectrometry: a novel protein biochip technology for detection of prostate cancer biomarkers in complex protein mixtures. Prostate Cancer Prostatic Dis. 1999;2:264-276. [PubMed] |
| 11. | Merchant M, Weinberger SR. Recent advancements in surface-enhanced laser desorption/ionization-time of flight-mass spectrometry. Electrophoresis. 2000;21:1164-1177. [PubMed] |
| 12. | Yip TT, Hutchens TW. Mapping and sequence-specific identification of phosphopeptides in unfractionated protein digest mixtures by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. FEBS Lett. 1992;308:149-153. [PubMed] |
| 13. | Jain KK. Role of proteomics in diagnosis of cancer. Technol Cancer Res Treat. 2002;1:281-286. [PubMed] |
| 14. | 高 春芳, 赵 光, 宋 国英, 李 冬晖, 王 秀丽, 许 洋, 马 龙化. 用飞行时间质谱技术筛选结直肠癌患者中特异性生物标记物的临床意义. 中华检验医学杂志. 2003;26:658-661. |
| 15. | Craven RA, Banks RE. Laser capture microdissection and proteomics: possibilities and limitation. Proteomics. 2001;1:1200-1204. [PubMed] |
| 16. | Wulfkuhle JD, McLean KC, Paweletz CP, Sgroi DC, Trock BJ, Steeg PS, Petricoin EF 3rd. New approaches to proteomic analysis of breast cancer. Proteomics. 2001;1:1205-1215. [PubMed] |
| 17. | Nhieu JT, Renard CA, Wei Y, Cherqui D, Zafrani ES, Buendia MA. Nuclear accumulation of mutated beta-catenin in hepatocellular carcinoma is associated with increased cell proliferation. Am J Pathol. 1999;155:703-710. [PubMed] |
| 18. | Peng SY, Lai PL, Hsu HC. Amplification of the c-myc gene in human hepatocellular carcinoma: biologic significance. J Formos Med Assoc. 1993;92:866-870. [PubMed] |
| 19. | Nishida N, Fukuda Y, Komeda T, Kita R, Sando T, Furukawa M, Amenomori M, Shibagaki I, Nakao K, Ikenaga M. Amplification and overexpression of the cyclin D1 gene in aggressive human hepatocellular carcinoma. Cancer Res. 1994;54:3107-3110. [PubMed] |
| 20. | Kuroki T, Fujiwara Y, Tsuchiya E, Nakamori S, Imaoka S, Kanematsu T, Nakamura Y. Accumulation of genetic changes during development and progression of hepatocellular carcinoma: loss of heterozygosity of chromosome arm 1p occurs at an early stage of hepatocarcinogenesis. Genes Chromosomes Cancer. 1995;13:163-167. [PubMed] |
| 21. | De Souza AT, Hankins GR, Washington MK, Fine RL, Orton TC, Jirtle RL. Frequent loss of heterozygosity on 6q at the mannose 6-phosphate/insulin-like growth factor II receptor locus in human hepatocellular tumors. Oncogene. 1995;10:1725-1729. [PubMed] |
| 22. | Nishida N, Fukuda Y, Kokuryu H, Sadamoto T, Isowa G, Honda K, Yamaoka Y, Ikenaga M, Imura H, Ishizaki K. Accumulation of allelic loss on arms of chromosomes 13q, 16q and 17p in the advanced stages of human hepatocellular carcinoma. Int J Cancer. 1992;51:862-868. [PubMed] |
| 23. | Thorgeirsson SS, Grisham JW. Molecular pathogenesis of human hepatocellular carcinoma. Nat Genet. 2002;31:339-346. [PubMed] |
| 24. | van 't Veer LJ, Dai H, van de Vijver MJ, He YD, Hart AA, Mao M, Peterse HL, van der Kooy K, Marton MJ, Witteveen AT. Gene expression profiling predicts clinical outcome of breast cancer. Nature. 2002;415:530-536. [PubMed] |
| 25. | Petricoin EF, Ardekani AM, Hitt BA, Levine PJ, Fusaro VA, Steinberg SM, Mills GB, Simone C, Fishman DA, Kohn EC. Use of proteomic patterns in serum to identify ovarian cancer. Lancet. 2002;359:572-577. [PubMed] |
| 26. | Rosty C, Christa L, Kuzdzal S, Baldwin WM, Zahurak ML, Carnot F, Chan DW, Canto M, Lillemoe KD, Cameron JL. Identification of hepatocarcinoma-intestine-pancreas/pancreatitis-associated protein I as a biomarker for pancreatic ductal adenocarcinoma by protein biochip technology. Cancer Res. 2002;62:1868-1875. [PubMed] |
| 28. | Oltvai ZN, Milliman CL, Korsmeyer SJ. Bcl-2 heterodimerizes in vivo with a conserved homolog, Bax, that accelerates programmed cell death. Cell. 1993;74:609-619. [PubMed] |
| 29. | Melle C, Kaufmann R, Hommann M, Bleul A, Driesch D, Ernst G, von Eggeling F. Proteomic profiling in microdissected hepatocellular carcinoma tissue using ProteinChip technology. Int J Oncol. 2004;24:885-891. [PubMed] |
| 30. | Melle C, Ernst G, Scheibner O, Kaufmann R, Schimmel B, Bleul A, Settmacher U, Hommann M, Claussen U, von Eggeling F. Identification of specific protein markers in microdissected hepatocellular carcinoma. J Proteome Res. 2007;6:306-315. [PubMed] |
| 31. | Ishibashi M, Fujita M, Nagai K, Kako M, Furue H, Haku E, Osamura Y, Yamaji T. Production and secretion of endothelin by hepatocellular carcinoma. J Clin Endocrinol Metab. 1993;76:378-383. [PubMed] |
| 32. | Scarlett CJ, Saxby AJ, Nielsen A, Bell C, Samra JS, Hugh T, Baxter RC, Smith RC. Proteomic profiling of cholangiocarcinoma: diagnostic potential of SELDI-TOF MS in malignant bile duct stricture. Hepatology. 2006;44:658-666. [PubMed] |