修回日期: 2007-04-11
接受日期: 2007-04-17
在线出版日期: 2007-06-28
目的: 研究组蛋白去乙酰酶抑制剂曲古菌素A (TSA)对结肠癌细胞Caco2增殖的影响和机制.
方法: 5-500 μg/L TSA处理结肠癌Caco2细胞0-5 d, 在药物处理前及36 h后用MTT法检测细胞增殖状况. 流式细胞仪检测细胞周期的改变. RT-PCR检测ID4 mRNA的表达.
结果: TSA在100 μg/L浓度以上可以明显抑制结肠癌Caco2细胞的增殖(P<0.05), 但在5和20 μg/L浓度时抑制作用不明显. 100 μg/L TSA可导致细胞G1期阻滞, 但没有诱导明显的细胞凋亡. RT-PCR显示20 μg/L TSA作用36 h后ID4 mRNA表达增强(P<0.05), 在100 μg/L其表达显著增强(P<0.01).
结论: TSA可以通过阻滞Caco2细胞的G1期和重新表达ID4来发挥抑癌作用.
引文著录: 张波, 陈剑英, 王国斌, 陈道达. 组蛋白去乙酰酶抑制剂对结肠癌细胞增殖和ID4基因表达的影响. 世界华人消化杂志 2007; 15(18): 2021-2025
Revised: April 11, 2007
Accepted: April 17, 2007
Published online: June 28, 2007
AIM: To investigate whether Trichostatin A (TSA) possesses anti-tumor activity against human colon cancer cells.
METHODS: The human colon cancer cell line Caco2 was treated with different concentrations of TSA, a potent and specific histone deacetylase inhibitor, for 0 to 5 d. The growth of Caco2 cells was observed by MTT assay before and after TSA treatment. The Caco2 cell cycle was analyzed by flow cytometry. The expression of ID4 mRNA was observed by reverse transcription-polymerase chain reaction.
RESULTS: Trichostatin A in a dose-dependent fashion significantly inhibited the proliferation of colon cancer cells at 100 μg/L (P < 0.05). The inhibition rate increased sharply from day 3 to 5 (57.3% to 70.2%). TSA treatment induced cell cycle arrest at the G1 phase but did not increase apoptotic cell death as shown by flow cytometry. ID4 mRNA was expressed in Caco2 cells after 20 μg/L TSA treatment, but it was only weakly detectable before treatment.
CONCLUSION: Our results demonstrated that TSA inhibited colon cancer cell growth in vitro, possibly through G1 cell cycle arrest and re-expression of the ID4 gene. This study suggested that TSA may be a potential therapeutic agent for the treatment of colon cancer.
- Citation: Zhang B, Chen JY, Wang GB, Chen DD. Effects of histone deacetylase inhibitor on colon cancer cell growth and ID4 gene expression. Shijie Huaren Xiaohua Zazhi 2007; 15(18): 2021-2025
- URL: https://www.wjgnet.com/1009-3079/full/v15/i18/2021.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i18.2021
核小体中心组蛋白的乙酰化(acetylation)和去乙酰化(deacetylation)是调节染色体结构和基因转录的重要修饰方法[1]. 具有转录活性的基因其中心组蛋白的乙酰化程度非常高, 相反转录抑制的基因则乙酰化程度很低. 组蛋白乙酰转移酶(histone acetyltransferase, HAT)和组蛋白去乙酰酶(histone deacetylase, HDAC)是调节染色体结构和基因表达的关键激酶[2-3]. HAT通过组蛋白乙酰化提高一些转录因子的活性, 以协助启动子区域进入转录复合体; HDAC则起着相反的作用[4-5]. 曲古菌素A(trichostatin A, TSA)是一种抗真菌药物, 也是强大的HDAC抑制剂, 研究显示他可以抑制一些肿瘤细胞的生长[6-9]. 我们探讨TSA调节结肠癌Caco2细胞增殖的机制.
Caco2细胞于含100 mL/L胎牛血清、100 kU/L青霉素、100 g/L链霉素的DMEM培养基(Gibco公司)中, 置含50 mL/L CO2, 37℃的培养箱. Trichostatin A (Sigma公司)溶解于二甲基亚砜(DMSO), 配制成1×104 μg/L的储存液, 在培养基中稀释成终浓度为5, 20, 100和500 μg/L的工作液.
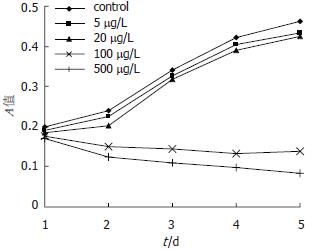
1.2.1 MTT法检测细胞增殖活性: 以每孔1×103个细胞接种于96孔板, 各组设6个复孔. 对照组细胞以常规DMEM培养, 药物处理组以含有5, 20, 100和500 μg/L TSA的DMEM中培养. 待细胞贴壁后, 每隔24 h检测1次. 每孔加入5 g/L MTT 20 μL, 置培养箱内温育4 h, 小心吸去培养基, 加入DMSO 150 μL, 37℃水平摇床振摇15 min, 待晶体充分溶解后, 置全自动酶标仪于490 nm波长处读取吸光度(A). 细胞增殖能力以平均吸光度(A)值分析, 以A值为纵坐标, 时间(d)为横坐标, 绘制生长曲线. 细胞生长抑制率% = (1-实验组平均A值/对照组平均A值)×100%, 以抑制率%为纵坐标, 时间(d)为横坐标, 绘制抑制率曲线.
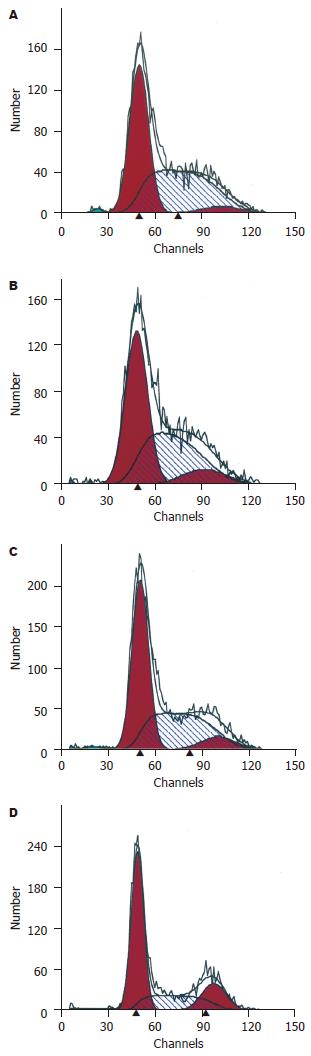
1.2.2 细胞周期的检测: 5, 20和100 μg/L TSA处理的细胞及对照组细胞均以1×106个接种于60 mm培养皿中, 各组设平行实验3个. 36 h后用胰蛋白酶消化分别收集对照组和处理组细胞, PBS洗涤细胞2次, 700 mL/L乙醇固定. 检测前离心弃乙醇, PI溶液(含1.5 g/L碘化丙锭, 10 g/L RNase A)于37℃避光染色3 h, 以流式细胞仪(BD公司)进行细胞周期检测.
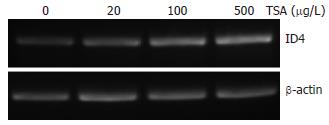
1.2.3 RT-PCR检测ID4基因的表达: 细胞总RNA的提取参照RNAiso试剂盒(TaKaRa公司)操作. 逆转录反应体系10 μL含5 mmol/L MgCl2, 1 mmol/L dNTPs, 167 nKaT RNase抑制剂, 42 nKaT AMV逆转录酶, 25 pmol随机引物及样本RNA, 逆转录反应条件: 30℃ 10 min, 42℃ 30 min, 95℃ 5 min. PCR反应体系50 μL含Taq酶21 nKaT, 特异性上下游引物各25 pmol. ID4基因PCR反应条件: 95℃ 15 min; 94℃ 30 s, 58℃ 30 s, 72℃ 30 s, 5个循环; 再将退火温度降至57℃进行32个循环. 扩增产物于25 g/L琼脂糖凝胶电泳分离. 目的基因ID4及内对照b-actin的引物由Primer5.0软件自行设计, 由Invitrogen公司合成. 引物序列如下: ID4 5'-ACTACATCCTGGACCTGC-3', 5'-GAATGCTGTCGCCCTGCT-3'(665-844), 产物片段长度180 bp. b-actin 5'-CGAAACTACCTTCAACTCCATCA-3', 5'-CGGACTCGTCATACTCCTGCT-3', 产物片段长度272 bp.
统计学处理 多样本均数比较采用单因素方差分析, SPSS10.0统计软件进行处理, P<0.05为统计学上差异有显著性.
5和20 μg/L的TSA处理结肠癌Caco2细胞5 d后, 与对照组比较细胞生长速度无显著性差异(P>0.05); 而100和500 μg/L TSA作用2 d后即明显抑制了细胞的生长(P<0.05). 100和500 μg/L组间比较, 细胞生长的抑制没有随着剂量的增加和时间的延长而更显著, 即在100 μg/L浓度以上没有出现剂量和时间的依赖性(图1). 5和20 μg/L组的细胞生长抑制率没有显著改变; 而100 μg/L组的抑制率在2-5 d分别为37.4%, 57.3%, 68.7%, 70.2%, 500 μg/L组的抑制率分别为48.8%, 67.4%, 76.7%, 81.8%.
TSA处理前结肠癌Caco2细胞处于G1, S和G2期的百分比分别为: 47.6%, 48.3%和4.2%; 5 μg/L TSA处理36 h后, 处于G1, S和G2期的百分比分别为: 48.5%, 41.9%和9.6%; 20 μg/L TSA处理36 h后, G1, S和G2期的百分比分别为: 51.2%, 41.0%和7.8%; 100 μg/L TSA处理36 h后, G1, S和G2期的百分比分别为: 55.6%, 25.6%和18.9%. 即100 μg/L TSA使结肠癌Caco2细胞周期出现G1期的阻滞, 而5和20 μg/L TSA与对照组比较没有影响Caco2的细胞周期. 各浓度组都没有显著的诱导Caco2细胞的凋亡(图2).
20, 100和500 μg/L TSA分别处理结肠癌Caco2细胞36 h, 对照组细胞ID4基因mRNA表达很弱, 20 μg/L TSA作用36 h后ID4基因的mRNA表达稍微增强, 而100 μg/L TSA使ID4基因的表达显著增强; 100和500 μg/L TSA组间比较无显著差异. β-actin的表达水平在各种浓度的TSA作用前后无显著改变(图3).
结肠癌是全球第3大恶性疾病, 其发病率在35-50/10万人, 在发达国家尤其显著, 发病率平均每年增长4.2%. 常见的高危因素有低纤维、高脂肪饮食, 家族史, 肥胖和炎性结直肠疾病等[10-11]. Vogelstein et al[12-13]证实的遗传突变累积学说是公认的结直肠癌发病的主要机制, 即前癌基因的激活和肿瘤抑制基因的失活所累积的遗传缺陷, 使结直肠腺瘤向结直肠癌进展的过程. 然而, 这些被证实的遗传缺陷不足以解释绝大多数的散发型结直肠癌的发病机制[14-15]. 近年研究发现表观遗传机制是调节一些重要基因功能的关键机制. 表观遗传是指通过DNA自身化学修饰方式从转录水平影响基因表达, 但不存在DNA序列改变的遗传改变. 表观遗传修饰包含3个方面: 组蛋白修饰, DNA甲基化和小RNA干扰[16-17]. 也就是说, 细胞可以只有一种遗传密码, 但由于表观遗传修饰的存在使蛋白质表达多种多样, 呈现出细胞分化后的不同形态. 核小体是人类染色体的基本组成单位, 由146个碱基对环绕组蛋白的十聚体构成. 组蛋白的共价修饰可以改变染色体的构象由紧密型变为松弛型, 包括乙酰化、甲基化、磷酸化和泛素化[18-19]. 这些可逆性的共价修饰使得基因的表达受到严格控制. HDAC通过调节核小体的构象改变, 在调节基因表达方面起着重要的作用. 在多种肿瘤中HDAC与细胞周期、细胞分化的调控密切相关[20-23].
ID蛋白(inhibition of DNA binding, ID)家族, 是一种独特的HLH(helix-loop-helix)蛋白家族成员. HLH是转录因子的基本结构, 以调节组织特异性基因的表达, 在细胞分化和胚胎发育过程中起着重要作用[24-25]. HLH蛋白必需通过同类或异类二聚化才具有DNA结合活性. ID蛋白含有HLH二聚化功能域, 但缺乏DNA结合功能域, 因此ID蛋白可以和HLH蛋白形成异类二聚体, 并抑制HLH蛋白与DNA的结合及转录[26]. 较早的研究发现ID4基因与少突胶质神经细胞的生长和分化相关[27], 最近的研究显示在30%的原发胃癌中, ID4基因的启动子区域被甲基化, 并导致该基因的低表达[28]. Umetani et al[29]发现, 在53%的原发性结直肠癌和73%的有肝转移的结直肠癌中, ID4基因的启动子区域被甲基化; 而在正常结肠组织和结直肠腺瘤中该基因启动子没有被甲基化. 同时这种异常的高甲基化可以通过甲基转移酶抑制剂逆转. 我们的研究发现, TSA在体外可以明显抑制结直肠癌Caco2细胞的增殖, 在100 μg/L浓度以上才表现出明显的生长抑制作用, 但在500 μg/L浓度时(数据没有显示)细胞出现明显的死亡趋势, 这说明TSA的抑制作用具有剂量依赖和相应的毒性作用. 为了寻找TSA抑癌作用的可能机制, 我们用流式细胞仪检测了不同浓度的TSA对细胞周期的影响, 结果发现100 μg/L TSA使该细胞出现G1期阻滞, 而100 μg/L浓度以下没有明显改变, 各浓度组均没有出现显著的细胞凋亡. 这提示TSA的抑癌作用与调节G1到S期转移的相关蛋白密切相关.
既然TSA与组蛋白乙酰化相关, 而组蛋白乙酰化和DNA异常甲基化是紧密相关的表观遗传修饰, 我们假设TSA也可以逆转一些表观遗传改变, 从而调节肿瘤相关基因的表达. ID4基因是新发现的候选抑癌基因, 其异常低表达与结直肠癌和乳腺癌的恶性进展有关. 我们发现100 μg/L浓度以上的TSA可以使Caco2细胞中ID4基因的表达恢复, 因而可以推测TSA恢复肿瘤抑制基因重新表达的作用, 可能是其发挥抑癌作用的关键. 值得进一步研究的是, TSA恢复ID4基因的表达是直接通过乙酰化作用, 还是间接通过去甲基化作用实现的. Ou et al[30]最新的研究发现, TSA可以广泛的诱导特定基因去甲基化并重新表达, 并证实组蛋白乙酰化和DNA去甲基化之间通过甲基结合蛋白(methyl-CpG binding proteins, MBDs)实现相互作用(crosstalk).
总之, 抑癌基因表观遗传沉默(epigenetic silencing)是其功能失活的第3种机制, 组蛋白去乙酰酶抑制剂TSA能够有效的逆转表观遗传沉默的抑癌基因重新表达, 这为结直肠的诊断和治疗提供了新的途径[31].
遗传突变的累积是结直肠癌发病的主要机制, 但对散发型结直肠癌, 肿瘤抑制基因的表观遗传沉默也是肿瘤发生的关键机制. 组蛋白去乙酰酶抑制剂曲古菌素A可明显抑制结直肠癌Caco2细胞的增殖, 并诱导ID4基因的重新表达, 提示组蛋白修饰也是抑癌基因功能失活的重要机制. 这种可逆性的表观遗传修饰为结直肠癌的治疗提供了新的契机.
本文采用MTT法和RT-PCR方法研究了组蛋白去乙酰酶抑制剂对结肠癌细胞增殖和ID4基因表达的影响, 为结肠癌的诊断和治疗提供了新的途径.
本文设计合理可靠, 结论较明确, 实验证据充足, 有一定的创新性和可读性, 较好地反映我国胃肠病学临床和基础研究的先进水平.
编辑: 何燕 电编:张敏
| 1. | Espino PS, Drobic B, Dunn KL, Davie JR. Histone modifications as a platform for cancer therapy. J Cell Biochem. 2005;94:1088-1102. [PubMed] |
| 2. | Strahl BD, Allis CD. The language of covalent histone modifications. Nature. 2000;403:41-45. [PubMed] |
| 3. | Villar-Garea A, Imhof A. The analysis of histone modifications. Biochim Biophys Acta. 2006;1764:1932-1939. [PubMed] |
| 4. | Lee KK, Workman JL. Histone acetyltransferase complexes: one size doesn't fit all. Nat Rev Mol Cell Biol. 2007;8:284-295. [PubMed] |
| 5. | Dey P. Chromatin remodeling, cancer and chemotherapy. Curr Med Chem. 2006;13:2909-2919. [PubMed] |
| 6. | Bouchain G, Delorme D. Novel hydroxamate and anilide derivatives as potent histone deacetylase inhibitors: synthesis and antiproliferative evaluation. Curr Med Chem. 2003;10:2359-2372. [PubMed] |
| 7. | Chiba T, Yokosuka O, Fukai K, Kojima H, Tada M, Arai M, Imazeki F, Saisho H. Cell growth inhibition and gene expression induced by the histone deacetylase inhibitor, trichostatin A, on human hepatoma cells. Oncology. 2004;66:481-491. [PubMed] |
| 8. | Kelly WK, Marks PA. Drug insight: Histone deacetylase inhibitors-development of the new targeted anticancer agent suberoylanilide hydroxamic acid. Nat Clin Pract Oncol. 2005;2:150-157. [PubMed] |
| 9. | Vigushin DM, Ali S, Pace PE, Mirsaidi N, Ito K, Adcock I, Coombes RC. Trichostatin A is a histone deacetylase inhibitor with potent antitumor activity against breast cancer in vivo. Clin Cancer Res. 2001;7:971-976. [PubMed] |
| 10. | Rose DJ, DeMeo MT, Keshavarzian A, Hamaker BR. Influence of dietary fiber on inflammatory bowel disease and colon cancer: importance of fermentation pattern. Nutr Rev. 2007;65:51-62. [PubMed] |
| 11. | Rennert G. Prevention and early detection of colorectal cancer-new horizons. Recent Results Cancer Res. 2007;174:179-187. [PubMed] |
| 12. | Vogelstein B, Kinzler KW. The multistep nature of cancer. Trends Genet. 1993;9:138-141. [PubMed] |
| 13. | Cho KR, Vogelstein B. Genetic alterations in the adenoma-carcinoma sequence. Cancer. 1992;70:1727-1731. [PubMed] |
| 14. | Komarova NL, Lengauer C, Vogelstein B, Nowak MA. Dynamics of genetic instability in sporadic and familial colorectal cancer. Cancer Biol Ther. 2002;1:685-692. [PubMed] |
| 15. | Parsons DW, Wang TL, Samuels Y, Bardelli A, Cummins JM, DeLong L, Silliman N, Ptak J, Szabo S, Willson JK. Colorectal cancer: mutations in a signalling pathway. Nature. 2005;436:792. [PubMed] |
| 16. | D'Alessio AC, Szyf M. Epigenetic tete-a-tete: the bilateral relationship between chromatin modifications and DNA methylation. Biochem Cell Biol. 2006;84:463-476. [PubMed] |
| 17. | Saito Y, Jones PA. Epigenetic activation of tumor suppressor microRNAs in human cancer cells. Cell Cycle. 2006;5:2220-2222. [PubMed] |
| 18. | Shilatifard A. Chromatin modifications by methylation and ubiquitination: implications in the regulation of gene expression. Annu Rev Biochem. 2006;75:243-269. [PubMed] |
| 19. | Martin C, Zhang Y. The diverse functions of histone lysine methylation. Nat Rev Mol Cell Biol. 2005;6:838-849. [PubMed] |
| 20. | Thiagalingam S, Cheng KH, Lee HJ, Mineva N, Thiagalingam A, Ponte JF. Histone deacetylases: unique players in shaping the epigenetic histone code. Ann N Y Acad Sci. 2003;983:84-100. [PubMed] |
| 21. | Chen YX, Fang JY, Zhu HY, Lu R, Cheng ZH, Qiu DK. Histone acetylation regulates p21WAF1 expression in human colon cancer cell lines. World J Gastroenterol. 2004;10:2643-2646. [PubMed] |
| 22. | Garcia-Manero G, Issa JP. Histone deacetylase inhibitors: a review of their clinical status as antineoplastic agents. Cancer Invest. 2005;23:635-642. [PubMed] |
| 23. | Mai A, Massa S, Rotili D, Cerbara I, Valente S, Pezzi R, Simeoni S, Ragno R. Histone deacetylation in epigenetics: an attractive target for anticancer therapy. Med Res Rev. 2005;25:261-309. [PubMed] |
| 24. | Ruzinova MB, Benezra R. Id proteins in development, cell cycle and cancer. Trends Cell Biol. 2003;13:410-418. [PubMed] |
| 25. | Sikder HA, Devlin MK, Dunlap S, Ryu B, Alani RM. Id proteins in cell growth and tumorigenesis. Cancer Cell. 2003;3:525-530. [PubMed] |
| 26. | Rotzer D, Krampert M, Sulyok S, Braun S, Stark HJ, Boukamp P, Werner S. Id proteins: novel targets of activin action, which regulate epidermal homeostasis. Oncogene. 2006;25:2070-2081. [PubMed] |
| 27. | Riechmann V, Sablitzky F. Mutually exclusive expression of two dominant-negative helix-loop-helix (dnHLH) genes, Id4 and Id3, in the developing brain of the mouse suggests distinct regulatory roles of these dnHLH proteins during cellular proliferation and differentiation of the nervous system. Cell Growth Differ. 1995;6:837-843. [PubMed] |
| 28. | Chan AS, Tsui WY, Chen X, Chu KM, Chan TL, Chan AS, Li R, So S, Yuen ST, Leung SY. Downregulation of ID4 by promoter hypermethy-lation in gastric adenocarcinoma. Oncogene. 2003;22:6946-6953. [PubMed] |
| 29. | Umetani N, Takeuchi H, Fujimoto A, Shinozaki M, Bilchik AJ, Hoon DS. Epigenetic inactivation of ID4 in colorectal carcinomas correlates with poor differentiation and unfavorable prognosis. Clin Cancer Res. 2004;10:7475-7483. [PubMed] |
| 30. | Ou JN, Torrisani J, Unterberger A, Provencal N, Shikimi K, Karimi M, Ekstrom TJ, Szyf M. Histone deacetylase inhibitor Trichostatin A induces global and gene-specific DNA demethylation in human cancer cell lines. Biochem Pharmacol. 2007;73:1297-1307. [PubMed] |
| 31. | Kondo Y, Issa JP. Epigenetic changes in colorectal cancer. Cancer Metastasis Rev. 2004;23:29-39. [PubMed] |