修回日期: 2007-02-10
接受日期: 2007-02-13
在线出版日期: 2007-04-28
目的: 分析Tec激酶区作用蛋白视黄酸诱导蛋白16 (RAI16)的蛋白质结构和功能信息.
方法: 利用相应的计算机软件及数据库,对RAI16 cDNA序列进行分析和比较,并对编码的蛋白质氨基酸序列及其二级结构进行分析和预测.
结果: RAI16 cDNA序列由2863个核苷酸组成,编码759氨基酸蛋白质, RAI16蛋白包括一段由31个氨基酸组成的信号肽, 没有跨膜区, 亚细胞定位于内质网上, 有多种二级结构形式,为一紧密包裹的球状蛋白质, 是一种分泌蛋白, 有多个磷酸化和蛋白酶切位点. RAI16蛋白为不稳定蛋白, 不稳定系数为53.11, 疏水性GRAVY (grand average of hydropathicity)值为-0.136.
结论: RAI16蛋白作为一种新的胞内信号蛋白, 很可能在肝癌细胞的诱导分化信号转导转录中与Tec协同发挥重要作用.
引文著录: 王阁, 邓婧, 杨进, 郑继军, 王红中, 胡庆, 张志敏, 陈川, 王东, 李增鹏. 视黄酸诱导蛋白16蛋白质结构功能分析. 世界华人消化杂志 2007; 15(12): 1342-1346
Revised: February 10, 2007
Accepted: February 13, 2007
Published online: April 28, 2007
AIM: To analyze the protein structure and function of Homo sapiens retinoic acid induced 16 (RAI16) cDNA nucleotides sequence.
METHODS: Using some related computer software and database, RAI16 cDNA sequence and the deduced amino acid (AA) sequence were analyzed and compared with those of other species, and the secondary structure and hydrophobicity of the protein were determined and predicted.
RESULTS: RAI16 cDNA sequence consisted of 2863 nucleotides encoding a 759-AA protein. RAI16 protein included a 31-AA signal peptide. The protein had no transmembrane domain, and its subcellular localization was in endoplasmic reticulum. The secondary structure contained several kinds of conformations. RAI16 protein appeared as a compact and globular domain, and was a secretory and hydrophilicity protein (grand average of hydropathicity: -0.136). The protein was also unstable (the unstable coefficient: 53.11), with many phosphorylation and proteinase sites.
CONCLUSION: As a new signal protein, RAI16 may be involved in the signal transduction and transcription of hepatoma cell differentiation by interacting with Tec protein.
- Citation: Wang G, Deng J, Yang J, Zheng JJ, Wang HZ, Hu Q, Zhang ZM, Chen C, Wang D, Li ZP. Analysis on the protein structure and function of retinoic acid induced 16. Shijie Huaren Xiaohua Zazhi 2007; 15(12): 1342-1346
- URL: https://www.wjgnet.com/1009-3079/full/v15/i12/1342.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v15.i12.1342
Tec(tyrosine kinase expressed in hepatocellular carcinoma)是一种存在于胞质内的非受体型蛋白酪氨酸激酶, 最初于肝癌组织中筛选得到, 他参与多种细胞因子的信号传递过程, 是多种细胞因子、辅助分子及受体型PTK信号转导过程中一个重要的靶分子. 我们进一步证实Tec参与肝再生、肝干细胞和肝癌细胞分化的信号转导过程[1-3]. 本课题组前期工作通过酵母双杂交技术在胎肝cDNA文库中筛选得到一种Tec相互作用蛋白, 即视黄酸诱导蛋白16(RAI16). 关于RAI16文献报告很少, 功能研究报告更是空白, 从其命名知晓, 为一种RA诱导蛋白. 本研究主要利用相应的计算机软件及一些有关的生物信息学站点, 对RAI16 cDNA序列进行分析和比较, 并对其编码的蛋白质氨基酸序列及其二级结构、功能域等进行分析和预测, 从中获得RAI16蛋白质结构方面的重要信息, 从而根据结构预测其未知新功能.
以NCBI上公布的RAI16做为研究对象. NCBI上蛋白质序列数据库及核酸序列数据库.
1.2.1 RAI16 cDNA的克隆及测序: 以Tec激酶结构域(KD)作为"钓饵"蛋白, 在酵母双杂交系统中筛选构建于转录激活结构域(AD)载体的人胎肝文库, 经营养缺陷选择、诱导筛选及初步鉴定, 筛库所得基因插段约2000 bp左右的文库质粒为一真阳性克隆, 随之亚克隆, 将其连接重组于pMD18-T质粒, 转化DH5α大肠杆菌, 提取重组质粒DNA, 由上海博亚生物技术公司完成测序.
1.2.2 cDNA核酸序列的比较和分析: 对已测出全长的cDNA阳性克隆序列, 通过生物信息学网站(http://www.ncbi.nlm.nih.gov/blast/), 在GenBank数据库中查询比较所测序列的同源性, 初步确认cDNA序列的性质及其相关信息.
1.2.3 蛋白质序列的同源性分析及比较: 分析RAI16蛋白质的开放阅读框架(ORF)并推译其cDNA编码的氨基酸序列. 通过BLAST方式, 查询比较RAI16蛋白质序列的同源性.
1.2.4 蛋白质二级结构及功能的初步预测: 基因的功能主要是通过其所翻译的蛋白质来体现, 在进化过程中, 蛋白质的顺序和结构在种间和种内的变化比相应核酸的变化要小. 因此通过分析蛋白质的功能基序、亚细胞定位、信号肽及其二级结构可以初步预测相应功能. 所用生物信息学网站有: 蛋白质功能基序分析: http://au.expasy.org/prosite/; 蛋白质二级结构预测: http://www.emblheidelberg.de/predictprotein/; 蛋白质信号肽及酶切位点预测: http://www.cbs.dtu.dk/services/SignalP/; 蛋白质跨膜区预测: http://www.cbs.dtu.dk/services/TMHMM-2.0/; 蛋白质亚细胞定位分析: http://psort.nibb.ac.jp/form2.html/; 蛋白质组成及疏水性分析: http://cn.expasy.org/tools/protparam.html/; 蛋白质亚细胞定位预测: http://psort.nibb.ac.jp/form2.html/; 蛋白质各组成及疏水性分析: http://cn.expasy.org/tools/protparam.html/; 蛋白酶切位点预测: http://www.expasy.org/tools/peptidecutter/.
测序发现插入片段全长2863个碱基对(base pair, bp), 他含有48 bp的5'端非翻译区、2232 bp组成的开放阅读框架及583 bp组成的3'非翻译区. 序列比对发现, 插入基因片段与人视黄酸诱导蛋白16(RAI16) cDNA的同源性最高, 为99%, 从而证实克隆的这一基因为RAI16(GenBank收录号为BC052237).
利用http://www.expasy.ch/网站, 把RAI16开放读码框(ORF)中的DNA顺序翻译成相应的氨基酸顺序, 再利用http://www.ncbi.nlm.nih.gov/BLAST的蛋白质数据库进行比较, 结果未见有意义的同源蛋白.
生物信息学分析提示[4], RAI16蛋白含6个蛋白质激酶C(PKC)磷酸化位点(164-166: TEK、271-273: SKK、336-338: SWR、435-437: TDR、468-470: TLR、590-592: SSR), 9个酪蛋白激酶Ⅱ(CK2)磷酸化位点(56-59: STDE、162-165: SVTE、164-167: TEKE、214-217: SHGD、507-510: SYED、523-526: SFLD、541-544: TSYD、573-576: TGYD、603-606: TPLD), 3个酪氨酸激酶(Tyr)磷酸化位点(73-81: KQMLDILVY、490-497: RNLEGRPY、629-635: RILDQPY), 6个N-豆蔻酰化位点(156-161: GGTASG、157-162: GTASGS、255-260: GTTESN、267-272: GLCQSK、482-487: GIIHSL、708-713: GQAPGE), 2个酰胺化位点(191-194: EGKK、196-199: VGRK), 3个酪氨酸硫酸化位点(501-515: GSPEPESYEDTLDLE、512-526: LDLEEDPYFTDSFLD、568-582: AFLEETGYDTYVHDA)以及1个ATP-GTP结合位点(540-547: ATSYDGKT).
成熟的RAI16蛋白可以形成α-螺旋、β-折叠及环状结构(LOOP)等多种二级结构形式, 其中, 以形成α-螺旋的区间居多. 通过生物信息学网站(http://www.emblheidelberg.de/predictprotein)得知: RAI16为一紧密包裹的球状蛋白质[5].
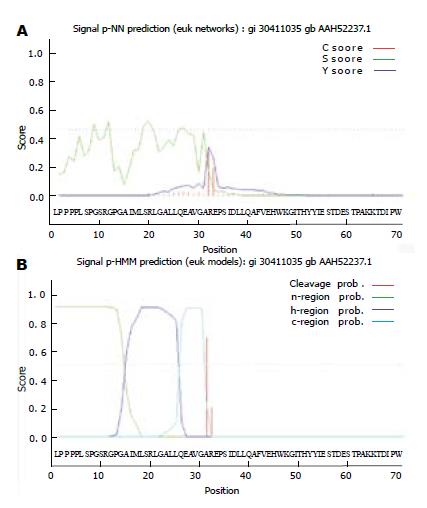
采用基于神经网络(NN, 图2A)的算法和隐藏的马尔可夫模型(HMM, 图2B)预测RAI16蛋白质的信号肽及其切割位点[6-7], 结果表明, RAI16蛋白具有一段由31 AA组成的信号肽, 且其切割位点位于30-31 AA之间. 推测为一种分泌蛋白.
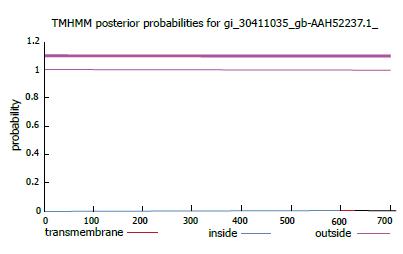
为了更直观的显示RAI16是否具有跨膜区, 借助expasy(http://cn.expasy.org/) 中的TMHMM工具 (http://www.cbs.dtu.dk/services/TMHMM-2.0/)[7], 预测结果如(图3).
通过工具PSORT(http://psort.nibb.ac.jp/form2.html)进行预测[8-10], 结果如下: 44.4%: endoplasmic reticulum(内质网); 22.2%: cytoplasmic(胞质); 11.1%: mitochondrial(线粒体); 11.1%: Golgi(高尔基体); 11.1%: nuclear(胞核).
为了更加全面地统计各参数, 用expasy中的工具ProtParam (http://cn.expasy.org/tools/protparam.html)[11-12], 结果如下: 氨基酸数目: 759, 分子量: 83834.8. 等电点: 5.31; 负电荷残基总数(Asp+Glu): 93; 正电荷残基总数(Arg+Lys): 62; 分子式: C3769H5901N999O1114S25; 原子总数: 11808; 半衰期: 5.5 h; 不稳定系数: 53.11, 该蛋白分类为不稳定蛋白; 脂肪系数: 94.23; Grand average of hydropathicity (GRAVY)亲水性评估: -0.136.
通过工具PeptideCutter (http://www.expasy.org/tools/peptidecutter)进行预测, 结果发现共有如下这些蛋白酶对RAI16有酶切作用: 精氨酸-C蛋白水解酶(Arg-C proteinase)、Asp-N内切酶(Asp-N endopeptidase)、Asp-N内切酶+N端亮氨酸(Asp-N endopeptidase+N-terminal Glu)、BNPS-Skatole、细胞凋亡蛋白酶1(caspase1)、高特异性糜蛋白酶(chymotrypsin-high specificity)、低特异性糜蛋白酶(chymotrypsin-low specificity)、梭菌蛋白酶(clostripain)、CNBr、凝血因子Xa (factor Xa)、甲酸(formic acid)、谷氨酰基肽链内切酶(glutamyl endopeptidase)、氧代苯甲酸(iodosobenzoic acid)、LysC、LysN、NTCB(2-nitro-5-thiocyanobenzoic acid)、胃蛋白酶(pH1.3)(pepsin)、胃蛋白酶(pH>2)(pepsin)、脯氨酸肽链内切酶(proline-endopeptidase)、蛋白酶K (proteinase K)、葡萄球菌肽酶I (staphylococcal peptidase I)、嗜热菌蛋白酶(thermolysin)、胰蛋白酶(trypsin).
1995年本课题组前期工作通过酵母双杂交技术在胎肝cDNA文库中筛选Tec相互作用蛋白, 得到一阳性克隆, 测序并经BLAST比对后, 确定为人源性RAI16基因, 已被GenBank收录, 编号为BC052237, 组织来源为脑Ⅳ型胶质细胞瘤.
RAI16 cDNA全长2863 bp, 含有48 bp的5'端非翻译区、2232 bp组成的开放阅读框架及583 bp组成的3'非翻译区, 在3'非编码区含有一个腺苷酸化位点(AATAAA)和长度为18个腺苷酸的poly(A). 编码框全长759 AA, 编码蛋白质分子量为83.8 kDa. PSORT结果提示, 该蛋白有很长的酪氨酸尾部, 并有一段9 AA长的过氧化物酶前导信号2(SKL2) (KLLLVRKQL at 698). 蛋白质同源性分析未见有意义的同源蛋白.
RAI16蛋白含有6个可能的PKC磷酸化位点, 9个可能的CK2位点, 3个Tyr磷酸化位点, 5个N-豆蔻酰化位点, 2个酰胺化位点, 3个酪氨酸硫酸化位点以及1个ATP-GTP结合位点. 这些磷酸化位点主要功能是参与和调节蛋白质的代谢、翻译后的修饰和加工, RAI16蛋白中这些磷酸化位点的存在提示RAI16的生物学活性有可能在细胞信号传导通路中接受多种信号的调控. 其中, 他具有多达18个不同蛋白激酶的磷酸化位点, 蛋白质的磷酸化和去磷酸化参与多种细胞功能的调节, 因此其失调可能与肝细胞的异常分化密切相关.
利用expasy中的工具ProtParam分析得到, RAI16蛋白等电点为5.31, 体外半衰期为5.5 h, 氨基酸组成成分中, 亮氨酸含量最高, N端序列考虑为亮氨酸. 蛋白不稳定性系数为53.11(Ⅱ级), 该蛋白分类为不稳定性蛋白. 蛋白脂质系数为94.23. 蛋白质疏水性的预测在蛋白质组学研究中是必不可少的, 通常根据蛋白质的GRAVY 值来预测. GRAVY值的范围在2与-2之间, 正值表明此蛋白为疏水蛋白, 负值表明为亲水蛋白. 从通过protparam工具统计的结果中可知, Grand average of hydropathicity(GRAVY)为-0.136, 这说明人源的RAI16为亲水蛋白. 分析RAI16蛋白二级结构发现, 成熟的RAI16蛋白可以形成α-螺旋、β-折叠及环状结构(LOOP)等多种二级结构形式, 其中大多为α-螺旋. 生物信息学网站PredictProtein提示该蛋白质为一包裹紧密的球状蛋白. 此外, 该蛋白无跨膜区, 具有一段由31个氨基酸组成的信号肽, 且其切割位点位于30-31 AA之间, 推测为一种分泌蛋白. 其亚细胞定位位于内质网上. 因此, 我们可以推测RAI16蛋白是一种定位于胞质内内质网上的分泌型的球形信号蛋白, RAI16蛋白很可能在肝干细胞和肝癌诱导分化过程中, 作为RA诱导蛋白被信号蛋白分子募集至胞质内, 通过磷酸化、去磷酸化、豆蔻酰化、酰胺化等修饰, 通过不同的活化形式和剪切方式以及胞质与胞核间的移位, 完成其在信号转导中的重要角色.
总之, 我们首次成功地克隆出视黄酸诱导蛋白16的cDNA序列, 由此推译出他的蛋白质一级结构, 并对其功能区、信号肽、亚细胞定位、二级结构及亲疏水性等进行了分析. 上述结果为深入研究RAI16的分子结构及其下一步的功能预测奠定了基础, 对其结构和功能的深入研究, 将有助于阐明RAI16在肝癌细胞诱导分化信号转导转录中的作用, 同时还将利于明确RAI16与Tec蛋白之间的相互作用机制, 从而进一步拓宽Tec的研究领域.
Tec是国内外研究较活跃的一个非受体型蛋白酪氨酸激酶, 他主要介导细胞信号传导. 近几年的研究发现Tec与肝细胞分化有关, 在肝癌细胞中具有高表达和特异性, 是肝干细胞和肝细胞增值、分化、凋亡信号转导路径中关键的蛋白激酶连接分子. 鉴于RAI16最初作为Tec相互作用蛋白被筛选克隆得到, 提示Tec和RAI16蛋白之间存在相互作用, 因此推测RAI16蛋白与细胞分化有关, 很可能在肝癌细胞的诱导分化信号转导转录中发挥重要作用. 探索RAI16蛋白的结构功能为进一步研究他们之间的相互作用关系提供了更广阔的空间.
近几年在对RA作用研究中发现了RAI系列基因, 但尚无具体完整的结构和功能研究的相关报道, 只是推测RAI系列蛋白可能是一种"新型"的胞内参与信号转导转录调控的蛋白质家族. RAI16蛋白作为家族成员之一, 了解其结构功能, 对认识整个家族特征具有重要的意义.
RAI16文献报告很少, 功能研究报告更是空白, 本研究利用相应的计算机软件及数据库, 对RAI16 cDNA序列进行分析和比较, 并对编码的蛋白质氨基酸序列及其二级结构进行了分析和预测, 从而获得RAI16的蛋白质结构信息, 为进一步探索RAI16的功能提供了理论依据.
对RAI16蛋白的功能预测提示RAI16可能是Tec激酶的底物分子, 他参与了细胞分化过程, 很可能在肝癌细胞的诱导分化信号转导转录中发挥重要作用. 对RAI16蛋白功能的更深入研究将为研究肿瘤细胞诱导分化信号转导分子机制提供新的思路和理论依据.
Tec: 最早是从肝癌组织中筛选出的一种重要的非受体型蛋白酪氨酸激酶, 他存在于胞质内, 主要在T细胞、B细胞、造血细胞及肝脏中表达. Tec是多种细胞因子、辅助分子及受体型蛋白酪氨酸激酶信号转导途径中的重要靶分子, 被上游分子激活后, 作用于下游底物使之磷酸化, 进行信号分子的逐级传递, 同时能够参与并调节多种生物学效应.
RAI16蛋白质作为一种新的包内信号蛋白, 可能在肝癌细胞的诱导分化信号转导转录中与Tec协同发挥重要作用. 本研究内容重要, 提供了有意义的信息, 文章科学性、创新性和可读性能较好的反映我国或国际肠胃病学临床和基础研究的先进水平.
电编: 郭海丽 编辑:张焕兰
| 1. | Qiu Y, Kung HJ. Signaling network of the Btk family kinases. Oncogene. 2000;19:5651-5661. [PubMed] [DOI] |
| 2. | Schmidt U, Boucheron N, Unger B, Ellmeier W. The role of Tec family kinases in myeloid cells. Int Arch Allergy Immunol. 2004;134:65-78. [PubMed] [DOI] |
| 3. | Miller AT, Berg LJ. New insights into the regulation and functions of Tec family tyrosine kinases in the immune system. Curr Opin Immunol. 2002;14:331-340. [PubMed] [DOI] |
| 4. | Mondal S, Jaishankar SP, Ramakumar S. Role of context in the relationship between form and function: structural plasticity of some PROSITE patterns. Biochem Biophys Res Commun. 2003;305:1078-1084. [PubMed] [DOI] |
| 5. | Xu W, Wang S, Wang G, Wei H, He F, Yang X. Identification and characterization of differentially expressed genes in the early response phase during liver regeneration. Biochem Biophys Res Commun. 2000;278:318-325. [PubMed] [DOI] |
| 6. | Bendtsen JD, Nielsen H, von Heijne G, Brunak S. Improved prediction of signal peptides: SignalP 3.0. J Mol Biol. 2004;340:783-795. [PubMed] [DOI] |
| 7. | Kall L, Krogh A, Sonnhammer EL. A combined transmembrane topology and signal peptide prediction method. J Mol Biol. 2004;338:1027-1036. [PubMed] [DOI] |
| 8. | Nakai K, Horton P. PSORT: a program for detecting sorting signals in proteins and predicting their subcellular localization. Trends Biochem Sci. 1999;24:34-36. [PubMed] [DOI] |
| 9. | Peuvel I, Peyret P, Metenier G, Vivares CP, Delbac F. The microsporidian polar tube: evidence for a third polar tube protein (PTP3) in Encephalitozoon cuniculi. Mol Biochem Parasitol. 2002;122:69-80. [PubMed] [DOI] |
| 10. | Guo J, Jin G, Meng L, Ma H, Nie D, Wu J, Yuan L, Shou C. Subcellullar localization of tumor-associated antigen 3H11Ag. Biochem Biophys Res Commun. 2004;324:922-930. [PubMed] [DOI] |
| 11. | Schneider M, Tognolli M, Bairoch A. The Swiss-Prot protein knowledgebase and ExPASy: providing the plant community with high quality proteomic data and tools. Plant Physiol Biochem. 2004;42:1013-1021. [PubMed] [DOI] |
| 12. | Gattiker A, Michoud K, Rivoire C, Auchincloss AH, Coudert E, Lima T, Kersey P, Pagni M, Sigrist CJ, Lachaize C. Automated annotation of microbial proteomes in SWISS-PROT. Comput Biol Chem. 2003;27:49-58. [PubMed] [DOI] |