修回日期: 2006-08-08
接受日期: 2006-08-14
在线出版日期: 2006-11-28
目的: 构建cagA基因缺失的中国幽门螺杆菌突变株.
方法: 运用基因同源重组方法将卡那霉素抗性基因(kmR)连接到PCR扩增cagA基因两端区域产生的2个基因片段之间, 并共同插人到pBluescript SKⅡ(-)载体的多克隆位点中, 构建出带卡那霉素抗性标志的缺失突变载体pBs-cagA-mutant. 将突变载体转化到含完整cagA基因的突变受体MEL-H. pylori 27菌株中. 卡那霉素筛选出cagA基因缺失的中国幽门螺杆菌突变株, 并经PCR和DNA测序鉴定.
结果: 构建的突变载体经限制性内切酶酶切分析显示, 产生的条带与设计结果完全一致. PCR方法扩增突变株cagA, kmR基因显示, cagA基因已被完整敲除掉, DNA测序证实筛选得到了cagA基因缺失的幽门螺杆菌突变株. 连续培养25代后证实, 幽门螺杆菌突变株具有良好稳定性.
结论: 首次成功构建出1株中国人来源的、cagA基因缺失的幽门螺杆菌突变株.
引文著录: 黄志刚, 段广才, 范清堂, 黄学勇. cagA基因缺失的中国幽门螺杆菌突变菌株的构建及鉴定. 世界华人消化杂志 2006; 14(33): 3190-3194
Revised: August 8, 2006
Accepted: August 14, 2006
Published online: November 28, 2006
AIM: To knock out the entire cagA gene of Chinese Helicobacter pylori strain via homologous recombination and construct a cagA-deleted mutant strain of Chinese H. pylori.
METHODS: Two DNA fragments locating in the upper and downstream of cagA gene were amplified and a kanamycin resistance gene of pEGFP-N2 between them were engineered into pBluescript SK II(-) plasmid (pBs-cagA-mutant). Electrotransformation of H. pylori cells with pBs-cagA-mutant resulted in isolation of kanamycin-resistant H. pylori transformants, which was identified by polymerase chain reaction (PCR) and sequencing analysis.
RESULTS: Restriction endonuclease analyses showed that pBs-cagA-mutant vector had been successfully recombined. The cagA-deleted status of Chinese H. pylori mutants was confirmed by PCR with primers specific for the genes of cagA and kanamycin resistance. After 25 generations of culture, the constructed Chinese H. pylori mutants were confirmed stable.
CONCLUSION: A cagA-negative mutants of Chinese H. pylori strain is firstly constructed, which can help to further study the role of cagA in the pathogenesis of H. pylori infection.
- Citation: Huang ZG, Duan GC, Fan QT, Huang XY. Construction and identification of Chinese mutant Helicobacter pylori strain with absent expression of cagA gene. Shijie Huaren Xiaohua Zazhi 2006; 14(33): 3190-3194
- URL: https://www.wjgnet.com/1009-3079/full/v14/i33/3190.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i33.3190
幽门螺杆菌(Helicobacter pylori, H. pylori)是一种可长期定植于人类胃黏膜的革兰氏阴性螺旋形微需氧菌, 在全世界范围内感染率超过50%. H. pylori是引起慢性胃炎和胃十二指肠溃疡的致病因子, 流行病学和动物实验证实H. pylori慢性感染是导致胃癌的重要危险因素. H. pylori致病的多样性与尿素酶、黏附因子、应激反应蛋白、脂多糖、空泡毒素以及细胞毒素相关基因A蛋白(cytotoxin-associated gene A, cagA)等毒力因子关系密切[1]. 其中cagA是H. pylori最重要的毒力因子之一[2]. 近来发现, cagA可通过Ⅳ型分泌系统转移进宿主细胞, 经由Src家族的酪氨酸激酶磷酸化修饰后, 与含Src同源序列SH2结构域的酪氨酸磷酸酶(tyrosine phosphatase 2, SHP-2)结合, 干扰细胞信号转导, 使细胞骨架排列紊乱[3]. 世界范围内不同H. pylori菌株cagA分子变异较大, 该变异可能导致不同菌株的cagA生物学作用乃至致病性不同[4]. 我国作为H. pylori感染较严重的国家之一, 临床分离株中超过90%含有cagA基因, 但对于毒力因子cagA的生物学功能不甚明了. 另外, 由于cagA阴性自然突变株与cagA阳性野生株之间缺乏除cagA基因以外的相同遗传背景, 所以无法对cagA基因功能做出真实判断. 基于以上原因, 本研究通过同源重组方法敲除掉幽门螺杆菌中国株MEL-H. pylori 27 cagA基因, 首次构建出cagA基因全部缺失的源自中国的幽门螺杆菌cagA阴性突变株, 为研究中国株幽门螺杆菌cagA基因的生物学功能及在毒力相关蛋白表达中的作用, 从而最终明确cagA基因与幽门螺杆菌致病性的关系等方面提供良好的实验前提.
含cagA基因的幽门螺杆菌菌株MEL- H. pylori 27(简称H. pylori 27)由本教研室从郑州市慢性萎缩性胃炎患者体内分离并保种. 克隆载体pBluescript SKⅡ(-)由军事医学科学院韩聚强博士惠赠, 质粒pEGFP-N2(含卡那霉素抗性基因)由河南省医学科学研究所范天黎博士惠赠. 大肠杆菌DH5α为本室保存. pyrobest DNA聚合酶、T4 DNA连接酶、DL15000 DNA Marker、l00 bp DNA Marker、限制性内切酶XhoⅠ、HindⅢ、PstⅠ和XbaⅠ为TaKaRa公司产品; 蛋白酶K、RNA酶A购自Sigma公司; 胰蛋白胨、酵母提取物购自OXOID公司; quick gel extraction kit购自Qiagen公司.
将H. pylori 27接种于含100 mL/L羊血的布氏平板培养基(万古霉素10 mg/L、多黏菌素B 2500 U/L、TMP 5 mg/L、两性霉素5 mg/L), 37 ℃, 微需氧条件下(100 mL/L CO2, 50 mL/L O2, 850 mL/L N2)培养3-4 d. 从培养基上刮取生长良好的H. pylori菌落, TE洗涤后采用酚/氯仿法提取基因组DNA, 紫外分光光度计测定DNA的浓度和纯度. 抽提的DNA溶于50 μL去离子双蒸水中, 置-20 ℃保存备用.
1.2.1 PCR扩增: 根据本室报道的H. pylori 27 cagA基因全序列(GenBank登录号: DQ306710)自行设计用于扩增cagA基因两翼的同源臂片段: P1: 5'-CGCTCGAGAGCCCTAAAGTTACTACAA-3'(含XhoⅠ位点), P2: 5'-GCAAGCTTTGTTTCTCCTTTACT-3'(含HindⅢ位点), 扩增片段长约720 bp; P3: 5'-GGCTGCAGAGGATTGAGGAATAC-3'(含PstⅠ位点), P4: 5'-CGTCTAGATCAAGTTGTCT AATTTAG-3'(含XbaⅠ位点), 扩增片段长约985 bp. 根据cagA基因编码区保守序列设计引物: P5: 5'-GGTCTGCCAAACAATCTTTTGCAG-3', P6: 5'-TCAAGTTGTCT AATTTAGCGTTG-3', 扩增片段长约760 bp. 根据质粒pEGFP-N2全序列设计用于扩增抗性筛选标记基因(卡那霉素)的引物: P7: 5'-GCAAGCTTATGATTGAACAAGATGGATTG-3'(含HindⅢ位点), P8: 5'-GGCTGCAGTCAGAAGAACTCGTCAAGAAG-3'(含PstⅠ位点), 扩增片段长约800 bp. 引物设计软件为Primer 5.0. 所有引物均由北京赛百盛公司合成. 以H. pylori 27基因组DNA为模板, 分别用P1, P2和P3, P4引物扩增cagA基因的两翼序列, 扩增产物分别记作FL1和FR2; 以质粒pEGFP-N2为模板, 用P7, P8引物扩增卡那霉素抗性基因, 扩增产物记作kmR. 反应体系50 μL, 扩增条件为: 94 ℃ 4 min×1; 94 ℃ 50 s, 54 ℃ 50 s, 72 ℃ 50 s×30; 72 ℃ 7 min×1, 产物于4 ℃保存备用.
1.2.2 突变载体的构建: 将酶切消化后的扩增片段: FL1/XhoⅠ+Hind Ⅲ, kmR/Hind Ⅲ+PstⅠ, FR2/ PstⅠ+XbaⅠ与载体pBluescript SKⅡ(-)/XhoⅠ+XbaⅠ进行体外连接, 连接体系40 μL, 目的片段与载体分子比为3:3:3:1, 16 ℃连接24 h后, 按常规方法转化感受态DH5α细胞. 限制性内切酶消化鉴定出阳性重组克隆, 记作pBS-cagA-mutant.
1.2.3 突变菌株的构建与鉴定: 参考Yuan et al[5]的方法, 自培养72 h的固体培养基上刮取H. pylori于1 mL 150 mL/L甘油-90 g/L蔗糖溶液, 4 ℃ 5000 r/min离心10 min(重复3次), 沉淀重悬于50 mL 150 mL/L甘油-90 g/L蔗糖溶液中, 调整细菌浓度为1010 CFU/L. 4 ℃放置10 min, 加质粒pBS-cagA-mutant 5 μg, 冰上静置5 min, 移入-20 ℃预冷的0.1 cm电击池中, 冰浴5 min, 电击池置电穿孔架上, 电击条件: 25 F, 1.8 kV, 200 Ω, 4.92 s, 电击后, 加SOC缓冲液100 μL, 将电转液均匀涂布于普通H. pylori平板上, 37 ℃微需氧培养48 h后, 刮取平板上菌落于无菌生理盐水中混匀, 转涂于含25 mg/L卡那霉素的H. pylori平板上, 37 ℃微需氧培养3-6 d, 挑取单个菌落接种于含25 mg/L卡那霉素的H. pylori平板上继续培养3-6 d, 刮取平板上的菌落进行尿素酶活性检测和细菌染色鉴定, 并且抽提基因组DNA进行鉴定: 以扩增cagA基因编码区序列引物P5, P6和卡那霉素抗性基因引物P7, P8, 结合cagA基因侧翼的引物P1, P4, 进行PCR扩增, 并以野生株H. pylori 27为对照. 将引物P1, P4的扩增产物克隆后, 送上海鼎安生物科技有限公司测序. 鉴定出的突变菌株记作H. pylori 27△cagA. H. pylori 27野生株和cagA基因缺失的H. pylori27△cagA突变株分别连续培养25代(突变株培养基中含25 mg/L卡那霉素)后, 再按上述条件行相关基因片段的PCR扩增, 以鉴定突变菌株稳定性.
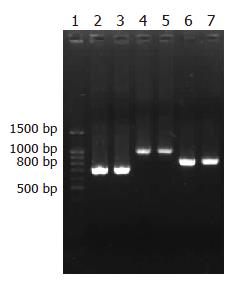
引物P1, P2扩增cagA基因编码区的上游序列, 扩增片段FL1长约720 bp; 引物P3, P4扩增cagA基因编码区的下游序列, 扩增片段FR2长约985 bp; 引物P7, P8扩增质粒pEGFP-N2的抗性筛选标记基因(卡那霉素)的编码序列, 扩增片段kmR长约800 bp(图1).
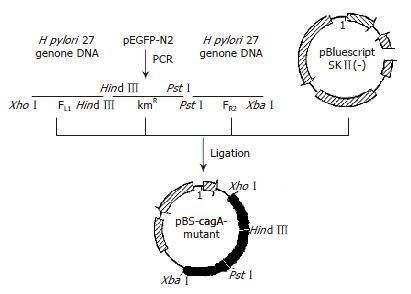
H. pylori cagA基因缺失突变的重组载体pBS-cagA-mutant的连接顺序为: 两侧同源臂是cagA基因编码区上游、下游两端区域的基因片段FL1和FR2, 中间为卡那霉素抗性基因kmR, 以上3个DNA片段与pBluescript SKⅡ(-)连接构建突变载体pBS-cagA-mutant, 模式图见图2. pBS-cagA-mutant经XhoⅠ+XbaⅠ双酶切产生2500 bp(FL1+kmR+FR2), 3000 bp片段; 经XhoⅠ+PstⅠ双酶切产生1520 bp(FL1+kmR), 3985 bp片段; XhoⅠ+Hind Ⅲ双酶切产生720 bp(FL1), 4785 bp片段(图3). 与设计结果完全相符.
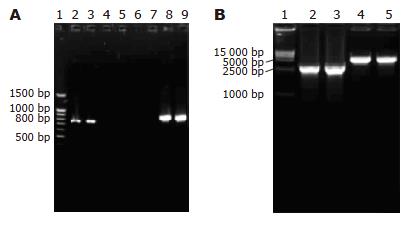
卡那霉素筛选出的cagA基因缺失突变株H. pylori 27△cagA及其对应的野生株H. pylori 27, 采用PCR方法进行鉴定, 以引物P5, P6扩增cagA基因片段, 以引物P7, P8扩增卡那霉素抗性基因片段, 结果显示, 突变株H. pylori27△cagA可以扩出长约800 bp的卡那霉素抗性基因片段, 而不能扩出cagA基因长约760 bp片段; 野生株H. pylori 27可以扩出长约760 bp的cagA基因片段, 而不能扩出卡那霉素抗性基因片段(图4A). 引物P1, P4的扩增结果显示, H. pylori 27野生株可扩出约5100 bp的片段(包括cagA基因完整编码区序列和5, 3非编码区序列以及cagA基因上下游结构基因的部分序列), H. pylori 27△cagA突变株仅能扩出约2500 bp的片段, 提示cagA基因完整编码区缺失(约3510 bp), 代之以800 bp的卡那抗性基因(图4B). 测序结果进一步证实同源重组成功, GenBank登录号: DQ789393(序列略). 突变菌株的稳定性鉴定: 野生株和突变株分别连续培养25代后, 其基因组DNA的PCR结果与上述相同, 表明cagA基因缺失突变株具有良好稳定性.
cagA是H. pylori最重要的毒力因子之一, 根据该蛋白有无可将H. pylori分成两型:Ⅰ型为高毒力株, 含cagA基因, 表达cagA和vacA蛋白, 具有空泡毒素活性; Ⅱ型为低毒力株, 不含cagA基因, 不表达cagA和vacA蛋白, 无空泡毒素活性[6]. 流行病学研究发现, H. pyloriⅠ型菌株可明显增加患严重性胃炎及胃癌的危险性[7-8]. 因此, cagA可能是一种重要的疾病相关蛋白, 在H. pylori致病过程中发挥重要作用. 编码cagA蛋白的cagA基因定位于被称为cag致病岛(cag pathogenicity island, cagPAI)的长约40 kb的基因群中, 其中包含约编码27-31种蛋白质的基因. cagPAI的部分序列可编码形成注射器样结构的Ⅳ型分泌系统, 该系统可以把cagA蛋白注入宿主细胞. 迄今为止, cagA是H. pylori毒力因子中唯一一个通过这种方式进入宿主细胞的蛋白质, 因此对于cagA蛋白的研究备受关注[2]. 为了更好的研究中国幽门螺杆菌cagA基因的功能, 本文根据同源重组的原理构建了cagA基因缺失突变株: 采用PCR方法扩增cagA基因两端区域目的基因片段FL1和FR2, 其中FL1包含有cagA基因完整的调控序列, 运用基因重组方法将卡那霉素抗性基因(kmR)连接到FL1和FR2两片段之间, 并共同构建到pBluescriptSKⅡ(-)载体的多克隆位点中, 构建出带卡那霉素抗性标志的cagA基因缺失突变的pBS-cagA-mutant载体. 将该载体转化大肠杆菌DH5α, 转化菌可以在氨苄青霉素/卡那霉素双抗性培养基中生长, 提示卡那霉素抗性基因可以在cagA基因调控序列作用下表达. 我们采用电穿孔转化技术, 将突变载体pBS-cagA-mutant转化进入野生株H. pylori 27菌体, 突变载体与野生株染色体之间发生同源重组, 使卡那霉素抗性基因(kmR)置换野生株的cagA基因, 随后因突变载体不能在H. pylori菌体内生长复制而丢失, 所以cagA基因缺失的突变株H. pylori 27△cagA得以构建. 本文所用的电穿孔技术是一种高效的基因转移方法, 且没有种属的特异要求, 被广泛用于各种细菌、细胞的基因转化和转染中. 但不同的目的菌株或细胞的转化体系中, 缓冲液不同, 所用电压、电阻、电容也不同[9]. 本研究实验过程中, 经反复比较不同条件的转化情况, 确定了最佳条件. 与Yuan et al[5]的条件略有不同, 可能与所用菌株不同有关.
检测突变株H. pylori 27△cagA中是否还存在cagA基因, 是最后认证cagA基因缺失突变株构建成功与否的关键. 我们对野生株和突变株的基因组DNA进行了PCR鉴定: 野生株可以扩出cagA基因片段, 而不能扩出卡那霉素抗性基因; 突变株未能扩出cagA基因片段, 但可扩出卡那霉素抗性基因, 提示cagA基因已经缺失. 另外, 用cagA基因两侧的引物进行长片段扩增, 也得到了相同的验证: 野生株可以扩出包含完整cagA基因和非编码序列的长约5100 bp片段, 而突变株仅能得到约2500 bp的基因片段, 提示cagA基因完整编码区(3510 bp)被卡那霉素抗性基因(800 bp)所取代. 以往国外虽有构建cagA基因失活突变株的报道, 但因均采用在cagA基因编码区中插入抗性基因使cagA基因失活, 进而研究cagA基因的相关功能, 但是由于残存cagA基因片段的存在, 必然会对研究结果产生一定影响, 不利于评价完整cagA基因的功能[10-11]. 另外, cagA基因失活突变株均是以西方菌株或国际参考株为基础进行构建[12], 而就cagA分子而言, 东西方菌株之间存在明显差异[13-14], 以西方菌株为基础的研究结果不能全面反映东方菌株cagA基因的生物学作用. 因此构建一株中国人群感染H. pylori的cagA基因缺失突变株具有重要的价值及意义. 本研究首次构建出一株中国人来源的、cagA全基因缺失的H. pylori突变株, 为进一步研究幽门螺杆菌中国株cagA基因的功能, 阐明其在H. pylori致病中的地位及作用奠定了实验基础. 目前, 有关H. pylori 27野生株与H. pylori27△cagA突变株的相关功能差异研究正在进行之中.
世界范围内不同H. pylori菌株cagA分子变异较大, 该变异可能导致不同菌株的cagA生物学作用乃至致病性不同. 我国作为H. pylori感染较严重的国家之一, 临床分离株中超过90%含有cagA基因, 但对于毒力因子cagA的生物学功能不甚明了. 另外, 由于cagA阴性自然突变株与cagA阳性野生株之间缺乏除cagA基因以外的相同遗传背景, 所以无法对cagA基因功能做出真实判断. 目前, 利用基因打靶技术, 敲除生物体内特定的靶基因已成为后基因组时代研究基因功能最直接和最有效的手段.
本文通过同源重组方法敲除幽门螺杆菌中国株MEL-H. pylori 27 cagA基因, 首次构建出cagA基因全部缺失的源自中国的幽门螺杆菌cagA阴性突变株, 为研究中国株幽门螺杆菌cagA基因的生物学功能及在毒力相关蛋白表达中的作用, 从而最终在明确cagA基因与幽门螺杆菌致病性的关系等方面提供良好的实验前提.
本文构建了一株中国人来源的、cagA基因缺失的幽门螺杆菌突变株, 为今后研究幽门螺杆菌cagA基因的功能做了一定的基础研究工作. 文章选题准确, 研究方法合理, 结果可靠.
电编: 张敏 编辑:张焕兰
| 1. | 严 英, 钱 利生. 幽门螺杆菌毒力因子的研究进展. 国外医学(流行病学传染病学分册). 2002;29:27-30. |
| 2. | Al-Ghoul L, Wessler S, Hundertmark T, Kruger S, Fischer W, Wunder C, Haas R, Roessner A, Naumann M. Analysis of the type IV secretion system-dependent cell motility of Helicobacter pylori-infected epithelial cells. Biochem Biophys Res Commun. 2004;322:860-866. [PubMed] [DOI] |
| 3. | Stein M, Bagnoli F, Halenbeck R, Rappuoli R, Fantl WJ, Covacci A. c-Src/Lyn kinases activate Helicobacter pylori CagA through tyrosine phosphorylation of the EPIYA motifs. Mol Microbiol. 2002;43:971-980. [PubMed] [DOI] |
| 4. | Hsu PI, Hwang IR, Cittelly D, Lai KH, El-Zimaity HM, Gutierrez O, Kim JG, Osato MS, Graham DY, Yamaoka Y. Clinical presentation in relation to diversity within the Helicobacter pylori cag pathogenicity island. Am J Gastroenterol. 2002;97:2231-2238. [PubMed] [DOI] |
| 5. | Yuan JP, Li T, Shi XD, Hu BY, Yang GZ, Tong SQ, Guo XK. Deletion of Helicobacter pylori vacuolating cytotoxin gene by introduction of directed mutagenesis. World J Gastroenterol. 2003;9:2251-2257. [PubMed] [DOI] |
| 6. | Censini S, Lange C, Xiang Z, Crabtree JE, Ghiara P, Borodovsky M, Rappuoli R, Covacci A. cag, a pathogenicity island ofHelicobacter pylori, encodes type I-specific and disease-associated virulence factors. Proc Natl Acad Sci USA. 1996;93:14648-14653. [PubMed] [DOI] |
| 7. | Nomura AM, Lee J, Stemmermann GN, Nomura RY, Perez-Perez GI, Blaser MJ. Helicobacter pylori CagA seropositivity and gastric carcinoma risk in a Japanese American population. J Infect Dis. 2002;186:1138-1144. [PubMed] [DOI] |
| 8. | Enroth H, Kraaz W, Engstrand L, Nyren O, Rohan T. Helicobacter pylori strain types and risk of gastric cancer: a case-control study. Cancer Epidemiol Biomarkers Prev. 2000;9:981-985. [PubMed] |
| 9. | Tilly K, Elias AF, Bono JL, Stewart P, Rosa P. DNA exchange and insertional inactivation in spirochetes. J Mol Microbiol Biotechnol. 2000;2:433-442. [PubMed] |
| 10. | Tummuru MK, Cover TL, Blaser MJ. Mutation of the cytotoxin-associated cagA gene does not affect the vacuolating cytotoxin activity of Helicobacter pylori. Infect Immun. 1994;62:2609-2613. [PubMed] |
| 11. | Crabtree JE, Xiang Z, Lindley IJ, Tompkins DS, Rappuoli R, Covacci A. Induction of interleukin-8 secretion from gastric epithelial cells by a cagA negative isogenic mutant of Helicobacter pylori. J Clin Pathol. 1995;48:967-969. [PubMed] [DOI] |
| 12. | Zeng X, He LH, Yin Y, Zhang MJ, Zhang JZ. Deletion of cagA gene of Helicobacter pylori by PCR products. World J Gastroenterol. 2005;11:3255-3259. [PubMed] [DOI] |
| 13. | Azuma T, Yamazaki S, Yamakawa A, Ohtani M, Muramatsu A, Suto H, Ito Y, Dojo M, Yamazaki Y, Kuriyama M. Association between diversity in the Src homology 2 domain-containing tyrosine phosphatase binding site of Helicobacter pylori CagA protein and gastric atrophy and cancer. J Infect Dis. 2004;189:820-827. [PubMed] [DOI] |
| 14. | Higashi H, Tsutsumi R, Fujita A, Yamazaki S, Asaka M, Azuma T, Hatakeyama M. Biological activity of the Helicobacter pylori virulence factor CagA is determined by variation in the tyrosine phosphorylation sites. Proc Natl Acad Sci USA. 2002;99:14428-14433. [PubMed] [DOI] |