修回日期: 2005-12-01
接受日期: 2005-12-08
在线出版日期: 2006-01-18
目的: 在肠毒素性大肠杆菌CS3菌毛表面构建10肽随机肽库.
方法: 首先将原有的单酶切CS3菌毛呈现载体改造为双酶切载体, 并证实改造后的载体能正确形成CS3菌毛. 同时设计合成2条寡核苷酸序列, 链1含有1个10肽随机编码序列(NNS)10, 链2可与链1的3'端互补. 两条链经过退火、延伸、酶切和回收与经过同样酶切的呈现载体连接, 连接产物纯化后分多次电击转化, 获得随机肽库. 随机挑选10个克隆进行测序并对测序结果进行分析.
结果: 获得1个库容量为1.8×106大小的随机肽库. 测序结果显示, 所构建肽库的基本框架与预期设计相符, 4个寡核苷酸出现的频率也与理论值相接近.
结论: 在肠毒素性大肠杆菌CS3菌毛表面成功构建库容量为1.8×106大小的10肽随机肽库. 为下一步利用肽库进行筛选奠定了基础.
引文著录: 刘向昕, 袁盛凌, 展德文, 郑继平, 刘纯杰, 王芃, 王令春, 张兆山. 大肠杆菌表面 CS3 菌毛展示随机肽库的构建. 世界华人消化杂志 2006; 14(2): 158-162
Revised: December 1, 2005
Accepted: December 8, 2005
Published online: January 18, 2006
AIM: To construct a CS3 fimbria-displayed random peptide library on the Escherichia coli cell surface
METHODS: Firstly, a new display vector with double restriction sites was reconstructed, and the ability of the new vector to form CS3 fimbriae on E. coli surface was confirmed by Western blot and transmission electron microscopy. Then two oligonucleotides were synthesized, one of which contained a random oligonucleotide encoding region (NNK)10 and the other was its complement. The two synthesized oligonucleotides was annealed and then extended by Klenow Fragment. The double-stranded oligonucleotides were digested by XhoT and BamHT and purified by PAGE, then inserted into the digested display vector. The ligation product was purified and electroporated into XL1-Blue. Ten randomly selected clones were sequenced and the sequences were analyzed..
RESULTS: A library with diversity of 1.8×106 was obtained. The sequencing results confirmed the basic frame of the constructed library to be correct and the bases of A, T, G, and C were distributed randomly.
CONCLUSION: A random ten-peptide library is displayed on the Escherichia coli cell surface using CS3 fimbriae display system.
- Citation: Liu XX, Yuan SL, Zhan DW, Zheng JP, Liu CJ, Wang P, Wang LC, Zhang ZS. Construction of a CS3 fimbria-displayed random peptide library on Escherichia coli cell surface. Shijie Huaren Xiaohua Zazhi 2006; 14(2): 158-162
- URL: https://www.wjgnet.com/1009-3079/full/v14/i2/158.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i2.158
众所周知, 疫苗是通过表面的多个优势抗原表位, 刺激机体产生免疫应答, 抵制外来病原体发挥作用的. 因而, 抗原表位的获得在疫苗研制中具有极原表位的主要方法之一. 所谓微生物表面展示随机肽库, 就是将随机编码的多肽序列与相应的载体(噬菌体、细菌、病毒和酵母等)蛋白融合并活性表达于其表面, 构成随机肽库. 利用生物淘洗等筛选方法, 即可快速有效地获得抗原表位. 在微生物表面展示随机肽库中, 噬菌体肽库比较成熟, 细菌展示肽库是继其后发展起来的, 其优势在于除了可用传统的生物淘洗法筛选外, 还可用荧光激活细胞分选(fluorescence-activated cell sorting, FACS)技术进行更快速更高效的筛选[1]. 另外, 在利用筛选出的抗原表位构建重组活菌苗的过程中, 细菌表面展示系统与噬菌体相比也有独特的优势, 包括可供选择的载体蛋白种类较多, 免疫佐剂作用明显以及易于构建多价疫苗等[2].
研究表明, 细菌的外膜蛋白、脂蛋白、菌毛蛋白(如K88, CS31A, P纤毛)以及鞭毛蛋白都能够作为外源蛋白的表面呈现载体. Lu et al[3]在1995年利用鞭毛为骨架构建FliTrxTM随机12肽库, 应用到抗原表位筛选[3]、蛋白磷酸化酶1结合位点确定[4]、细胞特异性结合肽[5]以及肿瘤成血管肽筛选研究[6]; Kjaergaard et al[7]利用菌毛FimH为骨架构建随机肽库, 并从中成功筛选得到Zn2+结合肽. 菌毛在细菌表面数量很多, 每根菌毛又含有500-1 000拷贝的菌毛蛋白, 而且具有较强的免疫原性, 特别适合于构建基因工程活菌疫苗. 肠毒素性大肠杆菌(enterotoxigenic Escherichia coli, ETEC)是引起婴幼儿和旅游者腹泻的主要病原菌, 他的致病性依赖于两类毒性因子, 即肠毒素和定居因子抗原(colonization factor antigens, CFAs). CS3是CFA/Ⅱ阳性菌株的共有抗原, 由60 Mu的大质粒编码[8]. 本实验室构建了CS3菌毛呈现载体, 并证明在包括信号肽在内的CS3的第72-73位氨基酸间插入从9肽到19肽的多种抗原表位后, 外源表位都能得到呈现[9]. 现利用此呈现载体并加以改造, 在大肠杆菌CS3菌毛上构建了细菌表面展示随机肽库, 为今后筛选抗原表位以及进一步构建重组活菌苗的研究奠定基础.
大肠杆菌DH5a和XL1-Blue为本实验室保存. CS3呈现载体pCSX72由本实验室构建. CS3单克隆抗体由瑞典Gobebourgs大学A.M.Svennerholm教授惠赠. 限制性内切酶、DNA聚合酶Ⅰ(Klenow大片段)以及T4 DNA连接酶等均为New England Biolabs产品. 质粒纯化试剂盒购自Promega公司, 琼脂糖凝胶DNA回收试剂盒和聚丙烯酰胺凝胶DNA回收试剂盒购自北京天为时代公司, HRP标记羊抗鼠IgG, DAB显色试剂盒购自北京中杉金桥生物公司. DNA分子质量标准pBR322/MspⅠ和DL15000分别为华美生物工程公司和大连宝生物公司产品, 低分子质量蛋白标准购自安玛西亚公司. 引物合成及测序分别由上海博亚生物技术公司和上海华诺生物科技公司完成. JOELJME 1200EXⅡ扫描电镜由军事医学科学院微生物流行病研究所提供.
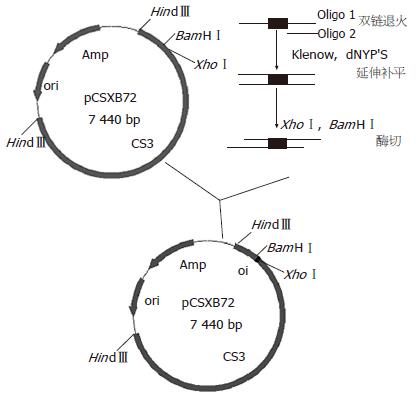
构建双酶切载体pCSXB72合成2条寡核苷酸序列: 5'CTAGACTCGAGACTAGTACGCGTGGATCCT 3'(含有XhoⅠ和BamHⅠ酶切位点)和5'TGAGCTCTGATCATGCGCACCTAGGAGATC 3'(含有XhoⅠ和BamHⅠ酶切位点). 将2条引物退火, 两端即形成XbaⅠ酶切位点, 与同样用XbaⅠ酶切并去磷酸化的载体pCSX72连接, 构建为包含XhoⅠ和BamHⅠ酶切位点的双酶切载体pCSXB72.
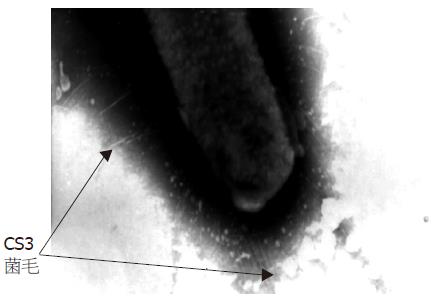
1.2.1 电镜下观察菌毛形成[10]: 收获在CFA平板上培养18-20 h的转化有pCSXB72的大肠杆菌XL1-Blue, PBS洗涤3遍后, 用适量的PBS重悬, 使A600 = 1.0; 吸取一滴细菌悬液滴加在喷有炭膜的铜网上, 放置1 min; 将菌液弃掉, 用PBS洗一遍, 稍干后用20 g/L磷钨酸负染20 s, 干燥后在扫描电镜下观察.
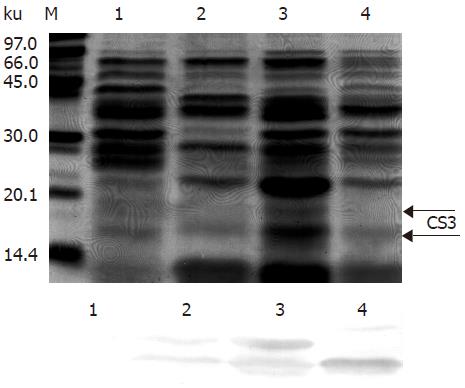
1.2.2 菌毛蛋白的粗提及Western印迹分析: 收获在CFA平板上培养的重组菌, 用PBS洗2遍, 重悬于适量的PBS中, 60 ℃水浴放置30 min, 涡悬振荡器剧烈振荡1 min, 12 000 g离心10 min, 上清即含有菌毛蛋白[11]. 菌毛蛋白经过SDS-PAGE(150 g/L分离胶和50 g/L积层胶)分离, 用湿式转移电泳的方法将蛋白质转移到硝酸纤维素膜上, 加封闭液(含50 g/L脱脂奶粉的PBST)室温缓慢摇动封闭1 h, 加CS3单克隆抗体结合2 h, PBST洗膜3次后, 加HRP标记羊抗鼠IgG室温孵育1 h, PBST洗膜3次后, 用DAB显色试剂盒显色.
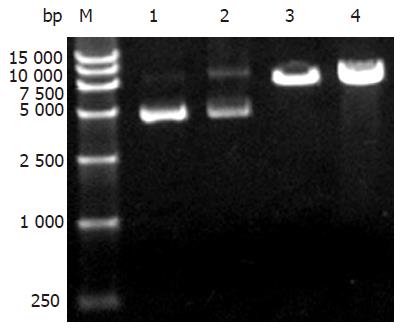
1.2.3 随机肽库的构建: 随机肽库构建[12]的基本流程如(图1), 具体操作: (1)线性化载体的制备. 提取质粒pCSXB72, 用XhoⅠ和BamHⅠ进行双酶切, 琼脂糖凝胶回收目的片段, 共需线性化载体约为12 mg, 冻存备用. (2)随机寡核苷酸插入片段的制备. 根据载体pCSXB72两端的XhoⅠ和BamHⅠ酶切位点情况以及构建随机肽库的要求, 设计合成两条随机寡核苷酸片段, 链1序列为5'GGACGCCTCGAG(NNS)10GGATCCGCGACCAGT 3', 其中NNS为随机序列(N为A、G、C、T四种碱基之一, S为G、C两种碱基之一), 两端为保护碱基和酶切位点; 链2序列为5'ACTGGTCGCGGATCC 3', 与链1的3'端互补. 在Eppendorf管中加入退火缓冲液[TE(pH 8.0)+100 mmol/L NaCl]12 mL, 100 mmol/L的链1, 链2各4 mL, 95℃加热10 min, 缓慢降至室温, 继续加入2.5 mmol/L的dNTP 10 mL, Klenow大片段10 mL, 10×Klenow缓冲液16 mL, 37℃ 40 min, 75℃ 15 min. XhoⅠ和BamHⅠ进行双酶切, PAGE(160 g/L)分离, 聚丙烯酰胺凝胶DNA回收试剂盒回收目的条带, 共需插入片段约300 ng. (3)连接及连接产物的纯化. 对酶切回收的载体与插入片段进行定量, 在300 mL反应体系以适当的连接比连接数管, 连接产物用酚: 氯仿(1:1)抽提2次, 氯仿抽提1次后, 进行乙醇沉淀, 最后分别溶于35 mL TE(pH 8.0)中. (4)电转化构建随机肽库. 电转化感受态细胞XL1-Blue的制备参照文献[13]. 取3 mL回收纯化的连接产物加入100 mL电转化感受态细胞中, 利用Bio-Rad公司的电转化仪进行电转化. 电击参数为: 电极间隙0.2 cm, 电压2.5 kV, 电容25 mF, 电阻200 W, 电击后尽快加入500 mL SOC培养基混匀, 将混合物转移至Eppendorf管中, 冰上放置, 如此共电击30次. 待全部样品转化完毕, 37℃振荡培养45 min, 取适当样品稀释后涂SOB平板. 其他加入到LB培养基30 mL中, 37℃振荡培养6 h, 取样进行平板计数. 其余加甘油至终浓度为250 mL/L, 分装成小管, -70 ℃冻存备用. (5)序列测定. 根据载体pCSXB72序列, 在插入位点上游设计测序引物, 5'CACTGTCAGCTATGAGTTCA3'. 取构建的肽库中适量样品稀释后涂平板, 随机挑选10个克隆, 进行序列测定, 对肽库进行基本框架验证及多样性评价.
由于单酶切载体自连率太高, 我们在原有载体的基础上构建了双酶切载体pCSXB72, 测序证明载体构建正确. 将pCSXB72转化XL1-Blue后在电镜下可观察到菌毛生长(图2), 进一步提取菌毛蛋白进行Western印迹实验, 证明新构建的载体能形成CS3菌毛(图3).
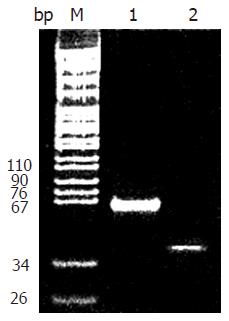
在构建肽库的过程中, 首先将载体进行双酶切(图4), 与经过同样酶切的随机寡核苷酸片段(图5)进行连接. 由于载体与插入片段的摩尔比是连接效率的关键因素, 我们分别将载体与插入片段以1:0, 1:3, 1:5, 1:10的摩尔比进行连接. 实验表明, 当载体与插入片段的摩尔比是1:5时, 连接效率最高, 此时与摩尔比为1:0的转化菌落数相比大于100倍, 因此采用1:5的摩尔比进行连接. 连接产物电转化后, 取样进行平板计数, 表明含有1.8×106个原始克隆, 即该随机肽库的库容量为1.8×106. 原始肽库扩增后再次进行平板计数为6.5×108, 这样每一克隆的拷贝数约为360, 所构建肽库的库容量可基本满足筛选的要求.
从构建好的随机肽库中, 任意挑选10个克隆进行测序(图6), 根据测序结果统计四种碱基出现频率(表1). 结果显示, 四种碱基出现情况与预先设计的NNS相符, 即第1, 2位碱基分别为A, C, G, T中的任一种, 第3位碱基为G/C中的任1种. 四种碱基出现频率与理论值相近. 而实测值与理论值的差异可能是由于所测序列较少, 当所测序列样本增大时, 这种差别就会减少到最低. 在氨基酸组成方面, 以上10个序列中包含了19种氨基酸, 只有苯丙氨酸未包括. 这些结果表明, 我们所构建肽库的基本框架以及寡核苷酸出现的频率均达到了肽库构建的要求. 同时, 从随机肽库中任意挑选一个克隆, 进行Western印迹分析, 证明在插入10肽随机序列后, 仍能正确形成CS3菌毛(图3).
| A | G | C | T | |
| N | 22.5 | 36.5 | 17.0 | 24.0 |
| S | 0 | 56.0 | 44.0 | 0 |
尽管随机肽库的构建方法目前已比较成熟, 但是不同的随机肽库、不同的展示载体, 具体方法往往不一样, 肽库构建的每一个过程都有可能影响其质量. 因此, 在肽库构建的过程中, 要通过提高每一步反应的效率, 构建具有更大库容量和多样性的肽库, 为筛选奠定良好的基础. 随机寡核苷酸片段的设计采用常用的NNS模式, 组成32种密码子, 既可以编码20种氨基酸和一种终止密码子(TAG), 又减少了随机寡核苷酸序列中出现终止密码子的频率. 由于前期研究中构建的单酶切呈现载体pCSX72自连现象严重, 因此首先将其改造为双酶切载体, 大大减少了自连现象, 提高了连接效率. 接着, 在对载体和随机寡核苷酸片段进行彻底的酶切、确定连接条件和转化条件等多个环节中, 都进行反复实验以摸索出最佳条件. 受细菌本身转化效率的影响, 构建细菌表面展示随机肽库的库容量较噬菌体随机肽库要小的多, 目前已报道的随机肽库容量多在105-107之间. 我们通过多次转化获得的随机肽库容量为1.8×106, 可基本满足下一步筛选的要求.
在革兰阴性菌表面展示蛋白, 需要重组蛋白能从胞质运输至外膜, 然后穿过外膜锚定于细菌外表面. 然而蛋白靶向并插入外膜的机制尚不清楚, 而且外源蛋白插入外膜可能对细胞产生毒性. 另外, 由于外膜中脂多糖的空间位阻效应, 可导致外源多肽表面展示效果不好, 进而影响展示的外源蛋白与抗体和小分子配体的相互作用. 如果将重组多肽展示于细菌表面附属结构, 如鞭毛或菌毛, 他们突出于细菌表面, 这样就可避免空间位阻效应. 同时, 外源蛋白插入的部位多为菌毛表面突出的环状结构, 这样使外源蛋白具有一定的空间构象依赖性, 更有利于与目标蛋白结合[14]. 菌毛展示的肽段在氨基酸组成方面有一些限制. 如展示的多肽相对来说是亲水性的, 而且很少出现半胱氨酸. 这可能是由于在水性环境中菌毛无法展示高度疏水的片段, 而半胱氨酸间形成的二硫键会干扰蛋白向细胞表面运输[15]. 在本研究中, 在测序的十个序列的100个氨基酸中疏水性氨基酸只有31个, 但出现半胱氨酸的个数为7个. 另外, 为保证外源蛋白的插入不会影响大分子结构蛋白的自我装配, 菌毛展示片段的大小也受到一定程度的限制. 从已有的研究来看, 不同菌毛的主要结构蛋白只能容纳中等大小的插入片段(10-30个氨基酸), 但对于次要结构蛋白, 如Ⅰ型菌毛FimH, 能容纳的肽段可大于100个氨基酸. 这与丝状噬菌体类似, 丝状噬菌体的主要结构蛋白pⅧ对于插入片段大小有严格的限制, 而次要结构蛋白pⅢ蛋白能容纳较大的片段. 实际上, 菌毛与丝状噬菌体的结构在许多方面相似, 在表面展示领域, 可以把菌毛看成是附着于表面的噬菌体[15]. CS3亚单位是CS3菌毛的主要结构蛋白, 本实验室的前期研究表明, 在包括信号肽在内的CS3的第72-73位氨基酸间插入从九肽到十九肽的多种抗原表位后, 外源表位都能得到呈现.
菌毛在抗原表位展示方面是很有吸引力的候选载体, 因为菌毛在细胞表面以极高的拷贝存在, 他们具有强免疫原性和黏附特性, 而且易于纯化[14]. 因此, 我们打算利用构建的CS3菌毛表面展示随机肽库, 筛选抗体的抗原表位, 并进一步用于构建基因工程活菌疫苗.
运用重组DNA技术在微生物表面呈现外源蛋白或多肽, 在微生物学、分子生物学、疫苗学和生物技术等方面得到了广泛的应用. 对噬菌体、细菌、真菌、病毒等都在进行这方面的研究. 噬菌体表面展示技术比较成熟, 但其本身存在一些限制. 细菌表面展示系统是继噬菌体表面展示系统之后发展起来的, 其呈现载体多样, 可根据不同的需要呈现不同的蛋白, 达到不同的目的. 近年来其应用领域已扩展到包括重组细菌疫苗, 多肽库筛选系统, 全细胞吸附剂, 重组全细胞催化剂以及作为诊断的细胞基础的固相试剂等.
噬菌体展示技术在肽库构建与筛选研究中获得极大成功, 细胞表面展示技术作为一种配体筛选方法, 是对噬菌体展示文库的有力补充. 一些细胞表面展示的多肽文库已构建成功并用于筛选高亲和力的多肽配体. 如基于E.coli鞭毛蛋白FliC所构建的FLITRX系统, 就被成功用于多肽文库的展示. 另外, 抗体片段也可以展示于细菌或酵母表面, 从而使抗体库的构建成为可能. 细胞表面展示文库的最大优点在于他可借助荧光激活细胞分选技术进行高通量筛选, 从而大大加快和简化筛选过程.
本研究中采用的CS3菌毛呈现载体是由我们自行构建的载体, 并进一步用于构建随机肽库, 因而具有良好的工作基础和自主知识产权; 在肽库构建过程中, 通过将其改造为双酶切载体和优化各种酶切, 连接和转化条件, 提高了库容量, 可基本满足下一步筛选的要求.
利用多肽库和体外筛选技术来识别和分离具有特定性质的多肽, 是生物学中一个快速发展的领域. 其应用领域包括: 抗原表位的筛选; 分离金属结合肽; 抗体亲和力成熟; 提高酶催化活性; 改变底物特异性; 筛选结合肽等. 由于菌毛在细胞表面以极高的拷贝存在, 他们具有强免疫原性和黏附特性, 而且易于纯化, 因此利用菌毛呈现的随机肽库可用于筛选抗原表位或金属结合肽, 并进一步用于构建基因工程活菌疫苗或重金属去除吸附剂.
该研究利用CS3菌毛呈现载体寡核苷酸分子克隆技术构建随机肽库. 获得一个库容量为1.8×106大小的肽库, 为筛选抗原表位以及进一步构建重组活菌苗的研究奠定基础. 另外, 本研究起点较高方法技术先进, 结果可靠, 具有一定的科学意义和实用价值.
编辑: 潘伯荣 审读: 张海宁 电编: 张敏
| 1. | 沈 倍奋. 分子文库. 北京: 科学出版社 2001; 100-115. |
| 2. | Georgiou G, Stathopoulos C, Daugherty PS, Nayak AR, Iverson BL, Curtiss R 3rd. Display of heterolo-gous proteins on the surface of microorganisms: from the screening of combinatorial libraries to live recombinant vaccines. Nat Biotechnol. 1997;15:29-34. [PubMed] |
| 3. | Lu Z, Murray KS, Van Cleave V, LaVallie ER, Stahl ML, McCoy JM. Expression of thioredoxin random peptide libraries on the Escherichia coli cell surface as functional fusions to flagellin: a system designed for exploring protein-protein interactions. Biotechnology. 1995;13:366-372. [PubMed] |
| 4. | Zhao S, Lee EY. A protein phosphatase-1-binding motif identified by the panning of a random peptide display library. J Biol Chem. 1997;272:28368-28372. [PubMed] |
| 5. | Herrmann A, Pieper M, Schrader J. Selection of cell specific peptides in a rat carotid injury model using a random peptide-presenting bacterial library. Biochim Biophys Acta. 1999;1472:529-536. [PubMed] |
| 6. | Brown CK, Modzelewski RA, Johnson CS, Wong MK. A novel approach for the identification of uni-que tumor vasculature binding peptides using an E. coli peptide display library. Ann Surg Oncol. 2000;7:743-749. [PubMed] |
| 7. | Kjaergaard K, Schembri MA, Klemm P. Novel Zn(2+)-chelating peptides selected from a fimbria-displayed random peptide library. Appl Environ Microbiol. 2001;67:5467-5473. [PubMed] |
| 8. | Jalajakumari MB, Thomas CJ, Halter R, Manning PA. Genes for biosynthesis and assembly of CS3 pili of CFA/II enterotoxigenic Escherichia coli: novel regulation of pilus production by bypassing an amber codon. Mol Microbiol. 1989;3:1685-1695. [PubMed] |
| 10. | Bakker D, van Zijderveld FG, van der Veen S, Oudega B, de Graaf FK. K88 fimbriae as carriers of heterologous antigenic determinants. Microb Pathog. 1990;8:343-352. [PubMed] |
| 11. | Der Vartanian M, Girardeau JP, Martin C, Rousset E, Chavarot M, Laude H, Contrepois M. An Escherichia coli CS31A fibrillum chimera capable of inducing memory antibodies in outbred mice following booster immunization with the entero-pathogenic coronavirus transmissible gastroenteritis virus. Vaccine. 1997;15:111-120. [PubMed] |
| 12. | Noren KA, Noren CJ. Construction of high-comple-xity combinatorial phage display peptide libraries. Methods. 2001;23:169-178. [PubMed] |
| 13. | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. New York:. Cold Spring Harbor Laboratory Press. 2001;1119-1122. |
| 14. | Samuelson P, Gunneriusson E, Nygren PA, Stahl S. Display of proteins on bacteria. J Biotechnol 2002; 96: 129-154. [PubMed] |
| 15. | Klemm P, Schembri MA. Fimbrial surface display systems in bacteria: from vaccines to random libra-ries. Microbiology. 2000;146:3025-3032. [PubMed] |