修回日期: 2006-04-15
接受日期: 2006-04-22
在线出版日期: 2006-04-28
目的: 研究Barrett食管(BE)和食管腺癌的基因表达谱, 筛查与食管腺癌相关的基因.
方法: 使用Dchip软件对已经在GEO数据库中公开的BE和食管腺癌Affymetrix芯片表达谱数据进行分析. 还原扫描图像进行独立核验, 并对基因和组织进行双向聚类, 最后用配对t检验筛查出在BE和食管腺癌中表达水平都发生变化的基因, 并进一步分析其功能.
结果: 24张Affymetrix芯片的杂交质量稳定, 被污染和发生交叉杂交的探针簇都少于5%. 对基因和样本的双向聚类表明, 大部分组织分类正确. 只有N8和A5位于错误的组织类型中. 对其余22张芯片再次分别进行配对t检验, 得到24个基因. 其中表达水平呈进行性上升的5个, 呈进行性下降的19个. 新检出的PITX1已在稍前不久的另一项研究中得到证实.
结论: 用新的分析方法研究已公开的表达谱芯片资料为研究肿瘤的发病机制提供了新的手段.
引文著录: 陈威震, 王雯. Barrett食管和食管腺癌的基因表达谱研究. 世界华人消化杂志 2006; 14(12): 1156-1160
Revised: April 15, 2006
Accepted: April 22, 2006
Published online: April 28, 2006
AIM: To investigate the changes of gene expre-ssion in Barrett esophagus and adenocarci-noma of esophagus.
METHODS: A dataset of gene expression profilings for 24 paired samples of normal esophageal epithelium, Barrett esophagus and adenocarcinoma of esophagus were downloaded from database GEO and analyzed with Dchip software. The dataset recovered to raw scanned image was firstly verified. Then the gene expression profilings were subjected to gene and sample clusterings to identify the contaminated samples. Finally the genes selected by pair-wise t-tests were reviewed for function.
RESULTS: The quality of GeneChip hybridizations was good, and the percentages of contaminated and cross-hybridized probe sets were both below 5%. The gene and sample clusterings showed that most of the GeneChips were grouped perfectly except N8 and A5 which were located in wrong clusters. Excluding the two tissue contaminated GeneChips, the others were subjected to pair-wise t-test. Twenty-four genes were finally selected, of which 5 were progressively up-regulated in Barrett esophagus and adenocarcinoma of esophagus, while 19 down-regulated. The newly discovered PITX1 that was step-wise down-regulated has been confirmed by a recent study.
CONCLUSION: It provides a new way for stu-dying the mechanisms of cancer to reanalyze the publicly accessible datasets of gene expression profiling with new methods.
- Citation: Chen WZ, Wang W. Changes of gene expression in Barrett esophagus and adenocarcinoma of esophagus. Shijie Huaren Xiaohua Zazhi 2006; 14(12): 1156-1160
- URL: https://www.wjgnet.com/1009-3079/full/v14/i12/1156.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v14.i12.1156
Barrett食管(BE)是食管鳞状上皮在长期胃酸作用下被胃的单层柱状上皮所取代的病变. 目前普遍认为食管腺癌几乎都由Barrett食管发展而来[1]. 这种癌前病变与肿瘤之间的特殊关系为研究食管腺癌的发病机制提供了条件. 基因芯片表达谱可以对肿瘤的基因表达水平进行高通量分析[2], 而用新的分析方法重新研究已在互联网上公开的表达谱资料可为了解致癌机制提供新的手段, 目前已被广泛应用于筛查恶性肿瘤相关基因[3-4]. 我们用Dchip软件对已在GEO数据库中公开的一组配对的BE和食管腺癌基因芯片资料重新进行了研究, 试图筛查出在食管鳞状上皮经由BE最终发展为食管腺癌的过程中, 始终发生作用的基因, 以期作为肿瘤标志物评估BE患者发生食管腺癌的风险, 并借以取代代价高而效率差的食管镜筛查, 进而作为治疗性分子靶点.
从美国国立生物信息中心(NCBI)GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载由Kimchi et al[5]提交的基因芯片数据GSE1420, 这其中包括来自8个食管腺癌患者的正常、Barrett食管、食管腺癌组织的共24张Affymetrix U133A芯片的图像扫描数据文件.
1.2.1 扫描图像的还原、标准化和质量核验: 从GO数据库(http://www.geneontology.org/index.shtml)下载基因本体论结构文件function.ontology, process.ontology, component.ontology. 另外从Affymetrix公司网站(http://www.affymetrix.com/index.affx)下载U133A芯片的注释文件. 将以上文件输入Dchip软件. 据此还原原始芯片扫描图像, 以总荧光强度为中位数的芯片N2为基准, 对所有芯片进行标准化. 随后选择PM-only模式分析得出各芯片中所有基因的表达水平. 根据同时生成的芯片分析报告判断芯片中探针交叉杂交和芯片污染的情况.
统计学处理 对24张基因芯片按Barrett食管/正常组织和食管腺癌/Barrett食管分别进行配对t检验(P<0.05), 对在两次比较中表达水平都发生变化的基因进行基因和组织的双向聚类以排除污染组织的影响. 对排除2张组织污染芯片外的22张芯片再次分别进行配对t检验(P<0.01), 检出在两次比较中表达水平都有变化的基因. 通过NCBI的Entrez gene数据库进行基因功能分析.
芯片分析报告显示每张芯片的探针交叉杂交和污染都小于5%, 芯片信号强度分布均匀, 质量可靠, 可进行进一步分析(表1).
| 芯片名 | 芯片代号总荧光强度(未标准化) | 基因表达率(%) | 芯片污染率(%) | 探针交叉杂交率(%) | |
| GSM23385 | N11 | 248 | 57.5 | 0.381 | 0.083 |
| GSM23386 | N2 | 213 | 58.1 | 0.229 | 0.048 |
| GSM23387 | N3 | 210 | 53 | 0.09 | 0.027 |
| GSM23388 | N4 | 213 | 46.5 | 0.233 | 0.086 |
| GSM23389 | N5 | 182 | 58.9 | 0.211 | 0.048 |
| GSM23390 | N6 | 256 | 59.7 | 0.117 | 0.1 |
| GSM23391 | N7 | 188 | 57.8 | 0.978 | 0.213 |
| GSM23392 | N8 | 207 | 62.8 | 4.434 | 0.173 |
| GSM23395 | B12 | 285 | 63.5 | 0.372 | 0.052 |
| GSM23396 | B2 | 301 | 63 | 0.736 | 0.204 |
| GSM23397 | B3 | 254 | 59.1 | 0.13 | 0.056 |
| GSM23398 | B4 | 199 | 57.6 | 0.969 | 0.249 |
| GSM23399 | B5 | 189 | 62.2 | 1.135 | 0.289 |
| GSM23400 | B6 | 142 | 57.7 | 2.899 | 0.127 |
| GSM23401 | B7 | 270 | 60.3 | 0.224 | 0.07 |
| GSM23402 | B8 | 226 | 60.8 | 0.355 | 0.14 |
| GSM23413 | A13 | 235 | 56.6 | 1.243 | 0.358 |
| GSM23417 | A2 | 191 | 61 | 2.634 | 0.199 |
| GSM23419 | A3 | 173 | 60.8 | 0.628 | 0.079 |
| GSM23422 | A4 | 239 | 64 | 1.019 | 0.16 |
| GSM23424 | A5 | 183 | 60.5 | 0.233 | 0.107 |
| GSM23425 | A6 | 265 | 64.6 | 1.71 | 0.284 |
| GSM23426 | A7 | 152 | 58 | 2.405 | 0.231 |
| GSM23427 | A8 | 225 | 62.4 | 0.772 | 0.055 |
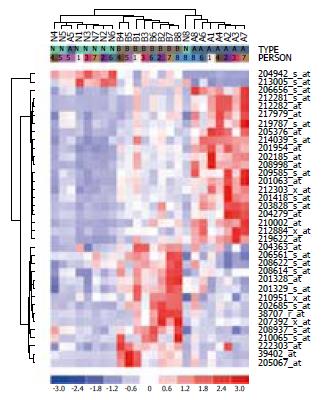
对Barrett食管/正常组织和食管腺癌/BE分别进行配对t检验(P<0.05), 检出36个基因在两次比较中表达水平都有变化. 对这36个基因进行基因和组织的双向聚类的结果表明: 大部分组织分类正确, 只有N8和A5位于错误的组织类型中, 应排除在进一步的分析之外(图1).
对排除污染组织外的22张芯片分别进行配对t检验(P<0.01), 再次检出在两次比较中表达水平都有变化的基因24个. 其中表达水平呈进行性上升的5个, 呈进行性下降的19个(表2).
| 探针簇 | 基因 | BE/正常组织a | 食管腺癌/BEb |
| 204942_s_at | ALDH3B2 | -2.36 | -6.48 |
| 219100_at | OBFC1 | -1.96 | -1.98 |
| 219165_at | PDLIM2 | -1.92 | -2.38 |
| 208502_s_at | PITX1 | -1.85 | -5.56 |
| 201118_at | PGD | -1.83 | -3.81 |
| 219764_at | FZD10 | -1.8 | -3.17 |
| 220569_at | PRO1728 | -1.7 | -2.33 |
| 215367_at | KIAA1614 | -1.68 | -2.63 |
| 220339_s_at | TPSG1 | -1.68 | -2.73 |
| 221295_at | CIDEA | -1.68 | -1.89 |
| 202575_at | CRABP2 | -1.66 | -7.81 |
| 219090_at | SLC24A3 | -1.65 | -2.22 |
| 201851_at | SH3GL1 | -1.59 | -1.71 |
| 209679_s_at | LOC57228 | -1.51 | -2.06 |
| 211667_x_at | V-J-C | -1.5 | -1.71 |
| 202096_s_at | BZRP | -1.47 | -1.81 |
| 212965_at | HIC2 | -1.46 | -1.78 |
| 221113_s_at | WNT16 | -1.45 | -1.48 |
| 216591_s_at | SDHC | -1.41 | -1.54 |
| 200039_s_at | PSMB2 | 1.4 | 1.44 |
| 218053_at | PRPF40A | 1.74 | 1.75 |
| 201954_at | ARPC1B | 1.99 | 1.6 |
| 208998_at | UCP2 | 2.45 | 1.98 |
| 202185_at | PLOD3 | 2.86 | 1.96 |
已知肿瘤发生是多种遗传事件共同作用的结果, 包括原癌基因表达、抑癌基因和错配修复基因缺失、非整倍体形成等等. 高通量表达谱分析提供了从整体水平了解肿瘤发生机制的途径. 因为表达谱涉及的基因往往成千上万, 而各种分析方法的敏感性和特异性各不相同, 用新的分析方法重新研究某特定表达谱资料完全有可能发现新的与肿瘤发生密切相关的基因表达水平变化. Dchip是专门开发用于Affymetrix芯片分析的多功能软件平台[6]. 他采用独特的反复建立探针簇表现模型的方法计算基因的表达水平, 排除了探针交叉杂交和芯片污染的影响, 从而保证了分析的可靠性. 另外还具有聚类、样本比较等功能. 已有多项研究表明Dchip的分析结果可靠[7-8], 并已广泛应用于Affymetrix芯片的研究. 我们采用Dchip软件分析一组食管腺癌患者的正常食管鳞状上皮、Barrett食管、食管腺癌组织的表达谱, 筛查在食管鳞状上皮经由Barrett食管最终发展为食管腺癌的过程中表达水平都发生变化的基因, 并探讨其作用机制.
为方便基因芯片数据的交流和独立核验, 基因芯片数据协会(MGED)制定了描述基因芯片实验的最低限度信息(MIAME)[9]. 符合这一标准的基因芯片实验数据提供了大量原始数据, 可供其他研究者还原扫描图像, 并独立核验芯片杂交的可靠性和进行进一步研究. 我们首先利用符合MIAME标准的图像扫描数据文件还原了原始扫描图像, 并对各芯片质量进行了评估. 独立核验表明所有芯片杂交可靠, 可进行进一步分析.
聚类是一种非监督学习的方法, 不需要已知类的信息, 结果完全依赖于数据本身. 研究表明基于基因表达谱的聚类分析可实现对肿瘤亚型的分类[10]. 我们对BE/正常组织和AD/BE分别进行配对t检验, 用在两次比较中表达水平都有变化的36个基因对组织样本进行聚类. 结果显示大部分组织分类正确, 只有N8和A5位于错误的组织类型中. 我们认为这可能是取材时的组织污染所致, 这与原文用功能组分类和二项分布法分析得出的结论一致. 排除以上两个样本后, 采用更为严格的标准(P<0.01)再次分别进行配对t检验, 得到24个基因. 其中表达水平呈阶梯性上升的5个, 呈进行性下降的19个.
成对同源异型结构域蛋白转录因子1(paired-like homeodomain transcription factor 1, PITX1)属于同源异形盒基因家族, 这是一段高度保守的约含180个碱基对的DNA基序, 他编码的同源结构域蛋白是DNA结合转录因子, 在器官发育和维持生物体的左右对称中发挥重要作用. 部分homeobox基因已被证实与肿瘤有关[11]. 我们的研究表明, 相对于正常食管鳞状上皮, PITX1的表达水平在BE和AD中呈进行性下降, 这一发现已在稍前不久的另一项研究中得到证实[12]. Kolfschoten et al[13]证实PITX1通过RAS-鸟苷三磷酸酶激活蛋白(RAS-GTPase-activating protein, RASAL1)抑制RAS活性而起到抑癌作用. PITX1在Barrett食管和食管腺癌的下降提示这一机制与在食管腺癌的发生密切相关. UCP2编码线粒体内膜上的阴离子通道蛋白. 缺氧时UCP2的过表达可以通过降低膜电压、减少活性氧簇(reactive oxygen species, ROS)而起到保护神经元的作用[14]. 肿瘤细胞代谢糖酵解往往占优势, 以减少线粒体呼吸时产生的ROS, 进而避免ROS介导的细胞凋亡. 研究表明UCP2在人类结肠腺癌中普遍升高[15], 并与恶性程度密切相关. UCP2在Barrett食管和食管腺癌组织中的进行性升高, 可能是迅速生长肿瘤逃避ROS介导的细胞凋亡的一种机制.
诱导调亡的DFFA类似因子A(cell death-inducing DFFA-like effector A, CIDEA)有诱导凋亡的作用, 但能被DFF45抑制[16]. CIDEA的C端部分是诱导凋亡的活性部分. CIDEA的N端与DFF45同源, 是DFF45发挥抑制作用的部位. 维甲酸类似物有明显的促进生长和分化的作用, 并已被成功地应用于治疗急性早幼粒白血病及其他恶性肿瘤[17]. 细胞维甲酸结合蛋白(cellular retinoic acid binding protein 2, CRABP2)被认为具有介导维甲酸入核与核内受体结合的作用. Barrett食管和食管腺癌中CIDEA和CRABP2的表达水平降低可能减弱细胞凋亡和维甲酸抑制肿瘤发生的作用.
总之, 我们通过用新的方法重新分析已公开的基因芯片表达谱发现了一些未能用原有方法检出的基因表达水平变化, 这些基因与细胞凋亡、氧化代谢、信号转导有关. 这些发现再次证明了联合应用不同统计方法对表达谱进行分析的重要意义.
在胃食管返流患者中约10%-20%合并BE. 相对正常人群而言, BE转变为食管腺癌的机会高出30-125倍.
高通量分子丰度检测技术除基因芯片外, 还有基因表达系列分析(serial analysisof gene expression, SAGE)、质谱分析蛋白质组学(mass spectro-metry proteomic technology).
通过重新分析业已公开的基因芯片表达谱发现了一些新的基因表达水平变化, 为研究肿瘤表达谱提供了新的手段.
筛查出的与食管腺癌发病相关的基因表达变化, 经验证后可能作为肿瘤标志物, 以取代代价高而效率差的食管镜筛查, 并可作为治疗性分子靶点.
基因表达谱: 是通过检测细胞内各种mRNA含量来了解各种基因在细胞内的表达情况. 基因表达谱的变化是疾病以及易感体质的重要标志. 通过比较不同环境下的细胞的基因表达谱, 可以发现某种或某些基因在这些过程中起到了关键性的作用;
Affymetrix基因芯片: 是美国Affymetrix公司采用独有的原位光刻专利技术生产的一种寡核苷酸芯片, 具有极高的特异性和灵敏度, 重复性好, 是国际上最常用的商用基因芯片.
本研究使用Dchip软件对已经在GEO数据库中公开的BE和食管腺癌Affymetrix芯片表达谱数据进行分析, 有一定创新性. 用新的分析方法研究已公开的表达谱芯片资料为研究肿瘤的发病机制提供了新的手段.
编辑: 张海宁 电编:张敏
| 1. | Cameron AJ. Epidemiology of columnar-lined esophagus and adenocarcinoma. Gastroenterol Clin North Am. 1997;26:487-494. [PubMed] |
| 2. | 吴 顺华, 郑 玉建, 成 军, 刘 妍, 张 跃新, 刘 开泰, 钟 彦伟, 吴 煜. 低剂量三氧化二砷对HepG2细胞基因表达谱调节的影响. 世界华人消化杂志. 2005;13:100-104. [DOI] |
| 3. | Sotiriou C, Wirapati P, Loi S, Harris A, Fox S, Smeds J, Nordgren H, Farmer P, Praz V, Haibe-Kains B. Gene expression profiling in breast cancer: understanding the molecular basis of histologic grade to improve prognosis. J Natl Cancer Inst. 2006;98:262-272. [PubMed] |
| 4. | Ein-Dor L, Kela I, Getz G, Givol D, Domany E. Outcome signature genes in breast cancer: is there a unique set? Bioinformatics. 2005;21:171-178. [PubMed] |
| 5. | Kimchi ET, Posner MC, Park JO, Darga TE, Kocher-ginsky M, Karrison T, Hart J, Smith KD, Mezhir JJ, Weichselbaum RR. Progression of Barrett's metaplasia to adenocarcinoma is associated with the suppression of the transcriptional progra-ms of epidermal differentiation. Cancer Res. 2005;65:3146-3154. [PubMed] |
| 6. | Li C, Wong WH. Model-based analysis of oligonu-cleotide arrays: expression index computation and outlier detection. Proc Natl Acad Sci USA. 2001;98:31-36. [PubMed] |
| 7. | Han ES, Wu Y, McCarter R, Nelson JF, Richardson A, Hilsenbeck SG. Reproducibility, sources of variability, pooling, and sample size: important considerations for the design of high-density oligo-nucleotide array experiments. J Gerontol A Biol Sci Med Sci. 2004;59:306-315. [PubMed] |
| 8. | Qin LX, Beyer RP, Hudson FN, Linford NJ, Morris DE, Kerr KF. Evaluation of methods for oligonucl-eotide array data via quantitative real-time PCR. BMC Bioinformatics. 2006;7:23. [PubMed] |
| 9. | Brazma A, Hingamp P, Quackenbush J, Sherlock G, Spellman P, Stoeckert C, Aach J, Ansorge W, Ball CA, Causton HC. Minimum information about a microarray experi-ment (MIAME)-toward standards for microarray data. Nat Genet. 2001;29:365-371. [PubMed] |
| 10. | Alizadeh AA, Eisen MB, Davis RE, Ma C, Lossos IS, Rosenwald A, Boldrick JC, Sabet H, Tran T, Yu X. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 2000;403:503-511. [PubMed] |
| 11. | Cillo C, Cantile M, Faiella A, Boncinelli E. Hom-eobox genes in normal and malignant cells. J Cell Physiol. 2001;188:161-169. [PubMed] |
| 12. | Lord RV, Brabender J, Wickramasinghe K, DeMeester SR, Holscher A, Schneider PM, Danen-berg PV, DeMeester TR. Increased CDX2 and decreased PITX1 homeobox gene expression in Barrett's esophagus and Barrett's-associated adeno-carcinoma. Surgery. 2005;138:924-931. [PubMed] |
| 13. | Kolfschoten IG, van Leeuwen B, Berns K, Mullen-ders J, Beijersbergen RL, Bernards R, Voorhoeve PM, Agami R. A genetic screen identifies PITX1 as a suppressor of RAS activity and tumorigenicity. Cell. 2005;121:849-858. [PubMed] |
| 14. | Mattiasson G, Shamloo M, Gido G, Mathi K, Toma-sevic G, Yi S, Warden CH, Castilho RF, Melcher T, Gonzalez-Zulueta M. Uncoupling protein-2 prevents neuronal death and diminishes brain dysfunction after stroke and brain trauma. Nat Med. 2003;9:1062-1068. [PubMed] |
| 15. | Horimoto M, Resnick MB, Konkin TA, Routhier J, Wands JR, Baffy G. Expression of uncoupling protein-2 in human colon cancer. Clin Cancer Res. 2004;10:6203-6207. [PubMed] |
| 16. | Inohara N, Koseki T, Chen S, Wu X, Nunez G. CIDE, a novel family of cell death activators with homology to the 45 kDa subunit of the DNA frag-mentation factor. EMBO J. 1998;17:2526-2533. [PubMed] |
| 17. | Hofmann WK, Ganser A, Seipelt G, Ottmann OG, Zander C, Geissler G, Hoffmann K, Hoffken K, Fischer JT, Isele G. Treatment of patients with low-risk myelodysplastic syndromes using a combination of all-trans retinoic acid, interferon alpha, and granulocyte colony-stimulating factor. Ann Hematol. 1999;78:125-130. [PubMed] |