修回日期: 2004-06-12
接受日期: 2004-06-17
在线出版日期: 2004-09-15
目的: 构建含荧光素酶(luciferase, luc)基因的短发夹状RNA(short hairpin RNA, shRNA)表达质粒, 并观察其在肝癌细胞Bel-7402中抑制外源荧光素酶的表达.
方法: 设计的两条反向重复的多聚核苷酸序列, 退火形成双链DNA, 再与双酶切后的载体PGE-1连接, 构建pshRNA-luc重组质粒, 与荧光素酶基因表达质粒pCMV-luc共转染肝癌细胞株BEL-7402, 检测其对荧光素酶表达的影响.
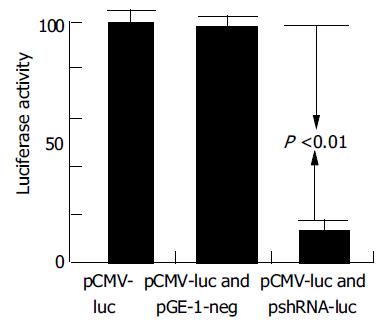
结果: PCR和DNA序列分析证实了重组质粒构建成功. pshRNA-luc对共转染的pCMV-luc中的荧光素酶具有明显的抑制作用, 抑制率可达86%(P <0.01).
结论: 构建的pshRNA-luc表达质粒能有效地抑制荧光素酶在肝癌细胞中的表达, 为RNA干扰用于肿瘤的基因治疗打下基础.
引文著录: 胡礼仪, 张有顺, 周新, 戴宗晴, 黄玲, 王菊. 短发夹RNA抑制外源荧光素酶在肝癌细胞中的表达. 世界华人消化杂志 2004; 12(9): 2045-2048
Revised: June 12, 2004
Accepted: June 17, 2004
Published online: September 15, 2004
AIM: To construct a recombinant plasmid containing short hairpin RNA of luciferase in order to suppress the expression of luciferase gene in hepatocellular carcinoma cells line Bel-7402.
METHODS: Two inverted repeating oligonucleotides, double-stranded DNA which dropped temperature were designed, and then cloned into PGE-1 vector digesting by the double restricted endoenzymes to generate the plasmids pshRNA-luc, pshRNA-luc and pCMV-luc, which were cotransfected into hepatocellular carcinoma cells line Bel-7402 to detect effect of luciferase expression.
RESULTS: The size of the PCR product was 652 bp. DNA sequencing showed the sequence of recombinant plasmids pshRNA-luc was successfully constructed. The recombinant plasmids suppressed the expression of luciferase gene by 86% in Bel-7402 cells (P <0.01).
CONCLUSION: Short hairpin RNA of luciferase can efficiently suppress its expression in hepatocellular carcinoma cells Bel-7402.
- Citation: Hu LY, Zhang YS, Zhou X, Dai ZQ, Huang L, Wang J. Inhibition of luciferase expression in hepatocellular carcinoma cells by short hairpin RNA. Shijie Huaren Xiaohua Zazhi 2004; 12(9): 2045-2048
- URL: https://www.wjgnet.com/1009-3079/full/v12/i9/2045.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i9.2045
RNA干扰(RNA interference, RNAi)是近年发展起来的一种研究基因功能的新方法, 他通过将双链RNA (double-stranded RNA, dsRNA) 导入细胞后, 在Dicer酶的作用下产生有活性的干扰性RNA(small interfering RNA, siRNA), 与该段RNA同源的mRNA产生特异性降解, 从而导致特异基因表达抑制的进化保守的转录后基因沉默现象[1-4]. 他具有高效性, 高特异性, 快速性等特点. 《Science》杂志将这一新发现评为2002年世界十大科学成就之首.RNA干扰在基因功能研究、抗肿瘤和抗病毒基因治疗等方面均得到了广泛应用[5-11]. 我们采取基因沉默策略, 利用带U6启动子的PGE-1载体, 构建在细胞内产生短发夹状RNA(short hairpin RNA, shRNA)的重组质粒, 并研究其在肝癌细胞中对外源性荧光素酶基因的抑制作用, 进而为应用RNA干扰技术来抑制外源基因的表达研究奠定基础.
SCS1大肠埃希氏菌菌株和表达质粒pGE-1、pCMV-luc购自 Stratagene 公司; TaqDNA聚合酶、T4DNA连接酶、AMV逆转录酶、质粒提取试剂、MgCl2、荧光素酶检测试剂盒均购自Promega公司; 限制性内切酶B am H I和X ba I购自MBI公司; Trizol试剂盒、 RPMI1640培养基、胎牛血清、低熔点琼脂糖购自Gibco公司; 转染试剂LyoVecTM为InvivoGen公司产品;DNA片段由上海生工生物工程有限公司合成. 人肝癌细胞株Bel-7402为华中科技大学同济医学院免疫教研室沈关心教授惠赠, 常规培养于含100 mL/L胎牛血清, 青、链霉素各100 mg/L于RPMI 1640培养基中, 培养条件为37 ℃、50 mL/L CO2. 待80%的细胞贴壁后备用.
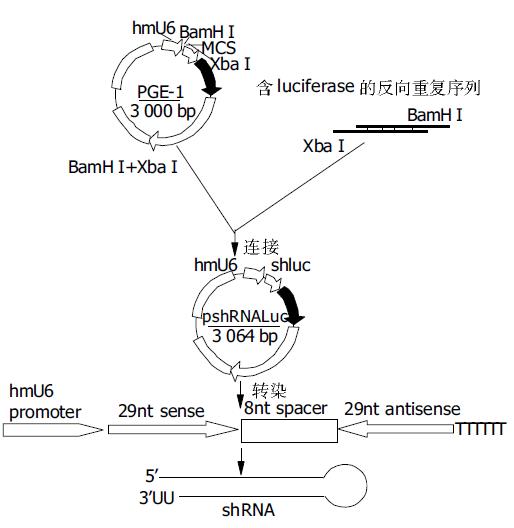
1.2.1 多聚核苷酸序列的设计及质粒的构建: 根据Luciferase mRNA选择目标序列, 按照shRNA的设计原则[12-13], 设计合成两条含Luciferase的反向重复序列, 并利用BLAST进行同源搜索, 确定其为特异性序列. 正义、反义序列分别为CGC GCT TGG TAG AGG TGG A和TCC ACC TCT ACC AAG CGC G. 正义链与反义链之间插入8 nt的回折序列GAAGCTTG; 在之后连接上6个"T", 此为RNA聚合酶Ⅲ的终止子, 另外在反向重复序列的两端分别保留Bam H I或X ba I的酶切位点. 将设计并合成好的多聚核苷酸, 各取1 mL, 另外加dH2O4 3 mL, 10×退火缓冲液5 mL, 93 ℃热冲击3 min, 然后在至少30 min内自然冷却至37 ℃, 即获得含Luciferase的退火双链DNA(double-stranded DNA, dsDNA)分子.然后将带U6启动子的PGE-1质粒进行扩增及提取后经电泳、紫外分光光度仪检测(A260/A280), 鉴定所提质粒的大小、纯度及含量.然后用限制性内切酶Bam H I和Xba I进行消化, 低熔点琼脂糖凝胶电泳法分离回收酶切片段, 再用苯酚、氯仿各抽提一次, 无水乙醇沉淀DNA. 酶切线性质粒和插入片段的连接条件: PGE-1线性载体1 mL, dsDNA 3 mL, rATP 1 mL, 10×Ligase buffer 1 mL, T4DNAligase 1 mL, ddH2O3 mL, 4 ℃过夜, 连接产物命名为pshRNA-luc.
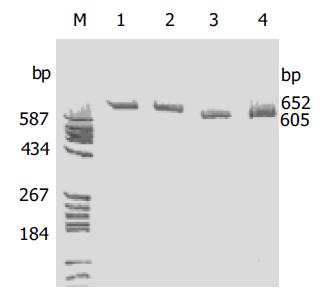
1.2.2 重组质粒鉴定: 取阳性质粒菌株和重组质粒菌株做PCR, 引物序列为上游5'- CGT CGA TTT TTG TGA TGC TCG TCAG-3', 下游3'-GAA GCA TTT ATC AGG GTT ATT GTC TCA TG-5', PGE-1扩增产物的分子大小为605 bp, pshRNA-luc为652 bp. PCR反应体系如下: 10×缓冲液4.0 mL, dNTP(25 mmol/L) 0.4 mL, 上、下游引物各取0.8 mL (10 mmol/L), Tween20(100 g/L)4 mL, DMSO 2.8 mL, TaqDNA酶2.0 U, 总体积40 mL, 挑取质粒菌株加入PCR反应管中, 其扩增条件为: 94 ℃变性3 min; 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸1 min, 30个循环后, 72 ℃延伸5 min. 取PCR产物和DNA Marker经50 g/L聚丙烯酰胺凝胶电泳, 银染观察并拍照.另外将PCR产物送上海博亚生物技术有限公司进行序列分析, 进一步证实DNA片段已克隆入载体中.
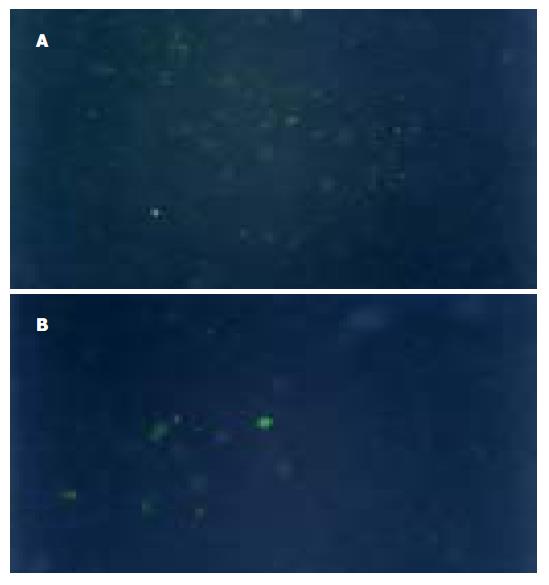
1.2.3 细胞转染及荧光素酶基因表达的检测: Bel-7402细胞1×105接种于6孔板, 37 ℃、50 mL/L CO2培养, 待80%细胞贴壁后, 换入无血清的RPMI 1640培养基800 mL, 同时分别加入AB混合液200 mL (A液:一定量的质粒稀释于100 mL无血清RPMI1640中; B液: 25 mL脂质体稀释于100 mL无血清RPMI 1640中, 混合AB液, 置室温孵育30 min), 培养5 h后加入小牛血清终止转染, 24 h后换完全培养基, 然后培养不同时间备用.实验设pCMV-luc单独转染组和pCMV-luc、pshRNA-luc共转染组以及pCMV-luc、pGE-1-neg共转染组, 实验重复3次. 质粒转染细胞培养24-72 h观察(仅48 h照相), 细胞单层直接在荧光显微镜下观察荧光素酶在细胞内表达的变化, 并拍照. 另外在转染细胞培养48 h后, 采用荧光素酶检测试剂盒, 按照其推荐的步骤进行操作, 用荧光素酶测试仪进行手动测值. 每个样本重复3次, 取平均值.
统计学处理 数据采用mean±SD表示, 用SPSS10.0统计软件进行t检验, 以P <0.05具有统计学差异.
pshRNA-luc重组质粒的构建过程以及其在细胞内从DNA模板产生shRNA的策略(见图1).
将阳性质粒菌株和重组质粒菌株分别做PCR, 取PCR产物0.5 mL用于50 g/L聚丙烯酰胺凝胶电泳, 银染结果显示, 在分子大小605 bp和652 bp处分别有一条清楚的带, 与预计的相符(图2). 另外DNA测序结果进一步证实了序列已克隆入载体中.
自1998年Fire et al[14]首次提出RNA干扰的概念以来, 他很快就成为分子生物学领域研究的主要技术手段之一, 在人类基因功能研究、信号转导及基因治疗方面已显现出了巨大的前景.目前利用RNAi 对乙型肝炎[15-16]、丙型肝炎[17]、白血病[18]、艾滋病[19]的研究已取得了一定进展, 成功地下调了相关基因的表达.研究表明, siRNA具有抑制作用强、稳定性高、细胞摄取相对容易等优点, 正逐渐取代反义核酸, 成为新的研究热点[20].诱导哺乳动物细胞RNAi的关键在于如何有效合成短干扰RNA, 目前siRNA来源有: (1)化学合成, (2)体外转录, (3)利用载体导入相应的发夹结构基因, 然后在启动子(主要为H1多聚酶Ⅲ和U6启动子)的作用下转录出shRNA, 进而直接在细胞内诱导RNAi. 虽然均有成功试验的报道[21-23], 但由于化学合成和体外转录费用较高, 转染效率低, 抑制作用短暂, 且需要多次重复导入等缺点, 因此限制了其应用和推广. 我们所构建的质粒产生shRNA的方法所产生的shRNA体内抑制目的基因的效果与合成siRNA相似, 但花费小、成本低, 可在细胞内稳定产生shRNA, 且在转染时还可克服合成siRNA可能造成的RNase污染[24].
由于荧光素酶作报告基因具有灵敏度高、易检测等特点, 所以他广泛应用于分子生物学研究中[25-26]. 我们构建了在细胞内产生荧光素酶shRNA的重组质粒, 并探讨其对所转染的报告基因荧光素酶表达水平的影响.结果表明: 转染pshRNA-luc后能有效并特异地抑制了外源荧光素酶在肝癌细胞中的表达, 其抑制率达80%以上(P <0.01), 与Ma et al[27]的研究结果相似. 我们的实验结果明确地显示, 在肝癌细胞中U6启动子转录短发状RNA介导的RNA干扰可以显著地降低相应序列基因的表达, 而且这种抑制是特异的, 说明我们的发夹样结构设计合理, 通过PGE-1载体构建对靶基因表达的shRNA重组质粒, 导入肿瘤细胞后, 在细胞内直接诱导RNAi的方法可行[28]. 这为在肝癌细胞系中利用RNAi来研究目的基因的功能奠定了基础, 也为利用导入含外源基因的短发夹RNA来进行肿瘤基因治疗提供理论依据. RNAi用于肿瘤的研究才刚刚开始, 但是已经显示出了巨大的潜力, 将成为肿瘤基因治疗的新希望[29-31].
| 3. | Agrawal N, Dasaradhi PV, Mohmmed A, Malhotra P, Bhatnagar RK, Mukherjee SK. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 2003;67:657-685. [PubMed] [DOI] |
| 5. | Wang QC, Nie QH, Feng ZH. RNA interference: antiviral weapon and beyond. World J Gastroenterol. 2003;9:1657-1661. [PubMed] [DOI] |
| 6. | Brummelkamp TR, Bernards R, Agami R. Stable suppression of tumorigenicity by virus-mediated RNA interference. Cancer Cell. 2002;2:243-247. [PubMed] [DOI] |
| 7. | Song E, Lee SK, Wang J, Ince N, Ouyang N, Min J, Chen J, Shankar P, Lieberman J. RNA interference targeting Fas protects mice from fulminant hepatitis. Nat Med. 2003;9:347-351. [PubMed] [DOI] |
| 12. | Tuschl T. Expanding small RNA interference. Nat Biotechnol. 2002;20:446-448. [PubMed] [DOI] |
| 13. | Elbashir SM, Harborth J, Weber K, Tuschl T. Analysis of gene function in somatic mammalian cells using small interfering RNAs. Methods. 2002;26:199-213. [PubMed] [DOI] |
| 14. | Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 1998;391:806-811. [PubMed] [DOI] |
| 15. | Tang N, Huang AL, Zhang BQ, Yan G, He TC. [Potent and specific inhibition of hepatitis B virus antigen expression by RNA interference]. Zhonghua Yixue Zazhi. 2003;83:1309-1312. [PubMed] |
| 16. | Shlomai A, Shaul Y. Inhibition of hepatitis B virus expression and replication by RNA interference. Hepatology. 2003;37:764-770. [PubMed] [DOI] |
| 17. | Krönke J, Kittler R, Buchholz F, Windisch MP, Pietschmann T, Bartenschlager R, Frese M. Alternative approaches for efficient inhibition of hepatitis C virus RNA replication by small interfering RNAs. J Virol. 2004;78:3436-3446. [PubMed] [DOI] |
| 18. | Scherr M, Battmer K, Winkler T, Heidenreich O, Ganser A, Eder M. Specific inhibition of bcr-abl gene expression by small interfering RNA. Blood. 2003;101:1566-1569. [PubMed] [DOI] |
| 19. | Jacque JM, Triques K, Stevenson M. Modulation of HIV-1 replication by RNA interference. Nature. 2002;418:435-438. [PubMed] [DOI] |
| 20. | Morrow T. Making sense of antisense and interference. Manag Care. 2003;12:62-63. [PubMed] |
| 21. | Donzé O, Picard D. RNA interference in mammalian cells using siRNAs synthesized with T7 RNA polymerase. Nucleic Acids Res. 2002;30:e46. [PubMed] [DOI] |
| 22. | Yu JY, DeRuiter SL, Turner DL. RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells. Proc Natl Acad Sci USA. 2002;99:6047-6052. [PubMed] [DOI] |
| 23. | Paddison PJ, Caudy AA, Bernstein E, Hannon GJ, Conklin DS. Short hairpin RNAs (shRNAs) induce sequence-specific silencing in mammalian cells. Genes Dev. 2002;16:948-958. [PubMed] [DOI] |
| 24. | Sui G, Soohoo C, Affar el B, Gay F, Shi Y, Forrester WC, Shi Y. A DNA vector-based RNAi technology to suppress gene expression in mammalian cells. Proc Natl Acad Sci USA. 2002;99:5515-5520. [PubMed] [DOI] |
| 25. | Liang XS, Lian JQ, Zhou YX, Nie QH, Hao CQ. Inhibitory effect of IRES specific inhibitor RNA on HCV IRES mediated protein translation. Shijie Huaren Xiaohua Zazhi. 2003;11:157-160. |
| 26. | Chen YC, Song C, Luo CQ. Short hairpin RNAs induced RNA interference in human cells. Ai Zheng. 2003;22:566-570. [PubMed] |
| 27. | Ma X, Lu X, Peng J, Tan S, Yuan Z, Yin F, Wang H, Wu X, Hou Y. [Inhibition of Luciferase expression in mammalian cells by AAV vector plasmid mediated Luciferase shRNA]. Zhonghua Shiyan He Linchuang Bingduxue Zazhi. 2002;16:253-255. [PubMed] |
| 28. | Miyagishi M, Taira K. Development and application of siRNA expression vector. Nucleic Acids Res Suppl. 2002;113-114. [PubMed] [DOI] |
| 29. | Cioca DP, Aoki Y, Kiyosawa K. RNA interference is a functional pathway with therapeutic potential in human myeloid leukemia cell lines. Cancer Gene Ther. 2003;10:125-133. [PubMed] [DOI] |
| 30. | Ichim TE, Li M, Qian H, Popov IA, Rycerz K, Zheng X, White D, Zhong R, Min WP. RNA interference: a potent tool for gene-specific therapeutics. Am J Transplant. 2004;4:1227-1236. [PubMed] [DOI] |
| 31. | Borkhardt A. Blocking oncogenes in malignant cells by RNA interference--new hope for a highly specific cancer treatment? Cancer Cell. 2002;2:167-168. [PubMed] [DOI] |