修回日期: 2004-03-25
接受日期: 2004-04-13
在线出版日期: 2004-07-15
目的: 应用cDNA微阵列从食管鳞癌配对组织中得到一批在食管鳞癌中差异表达的基因, 其中包括SLP-2. 他在食管鳞癌组织中的表达比其在配对的正常组织中高6倍以上. 验证SLP-2在食管鳞癌组织中高表达的结果, 构建组织表达谱, 同时对其进行生物信息学分析.
方法: 利用RT-PCR和Northern blot对SLP-2在食管鳞癌组织中高表达的结果进行验证, 构建组织表达谱. 利用CLUSTAL和SMART软件对SLP-2进行生物信息学分析.
结果: RT-PCR和Northern blot表明, SLP-2在食管鳞癌组织中高表达, 并在多种组织中表达.CLUSTAL软件分析表明, SLP-2蛋白是stomatin超家族的一员, 但缺少stomatin超家族所特有的N-端疏水性结构域. SMART 软件分析表明, SLP-2蛋白具有一个PHB结构域和一个螺旋丰富的区域.
结论: cDNA 微阵列是筛选差异表达基因的有力工具. SLP-2在食管鳞癌组织中高表达, 并在多种组织中都有表达. 生物信息学分析表明其缺少stomatin超家族所特有的N-端疏水性结构域, 具有一个 PHB结构域和一个螺旋丰富的区域, 为阐明SLP-2参与肿瘤发生发展的分子机制奠定了良好的基础.
引文著录: 张立勇, 王涛, 丁芳, 刘仲敏, 刘芝华, 李衍达. SLP-2基因在食管鳞癌中的差异表达及其生物信息学分析. 世界华人消化杂志 2004; 12(7): 1517-1521
Revised: March 25, 2004
Accepted: April 13, 2004
Published online: July 15, 2004
AIM: One of the differentially expressed genes, SLP-2 (stomatin-like protein 2, SLP-2), was obtained from esophageal squamous cell carcinoma (ESCC) patient-matched tissues using cDNA microarrays, which was overexpressed in ESCC tissues compared with their normal counterparts. This study was to confirm overexpression of SLP-2 in ESCC, to construct tissue expression pattern of SLP-2 in different embryonic tissues and to carry out bioinformatics analysis.
METHODS: Overexpression of SLP-2 in esophageal squamous cell carcinoma was confirmed by RT-PCR and Northern blot. Tissue expression pattern was constructed by RT-PCR. And the biological property was analyzed by bioinformatic softwares.
RESULTS: RT-PCR and Northern blot showed that SLP-2 was overexpressed in ESCC tissues and distributed in different embryonic tissues. Relatedness between members of the stomatin superfamily was compared using CLUSTAL procedure. Simple Modular Architecture Research Tool (SMART) software predicted that it possessed a PHB domain (prohibitin homologue) and a coiled coil.
CONCLUSION: cDNA microarray is a powerful tool for screening differentially expressed genes. SLP-2 is overex-pressed in ESCC tissues and expressed in different embryonic tissues. The biological property is analyzed by bioinformatic softwares. Our study lays a good foundation for elucidation the molecular mechanism of initiation and progression of ESCC.
- Citation: Zhang LY, Wang T, Ding F, Liu ZM, Liu ZH, Li YD. Differential expression and bioinformatics analysis of SLP-2 gene in esophageal squamous cell carcinoma. Shijie Huaren Xiaohua Zazhi 2004; 12(7): 1517-1521
- URL: https://www.wjgnet.com/1009-3079/full/v12/i7/1517.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i7.1517
深入研究食管癌发生发展的分子机制, 对于阐明肿瘤发生发展的机制、设计新的抗食管癌药物和有针对性地进行个体治疗至关重要.食管癌的发生发展是一个多因素、多基因参与的多步骤的复杂生物学过程[1-7]. 虽然在基因表达异常、分离表达失调蛋白和肿瘤排斥抗原等方面有所进展, 但对食管癌的发生发展机制的认识仍有待于进一步阐明[8-11]. 利用cDNA microarray技术发现了一些在食管癌中差异表达的基因[8,12-13]. 我们首次发现SLP-2(stomatin-like protein 2)在食管鳞癌中高表达. 目前尚无SLP-2与肿瘤相关研究的报道, 利用RT-PCR和Northern blot对SLP-2在食管鳞癌组织中高表达的结果进行验证, 构建组织表达谱. 利用CLUSTAL和SMART软件对SLP-2进行生物信息学分析, 为进一步阐明SLP-2参与肿瘤发生发展的分子机制打下了基础.
配对的食管鳞癌组织标本取自中国医学科学院协和医院和肿瘤医院, 由2位以上资深病理学专业人员确诊, 所有患者均未经放化疗. 胎儿组织取自中国医学科学院协和医院, 为6月龄女性. 所有组织经液氮速冻后, -80 ℃冰箱冻存备用. 从-80 ℃冰箱中取出食管癌组织及其配对的手术切除组织, 加入适量液氮后迅速研成粉末. 总RNA的提取按照TRIzol试剂盒(Gibco)说明书进行. 用DEPC处理的去离子水溶解RNA, 12 g/L甲醛变性琼脂糖凝胶电泳检查总RNA的完整性. 紫外分光光度计测定RNA的含量(A260值)和纯度(A260/A280比值)后, -80 ℃保存备用.
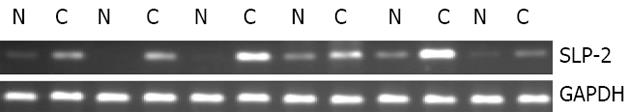
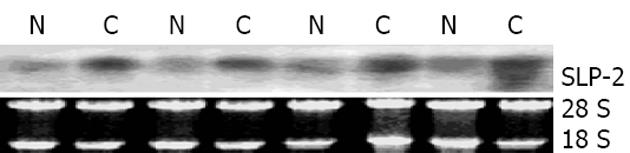
cDNA第一链的合成按照Transcriptase Super Script II TM Preamplification System for First Strand cDNA试剂盒(Gibco)说明书进行. PCR总反应体系为25 L, 其中含1-4 L模板, SLP-2上游引物序列为: 5'- GTGACTCTCGACAATGTAAC-3'; 下游引物序列为: 5'- TGATCTCATAACGGAGGCAG -3', 各0.4 moL/L, dATP, dCTP, dGTP, dTTP每种 0.2 mmoL/L, 1×PCR缓冲液, 1.5 mM MgCl2, 1.5 U Taq酶. PCR反应在PE9600型PCR仪上进行, 扩增条件为: 94 ℃预变性5 min; 94 ℃ 30 s, 57 ℃ 30 s, 72 ℃ 30 s, 27个循环, 72 ℃延伸5 min. 以GAPDH作为内对照. 扩增产物进行15 g/L琼脂糖凝胶电泳后置于Fluo-S MutiImager凝胶扫描仪中进行检测. Northern blot采用Primer-a-Gene (Promega)探针标记系统, 用-32P-dCTP标记的SLP-2片段作为探针, 按照说明书对食管鳞癌配对组织总RNA印迹进行杂交.
生物信息学分析从美国国家生物技术信息中心(National center for biotechnology information, NCBI)蛋白质数据库中调取stomatin超家族不同成员的氨基酸序列, 利用CLUSTAL软件对其进行同源性预测相关性分析. NCBI蛋白质数据库中调取SLP-2蛋白的氨基酸序列, 利用SMART软件对其进行结构域预测.
与相应配对正常食管上皮相比, SLP-2在食管鳞癌组织中表达上调(74%, 40/54, 图1). Northern blot结果表明, SLP-2在食管鳞癌组织中表达上调(77%, 10/13, 图2).
取新鲜胎儿的不同组织, 分别提取总RNA. 逆转录成cDNA后, 以SLP-2引物对cDNA进行半定量RT-PCR反应, 以GAPDH作为内对照. 结果表明, 在mRNA水平上, SLP-2在胎儿脑、肾脏、肺、胸腺、气管、食管黏膜、胃、肌肉、心脏、结肠、脾脏、小肠、胆囊等组织中广泛表达, 其中在脑和肾脏等组织中表达量很低(图3).
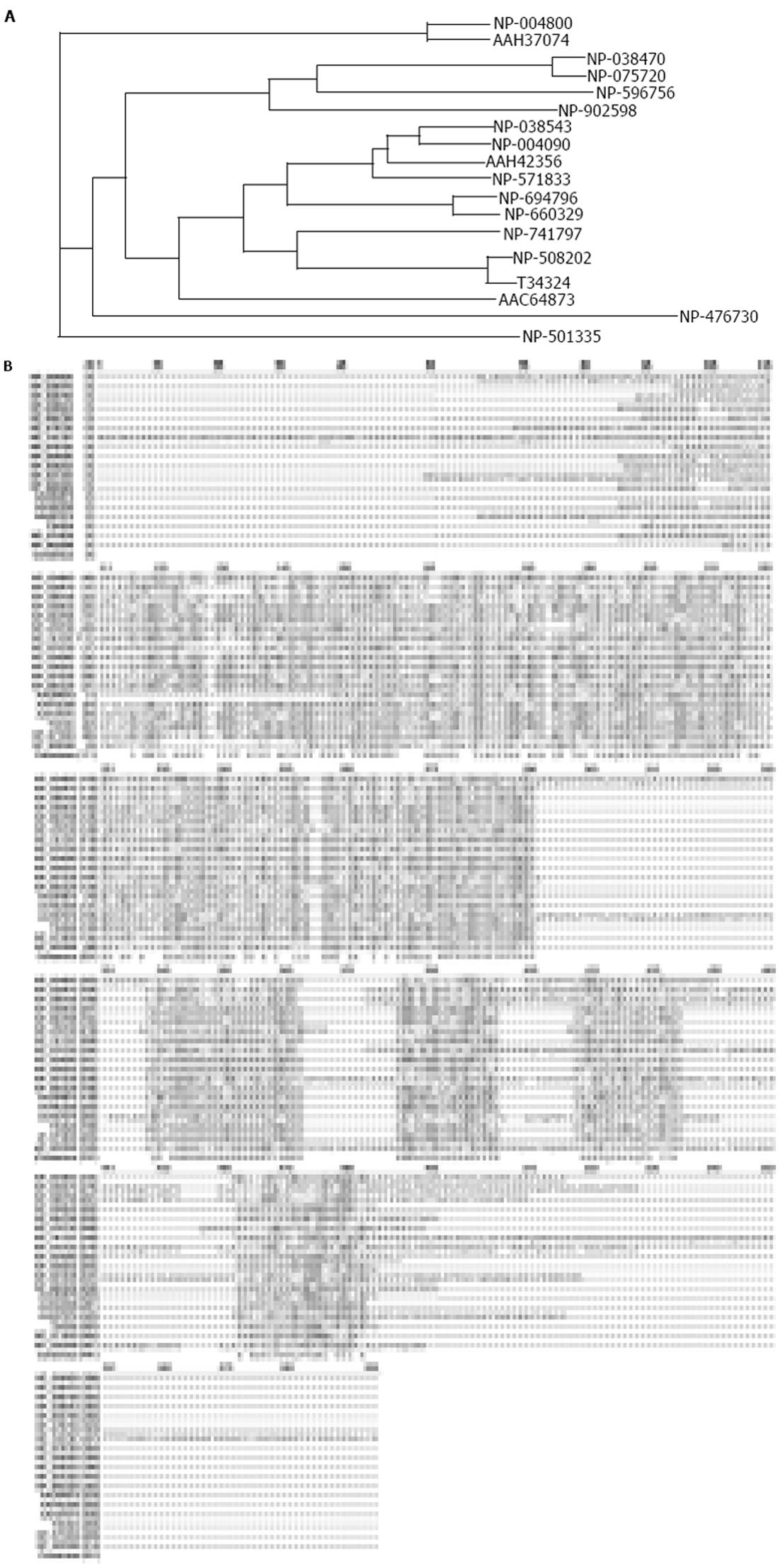
从NCBI蛋白质数据库中调取stomatin超家族不同成员的氨基酸序列(表1), 利用CLUSTAL软件对其进行相关性分析. 结果表明, 所有这些stomatin的同源蛋白和来自其他物种(如小鼠和斑马鱼)的stomatin在结构上都具有典型的N-端疏水性结构域和决定stomatin基因家族的一致氨基酸序列(如RX2(L/I/V)(S/A/N)X6(L/I/V)DX2TX2WG(L/I/V)(K/R/H)(L/I/V)X(K/R)(L/I/V)E(L/I/V)(K/R). SLP-2蛋白具有stomatin基因家族所共有的一致序列, 但不具有stomatin基因超家族所共有的N-端疏水性结构域(图4A, 4B).
| 蛋白质 | 登录号 | 物种来源 | 氨基酸数目 |
| stomatin | NP_004090 | Homo sapiens | 288 aa |
| stomatin | NP_038543 | Mus musculus | 284 aa |
| stomatin | NP_571833 | Danio rerio (zebrafish) | 284 aa |
| UNC-1 | NP_508202 | Caenorhabditis elegans | 285 aa |
| T34324 | Caenorhabditis elegans | 282 aa | |
| MEC-2 | NP_741797 | Caenorhabditis elegans | 481 aa |
| UNC-24 | NP_501335 | Caenorhabditis elegans | 415 aa |
| SLP-1 | NP_004800 | Homo sapiens | 398 aa |
| SLP-1 | AAH37074 | Mus musculus | 399 aa |
| SLP-1 | NP_476730 | Drosophila melanogaster | 322 aa |
| SLP-2 | NP_038470 | Homo sapiens | 356 aa |
| SLP-2 | NP_075720 | Mus musculus | 353 aa |
| SLP-3 | NP_660329 | Homo sapiens | 291 aa |
| SLP-3 | NP_694796 | Mus musculus | 287 aa |
| slp | AAC64873 | Rhizobium etli | 222 aa |
| AAH42356 | Xenopus laevis | 281 aa | |
| NP_596756 | Schizosaccharomyces pombe | 354 aa | |
| NP_902598 | Chromobacterium | 313 aa | |
| violaceum ATCC 12472 |
从NCBI蛋白质数据库中调取SLP-2蛋白的氨基酸序列, 利用SMART软件对其进行结构域预测. 结果表明, SLP-2蛋白具有一个PHB结构域(多肽链的52位氨基酸至211位氨基酸之间的肽段)和一个富含螺旋的区域(多肽链的231位氨基酸至252位氨基酸之间的肽段).
Stomatin基因定位于9q34.1, 编码Mr 31 000的整合膜蛋白[14-15]. Stomatin蛋白在结构上具有由23个氨基酸残基组成的特征性N-端疏水性结构域, 这个结构域附近的Cys29是主要的棕榈酰化位点[15-16]; 跨膜片段的远端氨基酸序列是亲水性的, 可能形成双向-折叠和-螺旋结构; C-端结构域朝向胞质[15]. Stomatin蛋白的Ser9可以被磷酸化, 含有这个磷酸化位点的N-端序列邻近N-端疏水性结构域, 并朝向胞质[15,17]. 在脊椎动物中发现的stomatin的同源物有SLP-1 (stomatin-like protein 1, SLP-1)、SLP-2和SLP-3(stomatin-like protein 3, SLP-3)[15,18]. SLP-1与UNC-24最为相似, 主要分布于脑, 而不出现于成熟红细胞中[15,19]. SLP-3是从嗅觉上皮中分离的嗅觉神经元蛋白[18]. 所有这些stomatin的同源蛋白和来自其他物种(如小鼠和斑马鱼)的stomatin在结构上都具有典型的N-端疏水性结构域和决定stomatin基因家族的一致氨基酸序列. 这表明, stomatin及其同源物可能作为离子通道和细胞骨架之间的连接分子, 从而影响通道的稳定性和质膜的组织[15].
SLP-2是从人心脏cDNA文库中得到的stomatin的同源物, 是stomatin基因超家族的一员[15,20]. SLP-2基因定位于9p13.1, 由10个外显子和9个内含子组成, 长约3 250 bp, 编码长约1.5 kb的mRNA, 指导有356个氨基酸组成的多肽链的合成[15,20]. SLP-2蛋白由3个主要的结构域组成: N-端-螺旋区、由-螺旋和-折叠交替组成的结构域和C-端结构域. SLP-2蛋白在人体组织中广泛分布.他具有stomatin基因家族所共有的一致序列, 但不具有stomatin基因超家族所共有的N-端疏水性结构域. 与人SLP-1不同, 他也出现在成熟红细胞膜中, 并且与血影蛋白-肌动蛋白细胞骨架相结合, 也可能与其他膜整合蛋白结合, 但其本身并不整合到膜双层中. SLP-2可能作为外周膜蛋白将stomatin或其他整合膜蛋白与细胞骨架联系起来, 从而调控离子通道的传导、鞘脂和富含胆固醇的脂质筏的组织[15,20].
SLP-2 mRNA编码区中有3个潜在的翻译起始位点, 可能负责组织特异性的翻译. COS 细胞的Western blot结果显示, SLP-2蛋白呈现3条带, Mr分别为45 500, 44 600和34 300[15,20], 表明SLP-2具有潜在的生物学功能.应用cDNA microarray技术比较分析了食管鳞癌/正常配对组织的基因表达谱, 首次发现SLP-2在食管鳞癌中表达上调. 目前尚无SLP-2参与肿瘤发生的相关报道.RT-PCR和Northern blot验证了在食管鳞癌组织中高表达, 并在多种组织中都有表达. SLP-2生物信息学分析表明, SLP-2蛋白具有一个PHB结构域, 这个结构域是prohibitin的同源物.这些都为阐明SLP-2与食管鳞癌发生发展的关系奠定了良好的基础.
编辑: N/A
| 2. | Zhang LY, Ying WT, Mao YS, He HZ, Liu Y, Wang HX, Liu F, Wang K, Zhang DC, Wang Y. Loss of clusterin both in serum and tissue correlates with the tumorigenesis of esophageal squamous cell carcinoma via proteomics approaches. World J Gastroenterol. 2003;9:650-654. [PubMed] [DOI] |
| 3. | Zhao XJ, Li H, Chen H, Liu YX, Zhang LH, Liu SX, Feng QL. Expression of e-cadherin and beta-catenin in human esophageal squamous cell carcinoma: relationships with prognosis. World J Gastroenterol. 2003;9:225-232. [PubMed] [DOI] |
| 4. | Wang HT, Kong JP, Ding F, Wang XQ, Wang MR, Liu LX, Wu M, Liu ZH. Analysis of gene expression profile induced by EMP-1 in esophageal cancer cells using cDNA Microarray. World J Gastroenterol. 2003;9:392-398. [PubMed] [DOI] |
| 5. | Zhang W, Bailey-Wilson JE, Li W, Wang X, Zhang C, Mao X, Liu Z, Zhou C, Wu M. Segregation analysis of esophageal cancer in a moderately high-incidence area of northern China. Am J Hum Genet. 2000;67:110-119. [PubMed] [DOI] |
| 6. | Wang DX, Li W. Advances on pathogenesis of esophageal cancer. Shijie Huaren Xiaohua Zazhi. 2000;8:1029-1030. |
| 7. | Hu SP, Yang HS, Shen ZY. Study on etiology of esophageal carcinoma: retrospect and prospect. Zhongguo Aizheng Zazhi. 2001;11:171-174. |
| 8. | Lu J, Liu Z, Xiong M, Wang Q, Wang X, Yang G, Zhao L, Qiu Z, Zhou C, Wu M. Gene expression profile changes in initiation and progression of squamous cell carcinoma of esophagus. Int J Cancer. 2001;91:288-294. [PubMed] [DOI] |
| 9. | Paweletz CP, Ornstein DK, Roth MJ, Bichsel VE, Gillespie JW, Calvert VS, Vocke CD, Hewitt SM, Duray PH, Herring J. Loss of annexin 1 correlates with early onset of tumorigenesis in esophageal and prostate carcinoma. Cancer Res. 2000;60:6293-6297. [PubMed] |
| 10. | Zhou G, Li H, DeCamp D, Chen S, Shu H, Gong Y, Flaig M, Gillespie JW, Hu N, Taylor PR. 2D differential in-gel electrophoresis for the identification of esophageal scans cell cancer-specific protein markers. Mol Cell Proteomics. 2002;1:117-124. [PubMed] [DOI] |
| 11. | Cui YP, Wang JB, Zhang XY, Bi MX, Guo LP, Lu SH. Using yeast two-hybrid system to identify ECRG2 associated proteins and their possible interactions with ECRG2 gene. World J Gastroenterol. 2003;9:1892-1896. [PubMed] [DOI] |
| 12. | Zhou J, Zhao LQ, Xiong MM, Wang XQ, Yang GR, Qiu ZL, Wu M, Liu ZH. Gene expression profiles at different stages of human esophageal squamous cell carcinoma. World J Gastroenterol. 2003;9:9-15. [PubMed] [DOI] |
| 13. | Su H, Hu N, Shih J, Hu Y, Wang QH, Chuang EY, Roth MJ, Wang C, Goldstein AM, Ding T. Gene expression analysis of esophageal squamous cell carcinoma reveals consistent molecular profiles related to a family history of upper gastrointestinal cancer. Cancer Res. 2003;63:3872-3876. [PubMed] |
| 14. | Stewart GW. Stomatin. Int J Biochem Cell Biol. 1997;29:271-274. [PubMed] [DOI] |
| 15. | Wang Y, Morrow JS. Identification and characterization of human SLP-2, a novel homologue of stomatin (band 7.2b) present in erythrocytes and other tissues. J Biol Chem. 2000;275:8062-8071. [PubMed] [DOI] |
| 16. | Snyers L, Umlauf E, Prohaska R. Cysteine 29 is the major palmitoylation site on stomatin. FEBS Lett. 1999;449:101-104. [PubMed] [DOI] |
| 17. | Salzer U, Ahorn H, Prohaska R. Identification of the phosphorylation site on human erythrocyte band 7 integral membrane protein: implications for a monotopic protein structure. Biochim Biophys Acta. 1993;1151:149-152. [PubMed] [DOI] |
| 18. | Goldstein BJ, Kulaga HM, Reed RR. Cloning and characterization of SLP3: a novel member of the stomatin family expressed by olfactory receptor neurons. J Assoc Res Otolaryngol. 2003;4:74-82. [PubMed] [DOI] |
| 19. | Seidel G, Prohaska R. Molecular cloning of hSLP-1, a novel human brain-specific member of the band 7/MEC-2 family similar to Caenorhabditis elegans UNC-24. Gene. 1998;225:23-29. [PubMed] [DOI] |
| 20. | Owczarek CM, Treutlein HR, Portbury KJ, Gulluyan LM, Kola I, Hertzog PJ. A novel member of the STOMATIN/EPB72/mec-2 family, stomatin-like 2 (STOML2), is ubiquitously expressed and localizes to HSA chromosome 9p13.1. Cytogenet Cell Genet. 2001;92:196-203. [PubMed] [DOI] |