修回日期: 2003-12-01
接受日期: 2003-12-16
在线出版日期: 2004-04-15
目的: 应用抑制性消减杂交(SSH)技术构建丙型肝炎病毒(HCV)NS3蛋白反式激活相关基因差异表达的cDNA消减文库, 克隆HCV NS3蛋白反式激活相关基因.
方法: 以HCV NS3表达质粒pcDNA3.1(-)-NS3转染HepG2细胞, 以空载体pcDNA3.1(-)为对照; 制备转染后的细胞裂解液, 从中提取mRNA并合成cDNA, 经RsaI酶切后将实验组cDNA分成两组, 分别与两种不同的接头衔接, 再与对照组cDNA进行两次消减杂交及两次抑制性PCR, 将产物与T/A载体连接, 构建cDNA消减文库, 并转染大肠杆菌进行文库扩增, 随机挑选克隆PCR后进行测序及同源性分析.
结果: 成功构建人HCV NS3蛋白反式激活相关基因差异表达的cDNA消减文库. 文库扩增后得到61个白色克隆, 进行菌落PCR分析, 均得到100-1 000 bp插入片段. 挑取30个插入片段测序分析, 得到30个已知功能基因序列.
结论: 筛选到的cDNA全长序列, 包括一些与细胞生长调节、物质代谢、免疫及细胞凋亡密切相关的蛋白编码基因, 推测了NS3TP2可能存在的调控机制的线索.
引文著录: 党晓燕, 成军, 邓红, 王建军, 杨倩, 刘妍, 纪冬, 王春花. 应用抑制性消减杂交技术克隆HCV NS3蛋白反式激活基因2的上调基因. 世界华人消化杂志 2004; 12(4): 847-850
Revised: December 1, 2003
Accepted: December 16, 2003
Published online: April 15, 2004
AIM: To clone and identify human genes transactivated by NS3TP2 by constructing a cDNA subtractive library with suppression subtractive hybridization technique.
METHODS: Suppression subtractive hybridization (SSH) and bioinformatics were used for screening and cloning of the target genes transactivated by NS3TP2 protein. The mRNA was isolated from HepG2 cells transfected pcDNA3.1(-)-NS3TP2 and pcDNA3.1(-) empty vector, respectively, and SSH method was employed to analyze the differentially expressed DNA sequence between the two groups. After restriction enzyme Rsa I digestion, small sizes cDNAs were obtained. Then tester cDNA was divided into two groups and ligated to the specific adaptor 1 and adaptor 2, respectively. The tester cDNA was hybridized with driver cDNA twice and underwent two times of nested PCR and then was subcloned into T/A plasmid vectors to set up the subtractive library. Amplification of the library was carried out with E.coli strain JM109. The cDNA was sequenced and analyzed in GenBank with Blast search after PCR.
RESULTS: The subtractive library of genes transactivated by NS3TP2 was constructed successfully. The amplified library contained 61 positive clones. Colony PCR showed that these clones contained 200-1000 bp inserts. Sequence analysis was performed in 30 clones, and the full length sequences were obtained with bioinformatics method. Altogether 21 coding sequences were identified.
CONCLUSION: The obtained sequences may be target genes transactivated by NS3TP2, among which some genes coding proteins involve cell cycle regulation, metabolism, immunity and cell apoptosis. Advanced experiments need to be done to prove this finding.
- Citation: Dang XY, Cheng J, Deng H, Wang JJ, Yang Q, Liu Y, Ji D, Wang CH. Screening and cloning of the target genes transactivated by human gene 2 transactivated by hepatitis C virus NS3 protein using suppression subtractive hybridization. Shijie Huaren Xiaohua Zazhi 2004; 12(4): 847-850
- URL: https://www.wjgnet.com/1009-3079/full/v12/i4/847.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v12.i4.847
丙型肝炎病毒(HCV)非结构蛋白(non-structural region)NS3是HCV基因组编码蛋白中具有重要作用的蛋白之一, 他由631个氨基酸残基(aa)组成. NS3区编码的蛋白包括解旋酶、三磷酸核苷酶 (NTPase)和丝氨酸蛋白酶. 其中解旋酶与RNA的解旋复制有关, 丝氨酸蛋白酶具有催化HCV病毒多蛋白的活性, 其在裂解加工过程中具有重要的作用[1-8]. Errington et al[8]研究还发现NS3可通过与核内蛋白质间相互作用, 进一步在转录水平上调节基因表达, 在HCV持续感染及肝细胞损害中发挥重要作用[2]. 因此HCV NS3对肝细胞的生长、代谢、细胞信号转导途径甚至是恶性转化产生重要影响. 本室利用抑制性消减杂交技术(suppression subtractive hybridization, SSH), 对于表达HCV NS3载体转染的HepG2细胞进行研究, 并结合生物信息学技术(bioinformatics)克隆了NS3蛋白反式激活作用的新靶基因, 其中之一命名为HCV NS3蛋白反式激活基因2(NS3TP2). 并继续应用抑制性消减杂交技术筛选与克隆NS3TP2反式激活的基因, 推测其在体内可能存在功能的线索, 为研究HCV的致病机制提供新的方向.
HepG2 细胞、感受态大肠杆菌DH5(我室保存), pcDNA3.1(-)真核表达载体(Invitrogen); FuGENE6 转染试剂(Roche), mRNA Purification试剂盒(Amersham Pharmacia Biotech), PCR-Select cDNA Subtraction试剂盒, 50×PCR Enzyme Mix、Advantage PCR Cloning试剂盒(Clontech), High Pure PCR Product Purification试剂盒(Boehringer Mannheim), T7、SP6通用引物及pGEM-Teasy载体(Promega).
1.2.1 消减杂交文库的建立及克隆分析: 分别将pcDNA3.1(-)-NS3及pcDNA3.1(-)空载体转染HepG2细胞, 48 h后提取mRNA, 按PCR-Select cDNA Subtraction试剂盒扩增差异表达的CDNA. 对于所获基因片段序列分析表明, 其中之一为新型基因片段, 与GenBank中注册的已知功能基因序列没有同源性, 利用表达序列标签(EST)序列的搜索和比对, 进行电子拼接, 根据基因起始密码子的Kozak规则和终止密码子下游保守的多聚腺苷酸信号序列, 确定新型基因序列. 从HepG2细胞提取总RNA, 以逆转录多聚酶链反应(RT-PCR)技术扩增获得该新基因的全长序列, 并测序证实, 命名为HCV NS3反式激活基因2(NS3TP2), 在GenBank中注册, 注册号为AY116970. NS3TP2基因的编码序列全长为1299个核苷酸(nt), 编码产物由432个氨基酸残基组成.
1.2.2 真核表达载体及细胞转染: HCV NS3蛋白反式激活基因2(NS3TP2)真核表达质粒pcDNA3.1(-)-NS3TP2 由本室构建. 用FuGENE6 转染试剂将2 g pcDNA3.1 (-)-NS3TP2及pcDNA3.1(-)空载体分别转染35 mm平皿HepG2 细胞, 48 h后收获细胞.
1.2.3 细胞mRNA提取: 使用mRNA Purification试剂盒, 直接提取转染了NS3表达质粒及空载体的HepG2细胞mRNA, 经琼脂糖凝胶电泳及分光光度计进行定性定量分析.
1.2.4 双链cDNA(dscDNA)合成: 用Clontech公司的PCR-SelectTM cDNA Subtraction Kit中的试剂, 以获得的mRNA为模板逆转录合成cDNA.
1.2.5 消减杂交文库的建立: 采用Clontech公司的PCR-SelectTM cDNA Subtraction et al, 常规SSH方法按说明书进行[3]: 转染了NS3表达质粒及空载体的HepG2 细胞cDNA分别标记为Tester和Driver, 经RsaⅠ(一种识别4碱基序列的内切酶)消化, 产生相对较短的平端片段, 纯化酶切产物. 将Tester的cDNA分为两份, 分别连接试剂盒提供的特殊设计的寡核苷酸接头Adapter 1和Adapter 2, 然后与过量的Driver cDNA进行杂交; 合并两种杂交产物后再与Driver cDNA作第2次杂交; 然后将杂交产物做选择性PCR扩增, 使Tester cDNA中特异性表达或高表达的片段得到特异性扩增.
1.2.6 克隆鉴定分析: 扩增产物与pGEM-Teasy载体连接, 转化JM109感受态细菌, 在含氨苄青霉素的LB/X-gal/IPTG培养板上, 37 ℃培养18 h. 挑取白色菌落, 增菌, 以pGEM-Teasy载体多克隆位点两端T7/SP6引物进行菌落PCR扩增, 证明含有插入片段后(100-1 000 bp), 测序(上海申友公司). 应用生物信息学将测得序列GenBank数据库进行同源性分析.
NS3TP2真核表达质粒pcDNA3.1(-)-NS3TP2经测序及酶切鉴定均正确.
使用高质量的mRNA是保证cDNA高产量的前提. 紫外分光检测显示, 转染了NS3表达质粒及空载体的HepG2 细胞mRNA分别为3.00 g和5.92 g, A260/A 280 =1.87. 1.0%琼脂糖凝胶电泳见 mRNA为大于0.5 kb清晰慧尾片状条带, 证实mRNA质优量足.
dscDNA与接头连接效率的高低是决定抑制性消减杂交成败的最关键步骤. 将连接有adaptor l和adaptor 2的两组dscDNA分别用两个基因特异性引物(G3PDH 3'-和5'-)与一个基因特异性引物(G3PDH 3')和一个PCR引物1进行28个循环扩增, 产物用2.0%琼脂糖凝胶电泳鉴定. 结果显示两组dscDNA扩增产物浓度相当, 说明dscDNA已与接头高效率连接.
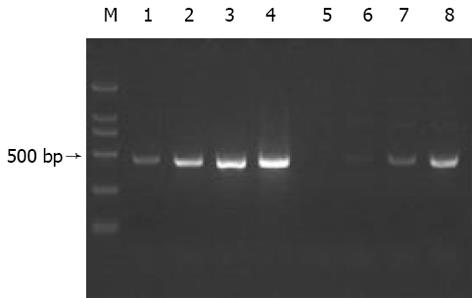
分别取l L抑制性及非抑制性PCR产物为模板, 用看家基因G3PDH引物进行PCR扩增, 分别在18、23、28、33次循环结束时从体系中吸取5 L进行电泳鉴定. 结果显示: 与非抑制性PCR产物相比, 抑制性PCR产物中G3PDH基因产物大大减少, 说明所构建的消减文库具有很高的消减效率(图1).
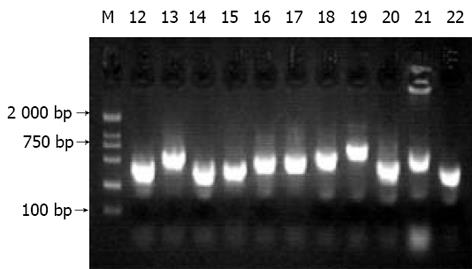
杂交产物经两轮PCR扩增后, 菌落PCR扩增结果显示为200-1 000 bp大小不等的插入片段, 所获得的61个克隆中几乎均含有插入片段, 这些条带可能代表差异表达的基因片段(图2).
挑选其中30个克隆测序, 与GenBank数据库进行初步比较. 有30个与已知基因的部分序列高度同源(93%-100%), 详细结果见表1.
| 已知的同源序列编码蛋白 | 相同克隆数 | 同源性(%) |
| 核糖体蛋白 | 10 | 99-100 |
| 锰超氧化物歧化酶 | 1 | 100 |
| 酪氨酸3单氧化酶激活蛋白 | 1 | 100 |
| 苯丙氨酸羟化酶 | 1 | 100 |
| 胰岛素样生长因子ⅡmRNA结合蛋白2 (IMP-2) | 1 | 93 |
| 肝细胞癌相关抗原64 | 1 | 99 |
| DEAD/H (Asp-Glu-Ala-Asp/His)框多肽15 (DDX15) | 1 | 100 |
| 肌动蛋白 | 1 | 100 |
| 载脂蛋白A-II | 1 | 100 |
| 纤连蛋白1(FN1) | 1 | 100 |
| 翻译控制的肿瘤蛋白1 | 1 | 99 |
| 白蛋白 | 1 | 100 |
| 溶酶体相关的跨膜蛋白4 | 1 | 100 |
| 真核翻译延长因子1 | 1 | 100 |
| 细胞色素C氧化酶亚单位IV同工型1 | 1 | 100 |
| 硒蛋白P | 1 | 100 |
| 电压依赖的阴离子通道1 | 1 | 96 |
| -2巨球蛋白 | 1 | 100 |
| 肿瘤排斥抗原1 | 1 | 97 |
| MLN51蛋白 | 1 | 100 |
| 酸性(亮氨酸富集)核磷蛋白32B | 1 | 100 |
| 克隆总计 | 30 |
众所周知, HCV NS3具有多种生物功能, 其中他的反式激活作用在HCV致病中发挥重要的作用. 为了进一步了解HCV NS3在丙型肝炎的发生及发展中的作用, 本中心首先利用抑制性消减杂交技术(suppression subtractive hybridization, SSH)结合生物信息学技术(bioinformatics)克隆了NS3蛋白反式激活作用的新靶基因, 其中之一命名为HCV NS3蛋白反式激活基因2(NS3TP2), 我们应用的抑制性消减杂交方法具有实验周期短、可靠性高、假阳性率低、易操作等优点, 能有效地分离扩增低丰度特异表达的基因, 可以在较短的时间内获得较理想的实验结果[3]. 我们将真核表达载体pcDNA3.1(-)-NS3TP2转染至肝母细胞瘤细胞系HepG2, 并以转染空白载体的相同细胞系作为对照, 以2种转染的细胞系中提取的mRNA为起始材料, 应用SSH方法成功地构建了NS3TP2反式激活相关基因差异表达的cDNA消减文库, 挑选30个克隆测序分析, 获得30个已知功能的基因序列.
在已知功能的基因序列中其中包括筛选到的纤连蛋白(FN), 他是一种多功能的广泛存在于胞质和细胞外基质中的糖蛋白. FN在促进细胞附着、迁移、分化、伤口愈合和肿瘤转移中发挥重要作用. 而且他是炎症修复时细胞外的主要成分. 慢性肝炎, 早期肝硬化的血浆FN往往升高, 反映纤维组织增生和结缔组织代谢增加. 纤维连接素的增加, 在肝损害时基质组成的改变中出现最早, 为I型胶原(CI)的沉积提供支架. 肝纤维化损伤时, 且FN最先增多, 通过纤维母细胞表面黏附受体将信息传入细胞内, 在肝纤维化中起"起搏点"样启动作用. FN还可增加丝分裂素激活蛋白激酶(MAPK)活动, 刺激激活蛋白-1(AP-1)活动, 即激活原癌基因c-fos和c-Jun的活动, 从而使 FN的表达与肝细胞癌(HCC)的分化、侵入和转移呈高度相关本实验中 HCV NS3上调FN的表达, 因此可以推测或许NS3与肝纤维化、肝癌的发生发展有关.
本实验的结果还表明HCV NS3可上调翻译控制的肿瘤蛋白(TCTP)的表达, TCTP也即人组胺释放因子(HRF), 其被在种系发生中高度保守的Tpt1基因所编码. TCTP在大量的真核生物中广泛表达[4-7], 他涉及细胞生长、急性变应反应且被被证实为抗凋亡蛋白[8]. 另有报道TPT1可上调肿瘤蛋白的表达[9-11], 因此, HCV NS3在HCV导致的高癌变率中或许发挥正向作用.
另外筛选到载脂蛋白AⅡ(apoAⅡ)是重要的脂类代谢物质, 已知HCV的病毒颗粒可被apoAⅡ包围, 这样HCV病毒颗粒即可通过肝细胞膜上的相应受体进入肝细胞中, 从而实现HCV的感染过程. 因此NS3或许有促进HCV感染的作用.
在已知功能的序列中, 还包括细胞内结构、细胞生长相关蛋白和参与细胞内代谢的蛋白基因, 如核糖体蛋白、真核翻译延长因子1、白蛋白、肌动蛋白、-2巨球蛋白、含硒蛋白P、锰超氧化物歧化酶、苯丙氨酸羟化酶及DEAD框多肽15等物质, 他们在细胞的生长、分化及各种物质代谢中发挥重要作用. 其中DEAD框蛋白质是具有保守性天冬-谷-丙-天冬氨酸基序的一类蛋白质, 他参与多种细胞生物学过程, 包括改变RNA的二级结构、调节翻译起始作用、细胞核和线粒体剪接、核糖体和剪接小体的装配等过程. 苯丙氨酸羟化酶可使苯丙氨酸转化为酪氨酸, 调控苯丙氨酸在体内的分解代谢.
通过应用抑制性消减杂交技术克隆HCV NS3蛋白反式激活基因2, 我们发现他与体内数种物质代谢、凋亡、信号转导等活动高度相关. 特别是他可上调与肝纤维化、癌变等高度相关因子, 为我们进一步阐明HCV感染及其引起的各种病变机制提供新的方向.
编辑: N/A
| 1. | 成 军, 杨 守纯. 现代肝炎病毒分子生物学. 北京: 人民军医出版社 1997; 37-42. |
| 2. | Errington W, Wardell AD, McDonald S, Goldin RD, McGarvey MJ. Subcellular localisation of NS3 in HCV-infected hepatocytes. J Med Virol. 1999;59:456-462. [PubMed] [DOI] |
| 3. | Kuang WW, Thompson DA, Hoch RV, Weigel RJ. Differential screening and suppression subtractive hybridization identified genes differentially expressed in an estrogen receptor-positive breast carcinoma cell line. Nucleic Acids Res. 1998;26:1116-1123. [PubMed] [DOI] |
| 4. | Yenofsky R, Cereghini S, Krowczynska A, Brawerman G. Regulation of mRNA utilization in mouse erythroleukemia cells induced to differentiate by exposure to dimethyl sulfoxide. Mol Cell Biol. 1983;3:1197-1203. [PubMed] [DOI] |
| 5. | Böhm H, Benndorf R, Gaestel M, Gross B, Nürnberg P, Kraft R, Otto A, Bielka H. The growth-related protein P23 of the Ehrlich ascites tumor: translational control, cloning and primary structure. Biochem Int. 1989;19:277-286. [PubMed] |
| 6. | MacDonald SM, Rafnar T, Langdon J, Lichtenstein LM. Molecular identification of an IgE-dependent histamine-releasing factor. Science. 1995;269:688-690. [PubMed] [DOI] |
| 7. | Thaw P, Baxter NJ, Hounslow AM, Price C, Waltho JP, Craven CJ. Structure of TCTP reveals unexpected relationship with guanine nucleotide-free chaperones. Nat Struct Biol. 2001;8:701-704. [PubMed] [DOI] |
| 8. | Li F, Zhang D, Fujise K. Characterization of fortilin, a novel antiapoptotic protein. J Biol Chem. 2001;276:47542-47549. [PubMed] [DOI] |
| 9. | Martínez-Chantar ML, García-Trevijano ER, Latasa MU, Martín-Duce A, Fortes P, Caballería J, Avila MA, Mato JM. Methionine adenosyltransferase II beta subunit gene expression provides a proliferative advantage in human hepatoma. Gastroenterology. 2003;124:940-948. [PubMed] [DOI] |
| 10. | LeGros HL Jr, Halim AB, Geller AM, Kotb M. Cloning, expression, and functional characterization of the beta regulatory subunit of human methionine adenosyltransferase (MAT II). J Biol Chem. 2000;275:2359-2366. [PubMed] [DOI] |
| 11. | Tuynder M, Susini L, Prieur S, Besse S, Fiucci G, Amson R, Telerman A. Biological models and genes of tumor reversion: cellular reprogramming through tpt1/TCTP and SIAH-1. Proc Natl Acad Sci U S A. 2002;99:14976-14981. [PubMed] [DOI] |