修回日期: 2002-08-20
接受日期: 2002-09-12
在线出版日期: 2003-07-15
通过TDI-FP法检测肝细胞癌组织中HBV DNA核心启动子区A1762 T和G1764 A双突变并分析二者之间的相关性, 同时建立一种新的突变检测方法.
以20例肝细胞癌组织为研究标本, 通过PCR扩增含有HBV DNA核心启动子区的DNA片段, 然后用TDI-FP法检测R110或TAMRA标记ddNTP的FP值, 鉴定nt1762和nt1764的基因型.
20例肝细胞癌组织中经HBV检测试剂盒鉴定后, 18例为HBV感染阳性, 阳性率为90%. 经TDI-FP检测, 其中有13例为T1762和A1764双突变型, 1例为T1762-A1764/A1762-G1764突变杂合型, 4例检测结果为阴性. 经测序, 阳性结果及突变杂合型与TDI-FP结果完全吻合, 阴性结果中2例在nt1762前有8个核苷酸缺失突变, 导致突变检测引物无法与模板结合, 故产生TDI-FP检测出现假阴性结果. 在肝细胞癌组织HBV DNA中, T1762-A1764突变阳性率为75%, 突变杂合型为5%, 野生型为10%.
在肝细胞癌组织中, HBV感染率非常高, 而且其核心启动子区出现nt1762和nt1764双突变率较高, 提示双突变与肝细胞癌关系密切; 同时本实验所建立的TDI-FP法操作快速简便, 可以进行高通量自动化操作, 具有进一步推广应用的前景.
引文著录: 吕贯廷, 卢冰, 白玉杰, 张剑, 阎小君. TDI-FP法分析肝细胞癌组织中HBV核心启动子双突变. 世界华人消化杂志 2003; 11(7): 916-919
Revised: August 20, 2002
Accepted: September 12, 2002
Published online: July 15, 2003
To detect the A1762 T and G1764 A paired mutations in the core promoter region of the HBV DNA in the hepatocellular carcinoma, to investigate the relationship between them, and to establish a new method for genotyping of mutation patterns.
The target DNA fragment of the HBV core promoter region which contains the paired mutations was amplified by polymerase chain reaction and the FP value of the R110- or TAMRA-labeled ddNTP was detected by TDI-FP assay to determine the nt1762 and nt1764 genotype.
Of 20 cases of hepatocellular carcinoma, 18 (90%) was positive for HBV infection. Through TDI-FP assay, there were 13 /20 for T1762 and A1764 paired mutations, 1/20 heterozygous for T1762-A1764/ A1762-G1764 4/20 negative for the TDI-FP assay. By sequencing, the positive and heterozygous mutations completely accorded with the TDI-FP assay. In the negative results, 2/4 had 8-nucleotide deletion mutation, thus the mutation detection primer could not complement with the template and gave rise to the "false" negative results. The rate of T1762-A1764 positive mutation rate of HBV DNA in heaptocellular carcinoma was 85%, heterozygous 5%, and wild-type 10%.
The HBV infection is very high in the heaptocellular carcinoma. The great frequency of the paired mutations in the core promoter indicates that the paired mutations may be significantly related to the pathogenesis of hepatocellular carcinoma. The established TDI-FP is suitable for high throughput and automated genotyping assays due to its rapid and simple manipulation.
- Citation: L GT, Lu B, Bai YJ, Zhang J, Yan XJ. Paired mutation in core promoter region of HBV in hepatocellular carcinoma by TDI-FP assay. Shijie Huaren Xiaohua Zazhi 2003; 11(7): 916-919
- URL: https://www.wjgnet.com/1009-3079/full/v11/i7/916.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v11.i7.916
乙肝病毒(hepatitis B virus, HBV)感染已经成为影响人们健康的主要威胁[1,2], 目前全世界大约有3亿5千万人为乙肝病毒携带者[1]. 我国在世界上属于HBV感染的高发区, HBsAg阳性携带者约占总人口的10%[3-5], HBV感染和许多肝脏疾病有关, 如急性肝炎、爆发性肝炎、无症状携带、慢性肝炎、肝硬化和肝细胞癌(hepatocellular carcinoma, HCC)等[1,6,7], 而且越来越多的证据表明, HBV还能够感染外周血单个核细胞, 与淋巴系统疾病有关, 诸如非霍奇金淋巴瘤[8]. 研究表明, 乙肝病毒感染是肝细胞癌的主要致病因素[9]. 在HBV感染高发区, 二者的关系尤为显著[10]. 乙肝病毒突变率很高, 大约每年在2 10个碱基中就有一个被置换[11]. 其中最重要、最常见的是在核心启动子(core promoter, CP)第二个AT丰富区的A1762 T和G1764 A突变, 二者经常同时出现, 与肝脏疾病关系如肝细胞癌较为密切[12-14]. 双突变可以减少HBeAg的分泌, 却又可以增加病毒前基因组的复制[15,16]. HBV DNA的双突变对其致癌机制的研究具有重要意义.
本研究采用TDI-FP(template-directed dye-terminator incorporation with fluorescence polarization, TDI-FP)[17]的方法来分析肝细胞癌组织中HBV的双突变基因型, 他简便易行, 可以实现高通量和自动化操作, 为进一步研究HBV DNA的突变及基因型与临床意义建立方法学基础.
由第四军医大学第一附属医院肝胆外科和第二附属医院传染科提供20例肝细胞癌组织(经第四军医大学病理科作病理切片鉴定), 其中男性9例, 女性11例. wallac VICTOR2TM 1 420 multilabel counter, 480 nm/30、535 nm/40、544nm/15、595AF60滤光片(PerkinElmer Life Sciences); MJ Research V2.0 384微孔板(MJ Research); Techgene PCR Amplifier (Techne); Taq DNA聚合酶(Takara); Wizard(r) Plus Minipreps DNA purification System, Wizard(r)PCR Preps DNA Purification System pGEM(r)T-easy Vector (Promega); AcycloPrimeTM-FP SNP Detection Kit(PerkinElmer Life Sciences); HBV DNA Detection Kit (Takara); 所用引物由上海生工公司合成, 其序列见(表1).
| 引物 | 序列(5'→3') | 位置(bp) | 长度 |
| P1 | 5'-GGTCAACGACCTGGATTGAG-3' | 1681-1700 | |
| P2 | 5'-GGCAGAGGTGAAAAAGTTGC-3' | 1818-1835 | 155 bp |
| P1762 | 5'-GAGTCGGGGGAGGAGCTGAGGTTAA-3'1 | 737-1761 | |
| P1764 | 5'-GTCGGGGGAGGAGCTGAGGTTAAAG-3'1 | 739-1763 |
1.2.1 肝细胞癌组织的处理及HBV鉴定 取5 g组织于1 mL生理盐水中, 反复冻融5次, 然后研磨成匀浆, 10000 rpm离心10 min, 取上清, 加EDTA(PH8.0)、蛋白酶K和SDS至终浓度分别为0.1 mmol/L、1%和150 μg/mL, 37 °C孵育20 h, 加入RNase至终浓度为20 μg/mL, 37 °C继续孵育1 h. 加等体积酚-氯仿-异戊醇(25:24:1), 混匀, 14000 rpm离心10 min; 重复抽提1次, 加入1/3体积的乙酸钠(3 mol/L)和2倍体积的冷无水乙醇, 14000 rpm离心5 min, 弃上清; 70%洗涤沉淀, 晾干后, 用100 μL TE溶解. 用HBV检测试剂盒对所提取的HBV DNA S基因进行巢式PCR (nested PCR)鉴定HBV.
1.2.2 目的片断的PCR扩增 取2 μL做PCR模板, 反应体系为: MgCl2、dNTPs (10 mM)和10×Buffer各2.5 μL, P1和P2各1 μL, Taq DNA聚合酶1 U, 加水补足25 μL. 反应条件为: 95 °C预变性3 min; 然后95 °C变性30 s, 55 °C退火30 s, 72 °C延伸30 min, 进行30个循环; 72 °C延伸5 min. 取5 μL PCR扩增产物在1.2%琼脂糖凝胶电泳, 溴化乙啶染色.
1.2.3 TDI-FP检测HBV A1762→T和G1764→A突变 MJ Research V2.0 384 微孔板每孔加入5 μL PCR扩增产物(50-500 fmol/5μL), 然后加入2 μL 1碢CR消化酶(核酸外切酶I和虾碱性磷酸酶), 37 °C反应1 h, 清除游离的PCR引物P1和P2及dNTPs; 80 °C水浴15 min, 失活PCR消化酶. 随后每孔加入13 μL AcycloPrime-FP混合物(AcycloPol聚合酶0.05 μL, 10×反应缓冲液2 μL, R110-ddATP和TAMRA-ddTTP混合液1 μL, P1762 0.5 μL, 加水补足13 μL). 在检测G1764A突变时, 操作步骤同前, 只是所加的荧光素标记的ddNTP及引物不同, 他们为R110-ddGTP、TAMRA-ddATP和P1764. 然后把微孔板置于热循环仪, 95 °C预变性2 min; 95 °C 15s, 63 °C 30 s, 共25个循环; 室温15 min. 微孔板在wallac 1 420 VICTOR2TM 1 420 multilabel counter上分别检测R110和TAMRA的FP值.
1.2.4 PCR扩增产物的克隆及测序 取3 μL胶回收的PCR扩增产物与pGEM(r)T-easy载体连接, 4 °C过夜; 按常规方法转化E. Coli DH5α感受态细菌, 加入20mL LB培养基37 °C快速振摇至A = 0.6. 提取质粒DNA, 用EcoRI酶切及PCR扩增鉴定. 另取20 μL送上海基康公司进行测序鉴定.
20例肝细胞癌患者中有18例为HBV感染阳性, 2例为阴性; HBV感染阳性率90%.
20例肝细胞癌组织扩增的含核心启动子区的PCR产物经TDI-FP检测后发现, 有13例为T1762突变型, 1例为T1762/A1762突变杂合型, 2例为A1762野生型, 4例检测结果为阴性(包括2例HBV感染阴性组织); 13例为A1764突变型, 1例为A1764/G1764突变杂合型, 2例G1764野生型, 4例检测结果为阴性(包括2例HBV感染阴性组织). 经分析, 有13例肝细胞癌组织HBV出现T1762和A1764双突变; 1例出现T1762-A1764/A1762-G1764杂合型; 2例肝细胞癌组织中无HBV检测, 故双突变检测为阴性; 还有2例HBV DNA感染阳性组织检测结果也为阴性, 原因未明.
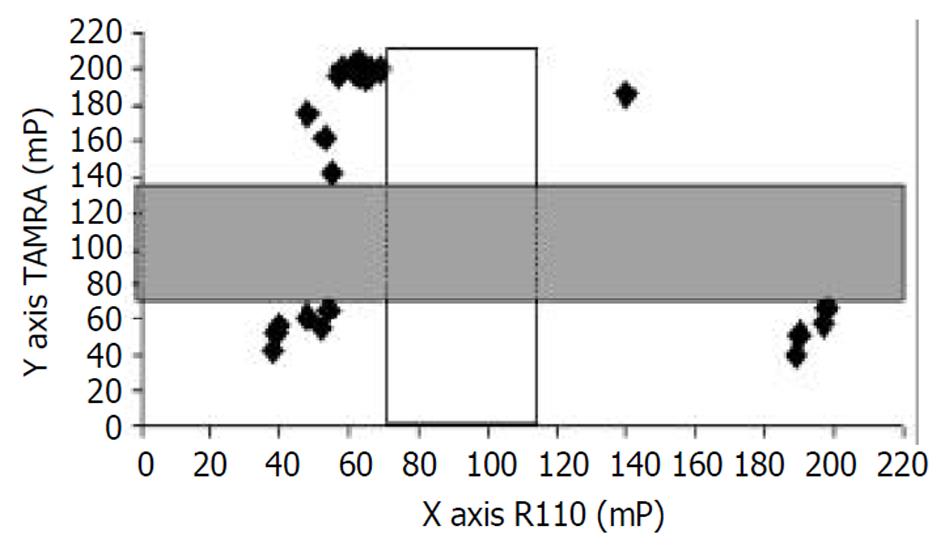
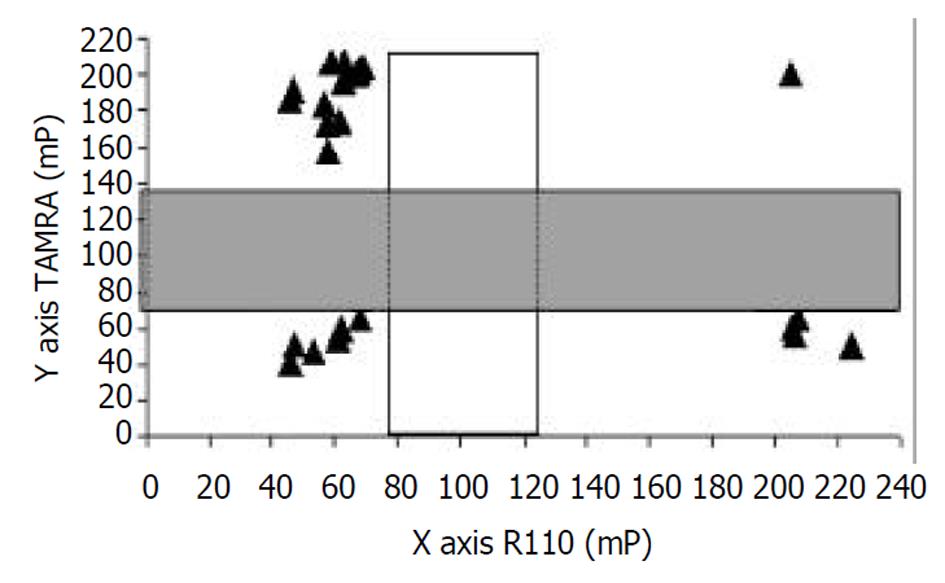
含核心启动子区的PCR产物与pGEM(r)T-easy载体重组克隆扩增后, 送上海基康公司测序, 测序结果显示, TDI-FP检测的13例T1762-A1764突变型及1例T1762-A1764/A1762-G1764杂合型与测序结果完全吻合. 2例TDI-FP检测为阴性的HBV DNA阳性组织在双突变位点前有8个核苷酸缺失突变, 突变检测引物无法与模板结合, 荧光素标记ddNTP无法加入而出现"假"阴性结果. 如果在突变位点设计正向和反向引物, 就可以提高检测的敏感性, 避免"假"阴性的产生. 20例肝细胞癌组织中, T1762-A1764突变型共15例, 其频率为75%; T1762-A1764/A1762-G1764杂合型为5%; 野生型为10%(图1、图2).
乙肝病毒是目前已知的可感染人体的最小的双链DNA病毒, 他以其前基因组RNA(pregenomic RNA)为模板进行逆转录反应而复制[18]. HBV因复制过程的特殊性, 他的自发突变率超过其他DNA病毒约4个数量级, 在过去的10多年中, 有关HBV突变临床意义的研究一直是热点之一[19]. 近10 a来, 一些国家男性人群中肝细胞癌的发生呈上升趋势, 在中国, 自1990年以来, 肝细胞癌已经成为第二位致死性癌症[20,21]. 肝细胞癌发生中, HBV的感染是一个非常重要的病因[22-24], 并在流行病学、免疫病理学和分子病理学等方面得到证实[23,25]. 但其致癌机制却不太清楚. 许多学者通过研究HBV DNA和宿主细胞染色体DNA的整合情况, 希望能找到有价值的线索, 目前他们已从人、土拨鼠、地松鼠和鸭的肝细胞癌组织染色体DNA中发现整合有HBV DNA, 但是这种整合是多发的和非特异的, 而且常常出现病毒和宿主染色体DNA的缺失和重排. 乙肝病毒核心启动子调控Pre-C RNA和C RNA的转录, 有研究表明, 慢性乙肝感染患者HBV DNA的核心启动子区常常出现A1762→T和G1764→A双突变[26,27]. 在HBV感染高发的国家里, 绝大多数的肝细胞癌发生于HBV携带者中, 并且在肝细胞癌HBV DNA中, 核心启动子发生双突变的频率非常高[28]. Edward Tabor et al发现江苏省启东县原发性肝细胞癌组织中85%有HBV T1762和A1764双突变序列, 同样的突变序列在慢性乙肝病毒感染者(非肝癌患者)中仅为11%. HBV DNA核心启动子区出现T1762和A1764双突变, 使核受体结合位点转换为肝细胞核转录因子-1(hepatocyte nuclear factor-1, HNF-1)的结合位点, 并且特异性地抑制了Pre-C RNA的转录. 双突变还使得与其重叠的X蛋白编码区的两个密码子发生改变. 有研究证明, 突变的X蛋白X(mt)和野生型X蛋白都有增强基因活化和加强HNF-1的DNA结合活性. 而少量的X(mt)是基因活化和HNF-1 DNA结合活性所需要的[26]. X蛋白可激活HNF-1, 使其从胞质转位到胞核, 与其结合位点结合[29]. 由于HBV复制增强, HBeAg表达降低, 宿主的免疫清除能力减弱, 导致宿主体内的HBV DNA有更多的机会整合到细胞染色体DNA上, 他们随染色体的复制而复制, 是细胞染色体DNA发生突变的机率增大, 当达到一定程度时, 就会导致肝细胞癌的发生.
本研究所采用的TDI-FP是TDI法(微测序(miniseq-uencing), 单核苷酸掺入法(single base incorporation), 引物延伸法(prime extension))的进一步发展和完善, 他以FP为检测手段的. 当荧光素分子被平面偏振光激发时, 他就在固定的平面发射出与其分子量成正比的偏振荧光(polarized fluorescence)[30,31], 这种现象称为荧光偏振(fluorescence polarization, FP). 如果荧光素分子量较小, 在溶液中自由旋转的速度较快, 他会向各个方向发出发射光, 荧光偏振不能保持. 可是当荧光素分子量较大, 旋转速度就会减慢, 于是在特定时间内向同一方向发出发射光, 荧光偏振就保持下来. 荧光偏振作为一种检测工具可以用来分析蛋白-蛋白、蛋白-多肽、蛋白质-DNA和其他种类的分子间相互作用[32]. 在单核苷酸多态性(single nucleotide polymorphism, SNP)分型方面, FP已经得到广泛应用[33], 如TDI-FP、FP-Taqman(r)和FP-Invader等. 其中FP-TDI 是最具前景的分析方法, 在DNA聚合酶催化下, 3'端紧临突变位点的引物加入一个与模板互补的荧光素标记的ddNTP, 使得荧光素的分子量增大许多倍, 通过检测荧光素分子的FP(单位为mP), 可以知道加入的ddNTP的种类, 根据互补原理推断出突变位点核苷酸的类型.
TDI-FP法的所有反应可以在同一个反应体系(如MJ Research V2.0 384微孔板)内分4步完成: (1) PCR扩增含有突变位点的目的DNA片断; (2)消化酶清除多余的引物和游离dNTPs, 避免其干扰随后的引物延伸反应; (3)加热失活消化酶; (4)加入DNA聚合酶、检测突变引物和荧光素标记的ddNTPs, 在热循环仪上进行引物延伸反应. 反应结束后, 把微孔板置于FP阅读仪上检测荧光素分子的FP[30]. TID-FP法具有以下优点: (1)仅需要热循环仪和可以做FP检测的仪器, 这些都非常便宜, 一般的实验室都可以承担; (2)不需要特别修饰的引物和试剂, 花费较低[34-36]; (3)该法非常容易优化; (4)FP-TDI分析以FP作为检测手段, 后者与荧光强度无关, 因此不需要把游离的荧光素标记双脱氧核苷酸从体系中分离[30].
由于TDI-FP法具有非常高的特异性和敏感性, 而且操作简便, 可以实现自动化和高通量检测, 在研究病原微生物基因突变、单核苷酸多态性、药物基因组学等方面具有广阔的应用前景.
| 1. | Kao JH, Wu NH, Chen PJ, Lai MY, Chen DS. Hepatitis B genotypes and the response to interferon therapy. J Hepatol. 2000;33:998-1002. [DOI] |
| 2. | Yan JC, Chen WB, Ma Y, Liu JH, Sun XH, Xu CJ. The study of the vascular diseases tissue and immunohistochemistry of HBV infected liver tissue. Shijie Huaren Xiaohua Zazhi. 2000;8:1205-1210. |
| 3. | Fang JN, Jin CJ, Cui LH, Quan ZY, Choi BY, Ki M, Park HB. A comparative study on serologic profiles of virus hepatitis B. World J Gastroenterol. 2001;7:107-110. [DOI] |
| 4. | Yan JC, Ma JY, Pan BR, Ma LS. The study of viral hepatitis B in China. Shijie Huaren Xiaohua Zazhi. 2001;9:611-616. |
| 5. | He XS, Huang JF, Chen GH, Fu Q, Zhu XF, Lu MQ, Wang GD, Guan XD. Orthotopic liver transplantation for fulminant hepatitis B. World J Gastroenterol. 2000;6:398-399. [DOI] |
| 6. | Wu J, Cheng ML, Ding YS, Liu RC, Li J, Wang WL, Hu L. Five year following-up surver of risk factor of virus hepatic cirrhosis. Shijie Huaren Xiaohua Zazhi. 2000;8:1365-1367. |
| 7. | Chen J. The molecular biochemistry study of pathogenesis of chronic viral hepatitis. Shijie Huaren Xiaohua Zazhi. 2002;10:125-128. |
| 8. | Li RL, Xu DZ, Nie QH, Yan YP, Zhang JX. Role of hepatitis virus infection in pathogenesis of B-cell non-Hodgkin's lymphoma. Shijie Huaren Xiaohua Zazhi. 2002;10:406-410. |
| 9. | Wang Y, Liu H, Zhou Q, Li X. Analysis of point mutation in site 1896 of HBV precore and its detection in the tissues and serum of HCC patients. World J Gastroenterol. 2000;6:395-397. [DOI] |
| 10. | Huang YX, Wu GH. The relationship between hepatitis B and hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi. 2001;9:682-685. |
| 11. | Kreutz C. Molecular, immunological and clinical properties of mutated hepatitis B viruses. J Cell Mol Med. 2002;6:113-143. [DOI] |
| 12. | Yap SF, Wong PW, Chen YC, Rosmawati M. The frequency of pre-core gene mutations in chronic hepatitis B infection: a study of Malaysian subjects. Southeast Asian J Trop Med Public Health. 2002;33:102-109. [PubMed] |
| 13. | Ogawa M, Hasegawa K, Naritomi T, Torii N, Hayashi N. Clinical features and viral sequences of various genotypes of hepatitis B virus compared among patients with acute hepatitis B. Hepatol Res. 2002;23:167-177. [DOI] |
| 14. | Hannoun C, Horal P, Lindh M. Long-term mutation rates in the hepatitis B virus genome. J Gen Virol. 2000;81:75-83. [PubMed] [DOI] |
| 15. | Ha-Lee YM, Lee J, Pyun H, Sohn J, Cho YJ, Kim Y. Sequence variations of hepatitis B virus promoter regions in persistently infected patients. Arch Virol. 2001;146:279-292. [DOI] |
| 16. | Grandjacques C, Pradat P, Stuyver L, Chevallier M, Chevallier P, Pichoud C, Maisonnas M, Trepo C, Zoulim F. Rapid detection of genotypes and mutations in the pre-core promoter and the pre-core region of hepatitis B virus genome: correlation with viral persistence and disease severity. J Hepatol. 2000;33:430-439. [DOI] |
| 17. | Akula N, Chen YS, Hennessy K, Schulze TG, Singh G, McMahon FJ. Utility and accuracy of template-directed dye-terminator incorporation with fluorescence-polarization detection for genotyping single nucleotide polymorphisms. Biotechniques. 2002;32:1072-1076. [PubMed] [DOI] |
| 18. | Bartholomeusz A, Locarnini S. Hepatitis B virus mutants and fulminant hepatitis B: fitness plus phenotype. Hepatology. 2001;34:432-435. [PubMed] [DOI] |
| 19. | Lan L, Wang YM. The influence of the genetic heterogeneity of HBV on the Interferon responsibility. Shijie Huaren Xiaohua Zazhi. 2002;10:696-698. |
| 20. | Tang ZY. Hepatocellular carcinoma-cause, treatment and metastasis. World J Gastroenterol. 2001;7:445-454. [DOI] |
| 21. | Shao XD, Wu KC, Guo XZ, Xu JH, Guo CC, Meng FP, Fan DM. Studies on expr ession of KAI1 gene in tissues of liver cirrhosis and hepatocellular carcinoma. Shijie Huaren Xiaohua Zazhi. 2001;9:1238-1241. |
| 22. | Chen WN, Oon CJ. Mutations and deletions in core promoter and precore stop codon in relation to viral replication and liver damage in Singaporean hepatitis B virus carriers. Eur J Clin Invest. 2000;30:787-792. [PubMed] [DOI] |
| 23. | Zhang WH, Zhu SN, Lu SL, Cong WM, Wu MC. HBV concomitant infection in HCV associated HCC. Shijie Huaren Xiaohua Zazhi. 2000;8:175-177. |
| 24. | Reid AE, Liang TJ. Association of hepatitis C virus and hepatocellular carcinoma in the United States. Princess Takamatsu Symp. 1995;25:41-49. [PubMed] |
| 25. | Lu YY, Li K, Cheng J, Wang L, Liu Y, Duan HJ, Zhang LX. Cloning and expression of hepatitis B virus X gene in yeast. Shijie Huaren Xiaohua Zazhi. 2002;10:15-18. |
| 26. | Li J, Xu Z, Zheng Y, Johnson DL, Ou JH. Regulation of hepatocyte nuclear factor 1 activity by wild-type and mutant hepatitis B virus X proteins. J Virol. 2002;76:5875-5881. [DOI] |
| 27. | Lin CL, Liao LY, Liu CJ, Chen PJ, Lai MY, Kao JH, Chen DS. Hepatitis B genotypes and precore/basal core promoter mutants in HBeAg-negative chronic hepatitis B. J Gastroenterol. 2002;37:283-287. [DOI] |
| 28. | Lin X, Qian GS, Lu PX, Wu L, Wen YM. Full-length genomic analysis of hepatitis B virus isolates in a patient progressing from hepatitis to hepatocellular carcinoma. J Med Virol. 2001;64:299-304. [DOI] |
| 29. | Guo SP, Wang WL, Zhai YQ, Zhao YL. Expression of nuclear factor-kappa B in hepatocellular carcinoma and its relation with the X protein of hepatitis B virus. World J Gastroenterol. 2001;7:340-344. [DOI] |
| 30. | Kwok PY. SNP genotyping with fluorescence polarization detection. Hum Mutat. 2002;19:315-323. [PubMed] [DOI] |
| 31. | Hsu TM, Chen X, Duan S, Miller RD, Kwok PY. Universal SNP genotyping assay with fluorescence polarization detection. Biotechniques. 2001;31:560-568. [PubMed] [DOI] |
| 32. | Tencza SB, Islam KR, Kalia V, Nasir MS, Jolley ME, Montelaro RC. Development of a fluorescence polarization-based diagnostic assay for equine infectious anemia virus. J Clin Microbiol. 2000;38:1854-1859. [PubMed] |
| 33. | Kricka LJ. Stains, labels and detection strategies for nucleic acids assays. Ann Clin Biochem. 2002;39:114-129. [PubMed] [DOI] |
| 34. | Kwok PY. Methods for genotyping single nucleotide polymorph- isms. Annu Rev Genomics Hum Genet. 2001;2:235-258. [PubMed] [DOI] |
| 35. | Nielsen K, Gall D. Fluorescence polarization assay for the diagnosis of brucellosis: a review. J Immunoassay Immunochem. 2001;22:183-201. [PubMed] [DOI] |
| 36. | Brennan MD. High throughput genotyping technologies for pharmacogenomics. Am J Pharmacog. 2001;1:295-302. [DOI] |