修回日期: 2020-05-09
接受日期: 2020-05-28
在线出版日期: 2020-06-08
甲基转移酶样蛋白(methyltransferase-like proteins, METTL)是一个大蛋白家族的一部分, 其特征是存在s-腺苷甲硫氨酸(s-adenosylmethionine, SAM)的结合域, SAM是甲基化反应的共同底物. 尽管这个蛋白家族的成员被显示或预测为RNA、DNA或蛋白质的甲基转移酶, 但大多数甲基转移酶的特征仍然很差, 识别这些潜在酶起作用的复合物有助于了解它们的功能和底物特异性. METTL蛋白家族与胃癌(gastric cancer, GC)的发生发展有密切作用, 明确其与GC的关系与GC的诊断治疗及预后有着十分重要的作用. 本研究是对METTL蛋白家族的作用机制及与GC的关系一次系统和全面的综述, 以期为进一步研究这些潜在的新型甲基转移酶和GC的诊治提供重要的资源.
核心提要: 甲基转移酶样蛋白(methyltransferase-like proteins, METTL)家族通过调节s-腺苷甲硫氨酸的浓度进行甲基化修饰, 家族成员可在哺乳动物中甲基化RNA、DNA或蛋白质, 其中主要进行RNA的甲基化修饰. 本文主要介绍METTL蛋白家族的分子作用机制以及家族成员在胃癌中的研究进展.
引文著录: 王婧, 王文安, 张安, 刘宏斌. 甲基转移酶样蛋白家族的分子作用机制及在胃癌中的研究进展. 世界华人消化杂志 2020; 28(11): 428-434
Revised: May 9, 2020
Accepted: May 28, 2020
Published online: June 8, 2020
Methyltransferase-like proteins (METTL) are part of a large protein family, which is characterized by the presence of an S-adenosylmethionine (SAM; a common substrate for methylation reactions) binding domain. Although members of this protein family have been shown or predicted as methyltransferases of RNA, DNA, or proteins, most methyltransferases are still poorly characterized. Identifying the complexes where these potential enzymes work can help to understand their function and substrate specificity. The METTL protein family is closely related to the occurrence and development of gastric cancer (GC), and its relationship with GC is of great importance in the diagnosis, treatment, and prognosis of GC. Here we give a systematic and comprehensive review of the mechanism of METTL protein family and its relationship with GC, with an aim to provide important resources for further research on these potential new methyltransferases and the diagnosis and treatment of GC.
- Citation: Wang J, Wang WA, Zhang A, Liu HB. Molecular mechanism of methyltransferase-like protein family: Relationship with gastric cancer. Shijie Huaren Xiaohua Zazhi 2020; 28(11): 428-434
- URL: https://www.wjgnet.com/1009-3079/full/v28/i11/428.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v28.i11.428
甲基化修饰广泛应用于调节RNA、DNA和蛋白质的活性, 在正常机体和疾病的多种生物学行为中起重要作用, 是近年来的研究前沿和热点. s-腺苷甲硫氨酸(s-adenosylmethionine, SAM)是几乎所有细胞甲基化反应的甲基供体, 因此需要细胞精确地维持SAM的水平, 但是目前对控制哺乳动物细胞内SAM丰度的机制却不甚了解. 细菌使用核糖开关直接将细胞内SAM水平与由蛋氨酸和ATP生成SAM的SAM合成酶的生产联系起来. 一些研究结果表明, 人SAM合成酶的表达也受转录后调节[1-4]. 核苷甲基转移酶(methyltransferase-like proteins, METTL)是蛋白质的一个多样化家族, 其特征是存在甲基转移酶样结构域和由中央的七链β-折叠结构形成的结构保守的SAM结合结构域. 到目前为止, DNA(胞嘧啶-5)甲基转移酶蛋白质家族已经得到了很好的研究, 并被鉴定为DNA (Dnmt1、3a和3b), 也被鉴定为RNA (Dnmt2)甲基转移酶[5,6]. METTL蛋白家族是通过调节SAM的浓度进而进行甲基化修饰. 通过使用无标记定量质谱系统研究了HeLa细胞中METTL蛋白家族成员的相互作用伴侣, 发现许多METTL蛋白在稳定的复合物之外发挥作用, 而包括METTL3、METTL14和METTL7B在内的其他蛋白家族成员则具有高置信度的相互作用伴侣[5,7-9]. 下文主要介绍METTL蛋白家族的分子作用机制以及其中的几个家族成员在胃癌(gastric cancer, GC)中的研究进展.
METTL蛋白家族的成员可在哺乳动物中甲基化RNA、DNA或蛋白质, 家族成员主要进行RNA的甲基化修饰.
METTL3和METTL14催化mRNA和主要miRNA中N6-甲基腺苷(N6-methyladenosine, m6A)的形成. 有趣的是, 要使METTL3具有活性, 必须与METTL14一起组成一个完整的体系[10]. 对于METTL7B, 研究鉴定了跨膜蛋白TMEM126A是高度信任的相互作用因子. TMEM126A本身是Myc原癌基因蛋白的靶标, 也是功能未知的线粒体膜蛋白, 该基因的缺陷可导致7型视神经萎缩. 这种与线粒体膜蛋白的相互作用可能暗示了METTL7B在线粒体RNA甲基化中的潜在功能. METTL8可以在体外和人类细胞中催化RNA中3-甲基胞苷的形成. 已经确定脯氨酰4-羟化酶亚单位α1是METTL8的交互因子, 可以推测METTL8将RNA修饰与转录调节结合在一起. 研究显示METTL1可以在与Trna[鸟嘌呤-N(7)-]-甲基转移酶非催化亚基WDR4形成复合体的情况下催化mRNA和tRNA中的7-甲基鸟苷形成[11-13]. 与此相反, 另一种新近鉴定出的m6A RNA甲基转移酶METTL16似乎不需要其他蛋白质来发挥其甲基转移酶活性. 通过对GFP-METTL16的纯化进行活性测试, 在体外RNA甲基转移酶测定中, 其纯化包含预期的总细胞RNA甲基转移酶活性, 这表明METTL16确实没有为保持活性所需的必需伴侣[14].
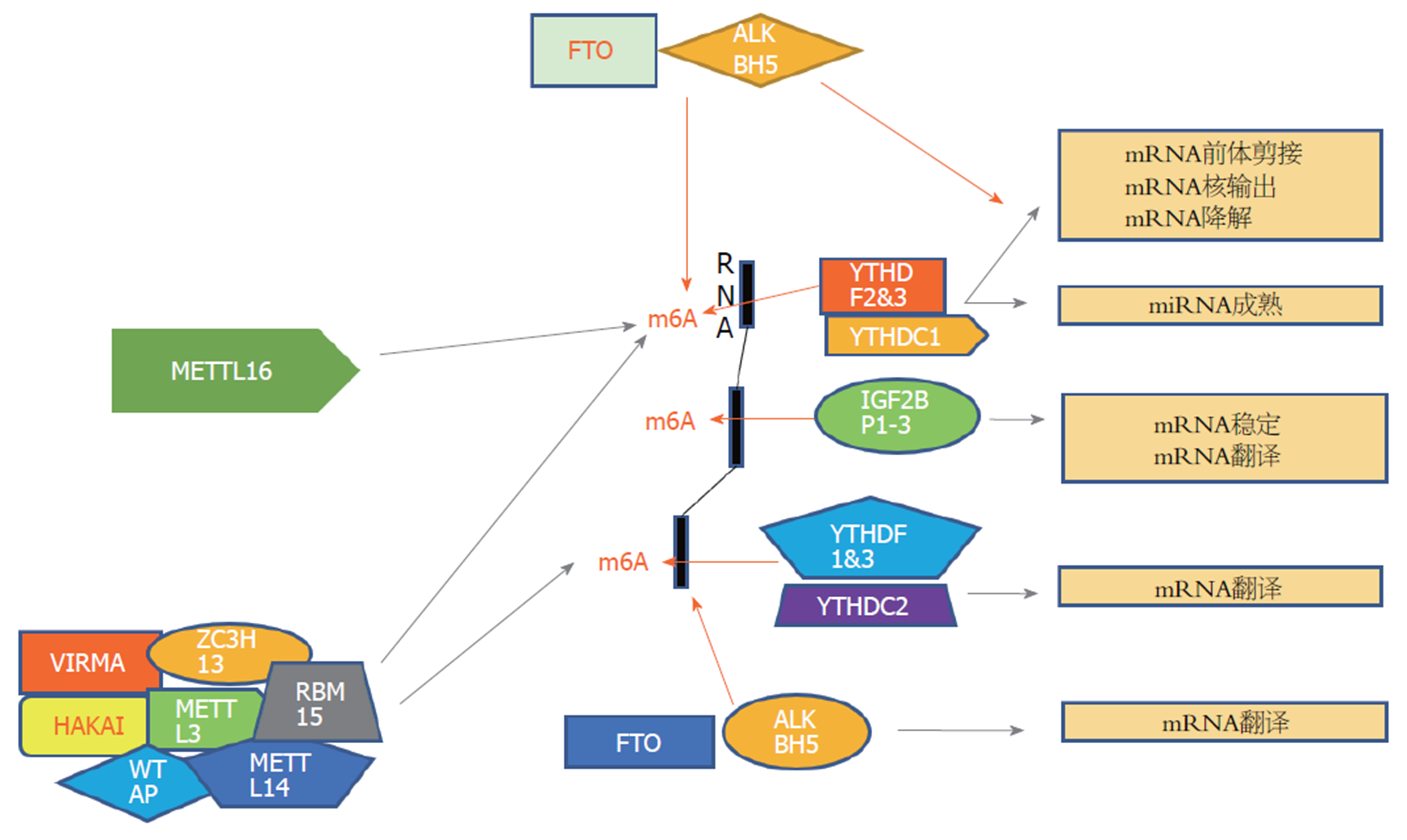
m6A被发现于20世纪70年代, 是腺嘌呤(A)第6位氮原子发生甲基化而形成的一种修饰, 是真核生物RNA最常见的甲基化修饰[18-20]. m6A是哺乳生物中mRNA和长链非编码RNA(long non-coding RNA, lncRNA)中最常见的内部修饰. M6A修饰受三种类型的酶调控: "写入程序"(甲基转移酶, 包括WTAP, RBM15和METTL3/14/16等), "阅读程序"(RNA结合蛋白和异质核糖核蛋白的YTH域, 包括YTHDF1/2/3等)和"橡皮擦"(脱甲基酶, 包括ALKBH5和FTO), 作用机制如图1所示. m6A甲基转移酶, 结合蛋白和脱甲基酶可以诱导癌基因转录表达, 癌细胞增殖, 分化阻滞, 侵袭, 转移, 肿瘤发生和对癌症治疗的抵抗力[21,22].
METTL3可与METTL14、WTAP形成一个稳定的异源二聚体, 对哺乳动物的mRNA进行m6A甲基化修饰, 这个复合体属于甲基转移酶类m6A调控蛋白[23-27]. METTL3和METTL14均有甲基转移酶活性, 但METTL14的催化活性要远高于METTL3. 尽管单独的METTL3或METTL14在体外表现出相对较弱的催化活性, METTL3-METTL4复合体则具有更高的催化能力. METTL3-METTL4复合体的结构中只有METTL3与SAM结合, 而METTL14起着底物识别的结构性作用, 因此, 异二聚体METTL3-METTL14复合体被认为是m6A甲基转移酶的催化结构域[28]. METTL3在催化中起到核心的作用, METTL14则起到支撑METTL3的结构的作用. 由METTL3-METTL4复合体介导的mRNA m6A修饰会导致mRNA衰减, 降低mRNA稳定性, 促进mRNA从细胞核输出到细胞质以及将5-UTR-甲基化的mRNA翻译成蛋白质. 敲低METTL3或METTL14会重新调节许多转录本的表达, 例如多能性因子Nanog, 并损害胚胎干细胞分化[29-33]. RNA甲基转移酶还通过调节miRNA表达来调节基因表达, METTL3甲基化pri-miRNA, 标记它们以进行识别和加工, 并以与细胞类型无关的方式增强整体miRNA的成熟度[34].
GC是消化道最多见的恶性肿瘤, 早期发现较为困难, 一般在偶然检查中发现, 往往已处于晚期, 给患者及家属带来巨大的精神和经济压力. GC的发病原因复杂, 它的发生、发展是一个由多种癌基因包括原癌和抑癌基因、促凋亡和抑凋亡基因共同参与的多阶段、多途径的过程. 从分子水平探讨GC发病机制是近几年来研究的热点之一, 基因水平的研究可以为GC的诊断、治疗和预后的判断开辟新的途径[35-38]. METTL3介导的m6A修饰存在于GC细胞中, 锌指MYM型含1 (zinc finger MYM-type containing 1, ZMYM1)是METTL3的真正m6A靶标. METTL3对ZMYM1 mRNA的m6A修饰依靠"阅读器"蛋白HuR依赖性途径增强了其稳定性. 此外, ZMYM1通过募集CtBP/LSD1/CoREST复合物结合并介导E-钙粘蛋白启动子的阻遏, 从而促进上皮-间质转化程序和转移. 揭示了METTL 3/ZMYM 1/E-钙粘蛋白信号作为抗GC转移策略中的潜在治疗靶点[39]. 通过shRNA转染敲低了人GC细胞系AGS和MKN45中的METTL3, RT-qPCR分析和蛋白质印迹分别验证了RNA干扰对mRNA和蛋白质水平的有效性, 发现METTL3可抑制AGS和MKN45细胞中的细胞增殖, 迁移和侵袭. 此外, METTL3减少GC细胞中的Bcl2和增加Bax和活性Caspase-3, 这表明细胞凋亡途径被激活. 机制研究表明METTL3导致人GC细胞中AKT信号通路失活, 包括AKT磷酸化水平降低以及下游效应子p70S6K和Cyclin D1的表达. 揭示了METTL3的下调会抑制人GC细胞的增殖和迁移, 并导致AKT信号通路失活, 这表明METTL3可能是治疗人GC的潜在靶标[40-46]. 研究发现, METTL14mRNA在GC组织中与癌旁组织相比, 表达水平显著降低. 通过RNAi干扰内源性METTL14表达后, 可显著促进GC细胞的增殖、侵袭和迁移. 结果说明METTL14可能在GC发生发展过程中起到抑癌基因作用, METTL14的抑癌作用也许能为GC临床分型诊断、治疗方案选择以及衡量愈合提供一个新的生物学标志[47,48].
METTL9是P53的一个下游基因, METTL9的染色体定位是在16p12-13, 由六个外显子和五个内含子组成, METTL9mRNA的开放阅读框在282-1130 bp处, 编码序列长度为849 bp. METTL9基因序列能与野生型P53蛋白结合, 与突变型P53蛋白几乎不结合. METTL9蛋白为亲水性蛋白, 存在一个跨膜区, 大约在42-79氨基酸片段, 没有信号肽[17,49]. METTL9是一种类甲基化转移酶, 属于DREVl转甲基酶基因家族, 与DREVl基因具有高相似性, 而且与野生的P53的内含子区域结合, 致使发生高甲基化, 导致肿瘤抑制因子等重要基因失活. 在该蛋白酶的作用下, 可能使DNA的SAM的甲基转移到CpG二核苷酸的胞嘧啶5位碳原子上, 生成5-甲基胞嘧啶, 最终导致DNA甲基化, 引起癌症的发生[6,16].
METTL9基因作为P53的一个下游基因在GC组织中高表达, 在正常组织中低表达并在GC组织中随组织分化程度减低表达成上升趋势[49]. 所以, 我们可以把METTL9当作肿瘤标记物对肿瘤风险进行评估, 进而得到预防策略, 对早期诊断及手术方式提供指导.
METTL16是鉴定出的第二种甲基腺苷(methyladenosine, mA)甲基转移酶, 其已知的底物包括U6小核核糖核酸(small nuclear RNA, snRNA)和编码转移相关肺腺癌转录物1 (metastasis-associated lung adenocarcinoma transcript 1, MALAT1)的人MAT2A mRNA[49-51]. METTL16在结构上与METTL3类似, 但具有一些独特的元素, 例如Rossmann折叠中的独特αB螺旋. 与METTL3-METTL14复合体相比, 全长METTL16形成同型二聚体, 一种天然的凝胶位移分析表明METTL16与MALAT1 RNA三螺旋结合. 此外, 尽管METTL16的结晶部分具有与全长多肽相同的活性, 但METTL3-METTL14复合体的催化结构域需要METTL3的N末端CCCH基序才能发挥全部活性, 可能需要额外的结构域才能将RNA底物正确定位在METTL3-METTL14复合体的催化腔内, 而METTL16的延伸环足以定向其RNA底物以进行mA修饰. 因此, 尽管METTL3和METTL16的催化活性可能具有相似的结构基础, 但由于延伸环的位置和其他RNA结合结构域的存在, 导致底物特异性差异很大[52-54]. METTL16诱导mRNA的3-UTR以及U6小核RNA的A43中的mA修饰, 该碱基在剪接过程中与前mRNA的5个剪接位点碱基配对, 这表明METTL16在mRNA稳定性和剪接中起重要作用. 有趣的是, 一项研究提出了METTL16在mRNA前剪接过程中发挥了额外的作用, 使METTL16可以作为无论是mA的"书写器"还是"阅读器". 作为mA的"书写器", METL16在SAM存在下迅速甲基化MAT2A mRNA, 导致内含子保留, 然后发生核降解[55,56]. 当SAM水平低时, MAT2A mRNA上METTL16的长期占据会增强保留内含子的剪接. 从大肠杆菌到人均发现METTL16同源物, 它们均具有N端甲基转移酶结构域. 作为SAM依赖性甲基转移酶, 预测METTL16具有高度保守的Rossmann折叠[57]. MALAT1核糖核酸三螺旋本身或邻近区域是METTL16的底物. MAT2A保留的内含子的剪接被快速诱导蛋氨酸耗竭后, 此效应需要一个保守的发夹(hp1), 我们进一步显示了它是METTL16 mA底物. 重要的是, 敲除METTL16可以消除蛋氨酸缺乏条件下对MAT2A剪接的诱导, 同时将METTL16强制结合到MAT2A 3UTR足以在Met充足的条件下促进剪接. 我们提出了一个模型, 其中MAT2A hp1上的METTL16占用促进了保留的内含子的剪接. 在高SAM含量下, METTL16迅速结合, 甲基化和解离, 有利于内含子保留. 在SAM限制条件下, METTL16不能有效地甲基化, 这会增加其在hp1上的停留时间并刺激保留的内含子的剪接. 我们得出的结论是METTL16是保守的U6 snRNA甲基转移酶, 它已经在脊椎动物中进化出了另一种功能, 通过转录后调节SAM合成酶基因的表达来控制SAM稳态[58-61].
虽然, 目前对于METTL16在GC中的研究还没有相关文献的报道, 但是METTL16相关的两个癌基因MAT2A和MALAT1已经被证实与GC的发生发展相关. 对癌组织及癌旁组织进行实时荧光定量 PCR与Western blot技术检测, 结果显示GC组织中MAT2A mRNA相对表达量明显高于癌旁正常胃组织, 表明MAT2A在GC中表达上调[59]. MALAT1基因在GC组织中呈高表达,下调MALAT1基因表达可有效抑制GC细胞增殖、迁移和侵袭力,为以MALAT1为靶点的GC基因靶向治疗奠定了基础[55].
目前, METTL蛋白家族引起了人们的极大兴趣, 因为该蛋白家族被认为涵盖了许多潜在的新型甲基转移酶. 但是, 对于许多METTL蛋白, 尚不清楚它们是否确实是活性酶, 以及它们的底物是什么: RNA, DNA或蛋白质. 最近发现, 在我们研究的酶中, 除METTL3-14复合体外, 还有METTL7B, METTL8和METTL16等都是RNA甲基转移酶. 在过去的几年中, 由METTL家族成员催化的新发现、新类型和新的RNA修饰位点的首次功能表征, 激发了对该蛋白家族的兴趣. METTL蛋白家族对GC的作用已经有一些研究进展, 但调控机制尚不明确, 对METTL蛋白家族的深入研究将对GC乃至所有肿瘤的诊疗有极其重要的意义.
学科分类: 胃肠病学和肝病学
手稿来源地: 甘肃省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): 0
D级 (一般): D
E级 (差): 0
科学编辑: 张晗 制作编辑:刘继红
| 1. | Murray B, Antonyuk SV, Marina A, Van Liempd SM, Lu SC, Mato JM, Hasnain SS, Rojas AL. Structure and function study of the complex that synthesizes S-adenosylmethionine. IUCrJ. 2014;1:240-249. [PubMed] [DOI] |
| 2. | Chik F, Machnes Z, Szyf M. Synergistic anti-breast cancer effect of a combined treatment with the methyl donor S-adenosyl methionine and the DNA methylation inhibitor 5-aza-2'-deoxycytidine. Carcinogenesis. 2014;35:138-144. [PubMed] [DOI] |
| 3. | Hashimoto Y, Zumwalt TJ, Goel A. DNA methylation patterns as noninvasive biomarkers and targets of epigenetic therapies in colorectal cancer. Epigenomics. 2016;8:685-703. [PubMed] [DOI] |
| 4. | Luo WP, Du YF, Huang J, Huang WQ, Xu M, Yan B, Mo XF, Zhang CX. [Effect of peripheral bloodgenomic DNA methylation on the relationship between methyl donor status and risk of breast cancer]. Zhonghua Liu Xing Bing Xue Za Zhi. 2017;38:537-541. [PubMed] [DOI] |
| 5. | Zhou KI, Pan T. Structures of the m(6)A Methyltransferase Complex: Two Subunits with Distinct but Coordinated Roles. Mol Cell. 2016;63:183-185. [PubMed] [DOI] |
| 6. | Okano M, Xie S, Li E. Cloning and characterization of a family of novel mammalian DNA (cytosine-5) methyltransferases. Nat Genet. 1998;19:219-220. [PubMed] [DOI] |
| 7. | Martin GH, Park CY. Meddling with METTLs in Normal and Leukemia Stem Cells. Cell Stem Cell. 2018;22:139-141. [PubMed] [DOI] |
| 8. | Dorsett M, Westlund B, Schedl T. METT-10, a putative methyltransferase, inhibits germ cell proliferative fate in Caenorhabditis elegans. Genetics. 2009;183:233-247. [PubMed] [DOI] |
| 9. | Dorsett M, Schedl T. A role for dynein in the inhibition of germ cell proliferative fate. Mol Cell Biol. 2009;29:6128-6139. [PubMed] [DOI] |
| 10. | Wang X, Feng J, Xue Y, Guan Z, Zhang D, Liu Z, Gong Z, Wang Q, Huang J, Tang C, Zou T, Yin P. Structural basis of N(6)-adenosine methylation by the METTL3-METTL14 complex. Nature. 2016;Jun 23; 534:575-578. [PubMed] [DOI] |
| 11. | Heyn H, Esteller M. An Adenine Code for DNA: A Second Life for N6-Methyladenine. Cell. 2015;161:710-713. [PubMed] [DOI] |
| 12. | van Nuland R, Smits AH, Pallaki P, Jansen PW, Vermeulen M, Timmers HT. Quantitative dissection and stoichiometry determination of the human SET1/MLL histone methyltransferase complexes. Mol Cell Biol. 2013;33:2067-2077. [PubMed] [DOI] |
| 13. | Ignatova VV, Jansen PWTC, Baltissen MP, Vermeulen M, Schneider R. The interactome of a family of potential methyltransferases in HeLa cells. Sci Rep. 2019;9:6584. [PubMed] [DOI] |
| 14. | Aoyama T, Yamashita S, Tomita K. Mechanistic insights into m6A modification of U6 snRNA by human METTL16. Nucleic Acids Res. 2020;48:5157-5168. [PubMed] [DOI] |
| 15. | Kweon SM, Chen Y, Moon E, Kvederaviciutė K, Klimasauskas S, Feldman DE. An Adversarial DNA N6-Methyladenine-Sensor Network Preserves Polycomb Silencing. Mol Cell. 2019;74:1138-1147.e6. [PubMed] [DOI] |
| 16. | Khalili M, Wong RJ. Underserved Does Not Mean Undeserved: Unfurling the HCV Care in the Safety Net. Dig Dis Sci. 2018;63:3250-3252. [PubMed] [DOI] |
| 18. | Guan K, Liu X, Li J, Ding Y, Li J, Cui G, Cui X, Sun R. Expression Status And Prognostic Value Of M6A-associated Genes in Gastric Cancer. J Cancer. 2020;11:3027-3040. [PubMed] [DOI] |
| 19. | Deng X, Su R, Weng H, Huang H, Li Z, Chen J. RNA N6-methyladenosine modification in cancers: current status and perspectives. Cell Res. 2018;28:507-517. [PubMed] [DOI] |
| 20. | Chen K, Wei Z, Zhang Q, Wu X, Rong R, Lu Z, Su J, de Magalhães JP, Rigden DJ, Meng J. WHISTLE: a high-accuracy map of the human N6-methyladenosine (m6A) epitranscriptome predicted using a machine learning approach. Nucleic Acids Res. 2019;47:e41. [PubMed] [DOI] |
| 21. | Lan Q, Liu PY, Haase J, Bell JL, Hüttelmaier S, Liu T. The Critical Role of RNA m6A Methylation in Cancer. Cancer Res. 2019;79:1285-1292. [PubMed] [DOI] |
| 22. | Zhang C, Fu J, Zhou Y. A Review in Research Progress Concerning m6A Methylation and Immunoregulation. Front Immunol. 2019;10:922. [PubMed] [DOI] |
| 23. | Liu N, Zhou KI, Parisien M, Dai Q, Diatchenko L, Pan T. N6-methyladenosine alters RNA structure to regulate binding of a low-complexity protein. Nucleic Acids Res. 2017;45:6051-6063. [PubMed] [DOI] |
| 24. | Lu N, Li X, Yu J, Li Y, Wang C, Zhang L, Wang T, Zhong X. Curcumin Attenuates Lipopolysaccharide-Induced Hepatic Lipid Metabolism Disorder by Modification of m6 A RNA Methylation in Piglets. Lipids. 2018;53:53-63. [PubMed] [DOI] |
| 25. | Zhou KI, Parisien M, Dai Q, Liu N, Diatchenko L, Sachleben JR, Pan T. N(6)-Methyladenosine Modification in a Long Noncoding RNA Hairpin Predisposes Its Conformation to Protein Binding. J Mol Biol. 2016;428:822-833. [PubMed] [DOI] |
| 26. | Yang Y, Hsu PJ, Chen YS, Yang YG. Dynamic transcriptomic m6A decoration: writers, erasers, readers and functions in RNA metabolism. Cell Res. 2018;28:616-624. [PubMed] [DOI] |
| 27. | Alarcón CR, Goodarzi H, Lee H, Liu X, Tavazoie S, Tavazoie SF. HNRNPA2B1 Is a Mediator of m(6)A-Dependent Nuclear RNA Processing Events. Cell. 2015;162:1299-1308. [PubMed] [DOI] |
| 28. | Cui Q, Shi H, Ye P, Li L, Qu Q, Sun G, Sun G, Lu Z, Huang Y, Yang CG, Riggs AD, He C, Shi Y. m6A RNA Methylation Regulates the Self-Renewal and Tumorigenesis of Glioblastoma Stem Cells. Cell Rep. 2017;18:2622-26. [PubMed] [DOI] |
| 29. | Wei W, Ji X, Guo X, Ji S. Regulatory Role of N6 -methyladenosine (m6 A) Methylation in RNA Processing and Human Diseases. J Cell Biochem. 2017;118:2534-2543. [PubMed] [DOI] |
| 30. | Dai D, Wang H, Zhu L, Jin H, Wang X. N6-methyladenosine links RNA metabolism to cancer progression. Cell Death Dis. 2018;9:124. [PubMed] [DOI] |
| 31. | Liu J, Yue Y, Han D, Wang X, Fu Y, Zhang L, Jia G, Yu M, Lu Z, Deng X, Dai Q, Chen W, He C. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. Nat Chem Biol. 2014;10:93-95. [PubMed] [DOI] |
| 32. | Mendel M, Chen KM, Homolka D, Gos P, Pandey RR, McCarthy AA, Pillai RS. Methylation of Structured RNA by the m6A Writer METTL16 Is Essential for Mouse Embryonic Development. Mol Cell. 2018;71:986-1000.e11. [PubMed] [DOI] |
| 33. | Sui X, Hu Y, Ren C, Cao Q, Zhou S, Cao Y, Li M, Shu W, Huo R. METTL3-mediated m6A is required for murine oocyte maturation and maternal-to-zygotic transition. Cell Cycle. 2020;19:391‐404. [PubMed] [DOI] |
| 34. | Bedi RK, Huang D, Eberle SA, Wiedmer L, Śledź P, Caflisch A. Small-Molecule Inhibitors of METTL3, the Major Human Epitranscriptomic Writer. Chem Med Chem. 2020;15:744-748. [PubMed] [DOI] |
| 35. | Wang P, Doxtader KA, Nam Y. Structural Basis for Cooperative Function of Mettl3 and Mettl14 Methyltransferases. Mol Cell. 2016;63:306-317. [PubMed] [DOI] |
| 36. | Huang J, Dong X, Gong Z, Qin LY, Yang S, Zhu YL, Wang X, Zhang D, Zou T, Yin P, Tang C. Solution structure of the RNA recognition domain of METTL3-METTL14 N6-methyladenosine methyltransferase. Protein Cell. 2019;10:272-284. [PubMed] [DOI] |
| 37. | Lin S, Choe J, Du P, Triboulet R, Gregory RI. The m(6)A Methyltransferase METTL3 Promotes Translation in Human Cancer Cells. Mol Cell. 2016;62:335-345. [PubMed] [DOI] |
| 38. | Du M, Zhang Y, Mao Y, Mou J, Zhao J, Xue Q, Wang D, Huang J, Gao S, Gao Y. MiR-33a suppresses proliferation of NSCLC cells via targeting METTL3 mRNA. Biochem Biophys Res Commun. 2017;482:582-589. [PubMed] [DOI] |
| 39. | Yue B, Song C, Yang L, Cui R, Cheng X, Zhang Z, Zhao G. METTL3-mediated N6-methyladenosine modification is critical for epithelial-mesenchymal transition and metastasis of gastric cancer. Mol Cancer. 2019;18:142. [PubMed] [DOI] |
| 40. | Taketo K, Konno M, Asai A, Koseki J, Toratani M, Satoh T, Doki Y, Mori M, Ishii H, Ogawa K. The epitranscriptome m6A writer METTL3 promotes chemo- and radioresistance in pancreatic cancer cells. Int J Oncol. 2018;52:621-629. [PubMed] [DOI] |
| 41. | Visvanathan A, Patil V, Arora A, Hegde AS, Arivazhagan A, Santosh V, Somasundaram K. Essential role of METTL3-mediated m6A modification in glioma stem-like cells maintenance and radioresistance. Oncogene. 2018;37:522-533. [PubMed] [DOI] |
| 42. | Cai X, Wang X, Cao C, Gao Y, Zhang S, Yang Z, Liu Y, Zhang X, Zhang W, Ye L. HBXIP-elevated methyltransferase METTL3 promotes the progression of breast cancer via inhibiting tumor suppressor let-7g. Cancer Lett. 2018;415:11-19. [PubMed] [DOI] |
| 43. | Barbieri I, Tzelepis K, Pandolfini L, Shi J, Millán-Zambrano G, Robson SC, Aspris D, Migliori V, Bannister AJ, Han N, De Braekeleer E, Ponstingl H, Hendrick A, Vakoc CR, Vassiliou GS, Kouzarides T. Promoter-bound METTL3 maintains myeloid leukaemia by m6A-dependent translation control. Nature. 2017;552:126-131. [PubMed] [DOI] |
| 44. | Li X, Tang J, Huang W, Wang F, Li P, Qin C, Qin Z, Zou Q, Wei J, Hua L, Yang H, Wang Z. The M6A methyltransferase METTL3: acting as a tumor suppressor in renal cell carcinoma. Oncotarget. 2017;8:96103-96116. [PubMed] [DOI] |
| 45. | Hu Y, Li Y, Huang Y, Jin Z, Wang C, Wang H, Xu J. METTL3 regulates the malignancy of cervical cancer via post-transcriptional regulation of RAB2B. Eur J Pharmacol. 2020;879:173134. [PubMed] [DOI] |
| 46. | Vu LP, Pickering BF, Cheng Y, Zaccara S, Nguyen D, Minuesa G, Chou T, Chow A, Saletore Y, MacKay M, Schulman J, Famulare C, Patel M, Klimek VM, Garrett-Bakelman FE, Melnick A, Carroll M, Mason CE, Jaffrey SR, Kharas MG. The N6-methyladenosine (m6A)-forming enzyme METTL3 controls myeloid differentiation of normal hematopoietic and leukemia cells. Nat Med. 2017;23:1369-1376. [PubMed] [DOI] |
| 47. | Ma JZ, Yang F, Zhou CC, Liu F, Yuan JH, Wang F, Wang TT, Xu QG, Zhou WP, Sun SH. METTL14 suppresses the metastatic potential of hepatocellular carcinoma by modulating N6 -methyladenosine-dependent primary MicroRNA processing. Hepatology. 2017;65:529-543. [PubMed] [DOI] |
| 48. | Weng H, Huang H, Wu H, Qin X, Zhao BS, Dong L, Shi H, Skibbe J, Shen C, Hu C, Sheng Y, Wang Y, Wunderlich M, Zhang B, Dore LC, Su R, Deng X, Ferchen K, Li C, Sun M, Lu Z, Jiang X, Marcucci G, Mulloy JC, Yang J, Qian Z, Wei M, He C, Chen J. METTL14 Inhibits Hematopoietic Stem/Progenitor Differentiation and Promotes Leukemogenesis via mRNA m6A Modification. Cell Stem Cell. 2018;22:191-205.e9. [PubMed] [DOI] |
| 49. | Hamey JJ, Wienert B, Quinlan KGR, Wilkins MR. METTL21B Is a Novel Human Lysine Methyltransferase of Translation Elongation Factor 1A: Discovery by CRISPR/Cas9 Knockout. Mol Cell Proteomics. 2017;16:2229-2242. [PubMed] [DOI] |
| 50. | Shima H, Matsumoto M, Ishigami Y, Ebina M, Muto A, Sato Y, Kumagai S, Ochiai K, Suzuki T, Igarashi K. S-Adenosylmethionine Synthesis Is Regulated by Selective N6-Adenosine Methylation and mRNA Degradation Involving METTL16 and YTHDC1. Cell Rep. 2017;21:3354-3363. [PubMed] [DOI] |
| 51. | Ruszkowska A, Ruszkowski M, Dauter Z, Brown JA. Structural insights into the RNA methyltransferase domain of METTL16. Sci Rep. 2018;8:5311. [PubMed] [DOI] |
| 52. | Dorsett M, Westlund B, Schedl T. Mett-10, a putative methyltransferase, inhibits germ cell proliferative fate in caenorhabditis elegans. Genetics. 2009;183(1), 233-47. [PubMed] [DOI] |
| 53. | Wen J, Lv R, Ma H, Shen H, He C, Wang J, Jiao F, Liu H, Yang P, Tan L, Lan F, Shi YG, He C, Shi Y, Diao J. Zc3h13 Regulates Nuclear RNA m6A Methylation and Mouse Embryonic Stem Cell Self-Renewal. Mol Cell. 2018;69:1028-1038.e6. [PubMed] [DOI] |
| 54. | Brown JA, Kinzig CG, DeGregorio SJ, Steitz JA. Methyltransferase-like protein 16 binds the 3'-terminal triple helix of MALAT1 long noncoding RNA. Proc Natl Acad Sci USA. 2016;113:14013-14018. [PubMed] [DOI] |
| 55. | Brown JA, Valenstein ML, Yario TA, Tycowski KT, Steitz JA. Formation of triple-helical structures by the 3'-end sequences of MALAT1 and MENβ noncoding RNAs. Proc Natl Acad Sci USA. 2012;109:19202-19207. [PubMed] [DOI] |
| 56. | Pendleton KE, Chen B, Liu K, Hunter OV, Xie Y, Tu BP, Conrad NK. The U6 snRNA m6A Methyltransferase METTL16 Regulates SAM Synthetase Intron Retention. Cell. 2017;169:824-835.e14. [PubMed] [DOI] |
| 57. | Shimazu T, Barjau J, Sohtome Y, Sodeoka M, Shinkai Y. Selenium-based S-adenosylmethionine analog reveals the mammalian seven-beta-strand methyltransferase METTL10 to be an EF1A1 lysine methyltransferase. PLoS One. 2014;9:e105394. [PubMed] [DOI] |
| 58. | Brannan KW, Jin W, Huelga SC, Banks CA, Gilmore JM, Florens L, Washburn MP, Van Nostrand EL, Pratt GA, Schwinn MK, Daniels DL, Yeo GW. SONAR Discovers RNA-Binding Proteins from Analysis of Large-Scale Protein-Protein Interactomes. Mol Cell. 2016;64:282-293. [PubMed] [DOI] |
| 59. | Martínez-Chantar ML, Latasa MU, Varela-Rey M, Lu SC, García-Trevijano ER, Mato JM, Avila MA. L-methionine availability regulates expression of the methionine adenosyltransferase 2A gene in human hepatocarcinoma cells: role of S-adenosylmethionine. J Biol Chem. 2003;278:19885-19890. [PubMed] [DOI] |