修回日期: 2019-11-20
接受日期: 2019-12-03
在线出版日期: 2019-12-08
消化道内生存有纷繁复杂的肠道菌群, 尤其以结肠中的细菌尤为复杂. 大量的实验表明, 肠道菌群的数量和肠道免疫系统的发育具有一定相互平行关系. 这对肠道功能的调节具有重要意义. 本文综述肠道微生物群的发生发展过程与肠道免疫系统发育特征的比较以及肠道微生物群与免疫系统对肠道功能的调节作用, 该研究为免疫相关疾病的治疗途径开辟了新的前景.
核心提要: 肠道菌群在机体的免疫系统的发生发展过程中发挥重要的作用, 而肠道免疫系统的发育与肠道菌群数量及多样性具有相关性, 该研究引起了科学家的高度重视.
引文著录: 王菲菲, 杨泽俊, 朱敏佳, 狄治杉, 尚宏伟, 徐敬东. 肠道菌群与肠道免疫系统的发育相关性进展. 世界华人消化杂志 2019; 27(23): 1446-1453
Revised: November 20, 2019
Accepted: December 3, 2019
Published online: December 8, 2019
The gut contains a complex array of intestinal bacteria, especially in the colon. A large number of studies have proved that the amount of intestinal flora and the development of the intestinal immune system have a certain parallel relationship, which is vital for the regulation for the intestinal function. This article reviews the correlation between the gut microbiota and the development of the intestinal immune system as well as the regulation of intestinal function by intestinal microbiota and immune system, with an aim to provide new clues to the treatment of immune-related diseases.
- Citation: Wang FF, Yang ZJ, Zhu MJ, Di ZS, Shang HW, Xu JD. Correlation between intestinal flora and gut immune system development. Shijie Huaren Xiaohua Zazhi 2019; 27(23): 1446-1453
- URL: https://www.wjgnet.com/1009-3079/full/v27/i23/1446.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v27.i23.1446
近来大量文献报道, 肠道菌群的构成影响机体黏膜免疫系统、免疫应答与免疫交流、免疫系统与其互惠互利. 这提示了二者的发育过程可能具有平行关系. 此外, 肠道菌群和免疫系统还调节肠神经网络[1]. 当肠道菌群紊乱, 可引起多种相关疾病. 本文将详述肠道菌群与肠道免疫系统的发育之间的关联性.
肠道里共生着约1013个细菌组成的菌落, 它们参与机体营养代谢, 产生身体必需维生素, 调节人体的健康状态, 其复杂的功能引起了科学家的关注. 尤其是肠道菌群与肠道相关疾病的关系成为研究热点[2]. 而肠道菌群的发育的过程有望为治疗相关疾病提供可能.
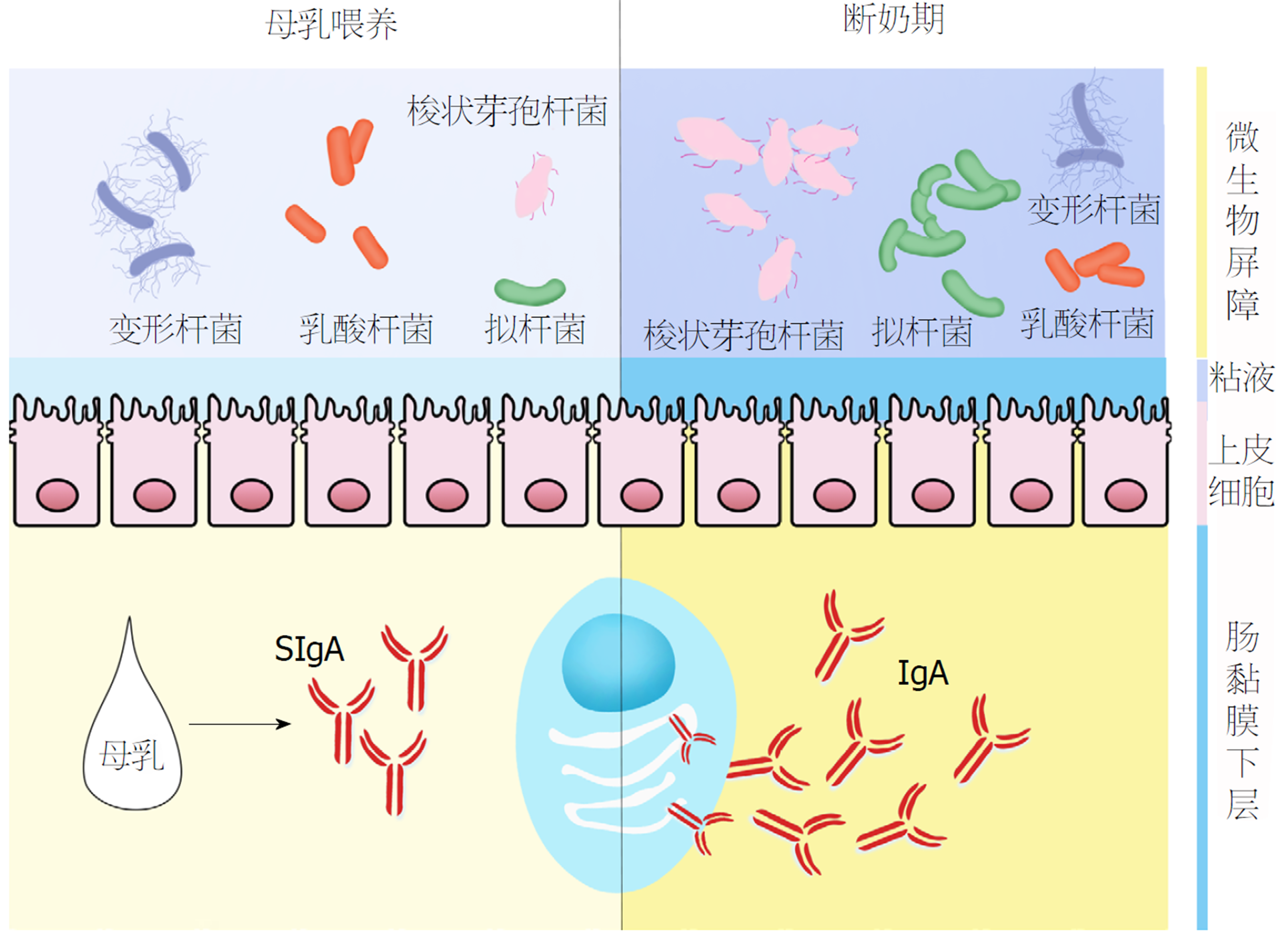
过去一直认为胎儿的消化道内保持无菌状态, 微生物群在出生时通过皮肤、口腔、肠道或阴道开始定殖[3]. 然而, 有研究表明微生物可能起源于子宫[4]. 但胎儿胃肠道微生物的起源、最初定殖的时间、不同菌种的比例还不清楚, 目前有"阴道播种"、来自阴道的位置上移或者是子宫起源等[5]. 值得注意的是, 这些微生物群落与羊水的微生物群相似[6], 很有可能是胎儿在子宫母体口腔微生物通过血液进入羊水, 胎儿吞咽羊水完成微生物定殖. 出生后, 胎儿突然暴露于富含各种细菌的外部环境使大量微生物群即刻定殖在肠腔[7], 此时肠道呈现以肠球菌和葡萄球菌主导的兼性厌氧和耐氧微生物[8]. 出生后数小时内由于阴道微生物从母体到婴儿垂直传播, 乳酸杆菌占主导地位[9]. 出生后1-7 d, 肠道菌群迅速而有序地"爆炸式"增殖形成双歧杆菌、拟杆菌和梭状杆菌为主的厌氧菌[10], 在出生后第7天形成主要由拟杆菌、放线菌和奇异杆菌定殖的生态系统(表1). 同在第7天时, 母乳中开始出现双歧杆菌为婴儿肠道和母乳之间共享[11], 这类特定菌株括还包括乳酸杆菌等有益菌和葡萄球菌、肠球菌等有害菌[12]. 出生后第30天, 婴儿的特殊微生物生态系统已趋向于以厚壁菌和拟杆菌为主导的成人胃肠道特征[13]. 第60天到第130天胃肠道微生物增殖减缓, 变为以双歧杆菌为主导而厚壁菌减少, 放线菌平均相对丰度增加的微生物生态系统[14]. 出生后第90天, 婴儿和母乳共享菌株在母乳和婴儿粪便中比例均很高[15], 提示母乳影响了婴儿肠道微生物的发展, 推测断奶期的菌群变化可能与婴儿对细菌的需求有关. 出生后170-290 d, 由于奶粉和豌豆等固体食物的摄入使机体对利用乳酸的细菌需求减少, 而有助合成维生素B、K与吸收碳水化合物的细菌需求加大[16], 同时断奶后小鼠不能继续获得母乳中SIgA以及低聚糖来辅助结肠中的发酵以产生短链脂肪酸[17], 以上种种原因导致断奶期变形杆菌和乳酸杆菌逐渐减少, 拟杆菌和梭状芽孢杆菌逐渐呈主导地位, 细菌总量显著增加[7,18](图1), 新生儿微生物生态系统逐步达到"肠型", 即由拟杆菌、普氏菌和厚壁菌主导的微生态系统[19]. 目前普遍认为, 其他一些因素也影响肠道及其微生物共生体的起源、组成和发展, 如孕期母亲的饮食[20]、抗生素[21]、分娩方式[22]以及母乳喂养等[23]. 在婴儿2岁时, 拟杆菌门和厚壁菌门丰度大于细菌总量的90%, 肠道菌群逐步发育为类似成人的水平[24,25].
| 肠道菌群 | 肠道免疫系统发育 | Ref. | |
| 起源 | 子宫内起源, 为厚壁菌门细菌(以乳酸菌为主) | 骨髓中的造血干细胞祖细胞产生淋巴细胞 | [4] |
| 妊娠7 wk | 无 | 表达CD34受体的T细胞祖细胞迁移至胸腺并分化成CD4+T细胞、CD8+T细胞和αβT细胞 | [28] |
| 妊娠8 wk | 无 | HSC定殖在未成熟胸腺上皮 | |
| 妊娠12 wk | 无 | 产生次级淋巴组织, 嗜中性粒细胞最初成熟 | [23] |
| 妊娠16-20 wk | 无 | HSC向BM转移, T细胞发育正在进行中并且T细胞移出以形成外周T细胞库 | |
| 妊娠31 wk | 无 | 中性粒细胞出现 | |
| 出生 | 肠腔中形成肠杆菌、葡萄球菌为主导的兼性厌氧和耐氧微生物 | NK细胞数量达到最高水平, CD4+CD25+Tregs丰富并且已经可以良好执行其功能 | [7,8] |
| 1-7 d | 微生物"爆炸式"增殖 | 中性粒细胞呈"指数式"增长, 单核细胞和巨噬细胞不断发育 | [9] |
| 7 d | 形成厚壁菌、拟杆菌、放线菌、变形杆菌等为主导的相对成熟的微生物生态系统 | 单核细胞、经典MΦ和MΦ均达到成熟状态 | [29] |
| 30 d | 厚壁菌和拟杆菌为主导, 趋于成人胃肠道的特征 | CD20+细胞占外周血B细胞的比例保持较高水平 | [10,55] |
| 60-130 d | 以长双歧杆菌和短双歧杆菌为主的放线菌平均相对丰度增加, 厚壁菌门减少 | [14] | |
| 170-290 d断奶期 | 双歧杆菌逐渐减少, 拟杆菌和厚壁菌逐渐呈主导地位, 细菌总量也显著增加 | CD19+B220lowB细胞进行免疫球蛋白基因重排、开始表达核基因重组激活基因2 | [11,30] |
胃肠道微生物是人和动物体内最复杂和最大的微生态系统. 成人肠道内含有超过500种细菌. 而新生儿出生时胃肠道是无菌的, 免疫系统几乎没有发育, 但很快由于细菌定植和其多样性的出现, 刺激肠道产生大量的淋巴细胞和淋巴组织, 以适应肠道生态环境的稳态[26].
肠道中固有淋巴细胞(innate lymphoid cells, ILCs)在胎儿出生时已发育完全, 作为入侵病原体的第一道防线. 胎儿期3 mo末, 中性粒细胞最初成熟, 树突状细胞在脐带血中发育并且初步成熟[23]. 出生时, 新生儿IL-6的水平急速上升, 使白介素(interleukin, IL)-6诱导性肝细胞产生C-反应蛋白(C-reactive protein, CRP)和脂多糖(lipopolysaccharide, LPS)[27]. 新生儿先天免疫细胞活性较成人低, 婴儿却拥有比成年期的更高活性的ILCs、更高水平的单核细胞和抗原呈递细胞(antigen presenting cell, APC)产生的IL-6, IL-10和IL-23[28], 这提示了肠道中固有淋巴细胞或许对特定情况有可变的敏感性. 出生后1-7 d, 单核细胞和巨噬细胞(macrophages, MΦ)不断发育, 在出生后的第7天达到成熟状态[29]. 在出生后的18-24 d, 有4%的CD19+B220lowB细胞中表达核基因重组激活基因(recombination activation gene, RAG)2, 但在小鼠出生后7 d内至5-6 wk, RAG2几乎不表达[30], 提示断奶期是骨髓中RAG调节V(D)J基因重排形成BCR的关键时期. 新生儿期, 肠道固有免疫系统具有一定的可塑性, 经典MΦ和MΦ未达到成熟状态, TLR4样受体4(Toll-like receptors, TLR4)的低表达和其不完善的信号通路导致组织修复不良、对潜在病原体的吞噬作用较差、生物活性分子分泌量不足令促炎细胞因子产生较少[31]. 新生儿期与胎儿期不同, 此时产生丰富的Th2细胞而合成少量Th1极化细胞因子, 其中包括肿瘤坏死因子(tumor necrosis factor, TNF)、γ-干扰素(interferon-γ, IFN-γ)和IL-12[32]. 出生7 d以后, 新生儿MΦ和APC中TNF、IL-12和IFN-γ的产生减少, 但IL-6、IL-10和IL-23的产生十分稳定[33]. 由此观之, 新生儿肠道免疫处于由分子和细胞成分网络精细调节中, 固有免疫通过识别"自身"和"非己", 不但成为机体抵御微生物侵袭的第一道防线, 而且参与特异性免疫应答, 甚至影响非感染性疾病的发生发展.
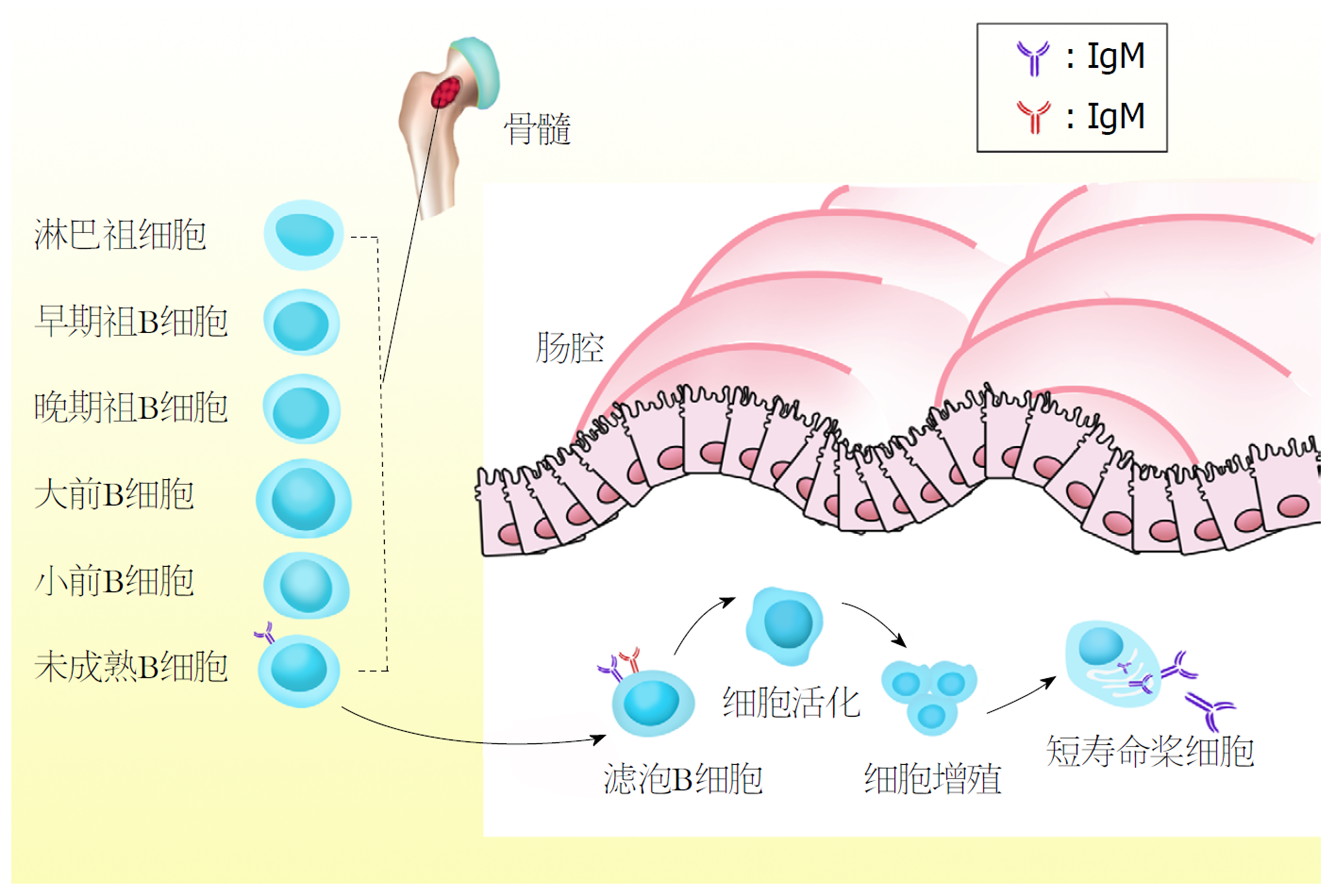
众所周知, B细胞体液免疫系统发挥重要作用, 而B细胞以持续合成和分泌抗体参与机体的体液免疫而备受关注. 成熟B细胞群主要有三类即B1细胞、边缘区B细胞、滤泡B细胞. 已经已知Notch通路等信号转导和表观遗传调控调节下[34], 转录因子IKA ROS、PU.1、E2A、早期B-细胞因子1(early B-cell factor1, EBF1)、配对盒5(paired box 5, PAX5)通过前馈、反馈环以及交叉拮抗作用相互连接, 构成复杂的网络来调节淋巴祖细胞产生B细胞功能性外周亚群[35]. 在胎儿肝脏中, B细胞是免疫细胞中最早发育的, 在RAG和IL-7信号的调节下, μH链和替代L链(SLCs)配对形成前B细胞受体(pre-BCR)[36-38]. miR-17-miR-92通过抑制PTEN基因(gene of phosphate and tension homology deleted on chromosome ten, PTEN)的表达, 促使pre-B细胞经历第一个自身反应检查点, 建立中枢耐受转化为未成熟B细胞[39]. 作为未成熟的"过渡性"B细胞到达脾脏即和CD21、CD22同时获得IgD, 成熟后进入卵泡或边缘区. 妊娠31 wk, 胎儿嗜中性粒细胞开始产生[40]. 在出生后1-7 d, 在粒细胞集落刺激因子(granulocyte colony-stimulating factor, GCSF)刺激下, 中性粒细胞呈"指数式"急剧增长[41], 出生后第7天, 中性粒细胞数量恢复到稳定水平, 但此时它们的杀菌功能、炎性刺激的反应较弱, 对内皮细胞的粘附性和趋化性较差[42], 这是由于B细胞共同受体低表达使B细胞的抗体反应能力较弱导致的. 此时B细胞在脾、卵泡外和生发中心增殖分化为PC(图2). 此时卵泡外分化相对不受控制, 产生分泌低亲和力抗体的短寿命浆细胞(short-lived plasma cell, SLPC)[43]. 然而, 生发中心允许外部信号整合以延迟浆细胞分化, 最终产生可以分泌高亲和力抗体的PC[44]. SIgM与B细胞上的FcμR相互作用, 促进早期B细胞活化[45], 通过iLS细胞和B细胞诱导发现B细胞发育的编程是根据转录特征进行排序, 首先是基因表达涉及抑制与多能性相关的增强子和与替代细胞谱系相关的增强子或启动子, 其次激活与细胞周期进程和细胞代谢相关的基因, 再次建立一个B细胞分化因子的调节通路, 如E2A、Forkhead转录因子(forkhead box O1, FoxO1), EBF1和PAX5[46]. 活化B细胞表达IL-6受体与Th2细胞产物IL-10共同作用, 增加PC数量、抑制Th1细胞产生细胞因子并诱导肠道PC分泌大量的IgG、SIgA和IgM. 记忆B细胞(memory B cell, MB)活化后, CD27和CD20相互作用使B细胞产生Ig的作用增强. 另外B细胞通过转录因子和信号分子完成分化的分子机制包括下游信号蛋白布鲁氏酪氨酸激酶(bruton tyrosine kinase, Btk)、TLR[47]、B淋巴细胞诱导成熟蛋白(B-lymphocyte induced maturation protein1, Blimp-1), X-框结合蛋白1(X-box binding protein 1, XBP-1)和干扰素调节因子4(interferon regulation factor 4, IRF4)等转录因子的相互作用[48], 其中尤为重要的是IRF4, 其指导生发中心(germinal center, GC)的B细胞进行细胞自主性的分化, 与XBP1、Blimp-1共同决定PC的特性和功能, IRF4瞬时爆发表达诱导表达Bcl-6的B细胞群产生[47]并介导PC分化[49]. 出生后, 新生儿接触大量外界抗原, IgG+和IgA+ B细胞比例大量增加. 当B细胞受到抗原刺激后, 通过Ig基因座中的类别转换, 依次分泌IgM、IgG、IgA. 在1岁时, 儿童的IgG水平已达到成人水平的约70%, 但仅为成人IgA水平的30%, 说明肠道的B细胞还需继续分化和成熟.
在子宫内, 胎儿免疫系统需要对母体同种异体抗原保持耐受. 出生后, 新生儿进入复杂抗原环境中. 在出生时, 肠腔中形成肠杆菌、葡萄球菌为主导的兼性厌氧和耐氧微生物, 同时CD4+CD25+Tregs数量丰富且可以良好执行其功能[50], 这是由于微生物定殖影响肠道CD4+或CD8+T细胞数、T细胞活化状态、CD4亚群和肠微结构[51]. 更有趣的是, 出生后1-7 d内, 肠腔内微生物"爆炸式"增殖, 肠道免疫细胞中性粒细胞也呈"指数式"增长, 伴有单核细胞和巨噬细胞的发育. 最终在出生后第7天肠道形成厚壁菌、拟杆菌、放线菌、变形杆菌等为主导的微生物生态系统, 同时单核细胞和巨噬细胞也达到成熟状态, 肠道免疫细胞与肠道菌群二者的成熟相互平行, 从而适应肠道复杂的环境[52-55].
在婴儿期之后, 肠道微生物群与免疫系统相互作用, 在平行发育中共同构建一个和谐有序的肠道微环境[56]. 断奶前, 肠道中的抗体来自母乳中的SIgA, B细胞不产生抗体, 肠道菌群以变形杆菌和乳酸杆菌为主导; 断奶后, 肠道中原本由母乳提供的SIgA缺失, 肠道菌群总量增加, 产生的代谢产物刺激B细胞分化为成熟的浆细胞, 产生IgA[57], 此时肠道菌群以梭状芽孢杆菌和拟杆菌为主导, 逐渐趋向成人肠道菌群[58]. 小鼠出生后第14-21天(断奶期), 小肠固有层绒毛和上皮内的B细胞和T细胞数目显著增多,与此同时, 肠道拟杆菌逐渐呈主导地位, 细菌总量也显著增加, 免疫细胞和肠道微生物的数量变化平行. 一项关于断奶期肠道免疫细胞和拟杆菌的研究可为上述平行性提供理论依据. 经多形拟杆菌或是肠道微生物群定殖的GF小鼠血管生成素(angiogenin 4, Ang4)可恢复至正常水平, 这表明在断奶期间诱导Ang4表达需要肠道微生物群, 推测断奶期由于母乳免疫球蛋白介导的被动免疫逐步减弱, 肠道菌群改变, 刺激肠道免疫细胞和抗菌成分的改变, 从而参与肠道上皮宿主免疫[42]. 免疫系统中PC和细菌的分布具有平行性, 从肠道近端到远端, 免疫系统中PC的逐渐增多, 在结肠内达到峰值. 与此相平行地, 从上到下, 细菌的数量逐渐递增, 十二指肠约为102 cfu/mL, 空肠约为102 cfu/mL, 近端回肠约为103 cfu/mL, 远端回肠约为107-108 cfu/mL, 结肠约为1011-1022 cfu/mL, 细菌数量与PC一样在结肠处达到峰值[59], 这是由于细菌数量呈通过抗原特异性和非特异性方式使得肠道免疫系统发育完善, 使得肠道菌群定植于肠道中, 此时肠道免疫系统已经具有维持宿主与这些高度多样化和不断发展的微生物的共生关系的手段[60].
在出生后的第一年, 肠道微生物群的组成不断成熟, 微生物可能影响宿主的免疫成熟的能力. 在此阶段, 如果肠道微生物的紊乱会导致哮喘、过敏和其他免疫疾病的发生. 因此, 肠道微生物可能是一个重要的环境因素, 决定了从健康到疾病的过渡. 事实上, 一些研究结果显示婴儿体内的微生物群落会因为出生方式、饮食、环境和抗生素使用等原因而有所不同, 而这种差异可能会影响这些孩子们患病的几率, 比如哮喘和过敏[61]. 而婴儿粪便中如含有拉希诺皮拉菌、韦荣球菌、粪细菌、罗斯氏菌四种肠道细菌, 即可较为准确的预测患哮喘的风险明显增加, 而且通过控制这些肠道细菌的组成还有可能找到预防儿童哮喘的方法[62]. 提示肠道菌群数量和丰度降低时, 其患哮喘的可能性增大.
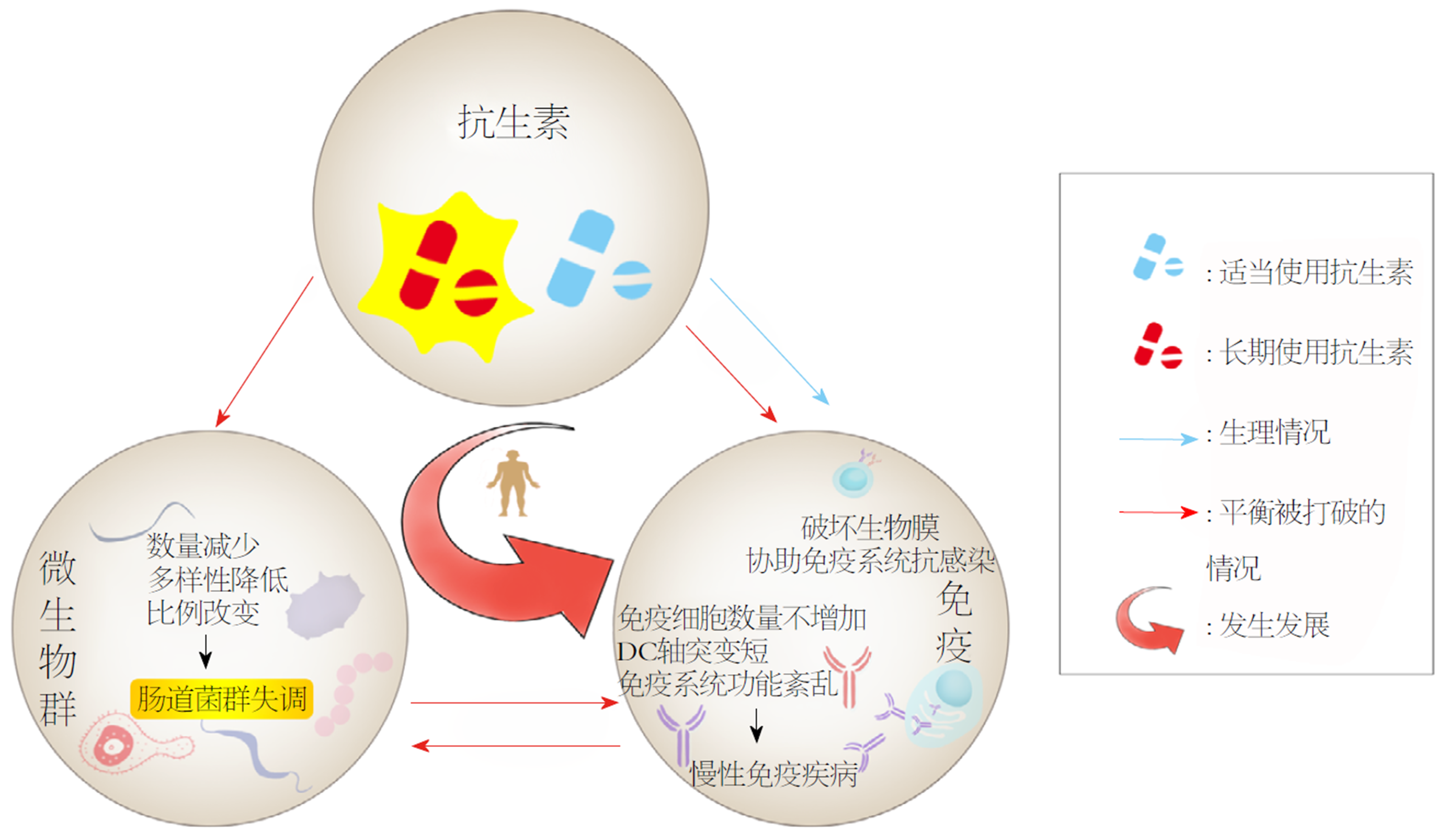
越来越多的医务工作者发现抗生素的滥用往往导致肠道菌群的失调. 实验证明围产期抗生素给药后第7天, 婴儿肠道中放线菌属和拟杆菌类比例降低, 而变形菌和厚壁菌比例增加, 出现肠道菌群失调[63], 出生后3-9 mo内瘤胃球菌(ruminococcus)和梭状杆菌(clostridium)数量大幅下降, 微生物群成熟延迟[10], T细胞、B细胞发生紊乱,中性粒细胞和巨噬细胞的数量均不再增加[64], DC轴突变小萎缩, 甚至无法在粪便中检测到微生物群而当肠道以假单胞菌属为主时, 多样性的减少尤为突出[65]. 在一项为期7 d的克林霉素疗程后2年, 人类粪便微生物依旧没有完全恢复并持续发生变化[66], 这提示即使是短暂的抗生素扰动也会导致微生物群恒久的改变. 当然, 也有相反的研究认为, 婴儿接触抗生素对肠道微生物组的影响并不明显[67]. 抗生素、肠道菌群和免疫三者之间复杂的相互作用引发了临床工作者的广泛关注, 而肠道菌群与慢性免疫疾病的相关性也对未来以广大临床工作者微生物为基础的诊断和治疗提供了更进一步的可能性. 许多炎症性疾病受到先天免疫系统和微生物菌群之间交叉信号转导异常的影响, 包括代谢类疾病、肿瘤疾病和自身免疫性疾病或自身炎症性疾病, 比如溃疡性结肠炎、自身免疫性肝病等[68]. 通过饮食干预及其调节微生物菌群与免疫系统相互作用, 机体菌群失衡, 此时应该多补充有益菌, 恢复菌群平衡, 强化先天免疫系统, 这样才有希望成为健康、强壮的"先天高手".
学科分类: 胃肠病学和肝病学
手稿来源地: 北京市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): D, D
E级 (差): 0
编辑: 王禹乔 电编:刘继红
| 1. | Shi N, Li N, Duan X, Niu H. Interaction between the gut microbiome and mucosal immune system. Mil Med Res. 2017;4:14. [PubMed] [DOI] |
| 2. | Jost T, Lacroix C, Braegger C, Chassard C. Impact of human milk bacteria and oligosaccharides on neonatal gut microbiota establishment and gut health. Nutr Rev. 2015;73:426-437. [PubMed] [DOI] |
| 3. | Obata Y, Pachnis V. The Effect of Microbiota and the Immune System on the Development and Organization of the Enteric Nervous System. Gastroenterology. 2016;151:836-844. [PubMed] [DOI] |
| 4. | Chu DM, Ma J, Prince AL, Antony KM, Seferovic MD, Aagaard KM. Maturation of the infant microbiome community structure and function across multiple body sites and in relation to mode of delivery. Nat Med. 2017;23:314-326. [PubMed] [DOI] |
| 5. | Stinson LF, Payne MS, Keelan JA. Planting the seed: Origins, composition, and postnatal health significance of the fetal gastrointestinal microbiota. Crit Rev Microbiol. 2017;43:352-369. [PubMed] [DOI] |
| 6. | Tanaka M, Nakayama J. Development of the gut microbiota in infancy and its impact on health in later life. Allergol Int. 2017;66:515-522. [PubMed] [DOI] |
| 7. | Del Chierico F, Vernocchi P, Petrucca A, Paci P, Fuentes S, Praticò G, Capuani G, Masotti A, Reddel S, Russo A, Vallone C, Salvatori G, Buffone E, Signore F, Rigon G, Dotta A, Miccheli A, de Vos WM, Dallapiccola B, Putignani L. Phylogenetic and Metabolic Tracking of Gut Microbiota during Perinatal Development. PLoS One. 2015;10:e0137347. [PubMed] [DOI] |
| 8. | Matsuki T, Yahagi K, Mori H, Matsumoto H, Hara T, Tajima S, Ogawa E, Kodama H, Yamamoto K, Yamada T, Matsumoto S, Kurokawa K. A key genetic factor for fucosyllactose utilization affects infant gut microbiota development. Nat Commun. 2016;7:11939. [PubMed] [DOI] |
| 9. | Hansen R, Scott KP, Khan S, Martin JC, Berry SH, Stevenson M, Okpapi A, Munro MJ, Hold GL. First-Pass Meconium Samples from Healthy Term Vaginally-Delivered Neonates: An Analysis of the Microbiota. PLoS One. 2015;10:e0133320. [PubMed] [DOI] |
| 10. | Bokulich NA, Chung J, Battaglia T, Henderson N, Jay M, Li H, D Lieber A, Wu F, Perez-Perez GI, Chen Y, Schweizer W, Zheng X, Contreras M, Dominguez-Bello MG, Blaser MJ. Antibiotics, birth mode, and diet shape microbiome maturation during early life. Sci Transl Med. 2016;8:343ra82. [PubMed] [DOI] |
| 11. | Aagaard K, Ma J, Antony KM, Ganu R, Petrosino J, Versalovic J. The placenta harbors a unique microbiome. Sci Transl Med. 2014;6:237ra65. [PubMed] [DOI] |
| 13. | Zhao XQ, Guo S, Lu YY, Hua Y, Zhang F, Yan H, Shang EX, Wang HQ, Zhang WH, Duan JA. Lycium barbarum L. leaves ameliorate type 2 diabetes in rats by modulating metabolic profiles and gut microbiota composition. Biomed Pharmacother. 2019;121:109559. [PubMed] [DOI] |
| 14. | de Muinck EJ, Trosvik P. Individuality and convergence of the infant gut microbiota during the first year of life. Nat Commun. 2018;9:2233. [PubMed] [DOI] |
| 15. | Makino H, Martin R, Ishikawa E, Gawad A, Kubota H, Sakai T, Oishi K, Tanaka R, Ben-Amor K, Knol J, Kushiro A. Multilocus sequence typing of bifidobacterial strains from infant's faeces and human milk: are bifidobacteria being sustainably shared during breastfeeding? Benef Microbes. 2015;6:563-572. [PubMed] [DOI] |
| 16. | Vallès Y, Artacho A, Pascual-García A, Ferrús ML, Gosalbes MJ, Abellán JJ, Francino MP. Microbial succession in the gut: directional trends of taxonomic and functional change in a birth cohort of Spanish infants. PLoS Genet. 2014;10:e1004406. [PubMed] [DOI] |
| 17. | Laursen MF, Bahl MI, Michaelsen KF, Licht TR. First Foods and Gut Microbes. Front Microbiol. 2017;8:356. [PubMed] [DOI] |
| 18. | Ma J, Prince AL, Bader D, Hu M, Ganu R, Baquero K, Blundell P, Alan Harris R, Frias AE, Grove KL, Aagaard KM. High-fat maternal diet during pregnancy persistently alters the offspring microbiome in a primate model. Nat Commun. 2014;5:3889. [PubMed] [DOI] |
| 19. | Costea PI, Hildebrand F, Arumugam M, Bäckhed F, Blaser MJ, Bushman FD, de Vos WM, Ehrlich SD, Fraser CM, Hattori M, Huttenhower C, Jeffery IB, Knights D, Lewis JD, Ley RE, Ochman H, O'Toole PW, Quince C, Relman DA, Shanahan F, Sunagawa S, Wang J, Weinstock GM, Wu GD, Zeller G, Zhao L, Raes J, Knight R, Bork P. Enterotypes in the landscape of gut microbial community composition. Nat Microbiol. 2018;3:8-16. [PubMed] [DOI] |
| 20. | Chassaing B, Vijay-Kumar M, Gewirtz AT. How diet can impact gut microbiota to promote or endanger health. Curr Opin Gastroenterol. 2017;33:417-421. [PubMed] [DOI] |
| 21. | Gosalbes MJ, Llop S, Vallès Y, Moya A, Ballester F, Francino MP. Meconium microbiota types dominated by lactic acid or enteric bacteria are differentially associated with maternal eczema and respiratory problems in infants. Clin Exp Allergy. 2013;43:198-211. [PubMed] [DOI] |
| 22. | Martin R, Makino H, Cetinyurek Yavuz A, Ben-Amor K, Roelofs M, Ishikawa E, Kubota H, Swinkels S, Sakai T, Oishi K, Kushiro A, Knol J. Early-Life Events, Including Mode of Delivery and Type of Feeding, Siblings and Gender, Shape the Developing Gut Microbiota. PLoS One. 2016;11:e0158498. [PubMed] [DOI] |
| 23. | Murphy K, Curley D, O'Callaghan TF, O'Shea CA, Dempsey EM, O'Toole PW, Ross RP, Ryan CA, Stanton C. The Composition of Human Milk and Infant Faecal Microbiota Over the First Three Months of Life: A Pilot Study. Sci Rep. 2017;7:40597. [PubMed] [DOI] |
| 24. | Carding S, Verbeke K, Vipond DT, Corfe BM, Owen LJ. Dysbiosis of the gut microbiota in disease. Microb Ecol Health Dis. 2015;26:26191. [PubMed] [DOI] |
| 25. | Milani C, Duranti S, Bottacini F, Casey E, Turroni F, Mahony J, Belzer C, Delgado Palacio S, Arboleya Montes S, Mancabelli L, Lugli GA, Rodriguez JM, Bode L, de Vos W, Gueimonde M, Margolles A, van Sinderen D, Ventura M. The First Microbial Colonizers of the Human Gut: Composition, Activities, and Health Implications of the Infant Gut Microbiota. Microbiol Mol Biol Rev. 2017;81. [PubMed] [DOI] |
| 26. | Sánchez B, Gueimonde M, Peña AS, Bernardo D. Intestinal microbiota as modulators of the immune system. J Immunol Res. 2015;2015:159094. [PubMed] [DOI] |
| 28. | Basha S, Surendran N, Pichichero M. Immune responses in neonates. Expert Rev Clin Immunol. 2014;10:1171-1184. [PubMed] [DOI] |
| 30. | Nagalingam NA, Kao JY, Young VB. Microbial ecology of the murine gut associated with the development of dextran sodium sulfate-induced colitis. Inflamm Bowel Dis. 2011;17:917-926. [PubMed] [DOI] |
| 31. | Förster-Waldl E, Sadeghi K, Tamandl D, Gerhold B, Hallwirth U, Rohrmeister K, Hayde M, Prusa AR, Herkner K, Boltz-Nitulescu G, Pollak A, Spittler A. Monocyte toll-like receptor 4 expression and LPS-induced cytokine production increase during gestational aging. Pediatr Res. 2005;58:121-124. [PubMed] [DOI] |
| 32. | Blahnik MJ, Ramanathan R, Riley CR, Minoo P. Lipopolysaccharide-induced tumor necrosis factor-alpha and IL-10 production by lung macrophages from preterm and term neonates. Pediatr Res. 2001;50:726-731. [PubMed] [DOI] |
| 33. | Yu JC, Khodadadi H, Malik A, Davidson B, Salles ÉDSL, Bhatia J, Hale VL, Baban B. Innate Immunity of Neonates and Infants. Front Immunol. 2018;9:1759. [PubMed] [DOI] |
| 34. | Rossi D. Notch signaling in peripheral T-cell lymphoma: revelations from immunohistochemistry. Leuk Lymphoma. 2014;55:480-481. [PubMed] [DOI] |
| 35. | Boller S, Grosschedl R. The regulatory network of B-cell differentiation: a focused view of early B-cell factor 1 function. Immunol Rev. 2014;261:102-115. [PubMed] [DOI] |
| 36. | Hardy RR, Hayakawa K. B cell development pathways. Annu Rev Immunol. 2001;19:595-621. [PubMed] [DOI] |
| 37. | Milne CD, Paige CJ. IL-7: a key regulator of B lymphopoiesis. Semin Immunol. 2006;18:20-30. [PubMed] [DOI] |
| 38. | Melchers F. The pre-B-cell receptor: selector of fitting immunoglobulin heavy chains for the B-cell repertoire. Nat Rev Immunol. 2005;5:578-584. [PubMed] [DOI] |
| 39. | Lai M, Gonzalez-Martin A, Cooper AB, Oda H, Jin HY, Shepherd J, He L, Zhu J, Nemazee D, Xiao C. Regulation of B-cell development and tolerance by different members of the miR-17∼92 family microRNAs. Nat Commun. 2016;7:12207. [PubMed] [DOI] |
| 42. | Nussbaum C, Gloning A, Pruenster M, Frommhold D, Bierschenk S, Genzel-Boroviczény O, von Andrian UH, Quackenbush E, Sperandio M. Neutrophil and endothelial adhesive function during human fetal ontogeny. J Leukoc Biol. 2013;93:175-184. [PubMed] [DOI] |
| 44. | Oracki SA, Walker JA, Hibbs ML, Corcoran LM, Tarlinton DM. Plasma cell development and survival. Immunol Rev. 2010;237:140-159. [PubMed] [DOI] |
| 45. | Nguyen TTT, Graf BA, Randall TD, Baumgarth N. sIgM-FcμR Interactions Regulate Early B Cell Activation and Plasma Cell Development after Influenza Virus Infection. J Immunol. 2017;199:1635-1646. [PubMed] [DOI] |
| 46. | Miyai T, Takano J, Endo TA, Kawakami E, Agata Y, Motomura Y, Kubo M, Kashima Y, Suzuki Y, Kawamoto H, Ikawa T. Three-step transcriptional priming that drives the commitment of multipotent progenitors toward B cells. Genes Dev. 2018;32:112-126. [PubMed] [DOI] |
| 47. | Ochiai K, Maienschein-Cline M, Simonetti G, Chen J, Rosenthal R, Brink R, Chong AS, Klein U, Dinner AR, Singh H, Sciammas R. Transcriptional regulation of germinal center B and plasma cell fates by dynamical control of IRF4. Immunity. 2013;38:918-929. [PubMed] [DOI] |
| 48. | Nutt SL, Taubenheim N, Hasbold J, Corcoran LM, Hodgkin PD. The genetic network controlling plasma cell differentiation. Semin Immunol. 2011;23:341-349. [PubMed] [DOI] |
| 49. | Sciammas R, Shaffer AL, Schatz JH, Zhao H, Staudt LM, Singh H. Graded expression of interferon regulatory factor-4 coordinates isotype switching with plasma cell differentiation. Immunity. 2006;25:225-236. [PubMed] [DOI] |
| 51. | Gomez de Agüero M, Ganal-Vonarburg SC, Fuhrer T, Rupp S, Uchimura Y, Li H, Steinert A, Heikenwalder M, Hapfelmeier S, Sauer U, McCoy KD, Macpherson AJ. The maternal microbiota drives early postnatal innate immune development. Science. 2016;351:1296-1302. [PubMed] [DOI] |
| 52. | Maynard CL, Elson CO, Hatton RD, Weaver CT. Reciprocal interactions of the intestinal microbiota and immune system. Nature. 2012;489:231-241. [PubMed] [DOI] |
| 53. | Wesemann DR, Portuguese AJ, Meyers RM, Gallagher MP, Cluff-Jones K, Magee JM, Panchakshari RA, Rodig SJ, Kepler TB, Alt FW. Microbial colonization influences early B-lineage development in the gut lamina propria. Nature. 2013;501:112-115. [PubMed] [DOI] |
| 54. | Ygberg S, Nilsson A. The developing immune system - from foetus to toddler. Acta Paediatr. 2012;101:120-127. [PubMed] [DOI] |
| 55. | Hannet I, Erkeller-Yuksel F, Lydyard P, Deneys V, DeBruyère M. Developmental and maturational changes in human blood lymphocyte subpopulations. Immunol Today. 1992;13:215, 218. [PubMed] [DOI] |
| 56. | Ley RE, Hamady M, Lozupone C, Turnbaugh PJ, Ramey RR, Bircher JS, Schlegel ML, Tucker TA, Schrenzel MD, Knight R, Gordon JI. Evolution of mammals and their gut microbes. Science. 2008;320:1647-1651. [PubMed] [DOI] |
| 57. | Arrieta MC, Stiemsma LT, Dimitriu PA, Thorson L, Russell S, Yurist-Doutsch S, Kuzeljevic B, Gold MJ, Britton HM, Lefebvre DL, Subbarao P, Mandhane P, Becker A, McNagny KM, Sears MR, Kollmann T; CHILD Study Investigators, Mohn WW, Turvey SE, Finlay BB. Early infancy microbial and metabolic alterations affect risk of childhood asthma. Sci Transl Med. 2015;7:307ra152. [PubMed] [DOI] |
| 58. | Ober C, Sperling AI, von Mutius E, Vercelli D. Immune development and environment: lessons from Amish and Hutterite children. Curr Opin Immunol. 2017;48:51-60. [PubMed] [DOI] |
| 59. | Sevelsted A, Stokholm J, Bønnelykke K, Bisgaard H. Cesarean section and chronic immune disorders. Pediatrics. 2015;135:e92-e98. [PubMed] [DOI] |
| 60. | Belkaid Y, Hand TW. Role of the microbiota in immunity and inflammation. Cell. 2014;157:121-141. [PubMed] [DOI] |
| 61. | Palmer C, Bik EM, DiGiulio DB, Relman DA, Brown PO. Development of the human infant intestinal microbiota. PLoS Biol. 2007;5:e177. [PubMed] [DOI] |
| 63. | Nogacka A, Salazar N, Suárez M, Milani C, Arboleya S, Solís G, Fernández N, Alaez L, Hernández-Barranco AM, de Los Reyes-Gavilán CG, Ventura M, Gueimonde M. Impact of intrapartum antimicrobial prophylaxis upon the intestinal microbiota and the prevalence of antibiotic resistance genes in vaginally delivered full-term neonates. Microbiome. 2017;5:93. [PubMed] [DOI] |
| 64. | Ray A, Basu S, Gharaibeh RZ, Cook LC, Kumar R, Lefkowitz EJ, Walker CR, Morrow CD, Franklin CL, Geiger TL, Salzman NH, Fodor A, Dittel BN. Gut Microbial Dysbiosis Due to Helicobacter Drives an Increase in Marginal Zone B Cells in the Absence of IL-10 Signaling in Macrophages. J Immunol. 2015;195:3071-3085. [PubMed] [DOI] |
| 65. | Dellacecca ER, Cosgrove C, Mukhatayev Z, Akhtar S, Engelhard VH, Rademaker AW, Knight K, Le Poole IC. Antibiotics drive microbial imbalance and vitiligo development in mice. J Invest Dermatol. 2019;. [PubMed] [DOI] |
| 66. | 李 娜, 赵 玉民, 陈 正元, 王 碧君, 罗 霞, 王 青, 何 国林, 熊 天琴, 桑 传兰. 抗生素诱导肠道菌群失调对肠黏膜屏障和肝脏功能的动态影响. 动物营养学报. 2019;31:1278-1287. [DOI] |
| 67. | Liu Y, Qin S, Song Y, Feng Y, Lv N, Xue Y, Liu F, Wang S, Zhu B, Ma J, Yang H. The Perturbation of Infant Gut Microbiota Caused by Cesarean Delivery Is Partially Restored by Exclusive Breastfeeding. Front Microbiol. 2019;10:598. [PubMed] [DOI] |
| 69. | Langdon A, Crook N, Dantas G. The effects of antibiotics on the microbiome throughout development and alternative approaches for therapeutic modulation. Genome Med. 2016;8:39. [PubMed] [DOI] |
| 70. | Götz F, Perconti S, Popella P, Werner R, Schlag M. Epidermin and gallidermin: Staphylococcal lantibiotics. Int J Med Microbiol. 2014;304:63-71. [PubMed] [DOI] |