修回日期: 2019-06-05
接受日期: 2019-06-25
在线出版日期: 2019-07-08
结直肠癌(colorectal cancer, CRC)是临床中最为常见的消化系统恶性肿瘤, 近年来长链非编码RNA(long-chain non-coding RNA, LncRNA)在CRC发生、发展及侵袭转移中发挥重要作用. MALAT1为新近发现的LncRNA, 其在CRC中的作用及与患者预后的关系并不十分清楚.
应用生物信息数据挖掘技术探讨LncRNA MALAT1在大肠癌中的差异表达及其临床意义.
应用BioGPS数据库分析正常肠上皮中MALAT1的表达情况. 在肿瘤芯片表达数据库Oncomine中荟萃分析大肠癌组织与正常肠上皮组织MALAT1的差异表达并分析MALAT1高、低表达与患者生存期是否存在相关性. 在STRING数据库中应用蛋白相互作用网络分析与MALAT1相互作用及共表达的相关蛋白.
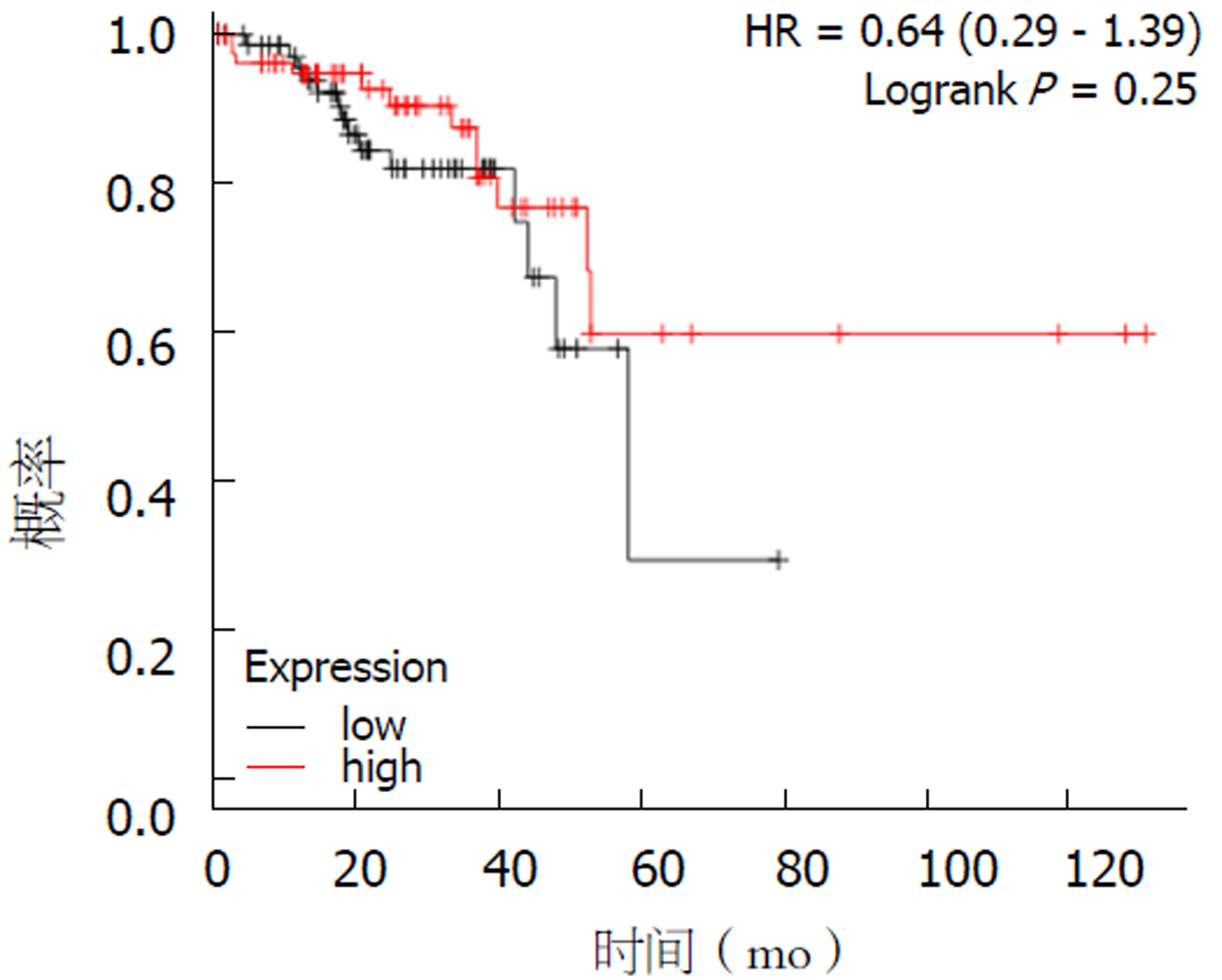
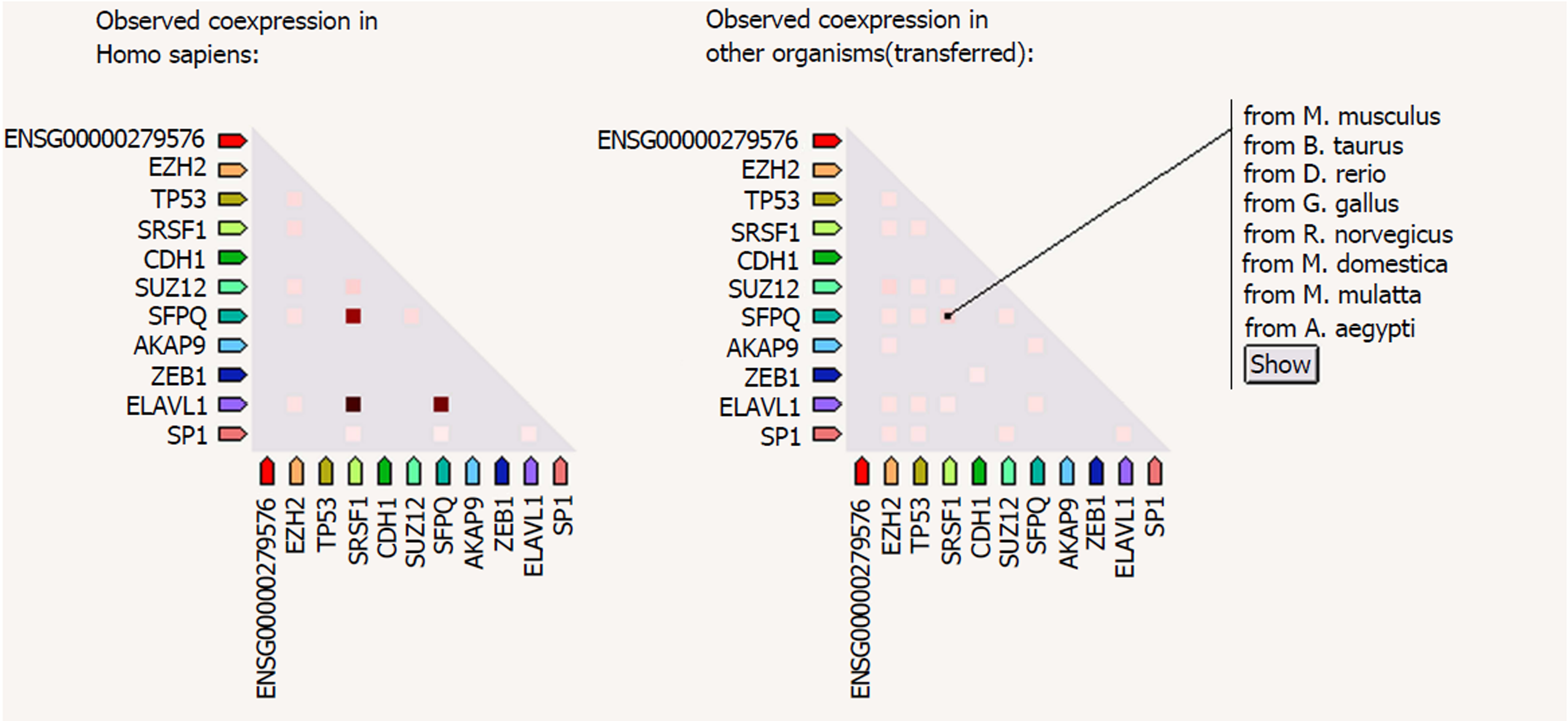
MALAT1在正常大肠癌组织中的相对表达量较低. Oncomine数据库中, 大肠癌差异表达的研究8项, 其中6项在大肠癌中高表达, 2项低表达. 比较大肠癌与对应正常组织的相关芯片数据为18个. 大肠癌组织中MALAT1表达水平显著高于正常组织(P = 0.027). 按结肠癌与直肠癌进亚组分析, 结肠癌组织与正常组织MALAT1表达水平无统计学差异(P = 0.149), 而直肠癌组织中MALAT1表达水平明显高于对应正常组织(P = 1.04 E-5). 在Kaplan-Meier Plotter数据平台中分析MALAT1高、低表达组总生存期分别为41.93 mo和52.2 mo, 差异无统计学意义(HR = 0.64, 95%CI: 0.29-1.39, P = 0.25). String数据库中分析与MALAT1可能相互作用的蛋白, 结果显示MALAT1与TP53, SUZ12, CDK4, KDMA, 等具有相互作用关系. 共表达分析显示MALAT1与EZH2, TP53, SRSF1等基因具有共表达关系, 提示这些基因在功能上可能具有相似性.
大肠癌组织中MALAT1基因表达表达水平明显上调, 但MALAT1表达水平与患者预后并无相关性. MALAT1与TP53, SUZ12, CDK4, KDMA蛋白相互作用, 上述相互作用蛋白包括多梳蛋白家族, 周期蛋白依赖性蛋白激酶等, 与肿瘤的基因表达、转录调控及细胞分裂有关.
核心提要: 本文通过生物信息学数据深入挖掘, 初步探讨并明确了长链非编码RNA MALAT1在结直肠癌(colorectal cancer, CRC)中表达水平、相关信号通路及其与患者预后的关系. 研究认为, MALAT1在CRC患者癌组织中的表达水平明显高于癌旁正常组织. MALAT1与TP53, SUZ12, CDK4, KDMA等具有相互作用关系且MALAT1与EZH2, TP53, SRSF1等基因具有共表达关系, 提示这些基因在功能上可能具有相似性. 但由于样本量、检测方法等原因, 研究结果并未发现MALAT1表达水平与CRC患者预后有关. 因此, MALAT1表达水平与CRC患者预后的关系有待进一步研究.
引文著录: 倪雅懿, 薛丽华, 张培, 朱广博. 长链非编码RNA MALAT1在大肠癌中的表达及其临床意义: 基于多基因表达数据库分析. 世界华人消化杂志 2019; 27(13): 814-821
Revised: June 5, 2019
Accepted: June 25, 2019
Published online: July 8, 2019
Colorectal cancer (CRC) is the most common malignant tumor of the digestive system. Long-chain non-coding RNAs (lncRNAs) play an important role in the occurrence, development, invasion and metastasis of CRC. MALAT1 is a newly discovered lncRNA, and its role in CRC and its relationship with prognosis are not completely clear.
To explore the differential expression of the lncRNA MALAT1 in CRC and its clinical significance by using bioinformatics data mining technology.
BioGPS database was used to analyze the expression of MALAT1 in normal intestinal epithelium. Oncomine was used to meta-analyze the differential expression of MALAT1 in CRC tissue and normal intestinal epithelium tissue, and to analyze the difference of survival time between patients with high and low expression of MALAT1. Protein interaction network analysis was performed based on the STING database to analyze the proteins that potential interact with MALAT1.
The relative expression of MALAT1 in normal colorectal tissues was low. In Oncomine database, there were eight studies on differential expression of CRC, six of which suggested high expression of MALAT1 in CRC and two suggested low expression. The microarray data of 18 CRC and matched normal tissues were compared. The expression of MALAT1 in CRC tissues was significantly higher than that in normal tissues (P = 0.027). There was no significant difference in MALAT1 expression between colon cancer and normal tissues (P = 0.149), but the expression of MALAT1 in rectal cancer was significantly higher than that in normal tissues (P = 1.04 E-5). Kaplan-Meier Plotter analysis demonstrated that the overall survival time of the high and low MALAT1 expression groups was 41.93 months and 52.2 months, respectively, with no significant difference (HR = 0.64, 95%CI: 0.29-1.39, P = 0.25). The possible interaction proteins of MALAT1 were analyzed based on the String database. The results showed that MALAT1 interacts with TP53, SUZ12, CDK4, KDMA, etc. Co-expression analysis showed that MALAT1 protein was co-expressed with EZH2, TP53, SRSF1, and other genes, suggesting that these genes may have similar functions.
The expression level of MALAT1 gene in CRC tissues is significantly up-regulated, but there is no correlation between MALAT1 expression and the prognosis of patients. MALAT1 interacts with TP53, SUZ12, CDK4, and KDMA proteins. These interacting proteins include Polycomb-group proteins, cyclin-dependent protein kinase and so on, which are related to gene expression control, transcriptional regulation, and cell division.
- Citation: Ni YY, Xue LH, Zhang P, Zhu GB. MALAT1 gene expression in colorectal cancer and its clinical significance: Data mining based on multiple gene expression databases. Shijie Huaren Xiaohua Zazhi 2019; 27(13): 814-821
- URL: https://www.wjgnet.com/1009-3079/full/v27/i13/814.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v27.i13.814
大肠癌是全世界范围内最为常见的消化系统恶性肿瘤之一, 也是肿瘤相关死亡的重要原因. 2015年我国常见肿瘤的流行病调查显示, 当年我国大肠癌新发病例为376300例, 因大肠癌死亡的人数为19100例, 大肠癌已成为我国居民死亡的重要原因之一[1]. 随着环境和饮食习惯的改变, 我国结直肠癌(colorectal cancer, CRC)的发病率和死亡率分布具有以下特点: 男性高于女性, 城市高于农村, 发病率和死亡率均随年龄增长而上升. 大肠癌患者早期行根治术, 预后较好, 5年生存率较高, 而晚期已发生转移的患者预后较差. 因此, 探寻CRC侵袭、转移的分子机制及预后预测指标成为研究热点.
近年来大量文献报道, 长链非编码RNA(long-chain non-coding RNA, LncRNA)在剂量补偿效应、表观遗传调控、细胞周期调控、细胞分化、细胞凋亡衰老及肿瘤发生、发展调控方面发挥重要作用[2,3]. LncRNA MALAT1(metastasis associated lung adenocarcinoma transcript 1)是近年来研究的较多的肿瘤转移相关因子. 其在肺腺癌中高表达并与肺腺癌细胞侵袭转移能力有关. 而MALAT1在大肠癌中的表达情况及其临床意义鲜有报道.
MALAT1为近年来发现的LncRNA, 既往研究认为其与肿瘤的转移有关. 我们首先在BioGPS(http://biogps.org/#goto=welcome)数据库中分析人类正常肠上皮组织中MALAT1的表达情况; 然后在肿瘤基因表达芯片数据库Oncomie(https://http://www.oncomine.org/)中分析大肠癌组织与正常肠上皮是否存在MALAT1的差异表达(荟萃分析多个基因芯片数据); 同时根据高、低表达分为2组, 并计算MALAT1高表达与低表达组有无生存期差异, 数据来源与Kaplan-Meier Plotter(http://kmplot.com/analysis/); 最后应用蛋白相互作用数据库STRING(https://string-db.org/cgi/input.pl)预测MALAT1可能相互作用的蛋白及共表达情况.
1.2.1 BioGPS数据库: 在BioGPS数据库中输入MALAT1, 检索出人体大部分组织中MALAT1的相对表达量.
1.2.2 Oncomine数据库: Oncomie数据库检索MALAT1基因, 分析MALAT1基因在人体肿瘤与对应的正常组织中的相对表达情况. 设置条件为: (1)大肠癌(colorectal carcinoma/colon cancer/rectal cancer); (2)标本来源为手术切除标本; (3)组织对比为大肠癌组织与正常肠上皮组织; (4)数据类型为mRNA; (5)显著性P值<0.0001; (6)表达差异为基因上调或下调大于2倍; (7)基因排序为前10%.
1.2.2 Kaplan-Meier Plotter数据库: 在该数据总根据MALAT1在癌及正常组织中的总体中位表达水平分为高、低表达组. 应用生存曲线, 分析两组的总生存有无差异.
1.2.3 String 数据库蛋白相互作用分析: 在该数据库中, 对MALAT1蛋白相关的蛋白网络进行分析, 筛选条件为相关性大于0.9. 同时进行共表达分析, 绘制共表达和相关网络图.
统计学处理 数据分析采用STATA 10.0软件完成, 计量资料应用mean±SD表示, 应用t检验; 计数采用率表示, 应用chi-square检验; 生存分析首先绘制生存曲线, 并采用log-rank检验, P<0.05为存在统计学差异.
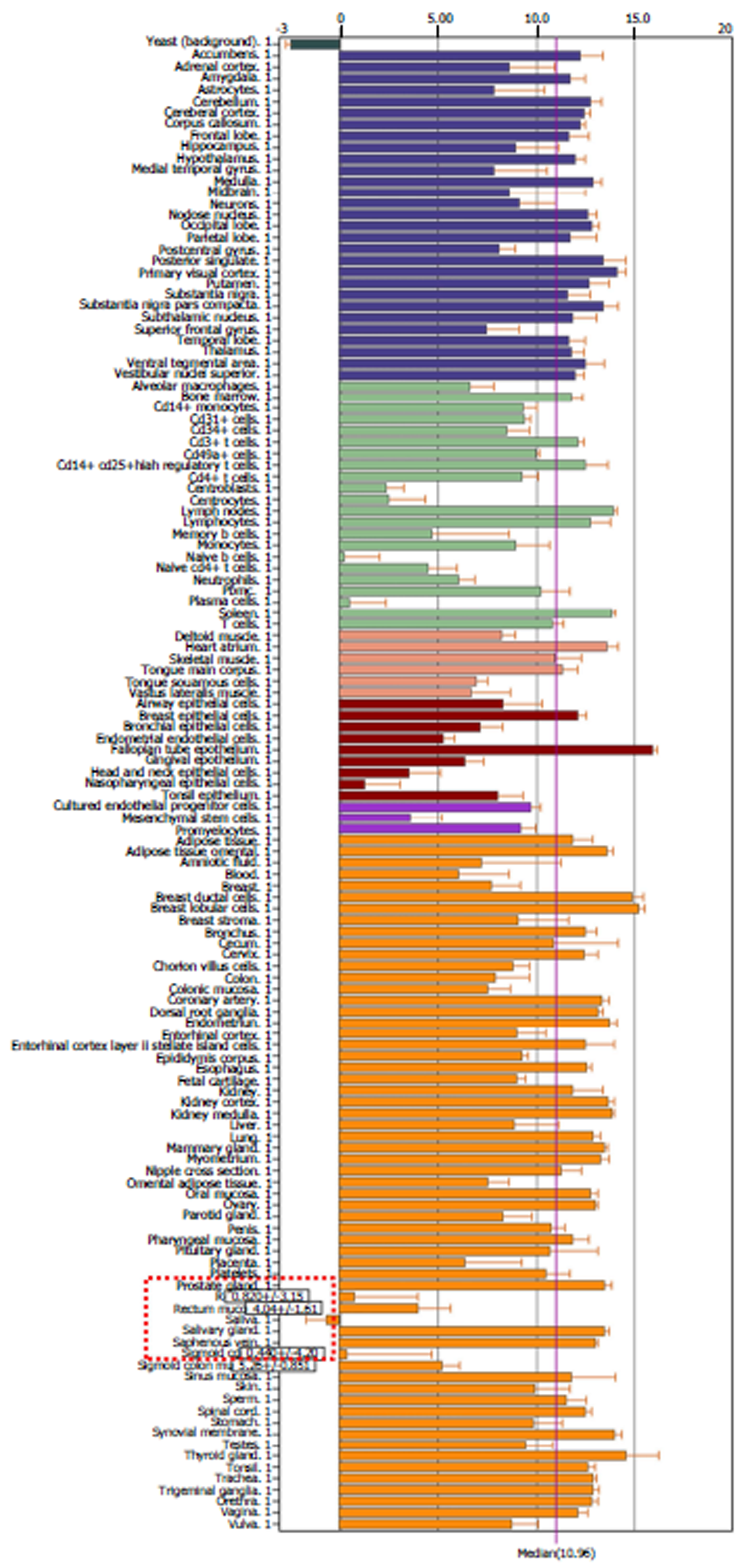
BioGPS数据库分析正常结直肠组织中MALAT1的相对表达情况. MALAT1在正常结直肠组织中的相对表达量较低(图1). Oncomine数据库中, LncRNA MALAT1基因mRNA肿瘤: 正常组织表达研究220项, 其中高表达13项, 低表达13项. 大肠癌中差异表达的研究8项, 其中6项在肠癌中高表达, 2项低表达(图2A和表1). 比较大肠癌与对应正常组织的相关芯片数据为18个(来自9项研究[4-12]), 整体分析显示, 大肠癌组织中MALAT1表达水平显著高于正常组织(P = 0.027)(图2B). 按结肠癌与直肠癌进亚组分析, 结肠癌组织与正常组织MALAT1表达水平无统计学差异(P = 0.149)(图2C). 而直肠癌组织中MALAT1表达水平明显高于对应正常组织(P = 1.04 E-5)(图2D).
| 分组 | 研究数(n) | 高表达数(n) | Median Rank | P值 |
| 结直肠肿瘤 | 18 | 5 | 6460.0 | 0.027 |
| 结肠肿瘤 | 12 | 1 | 8049.0 | 0.149 |
| 直肠肿瘤 | 6 | 4 | 563.0 | 1.04 E-5 |
在Kaplan-Meier Plotter数据平台中分析大肠癌患者MALAT1高低表达与患者生存期的关系, MALAT1高、低表达组总生存期分别为41.93 mo和52.2 mo, 差异无统计学意义(HR = 0.64, 95%CI: 0.29-1.39, P = 0.25)(图3).
lncRNA是一类长度大于200nt的非编码RNA转录本, 曾被认为是转录"噪音". 最初被认为由于其不具备开放阅读框ORF无法被进一步翻译成蛋白质而无生物学功能, 是转录过程中的副产品. 然而随着研究的深入, 越来越多的报道认为lncRNA可参与调控许多重要的生物学过程, 包括细胞分化、增殖、凋亡、多能干细胞重编程等[13]. 目前认为lncRNA的作用机制主要有: 转录并招募相关因子干扰蛋白质编码基因的表达; 通过组蛋白修饰等介导染色质重构, 从而影响下游基因的表达; 通过与mRNA形成互补双链对转录后的mRNA进行选择性剪接; 作为内源性siRNA发挥作用; 通过与相关蛋白相互作用, 调节其定位和/或其分子结构从而影响其活性[14-17].
MALAT1是较早发现的一类与转录本加工有关的lncRNA. 2003年Ji等[18]在对非小细胞肺癌患者的组织标本进行研究时发现, 在转移患者的组织中MALAT1表达水平明显高于非转移组织. 此后的功能学研究显示, MALAT1由DNA转录后的3'端加工而来, 其主要位于剪切斑点附近[19]. MALAT1可与丝氨酸/精氨酸(Ser/Arg)剪接因子间存在相互作用, 并还通过调控剪接因子在剪接斑点中的分布和磷酸化水平, 使mRNA前体的选择性剪接模式发生改变从而达到调控转录本剪接模式的分子生物学功能. MALAT1被认为是与肺癌转移相关的重要lncRNA. 在肺癌转移病灶中, MALAT1的表达水平显著增高, 因而推测其与肺癌的发生转移有关.
MALAT1在肺癌中的研究较多, 且大多数研究认为MALAT1高表达与肺癌的恶性生物学行为及预后不良有关[20-22]. 但MALAT1在肠癌中的表达情况及其与患者的预后关系报道并不多见, 且各研究的结论也并不完全一致. 杨孜欢等[23]人分析了LncRNA MALAT1表达与CRC侵袭和转移的关系, 结果认为MALAT1低表达与CRC侵袭和转移密切相关, 并且这一相关性是通过调控Rac1b的表达实现的. 而姚坤厚等[24]人的研究认为CRC中异常高表达的MALAT1可促进细胞迁移侵袭, 该功能可能是通过调节上皮-间充质转化实现的. 上述两个研究在MALAT1在肠癌中的表达水平方面得出了相反的结论. 因此, 在本研究中, 我们对多个数据库中关于MALAT1在肠癌中的表达情况及临床意义进行了深入挖掘, 以期明确MALAT1在肠癌中的真实表达情况. 研究结果认为, 大肠癌组织中MALAT1基因表达表达水平明显上调, 但MALAT1表达水平与患者预后并无相关性. MALAT1蛋白与TP53, SUZ12, CDK4, KDMA蛋白相互作用, 上述相互作用蛋白包括多梳蛋白家族, 周期蛋白依赖性蛋白激酶等, 与肿瘤的基因表达控制、转录调控及细胞分裂有关. 本研究为进一步分析MALAT1在肠癌中的表达、生物学功能及与其他基因相互作用的信号通路提供了依据. 同时也在一定程度上证实MALAT1作为大肠癌预后分子标志物的可行性不强.
长链非编码RNA(Long-chain non-coding RNA, LncRNA)在肿瘤中的作用为近年研究热点, MALAT1在结直肠癌(colorectal cancer, CRC)中的表达及其相关分子生物学功能不清. 其与患者预后关系仍存有争议. 因此, 我们采用生物信息学方法, 探讨MALAT1在CRC患者癌组织和癌旁组织中的差异表达、相关信号通路及与患者的关系.
本文通过分析基因芯片表达数据库中关于MALAT1表达与CRC的关系, 明确其在CRC患者组织中的表达、相关信号通路及与预后的相关性.
明确LncRNA在肠癌中的差异表达, MALAT1在肠癌发生中的信号通路相关蛋白及其在患者预后评价中的作用.
BioGPS数据库分析正常肠上皮中MALAT1的表达情况. 然后再Oncomine数据库中综合分析大肠癌组织与正常肠上皮组织MALAT1的差异表达, 并分析MALAT1高、低表达与患者生存期是否存在相关性. 应用STRING数据库中蛋白相互作用网络分析与MALAT1相互作用及共表达的相关信号通路蛋白.
大肠癌组织中MALAT1表达水平显著高于正常组织. MALAT1高、低表达组总生存期无统计学差异. MALAT1与TP53, SUZ12, CDK4, KDMA, 等具有相互作用关系而与EZH2, TP53, SRSF1等基因具有共表达关系.
CRC患者癌组织中MALAT1基因表达表达水平明显上调, 但与患者预后并无明显相关性. MALAT1与TP53, SUZ12, CDK4, KDMA蛋白相互作用并与细胞的转录调控及分裂有关.
MALAT1高低表达是否有肠癌患者预后相关仍存有争议, 需要高质量前瞻性研究进行证实. MALAT1具体调控肠癌细胞发生、发展和侵袭转移的分子机制有待相关分子生物学研究进一步验证.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): 0
E级 (差): 0
编辑: 崔丽君 电编:刘继红
| 1. | Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66:115-132. [PubMed] [DOI] |
| 2. | Deniz E, Erman B. Long noncoding RNA (lincRNA), a new paradigm in gene expression control. Funct Integr Genomics. 2017;17:135-143. [PubMed] [DOI] |
| 3. | Bink DI, Lozano-Vidal N, Boon RA. Long Non-Coding RNA in Vascular Disease and Aging. Noncoding RNA. 2019;5. [PubMed] [DOI] |
| 4. | Kaiser S, Park YK, Franklin JL, Halberg RB, Yu M, Jessen WJ, Freudenberg J, Chen X, Haigis K, Jegga AG, Kong S, Sakthivel B, Xu H, Reichling T, Azhar M, Boivin GP, Roberts RB, Bissahoyo AC, Gonzales F, Bloom GC, Eschrich S, Carter SL, Aronow JE, Kleimeyer J, Kleimeyer M, Ramaswamy V, Settle SH, Boone B, Levy S, Graff JM, Doetschman T, Groden J, Dove WF, Threadgill DW, Yeatman TJ, Coffey RJ, Aronow BJ. Transcriptional recapitulation and subversion of embryonic colon development by mouse colon tumor models and human colon cancer. Genome Biol. 2007;8:R131. [PubMed] [DOI] |
| 5. | National Cancer Institute. The Cancer Genome Atlas - Colon and Rectum Adenocarcinoma Gene Expression Data. Available from: http://tcga-data.nci.nih.gov/tcga/. |
| 6. | Zou TT, Selaru FM, Xu Y, Shustova V, Yin J, Mori Y, Shibata D, Sato F, Wang S, Olaru A, Deacu E, Liu TC, Abraham JM, Meltzer SJ. Application of cDNA microarrays to generate a molecular taxonomy capable of distinguishing between colon cancer and normal colon. Oncogene. 2002;21:4855-4862. [PubMed] [DOI] |
| 7. | Gaspar C, Cardoso J, Franken P, Molenaar L, Morreau H, Möslein G, Sampson J, Boer JM, de Menezes RX, Fodde R. Cross-species comparison of human and mouse intestinal polyps reveals conserved mechanisms in adenomatous polyposis coli (APC)-driven tumorigenesis. Am J Pathol. 2008;172:1363-1380. [PubMed] [DOI] |
| 8. | Graudens E, Boulanger V, Mollard C, Mariage-Samson R, Barlet X, Gémy G, Couillault C, Lajémi M, Piatier-Tonneau D, Zaborski P, Eveno E, Auffray C, Imbeaud S. Deciphering cellular states of innate tumor drug responses. Genome Biol. 2006;7:R19. [PubMed] [DOI] |
| 9. | Hong Y, Downey T, Eu KW, Koh PK, Cheah PY. A 'metastasis-prone' signature for early-stage mismatch-repair proficient sporadic colorectal cancer patients and its implications for possible therapeutics. Clin Exp Metastasis. 2010;27:83-90. [PubMed] [DOI] |
| 10. | Sabates-Bellver J, Van der Flier LG, de Palo M, Cattaneo E, Maake C, Rehrauer H, Laczko E, Kurowski MA, Bujnicki JM, Menigatti M, Luz J, Ranalli TV, Gomes V, Pastorelli A, Faggiani R, Anti M, Jiricny J, Clevers H, Marra G. Transcriptome profile of human colorectal adenomas. Mol Cancer Res. 2007;5:1263-1275. [PubMed] [DOI] |
| 11. | Gaedcke J, Grade M, Jung K, Camps J, Jo P, Emons G, Gehoff A, Sax U, Schirmer M, Becker H, Beissbarth T, Ried T, Ghadimi BM. Mutated KRAS results in overexpression of DUSP4, a MAP-kinase phosphatase, and SMYD3, a histone methyltransferase, in rectal carcinomas. Genes Chromosomes Cancer. 2010;49:1024-1034. [PubMed] [DOI] |
| 12. | Skrzypczak M, Goryca K, Rubel T, Paziewska A, Mikula M, Jarosz D, Pachlewski J, Oledzki J, Ostrowski J. Modeling oncogenic signaling in colon tumors by multidirectional analyses of microarray data directed for maximization of analytical reliability. PLoS One. 2010;5. [PubMed] [DOI] |
| 13. | Pan Y, Li C, Chen J, Zhang K, Chu X, Wang R, Chen L. The Emerging Roles of Long Noncoding RNA ROR (lincRNA-ROR) and its Possible Mechanisms in Human Cancers. Cell Physiol Biochem. 2016;40:219-229. [PubMed] [DOI] |
| 14. | Lu S, Su Z, Fu W, Cui Z, Jiang X, Tai S. Altered expression of long non-coding RNA GAS5 in digestive tumors. Biosci Rep. 2019;39. [PubMed] [DOI] |
| 15. | D'Angelo E, Agostini M. Long non-coding RNA and extracellular matrix: the hidden players in cancer-stroma cross-talk. Noncoding RNA Res. 2018;3:174-177. [PubMed] [DOI] |
| 16. | Li L, Wang Y, Zhang X, Huang Q, Diao Y, Yin H, Liu H. Long non-coding RNA HOXD-AS1 in cancer. Clin Chim Acta. 2018;487:197-201. [PubMed] [DOI] |
| 17. | Li S, Li J, Chen C, Zhang R, Wang K. Pan-cancer analysis of long non-coding RNA NEAT1 in various cancers. Genes Dis. 2017;5:27-35. [PubMed] [DOI] |
| 18. | Ji P, Diederichs S, Wang W, Böing S, Metzger R, Schneider PM, Tidow N, Brandt B, Buerger H, Bulk E, Thomas M, Berdel WE, Serve H, M¨¹ller-Tidow C. MALAT-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer. Oncogene. 2003;22:8031-8041. [PubMed] [DOI] |
| 19. | Wang J, Su L, Chen X, Li P, Cai Q, Yu B, Liu B, Wu W, Zhu Z. MALAT1 promotes cell proliferation in gastric cancer by recruiting SF2/ASF. Biomed Pharmacother. 2014;68:557-564. [PubMed] [DOI] |
| 20. | Guo F, Jiao F, Song Z, Li S, Liu B, Yang H, Zhou Q, Li Z. Regulation of MALAT1 expression by TDP43 controls the migration and invasion of non-small cell lung cancer cells in vitro. Biochem Biophys Res Commun. 2015;465:293-298. [PubMed] [DOI] |
| 21. | Guo F, Yu F, Wang J, Li Y, Li Y, Li Z, Zhou Q. Expression of MALAT1 in the peripheral whole blood of patients with lung cancer. Biomed Rep. 2015;3:309-312. [PubMed] [DOI] |
| 22. | Shen L, Chen L, Wang Y, Jiang X, Xia H, Zhuang Z. Long noncoding RNA MALAT1 promotes brain metastasis by inducing epithelial-mesenchymal transition in lung cancer. J Neurooncol. 2015;121:101-108. [PubMed] [DOI] |