修回日期: 2017-07-21
接受日期: 2017-08-01
在线出版日期: 2017-08-28
筛选先天性巨结肠(hirschsprung disease, HSCR)患者病变肠组织和正常肠组织的差异表达基因.
选取2例全结肠型HSCR、1例长段型HSCR患者, 从福尔马林固定后石蜡包埋样本中提取RNA, 采用去除核糖体的方法构建cDNA文库后, 应用RNA-Seq全面分析3对肠组织样本的转录组, 筛选出病变组织和正常组织的差异表达基因. 应用GO(gene ontology)方法分析差异表达基因的功能聚类, 应用KEGG(kyoto encyclopedia of genes and genomes)信号通路富集性方法分析差异表达基因相关的信号通路.
3对标本的总RNA提取质量合格, RNA-Seq测序数据质量合格, 测序数据与人类基因组参考基因对比匹配率合格, 样品间基因表达相关性合格. 3对病变组织和正常组织共有1382个差异表达基因, 包括899个表达一致上调的基因、483个表达一致下调的基因. 1382个差异表达基因被分为383个功能聚类. KEGG分析表明这些差异表达基因参与胆汁分泌、蛋白质消化与吸收、脂肪消化与吸收、维生素A代谢、类固醇激素生物合成、淀粉和蔗糖代谢等信号通路.
本研究首次利用RNA-Seq技术对先天性巨结肠病变组织和正常组织中的差异表达基因进行了全面分析, 可为推动疾病发病机制的研究提供新思路.
核心提要: 基于全转录组测序技术, 作者通过对先天性巨结肠患者病变组织和正常组织中的差异表达基因进行全面分析, 初步发现了几个重要的转录因子家族成员可能参与疾病发生.
引文著录: 姜茜, 李颀, 张震, 肖萍, 王慧, 李龙. 基于RNA-Seq技术的先天性巨结肠转录组学. 世界华人消化杂志 2017; 25(24): 2200-2208
Revised: July 21, 2017
Accepted: August 1, 2017
Published online: August 28, 2017
To screen the differentially expressed genes (DEGs) in aganglionic and ganglionated colon tissues of hirschsprung disease (HSCR) patients.
RNA was extracted from paired-colon samples (formalin-fixed, paraffin-embedded sections) of three HSCR patients (two affected by total colonic aganglionosis and one by long-segment HSCR) and purified. Ribosomal RNA was eliminated before generating the cDNA library. After RNA-sequencing, the gene ontology (GO) analysis was performed to uncover the biological functions of DEGs. The kyoto encyclopedia of genes and genomes (KEGG) pathway enrichment analysis was applied to find out the related signal pathways.
There were a total of 1382 significantly commom DEGs in the three paired samples, including 899 consistently up-regulated DEGs and 483 consistently down-regulated DEGs. The 1382 DEGs were categorized into 383 functional categories by GO analysis. The KEGG pathway analysis suggested 25 significant signal pathways, including bile secretion, protein digestion and absorption, fat digestion and absorption, retinol metabolism, steroid hormone biosynthesis, and starch and sucrose metabolism.
In the current study, the DEGs of aganglionic and ganglionated colon tissues were thoroughly analyzed for the first time, which will shed new light on the pathogenesis of HSCR.
- Citation: Jiang Q, Li Q, Zhang Z, Xiao P, Wang H, Li L. Screening of differentially expressed genes in Hirschsprung disease based on RNA-Seq technology. Shijie Huaren Xiaohua Zazhi 2017; 25(24): 2200-2208
- URL: https://www.wjgnet.com/1009-3079/full/v25/i24/2200.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i24.2200
先天性巨结肠(hirschsprung disease, HSCR)又称肠无神经节细胞症, 是由胚胎期肠神经嵴细胞迁移障碍导致的先天性消化道畸形[1-3]. 活产儿发病率平均1/5000, 遗传因素占主导地位且发病机制复杂[4,5]. 临床首诊患儿中相当一部分是月龄<3 mo的新生儿或小婴儿, 其临床表现多样、诊断难度大, 因腹膜炎、感染中毒性休克、巨结肠危象、严重出血、小肠结肠炎等各种情况死亡者多. 虽然近年来随着外科治疗手段的不断改进使得本病死亡率有所降低, 但仍有很多患儿在治疗后出现诸如反复肠炎、粪污、持续性难治性便秘等一系列并发症, 造成进一步处理困难甚至造成患儿难以矫治的心理问题, 严重影响其生长发育和生活质量, 给社会和家庭都带来了沉重的经济负担. 我国平均每年新增患者3500例, 随着"全面两孩"政策的正式实施和高龄产妇的逐年增加, 这一患病人群仍会以较高的速度不断扩增, 是名副其实的不能被忽视的罕见病. 全转录组测序技术(RNA sequencing, RNA-Seq)是转录组测序的新一代技术, 已成为多物种转录组研究中不可或缺的一个强大平台[6-9]. 近年来, RNA-Seq技术已被广泛应用于多种人类复杂疾病、恶性肿瘤、出生缺陷等疾病的研究, 无论对于生物标志物的寻找还是对于探寻新的发病机制、制定个体化治疗方案、精准修正干预措施都有重要意义[10-13].
3例HSCR患者病变段肠组织(免疫组织化学染色确定无神经节细胞)及正常段肠组织(免疫组织化学染色确定有神经节细胞)石蜡包埋样本为首都儿科研究所附属儿童医院普通外科及病理科手术切除后制作、保存. 其中2例全结肠型HSCR患者(编号: H0116, 男性, 11 mo; 编号: H0127, 女性, 3 mo), 1例长段型HSCR患者(编号: H0129, 女性, 10 mo). 石蜡包埋组织切片总RNA提取试剂盒(货号: DP439, 天根生化科技北京有限公司, 中国); Ribo-zero™ rRNA Removal Kit (Epicentre, 麦迪逊, 威斯康星州, 美国); NEBNext® Ultra™ Directional RNA Library Prep Kit(NEB, 伊普斯威奇, 马萨诸塞州, 美国).
1.2.1 高通量测序: RNA测序由北京诺禾致源生物信息科技有限公司完成, 主要步骤包括: 3对肠组织样本各切取15片, 提取总RNA; 对样品进行质量检测后去除核糖体, 纯化、沉淀后进行文库构建. 质检合格的cDNA文库进行HiSeq上机测序.
1.2.2 生物信息学分析: 由于福尔马林固定过程会对样品的DNA和RNA产生明显破坏作用, 故在正式筛选差异表达基因之前, 我们首先对测序原始数据进行了质量评估, 包括: 测序错误率分布检查(Phred分值20对应的碱基正确识别率为99%, Phred分值30对应的碱基正确识别率为99.9%)、GC含量分布检查、测序数据过滤(去掉带接头的原始序列以及低质量的原始序列). 其中raw reads为原始数据中的reads数; clean reads为原始数据过滤后的reads数, 此系列数据是后续生物信息学分析的基础; clean bases为原始数据过滤后的碱基数; 错误率是通过公式Qphred = -10log10(e)计算得到的; GC含量显示clean reads中G与C占4种碱基的百分比. 然后我们应用STAR软件对RNA-Seq测序数据进行比对分析. Total reads为用于比对分析的clean reads数; total mapped reads为比对到基因组上的reads数; uniquely mapped reads为比对到基因组单一位置的reads数; multiple mapped reads为比对到基因组多个位置的reads数; total mapping rate为比对到基因组上的reads占总clean reads数的百分比; uniquely mapping rate为比对到基因组单一位置的reads数占总clean reads数的百分比; multiple mapping rate为比对到基因组多个位置的reads数占总clean reads数的百分比. 正常情况下测序产生的序列能够定位到参考基因组的百分比应高于70%, 其中具有多个比对位置的序列占总体的百分比通常不超过10%. 使用HTSeq软件进行基因水平的定量分析, 并采用FKPM(fragments per kilo bases per million reads, 来自某一基因每千碱基长度的测序片段数目, 是目前最常用的基因表达水平估算方法)估算样本中的基因表达量. 计算组内及组间样本的相关性系数绘制成热图(Pearson相关系数图)以直观显示组间样本差异及组内样本重复情况.
1.2.3 差异表达基因分析: 分别对HSCR患者病变段肠组织及正常段肠组织的基因表达水平进行对比, 最终找到共同的差异表达基因.
统计学处理 利用edgeR软件进行基因表达差异的显著性分析, 将Padj(多重假设检验校正后的P值)<0.05、Foldchange(倍数改变)绝对值>2作为差异显著性标准.
如表1所示, 所有样本的Q20在96.59%-97.31%之间, Q30在90.78%-93.14%之间(Q20、Q30分别显示Phred数值>20、30的碱基占总体碱基的百分比), GC含量分布的差异不大.
| 样品名称 | Raw reads | Clean reads | Clean bases | 错误率(%) | Q20(%) | Q30(%) | GC含量(%) |
| H0129_BB | 30731849 | 26953378 | 8.09G | 0.02 | 97.13 | 92.87 | 46.91 |
| H0127_BB | 29919105 | 27594122 | 8.28G | 0.02 | 97.31 | 93.14 | 46.83 |
| H0116_BB | 38469223 | 31466683 | 9.44G | 0.02 | 97.24 | 93.01 | 43.31 |
| H0129_ZC | 35545978 | 31402169 | 9.42G | 0.02 | 96.98 | 92.50 | 46.71 |
| H0127_ZC | 37290696 | 32446763 | 9.73G | 0.02 | 96.97 | 92.52 | 48.29 |
| H0116_ZC | 27778974 | 26742224 | 8.02G | 0.02 | 96.59 | 90.78 | 46.40 |
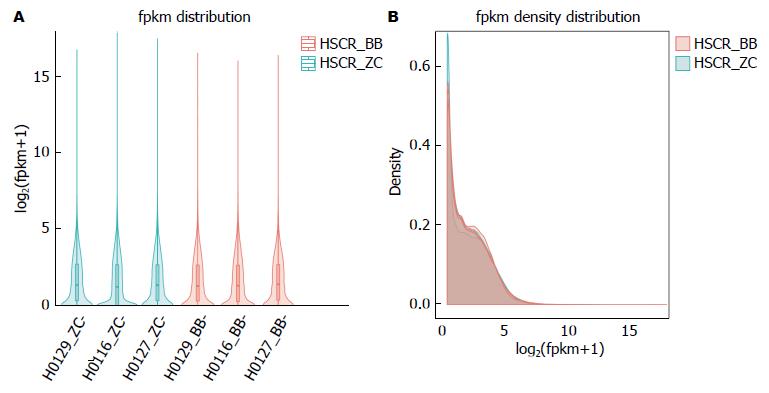
全部3对肠组织样本测序数据与人类基因组参考序列比对情况如表2. 本研究所有样本的total mapping rate为73.86%-89.89%, multiple mapping rate为2.27%-5.43%. 计算各样本所有基因或转录本的表达值(FPKM)后, 通过绘制分布图和密度重叠图来展示和比较不同样本间基因表达水平的分布情况. 如图1所示, 本研究所有样本的总体基因表达水平相当, 无明显差异.
| 样品名称 | H0129_BB | H0127_BB | H0116_BB | H0129_ZC | H0127_ZC | H0116_ZC |
| Total reads | 26953378 | 27594122 | 31466683 | 31402169 | 32446763 | 23766915 |
| Total mapped reads | 20869303 | 22462832 | 24245772 | 24920590 | 23963792 | 21364363 |
| Uniquely mapped reads | 20174846 | 21835711 | 23422760 | 24129028 | 23065295 | 20073384 |
| Multiple mapped reads | 694457 | 627121 | 823012 | 791562 | 898497 | 1290979 |
| Total mapping rate | 77.43% | 81.40% | 77.06% | 79.36% | 73.86% | 89.89% |
| Uniquely mapping rate | 74.85% | 79.13% | 74.44% | 76.84% | 71.09% | 84.46% |
| Multiple mapping rate | 2.58% | 2.27% | 2.62% | 2.52% | 2.77% | 5.43% |
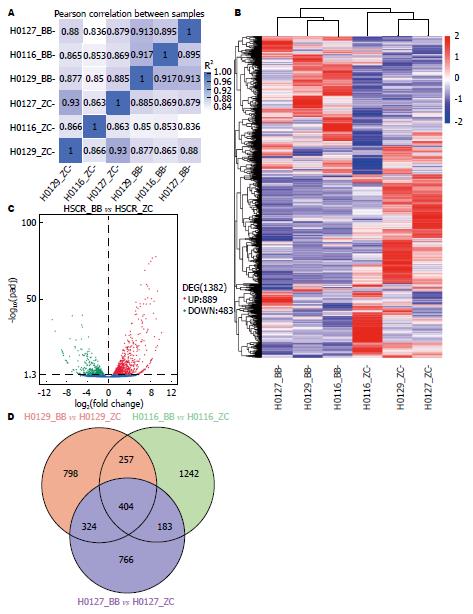
在3对先天性巨结肠患者肠组织中, 样品间基因表达相关系数均>0.8(图2A). 所有差异表达基因层次聚类热图分析表明3个病变段肠组织中基因表达变化模式类似, 3个正常段肠组织中基因表达变化模式类似(图2B). 火山图分析表明病变段肠组织与正常段肠组织间有统计学意义的差异表达基因的分布情况. 与相应的病变段肠组织相比, HSCR患者正常段肠组织的差异表达基因有1382个, 其中上调899个、下调483个(图2C). 从差异基因韦恩图中可以看出每对样本各自以及他们之间重叠的差异表达基因个数, 3例患者的肠组织中共检出404个共同差异表达基因(图2D). 按照生物学功能对表达差异存在显著性的基因集进行分类注释后, 我们发现, 很多基因都属于转录因子家族成员, 如: 同源异型盒基因家族, 锌指超家族, helix-loop-helix家族以及细胞核激素受体家族等. 与正常段肠组织相比, 患者病变段肠组织中表达量显著下降的基因主要包括: SLC10A2、SLC5A12、DPEP1、APOA1、SLC6A19、SLC2A2、MEP1B, MS4A10, CYP3A4及CCL25等(P值介于7.28E-83和1.02E-59之间), 最多的相差676倍, 最少的也相差74倍之多. GO分析表明1382个差异表达基因归属于383个功能聚类. KEGG分析表明这些差异表达基因主要富集到25个信号通路上(表3), 参与胆汁分泌、蛋白质消化与吸收、脂肪消化与吸收、维生素A代谢、类固醇激素生物合成、淀粉和蔗糖代谢等生物学过程.
| 序号 | 通路ID | 通路 | P值 | 校正P值 | 基因数 |
| 1 | hsa04976 | Bile secretion | 1.7E-09 | 2.8E-07 | 22 |
| 2 | hsa04974 | Protein digestion and absorption | 3.0E-09 | 2.8E-07 | 23 |
| 3 | hsa04975 | Fat digestion and absorption | 3.6E-08 | 2.2E-06 | 16 |
| 4 | hsa00830 | Retinol metabolism | 2.1E-07 | 9.8E-06 | 17 |
| 5 | hsa04977 | Vitamin digestion and absorption | 3.2E-07 | 1.2E-05 | 11 |
| 6 | hsa01100 | Metabolic pathways | 4.1E-07 | 1.3E-05 | 121 |
| 7 | hsa00980 | Metabolism of xenobiotics by cytochrome P450 | 1.4E-06 | 3.7E-05 | 17 |
| 8 | hsa00982 | Drug metabolism - cytochrome P450 | 2.3E-06 | 5.3E-05 | 17 |
| 9 | hsa00140 | Steroid hormone biosynthesis | 4.1E-06 | 8.6E-05 | 14 |
| 10 | hsa00500 | Starch and sucrose metabolism | 1.0E-05 | 1.9E-04 | 13 |
| 11 | hsa04973 | Carbohydrate digestion and absorption | 1.4E-05 | 2.4E-04 | 12 |
| 12 | hsa00591 | Linoleic acid metabolism | 3.4E-05 | 5.4E-04 | 10 |
| 13 | hsa03320 | PPAR signaling pathway | 9.6E-05 | 1.4E-03 | 15 |
| 14 | hsa00330 | Arginine and proline metabolism | 1.1E-04 | 1.5E-03 | 13 |
| 15 | hsa00590 | Arachidonic acid metabolism | 2.0E-04 | 2.5E-03 | 13 |
| 16 | hsa04614 | Renin-angiotensin system | 6.6E-04 | 7.8E-03 | 6 |
| 17 | hsa02010 | ABC transporters | 9.1E-04 | 1.0E-02 | 10 |
| 18 | hsa00910 | Nitrogen metabolism | 9.9E-04 | 1.0E-02 | 7 |
| 19 | hsa00100 | Steroid biosynthesis | 1.9E-03 | 1.8E-02 | 6 |
| 20 | hsa00380 | Tryptophan metabolism | 2.5E-03 | 2.4E-02 | 9 |
| 21 | hsa00051 | Fructose and mannose metabolism | 4.0E-03 | 3.6E-02 | 8 |
| 22 | hsa00340 | Histidine metabolism | 4.3E-03 | 3.7E-02 | 7 |
| 23 | hsa04964 | Proximal tubule bicarbonate reclamation | 5.4E-03 | 4.4E-02 | 6 |
| 24 | hsa04020 | Calcium signaling pathway | 6.1E-03 | 4.8E-02 | 22 |
| 25 | hsa00360 | Phenylalanine metabolism | 6.3E-03 | 4.8E-02 | 5 |
HSCR是一种与遗传有关的多基因、多因素参与的复杂疾病, 可散发, 也可呈家族性遗传. 目前已经鉴定的14个HSCR相关易感基因主要来自于肠神经系统(enteric nervous system, ENS)发育中起主要作用的2个信号通路(RET、EDNRB), 以及可调控、影响这些基因表达的转录因子的编码基因和其他几个在神经系统发育中已明确具有重要作用的基因[14-17]. 近年来通过连锁分析、全基因组关联分析、基因敲除以及动物模型研究发现并确定了这些基因携带的某些高外显率、罕见致病突变以及低外显率、常见单核苷酸多态性, 可以解释部分表型与基因型之间的关联. 尽管如此, 在所有已知易感基因累积发现的突变仅能解释不足15%的患者的发病原因, 高度提示其他致病基因或危险因子的存在[18].
HSCR属于典型的肠神经系统发育异常性疾病. ENS是外周神经系统的重要组成部分, 由数目众多的神经元、胶质细胞及纵横交织的神经纤维组成复杂的网络, 能够独立于脑自主调节肠道的分泌、运动、吸收、血运、免疫等功能. 由于含有与中枢神经系统相似的细胞类型及神经递质, 表达的基因也大部分重合, 且发育关键期大多数神经生长因子的时-空特异性表达模式及其调控途径也极其相似, ENS又被形象地称为"第二个大脑". 组成ENS的神经细胞均来源于神经嵴细胞, 后者在胚胎期从神经轴的两个不同水平进入肠道[称为肠神经嵴细胞(enteric neural crest cell, ENCC)], 在肠间充质中迁移、增殖、定居和分化, 最终聚集成规律排列的神经节并发出突起相互联系, 形成两个重要的神经丛, 完成信号传递以及靶细胞的支配. 这一发育过程具有严格的时间窗, 不仅取决于ENCC的内部特质, 也受到肠道微环境中多种外部信号分子的引导和影响, 缺一不可[19,20]. 本研究通过对比3例HSCR患者病变段与正常段肠组织标本中的基因表达水平, 发现了多个转录因子家族编码基因可能参与疾病发生. 其中, 同源异型盒基因(homeobox genes, Hox genes)编码一个高度保守的转录因子家族, 在决定胚胎期细胞的定向分化、增殖以及调控机体组织器官的发育过程中都发挥关键作用. 人类含有39个HOX基因, 形成4个基因簇(HOXA-HOXD), 分布在4条染色体上[21-23]. 我们在HSCR患者病变肠组织中发现的异常表达基因包括HOXA11、HOXC8、HOXA13、HOXB6、HOXD9、HOXD10、HOXD11、HOXD13、HOXB13、HOXD12、HOXC5、HOXC6等. 锌指转录因子广泛参与调控胚胎发育和肿瘤发生过程. 不同的家族成员已显示在许多信号的级联放大过程中起作用, 包括左右体轴识别、附肢形成、神经分化和细胞命运决定等[24-26]. 此外, helix-loop-helix家族成员和细胞核激素受体家族也都在胚胎发育期具有重要的调控作用[27-29].
全部1382个差异表达基因富集于383个功能聚类、25条信号通路, 主要参与胆汁分泌、蛋白质消化与吸收、脂肪消化与吸收、维生素A代谢、类固醇激素生物合成、淀粉和蔗糖代谢等生物学过程. 蛋白质、碳水化合物、脂类是机体能量代谢的重要底物, 其稳态与平衡对于胚胎早期发育的正常进行至关重要. 瑞典研究人员2016年进行的一项回顾性病例-对照研究结果显示, 肥胖女性(或至少孕期体质量指数超标)后代罹患HSCR的风险可显著增加[30]. 鉴于此, 我们推测, 无论是由于母体物质代谢紊乱还是个体基因突变等原因导致的胚胎发育早期能量代谢失常都有可能干扰ENS的正常发育, 并最终引起HSCR的发生.
总之, 本研究运用新的RNA-Seq技术对先天性巨结肠症差异表达基因进行了筛选, 初步定位出新的候选基因和相关分子信号通路. 下一步我们将对这些候选基因进行表达验证及体内、外功能验证, 同时增加筛选标本量, 以期通过推动疾病关键基因的定位来揭示HSCR发生发展的新机制及精准防治手段.
先天性巨结肠(hirschsprung disease, HSCR)是由胚胎期肠神经嵴细胞迁移障碍导致的先天性消化道畸形, 病因复杂, 遗传因素占主导地位. 全转录组测序技术近年来已被广泛应用于多种人类复杂疾病的研究, 无论对于生物标志物的寻找还是对于探寻新的发病机制、制定个体化治疗方案都有重要意义.
RNA-Seq技术为很多出生缺陷疾病的研究提供了强大的平台. 本研究首次将该技术应用于先天性巨结肠, 将差异基因表达与生物信息学分析相结合, 有可能为新的致病基因寻找提供新思路.
瑞典研究人员发现肥胖女性(或孕期体质量指数超标)后代罹患先天性巨结肠的风险可显著增加. 基于本文的研究结果, 大家推测: 无论是由于母体物质代谢紊乱还是个体基因突变等原因导致的胚胎发育早期能量代谢失常都有可能干扰肠神经系统的正常发育, 并最终引起疾病发生.
本文基于RNA-Seq技术, 同时运用差异基因表达和多种生物信息学分析策略对先天性巨结肠进行研究, 通过功能聚类和信号通路富集等方法筛选可能的新的致病基因单元. 目前国内外尚未见与本文报道相似的研究路线及结果.
本文发现先天性巨结肠患者病变肠组织中的很多异常表达基因都属于转录因子家族成员, 如: 同源异型盒基因家族, 锌指超家族, helix-loop-helix家族以及细胞核激素受体家族等. 这些基因很可能参与了肠神经系统的正常发育调控及病理状态下症状的产生.
转录组测序的研究对象: 特定细胞或组织在某一功能状态下所能转录出来的所有RNA的总和, 主要包括: mRNA、small RNA和non-coding RNA等. 转录组研究能够全面、快速地获得某一物种特定组织或器官在某一状态下的几乎所有转录本的序列信息, 已广泛应用于基础研究、临床诊断和药物研发等领域.
冯杰雄, 教授, 主任医师, 华中科技大学同济医学院附属同济医院小儿外科; 施诚仁, 教授, 上海交通大学医学院附属新华医院小儿外科
本文采用RNA-Seq技术对先天性巨结肠症差异表达基因进行筛选, 以初步定位出新的候选基因和相关分子信号通路, 对进一步研究有一定意义.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 北京市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B
C级 (良好): C
D级 (一般): 0
E级 (差): 0
编辑: 闫晋利 电编:杜冉冉
| 1. | Schriemer D, Sribudiani Y, IJpma A, Natarajan D, MacKenzie KC, Metzger M, Binder E, Burns AJ, Thapar N, Hofstra RM. Regulators of gene expression in Enteric Neural Crest Cells are putative Hirschsprung disease genes. Dev Biol. 2016;416:255-265. [PubMed] [DOI] |
| 2. | Moore SW. Advances in understanding functional variations in the Hirschsprung disease spectrum (variant Hirschsprung disease). Pediatr Surg Int. 2017;33:285-298. [PubMed] [DOI] |
| 3. | Bondurand N, Southard-Smith EM. Mouse models of Hirschsprung disease and other developmental disorders of the enteric nervous system: Old and new players. Dev Biol. 2016;417:139-157. [PubMed] [DOI] |
| 4. | Gui H, Tang WK, So MT, Proitsi P, Sham PC, Tam PK, Ngan ES, Cherny SS, Garcia-Barceló MM. RET and NRG1 interplay in Hirschsprung disease. Hum Genet. 2013;132:591-600. [PubMed] [DOI] |
| 5. | Amiel J, Sproat-Emison E, Garcia-Barcelo M, Lantieri F, Burzynski G, Borrego S, Pelet A, Arnold S, Miao X, Griseri P. Hirschsprung disease, associated syndromes and genetics: a review. J Med Genet. 2008;45:1-14. [PubMed] [DOI] |
| 6. | Wu H, Wei L, Fan F, Ji S, Zhang S, Geng J, Hong L, Fan X, Chen Q, Tian J. Integration of Hippo signalling and the unfolded protein response to restrain liver overgrowth and tumorigenesis. Nat Commun. 2015;6:6239. [PubMed] [DOI] |
| 7. | Duan Q, Xiao Y, Zhu L, Liu Z, Mao X, Zhou Z, Liao C, Cai J, Huang F, Liu Z. BET bromodomain is a novel regulator of TAZ and its activity. Biochim Biophys Acta. 2016;1859:1527-1537. [PubMed] [DOI] |
| 8. | Wang H, Diao D, Shi Z, Zhu X, Gao Y, Gao S, Liu X, Wu Y, Rudolph KL, Liu G. SIRT6 Controls Hematopoietic Stem Cell Homeostasis through Epigenetic Regulation of Wnt Signaling. Cell Stem Cell. 2016;18:495-507. [PubMed] [DOI] |
| 9. | Li A, Liu D, Wu J, Zhao X, Hao M, Geng S, Yan J, Jiang X, Zhang L, Wu J. mRNA and Small RNA Transcriptomes Reveal Insights into Dynamic Homoeolog Regulation of Allopolyploid Heterosis in Nascent Hexaploid Wheat. Plant Cell. 2014;26:1878-1900. [PubMed] [DOI] |
| 10. | Bao ZS, Chen HM, Yang MY, Zhang CB, Yu K, Ye WL, Hu BQ, Yan W, Zhang W, Akers J. RNA-seq of 272 gliomas revealed a novel, recurrent PTPRZ1-MET fusion transcript in secondary glioblastomas. Genome Res. 2014;24:1765-1773. [PubMed] [DOI] |
| 11. | Lai FP, Lau ST, Wong JK, Gui H, Wang RX, Zhou T, Lai WH, Tse HF, Tam PK, Garcia-Barcelo MM. Correction of Hirschsprung-Associated Mutations in Human Induced Pluripotent Stem Cells Via Clustered Regularly Interspaced Short Palindromic Repeats/Cas9, Restores Neural Crest Cell Function. Gastroenterology. 2017;153:139-153.e8. [PubMed] [DOI] |
| 12. | Zhao D, Mokhtari R, Pedrosa E, Birnbaum R, Zheng D, Lachman HM. Transcriptome analysis of microglia in a mouse model of Rett syndrome: differential expression of genes associated with microglia/macrophage activation and cellular stress. Mol Autism. 2017;8:17. [PubMed] [DOI] |
| 13. | Jensen VL, Carter S, Sanders AA, Li C, Kennedy J, Timbers TA, Cai J, Scheidel N, Kennedy BN, Morin RD. Whole-Organism Developmental Expression Profiling Identifies RAB-28 as a Novel Ciliary GTPase Associated with the BBSome and Intraflagellar Transport. PLoS Genet. 2016;12:e1006469. [PubMed] [DOI] |
| 14. | Yang D, Yang J, Li S, Jiang M, Cao G, Yang L, Zhang X, Zhou Y, Li K, Tang ST. Effects of RET, NRG1 and NRG3 Polymorphisms in a Chinese Population with Hirschsprung Disease. Sci Rep. 2017;7:43222. [PubMed] [DOI] |
| 15. | Chatterjee S, Kapoor A, Akiyama JA, Auer DR, Lee D, Gabriel S, Berrios C, Pennacchio LA, Chakravarti A. Enhancer Variants Synergistically Drive Dysfunction of a Gene Regulatory Network In Hirschsprung Disease. Cell. 2016;167:355-368.e10. [PubMed] [DOI] |
| 16. | Gui H, Schriemer D, Cheng WW, Chauhan RK, Antiňolo G, Berrios C, Bleda M, Brooks AS, Brouwer RW, Burns AJ. Whole exome sequencing coupled with unbiased functional analysis reveals new Hirschsprung disease genes. Genome Biol. 2017;18:48. [PubMed] [DOI] |
| 17. | Jiang Q, Arnold S, Heanue T, Kilambi KP, Doan B, Kapoor A, Ling AY, Sosa MX, Guy M, Jiang Q. Functional loss of semaphorin 3C and/or semaphorin 3D and their epistatic interaction with ret are critical to Hirschsprung disease liability. Am J Hum Genet. 2015;96:581-596. [PubMed] [DOI] |
| 18. | Eichler EE, Flint J, Gibson G, Kong A, Leal SM, Moore JH, Nadeau JH. Missing heritability and strategies for finding the underlying causes of complex disease. Nat Rev Genet. 2010;11:446-450. [PubMed] [DOI] |
| 19. | Sasselli V, Pachnis V, Burns AJ. The enteric nervous system. Dev Biol. 2012;366:64-73. [PubMed] [DOI] |
| 20. | Gershon MD, Tennyson VM. Microenvironmental factors in the normal and abnormal development of the enteric nervous system. Prog Clin Biol Res. 1991;373:257-276. [PubMed] |
| 21. | Garcia-Fernàndez J. The genesis and evolution of homeobox gene clusters. Nat Rev Genet. 2005;6:881-892. [PubMed] [DOI] |
| 22. | Bhatlekar S, Fields JZ, Boman BM. HOX genes and their role in the development of human cancers. J Mol Med (Berl). 2014;92:811-823. [PubMed] [DOI] |
| 23. | Novakovic B, Fournier T, Harris LK, James J, Roberts CT, Yong HEJ, Kalionis B, Evain-Brion D, Ebeling PR, Wallace EM. Increased methylation and decreased expression of homeobox genes TLX1, HOXA10 and DLX5 in human placenta are associated with trophoblast differentiation. Sci Rep. 2017;7:4523. [PubMed] [DOI] |
| 24. | Nieto MA. The snail superfamily of zinc-finger transcription factors. Nat Rev Mol Cell Biol. 2002;3:155-166. [PubMed] [DOI] |
| 25. | Chong CE, Venugopal P, Stokes PH, Lee YK, Brautigan PJ, Yeung DTO, Babic M, Engler GA, Lane SW, Klingler-Hoffmann M. Differential effects on gene transcription and hematopoietic differentiation correlate with GATA2 mutant disease phenotypes. Leukemia. 2017; Jun 23. [Epub ahead of print]. [PubMed] [DOI] |
| 26. | Wlodarski MW, Collin M, Horwitz MS. GATA2 deficiency and related myeloid neoplasms. Semin Hematol. 2017;54:81-86. [PubMed] [DOI] |
| 27. | Kömüves LG, Hanley K, Jiang Y, Elias PM, Williams ML, Feingold KR. Ligands and activators of nuclear hormone receptors regulate epidermal differentiation during fetal rat skin development. J Invest Dermatol. 1998;111:429-433. [PubMed] [DOI] |
| 28. | Rossi M, Spichty M, Attorri L, Distante C, Nervi C, Salvati S, Vitelli L. Eicosapentaenoic acid modulates the synergistic action of CREB1 and ID/E2A family members in the rat pup brain and mouse embryonic stem cells. Biochim Biophys Acta. 2017;1860:870-884. [PubMed] [DOI] |
| 29. | Zhang Q, Zagozewski J, Cheng S, Dixit R, Zhang S, de Melo J, Mu X, Klein WH, Brown NL, Wigle JT. Regulation of Brn3b by DLX1 and DLX2 is required for retinal ganglion cell differentiation in the vertebrate retina. Development. 2017;144:1698-1711. [PubMed] [DOI] |
| 30. | Löf Granström A, Svenningsson A, Hagel E, Oddsberg J, Nordenskjöld A, Wester T. Maternal Risk Factors and Perinatal Characteristics for Hirschsprung Disease. Pediatrics. 2016;138:e20154608. [PubMed] [DOI] |