修回日期: 2017-02-15
接受日期: 2017-02-27
在线出版日期: 2017-04-08
利用IncuCyte ZOOM系统筛选肿瘤细胞单克隆和克隆形成能力检测.
采用差速消化与差速贴壁分离培养原代肿瘤细胞; 在有限稀释法基础上, 采用IncuCyte ZOOM系统的实时动态追踪和全孔成像技术对肿瘤细胞进行单克隆筛选及克隆形成能力分析.
从30例肿瘤组织中培养6株原代肿瘤细胞(TJ3ZX-02至07), 进一步从5株原代肿瘤细胞(TJ3ZX-03至07)中成功筛选出89个持续增殖的单克隆, 其中67个克隆扩增培养并冻存; 并且通过时序图准确有效地排除了18个不能持续增殖的单克隆和28个由2个及以上细胞组成的多克隆. 2例单克隆株(TJ3ZX-06-B11, TJ3ZX-07-H11)克隆形成能力分析表明, 14 d时平皿方法的克隆形成率(35.17%, 13.17%)高于IncuCyte ZOOM 96孔全孔成像(23.13%, 5.51%), 且差异有统计学意义(P<0.05); 延长至21 d时全孔成像的克隆形成率为(35.63%, 13.22%), 与平皿方法比较无统计学差异.
IncuCyte ZOOM系统能够简便、准确、省时省力地实现单克隆细胞筛选和克隆形成率检测.
核心提要: 利用IncuCyte ZOOM系统进行原代肿瘤细胞的单克隆筛选和克隆形成力检测, 该方法使克隆筛选更加简便、准确, 时序性图像可判断细胞团来源等诸多优点, 为更好的研究肿瘤细胞异质性等生物学特性提供强有力的技术手段.
引文著录: 刘长政, 焦晓磊, 高敦芹, 邢龙彬, 刘辉, 骆莹, 高英堂. 实时动态活细胞成像系统在单克隆筛选和克隆形成能力检测中的应用. 世界华人消化杂志 2017; 25(10): 881-890
Revised: February 15, 2017
Accepted: February 27, 2017
Published online: April 8, 2017
To screen single tumor cell clones and evaluating their colony-forming ability by IncuCyte ZOOM.
Primary tumor cells were isolated by differential digestion and differential adherence method. On the basis of limited dilution, dynamic real-time tracking technology and full aperture imaging technology were used to track single cell clones and evaluate their colony-formation ability.
Six lines of primary tumor cells (TJ3ZX-02 to 07) were isolated from 30 tumor tissues, and 89 persistently proliferative tumor cell clones were screened from five primary tumor cell lines (TJ3ZX-03 to 07), of which 67 were expanded and cryopreserved. Eighteen monoclonal cell lines were excluded due to the lack of expansion ability, and 28 polyclonal cell lines were excluded because of consisting of two or more cell types as revealed by the Sequence Diagram. The analysis of clone-forming ability of two monoclonal cell strains (TJ3ZX-06-B11, TJ3ZX-07-H11) showed that the clone-forming rates for the plate method (35.17%, 13.17%) were significantly higher than those for IncuCyte ZOOM (23.13%, 5.51%) at 14 d (P < 0.05), although there was no significant difference at 21 d (35.63% and 13.22% for IncuCyte ZOOM).
IncuCyte ZOOM is simple, accurate and time-saving for screening single clones and measuring their colony-forming ability.
- Citation: Liu CZ, Jiao XL, Gao DQ, Xing LB, Liu H, Luo Y, Gao YT. Real-time live-cell analysis system for screening single tumor cell clones and analyzing their colony-forming ability. Shijie Huaren Xiaohua Zazhi 2017; 25(10): 881-890
- URL: https://www.wjgnet.com/1009-3079/full/v25/i10/881.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v25.i10.881
肿瘤异质性是恶性肿瘤的重要特征之一, 目前多数研究者认为肿瘤异质性和肿瘤干细胞的起源与分化密切相关[1], 其分子机制和临床应用潜能已成为近年来研究者所关注的热点[2-4]. 因此, 获取并分析大量单个癌细胞及其衍生的单克隆细胞群的生物学特征将是研究肿瘤异质性亟待解决的问题.
目前, 常规的细胞检测方法多为终点法, 即仅仅给出最终结果, 并且往往需要标记、固定或破碎细胞[5-7]; 该方法明显的缺点就是无法对细胞的生长过程做出全程的动态监测和分析[8,9]. 因此, 越来越多具有多种功能、实时观测细胞生长状态的活细胞成像系统用于细胞学检测, 其中美国Essen公司开发的第二代长时间实时动态活细胞成像分析仪(IncuCyte ZOOM)具有高通量(可同时实时监测6块细胞培养板/皿/瓶)、与各型号培养箱兼容可实现长达数月的监测、4倍物镜下对6至384孔培养板均可进行全孔成像以及强大的分析软件等优点, 可实现单克隆筛选、监测报告基因[10]、细胞增殖、迁移与侵袭[11]、细胞凋亡[12]、免疫细胞杀伤[13]、细胞质控、细胞毒性、监测干细胞生长及重编程、三维肿瘤球体成像等十余种细胞检测方法[14-16]. 基于该系统的优越性, 我们在有限稀释法的基础上进行了原代肿瘤细胞的单克隆筛选和克隆形成率的检测研究.
收集在天津市第三中心医院肝胆外科治疗的肝癌患者30例, 取其癌组织培养原代肿瘤细胞, 所有标本取材均通过天津市第三中心医院伦理委员会审核, 家属知情同意并签字. 实验所需的仪器与试剂主要有: 第二代长时间实时动态活细胞成像分析仪(IncuCyte ZOOM)购自美国Essen Bioscience公司; 荧光倒置显微镜(Olympus, Ix71)购自日本Olympus公司; 冷冻离心机(3-18K)购自德国Sigma公司; 细胞培养箱购自美国Thermo公司; DMEM/F12培养液、肝裂解液、100X抗菌-抗真菌剂、Falcon细胞筛网(40 μm)、0.25%胰蛋白酶(Trypsin-EDTA)均购于美国Life公司; 红细胞裂解液购于天津市灏洋生物制品科技有限公司; 胎牛血清购于以色列Biological Industries公司.
1.2.1 原代肿瘤细胞的分离与培养: 生理盐水充分清洗肿瘤组织, 将肝裂解液用注射器点状注射、消化组织5 min, 充分剪碎并吹打, 将细胞悬液过40 μm滤网, 1000 r/min离心5 min弃上清; 加入5 mL溶血素37 ℃静置5 min去除红细胞, 1000 r/min离心5 min弃上清收获细胞; 细胞接种于25 cm2培养瓶中, 加入DMEM/F-12培养液: 含5%胎牛血清、1X抗菌-抗真菌剂, 置于37 ℃、50 mL/L CO2培养箱中培养[17]. 待细胞贴壁后, 依据细胞生长状况进行半量或全量换液.
1.2.2 差速消化与差速贴壁: 根据肿瘤细胞和成纤维细胞等基质细胞对胰蛋白酶的敏感性不同及贴壁的速度差异, 采取消化、终止、再消化、终止, 分别收集细胞离心, 加DMEM/F-12培养液重悬细胞, 分别加入2个六孔板的A孔中贴壁15-30 min, 轻轻晃动培养板将上清液(含未贴壁的悬浮细胞)轻轻吸出转入B孔, B孔悬浮细胞贴壁15-30 min后, 再重复上一步骤将未贴壁的悬浮细胞转入C孔, 根据细胞数量和贴壁情况可重复多次, 尽可能纯化癌细胞.
1.2.3 利用IncuCyte ZOOM筛选单克隆细胞: 取对数生长期细胞消化、离心, 血球计数板计数, 将细胞梯度稀释至1×102个/10 mL, 充分悬浮, 100 μL/孔(理论值1个细胞)铺于96孔板中; 将铺好细胞的96孔板置于培养箱中的IncuCyte ZOOM内, 选取4×相差物镜, 设置全孔拍摄, 每隔4 h循环拍摄模式, 利用系统自带分析软件(IncuCyteZOOM 2015)对只有一个细胞的孔添加电子标记, 实时追踪肿瘤克隆细胞的生长状况, 根据克隆的生长状态消化克隆并扩大培养及冻存.
1.2.4 克隆形成能力检测: 挑选2例患者的单克隆株, 取对数生长期细胞梯度稀释至1×103个/ml. (1)IncuCyte ZOOM克隆形成能力检测: 取500 μL细胞稀释液加D-MEM/F-12培养液稀释至10 mL, 充分悬浮, 每孔加100 μL(理论值5个细胞)铺于96孔板中; 再将96孔板置于IncuCyte ZOOM内, 设置方法同上, 14 d后终止拍摄. 分析图片, 统计克隆形成率, 其中以≥50个细胞的克隆为阳性克隆, 克隆形成率(%) = 克隆数/接种细胞数×100%; (2)平皿克隆形成能力检测: 取200 μL细胞稀释液接种于6 cm培养皿补加DMEM/F-12培养液至3 mL; 14 d后, HE染色计算克隆数和克隆形成率, 各使用3个平皿.
统计学处理 采用SPSS20.0软件分析数据, χ2检验, P<0.05为差异有统计学意义.
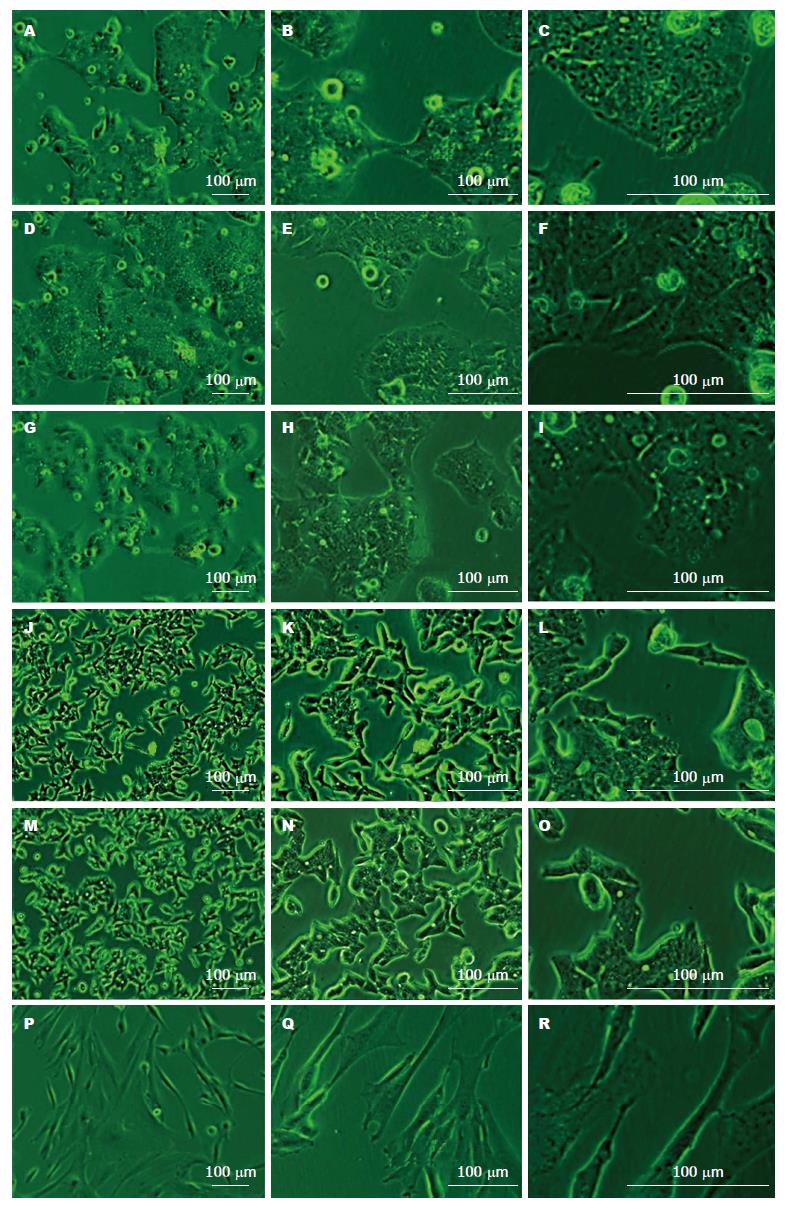
从每例癌组织中分离的细胞经过2-6 mo的差速消化与差速贴壁培养, 共培养出6株原代肿瘤细胞, 分别命名为TJ3ZX-02至07. 细胞形态如图1所示: TJ3ZX-02、03、04为上皮样细胞, 呈现多边形、铺路石状, 且细胞集落生长, 细胞间接触紧密; TJ3ZX-05、06形态相似, 细胞个体较小, 呈梭形、三角形、多角形等多种形态; TJ3ZX-07细胞形体细长, 细胞之间间隙大.
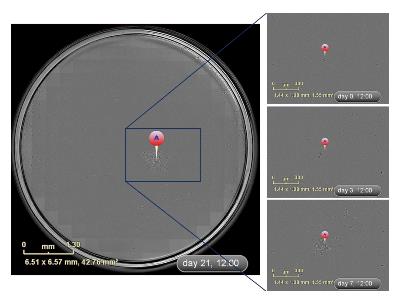
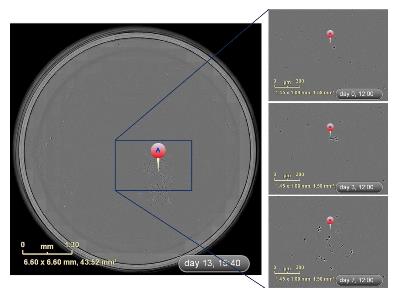
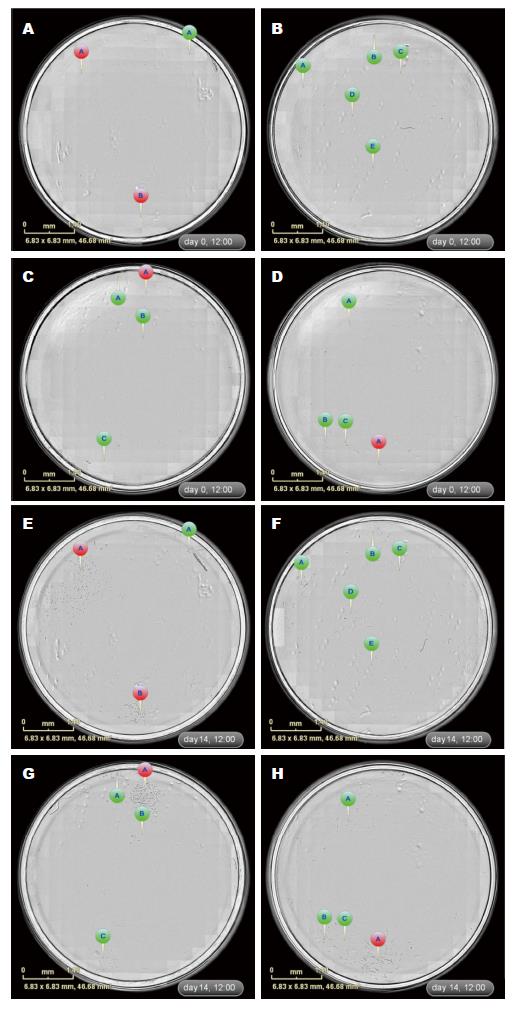
ZOOM单细胞克隆筛选 IncuCyte ZOOM筛选单克隆的最大优势是能够全孔、全程的动态实时成像观察, 即用时序性图像可以非常准确地判定克隆来源于单个细胞. 如图2所示, 通过时间轴回溯追踪发现TJ3ZX-06细胞12 h能贴壁, 24 h开始分裂, 3 d克隆逐渐变大, 7 d后能明显观察到克隆团, 细胞排列紧密、呈集落生长, 21 d左右重叠生长. TJ3ZX-07(图3)细胞形体细长, 个体较TJ3ZX-06大, 运动能力强, 细胞间隙大, 细胞分散, 克隆团呈松散状, 和TJ3ZX-06相比同等面积下细胞数目少. 另外, 通过该系统可准确鉴别出假单克隆, 如图4所示, 在培养13 d时貌似是1个单克隆, 但通过时间轴回溯追踪发现实际上是由4个细胞组成的多克隆.
在6株原代肿瘤细胞中, TJ3ZX-02由于单个细胞难以贴壁重复3次均未获得单细胞克隆, 其余5株成功获得单细胞克隆, 并通过时间轴回溯追踪分析准确排除了由2个及以上相邻细胞增殖形成的假单克隆. 5株原代肿瘤细胞单克隆筛选结果如表1所示, 通过实时追踪发现一部分单个细胞(89个)能形成持续增殖的克隆团并能扩大培养, 进一步通过2-3次传代排除可能的细菌或支原体污染、挑选细胞生长状态良好的67个单克隆株进行冻存; 另一部分单个细胞(18个)形成的克隆不能持续增殖, 细胞在增殖2-4 wk后出现停滞, 细胞出现衰老或凋亡; 此外, 还有28个由2个及以上细胞组成的多克隆.
| 细胞编号 | 培养时间(d) | 持续增殖的克隆数 | 不能持续增殖的克隆数 | 多克隆的孔数 |
| TJ3ZX-02 | 15 | 0 | 0 | 0 |
| TJ3ZX-03 | 35 | 7 | 4 | 1 |
| TJ3ZX-04 | 22 | 24 | 9 | 4 |
| TJ3ZX-05 | 14 | 29 | 1 | 3 |
| TJ3ZX-06 | 40 | 15 | 0 | 15 |
| TJ3ZX-07 | 38 | 14 | 4 | 5 |
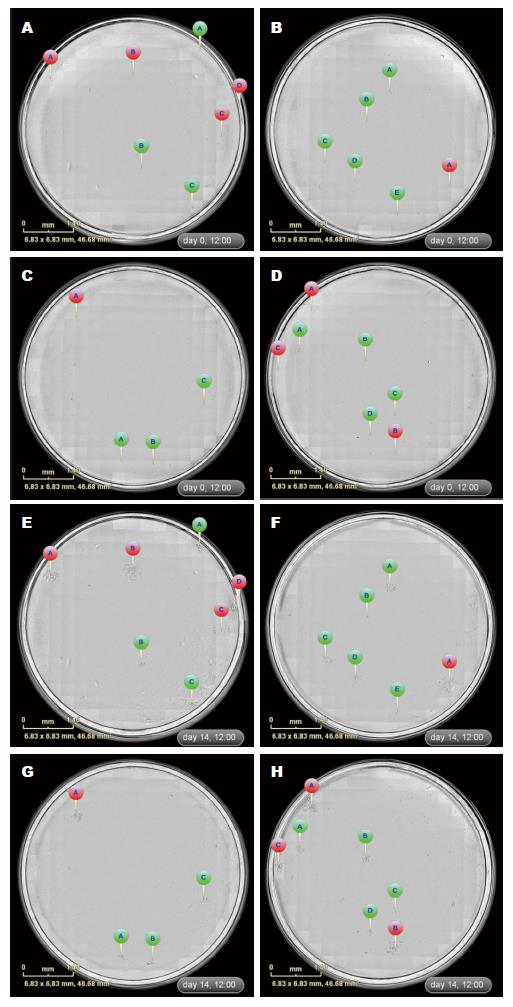
2.3.1 IncuCyte ZOOM克隆形成能力分析: 采集的全孔成像图(图5, 6), 通过时间轴回溯追踪发现: TJ3ZX-06-B11单克隆株在96孔板上总计形成237个单克隆, 以培养14 d时计算, 其中细胞数≥50个细胞的克隆数为111个, 即克隆形成率为23.13%(111/96×5), 另有细胞数<50的克隆126个; TJ3ZX-07-H11单克隆株在96孔板上总计形成103个克隆, 其中8个为多克隆(起始细胞2至5个, 合计26个细胞), 由于其培养2 wk时难以准确计数予以排除, 细胞数≥50个细胞的克隆数为25个, 即克隆形成率为5.51%[25/(96×5-26)], 另有细胞数<50的克隆70个.
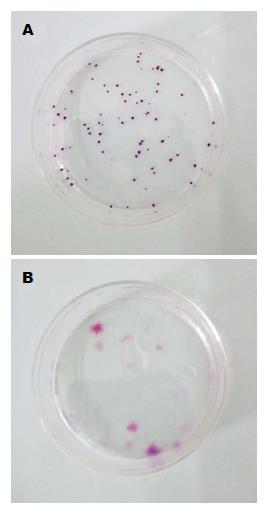
2.3.2 传统的平皿克隆形成能力分析: 克隆培养14 d时, 对TJ3ZX-06-B11和TJ3ZX-07-H11 2个单克隆株HE染色(图7), 统计克隆数. 对于TJ3ZX-06-B11单克隆株, 3个平皿合计细胞数≥50个细胞的克隆数为211个, 即克隆形成率为35.17%(211/600), 另有细胞数<50的克隆38个; 对于TJ3ZX-07-H11单克隆株, 3个平皿合计细胞数≥50个细胞的克隆79个, 即克隆形成率为13.17%(79/600), 另有细胞数<50的克隆14个.
2.3.3 两种方法对克隆形成能力检测的比较: 14 d时传统方法的克隆形成率比IncuCyte ZOOM 96孔全孔成像的克隆形成率高, 且差异有统计学意义(TJ3ZX-06-B11: χ2 = 18.478, P = 0.000017; TJ3ZX-07-H11: χ2 = 19.408, P = 0.00011). 考虑到利用IncuCyte ZOOM 96孔全孔成像分析克隆形成能力尚处于探索阶段, 并且每个单孔中初始仅有平均5个细胞, 远少于传统方法的200个细胞在一个平皿内, 由此造成的细胞之间旁分泌作用等微环境并不相同. 同时, IncuCyte ZOOM可以持续动态观察细胞, 因此, 我们进一步分析21 d时全孔成像的结果, 发现TJ3ZX-06-B11增加了细胞数≥50个的克隆数60个, TJ3ZX-07-H11增加了细胞数≥50个的克隆数35个, 重新计算二者的克隆形成率分别为35.63%[(111+60)/96×5]、13.22%[(25+35)/(96×5-26)], 分析二者无统计学差异(TJ3ZX-06-B11: χ2 = 0.25, P = 0.876; TJ3ZX-07-H11: χ2 = 0.106, P = 0.745).
随着肿瘤异质性研究受到越来越多的重视, 急需一种准确的单克隆细胞筛选方法研究肿瘤异质性等生物学特性. 常用的单细胞克隆技术[18-20]需要研究人员在显微镜下对96孔的每个孔及其孔内每个区域进行仔细观察、标记含单个细胞的孔, 其缺点是耗时费力、反复从培养箱中取出观察易污染、由于细胞形态和人为因素等原因难以保证单克隆筛选的准确性[21-23]. 细胞成像技术的发展为单克隆的筛选提供了新的思路[24,25], IncuCyte ZOOM可以在有限稀释法基础上实现自动化的单克隆筛选, 通过对96孔板设置全孔、循环拍摄的模式来筛选单克隆细胞, 克隆筛选的整个过程除培养板补液外, 细胞无需离开培养箱, 始终处于一个最适生长环境, 通过连续显微拍照, 对培养克隆进行动态监测, 并可远程控制、数据读取与分析; 通过在时间轴回溯中判断细胞团是否来源于一个细胞, 并可以在图像、孔板上标记单克隆位置, 便于挑选, 其优点显而易见.
利用IncuCyte ZOOM在5株原代肿瘤细胞中成功获得89个单细胞克隆, 并挑选细胞生长状态良好的67个单克隆进行冻存, 为进一步研究单克隆细胞群的生物学特征和肿瘤细胞异质性奠定了较好的基础. 在筛选单克隆细胞时我们发现像TJ3ZX-07运动能力强的细胞呈不定向分散生长, 与TJ3ZX-02至06细胞聚集成团呈集落式生长的方式明显不同, 像这种运动能力强的细胞在培养过程中可能会使相邻的克隆相互渗透生长到一起, 传统的人工方法由于是通过肉眼来观察会误认为是一个克隆团(图4), 从而使筛选结果不准确且难以发现. 但是利用IncuCyte ZOOM可以整理分析整个过程拍摄的照片, 并在每个克隆上加上电子标签, 可视化标签存在于整个拍摄过程中所设定的任意时间点上、使监测克隆更加简便、准确、随时记录克隆生长状况, 通过时序性图像判断细胞团是否来源于一个细胞, 同时我们也可以利用标记工具(Marker Tool)在培养器皿上有克隆的位置做上物理标记, 便于筛选结束时能够对多克隆的孔准确的判断挑选单克隆细胞, 并对克隆进行扩大培养, 提高克隆筛选结果的准确性.
另一部分单个细胞(18个)不能持续增殖, 细胞在增殖2-4 wk后出现停滞, 细胞出现衰老或凋亡. 我们推测有两种可能: (1)这些细胞可能不是癌细胞, 从瘤组织经差速消化与差速贴壁获取的原代肿瘤细胞中依旧含有少量成纤维细胞等间质细胞; (2)肿瘤干细胞理论能够解释这种现象, 原代肿瘤细胞中只有少部分的细胞具有干细胞特性, 即并非每个肿瘤细胞都具有无限增殖能力, 只有肿瘤干细胞能够无限制地生长、在体外克隆形成实验中可以形成克隆并在体内成瘤[26-28].
克隆形成率是反映细胞群体依赖性和增殖能力两个性状的重要指标[29]. 本文首次利用IncuCyte ZOOM全孔成像技术分析克隆形成能力, 与传统的平皿方法比较, 其优点在于自动化程度高、省时省力、实时动态追踪排除假单克隆; 但也发现一个问题, 在培养条件相同(相同材质的培养板、培养液、同一培养箱和同一人同时操作)的情况下, 按经典平皿克隆形成实验的要求细胞培养时间为2 wk时, IncuCyte ZOOM 96孔全孔成像的克隆形成率明显低于传统方法, 再延长培养一周时两种方法的克隆形成率趋于一致, 分析原因可能与细胞之间旁分泌作用密切相关, 由于96孔全孔成像分析每个单孔中初始仅有平均5个细胞, 而传统方法是200个细胞同在一个平皿内, 因此, 二者在旁分泌作用上可能是明显不同的. 考虑到利用克隆形成能力尚处于探索阶段, 如何更合理地利用96孔全孔成像技术分析克隆形成率需要更多细胞系的重复实验验证.
总之, 在有限稀释法基础上, 我们成功地利用IncuCyte ZOOM系统进行了原代肿瘤细胞的单克隆筛选和克隆形成率检测, 该方法能对克隆实时监测, 远程控制、使克隆筛选更加方便、准确、省时省力, 并能导出筛选克隆时视频作为证据等诸多优点, 为更好的研究肿瘤细胞生物学特性和细胞异质性提供了强有力的技术手段, 对以个性化治疗为目标的精准医学模式具有重要价值.
肿瘤异质性已成为近年来研究者关注的热点, 获取并分析大量单个癌细胞及其衍生的单克隆细胞群的生物学特征成为研究肿瘤异质性亟待解决的问题, 而IncuCyte ZOOM为肿瘤异质性的研究提供了一种新的技术手段.
目前, 常用的单克隆筛选和克隆形成率检测方法费时费力且难以保证准确性, 寻找一种能准确简便的方法成为亟待解决的问题, 本文基于IncuCyte ZOOM系统对克隆筛选和克隆形成率检测进行了阐述.
已有研究报道, 采用IncuCyte ZOOM评估细胞迁移与增殖率、细胞凋亡及监测巨噬细胞吞噬作用, 但利用IncuCyte ZOOM系统进行了原代肿瘤细胞的克隆筛选和克隆形成力的检测却鲜有报道.
实验采用IncuCyte ZOOM进行单克隆筛选和克隆形成力检测与传统方法相比, 可通过全孔成像、循环拍摄的模式, 实时动态的监测克隆的生长状态, 并通过时序性图像判断细胞团来源, 排除假单克隆的干扰, 提高准确性.
利用IncuCyte ZOOM系统能对克隆实时监测, 远程控制、使克隆筛选更加方便、准确, 可作为筛选单克隆细胞和克隆形成能力检测的一种新方法, 为更好的研究肿瘤细胞生物学特性及以个性化治疗为目标的精准医学具有重要价值.
克隆形成率: 指细胞被制成分散的悬液, 以低密度接种后, 单个细胞生长增殖并形成细胞小群(克隆)的比率.
陈绍勤, 副教授, 主任医师, 福建医科大学附属第一医院胃肠外科二病区; 韩双印, 主任医师, 郑州大学人民医院消化内科; 李云龙, 副教授, 哈尔滨医科大学附属二院普通外科; 王雅棣, 教授, 主任医师, 北京军区总医院放疗科
文章阐述了利用IncuCyte ZOOM系统筛选肿瘤细胞单克隆和克隆形成能力, 结论较明确, 证据充足, 统计学方法使用恰当, 条理分明, 简明而有特色, 有科学性和先进性, 能够反映该领域的先进水平.
手稿来源: 自由投稿
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): B, B
C级 (良好): C, C
D级 (一般): 0
E级 (差): 0
编辑: 马亚娟 电编:胡珊
| 1. | Valent P, Bonnet D, De Maria R, Lapidot T, Copland M, Melo JV, Chomienne C, Ishikawa F, Schuringa JJ, Stassi G. Cancer stem cell definitions and terminology: the devil is in the details. Nat Rev Cancer. 2012;12:767-775. [PubMed] [DOI] |
| 2. | Gerlinger M, Rowan AJ, Horswell S, Larkin J, Endesfelder D, Gronroos E, Martinez P, Matthews N, Stewart A, Tarpey P. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N Engl J Med. 2012;366:883-892. [PubMed] [DOI] |
| 3. | Logothetis CJ. Re: intratumor heterogeneity and branched evolution revealed by multiregion sequencing. Eur Urol. 2013;64:170. [PubMed] [DOI] |
| 4. | Bedard PL, Hansen AR, Ratain MJ, Siu LL. Tumour heterogeneity in the clinic. Nature. 2013;501:355-364. [PubMed] [DOI] |
| 5. | Carroll S, Al-Rubeai M. The selection of high-producing cell lines using flow cytometry and cell sorting. Expert Opin Biol Ther. 2004;4:1821-1829. [PubMed] [DOI] |
| 6. | Powell KT, Weaver JC. Gel microdroplets and flow cytometry: rapid determination of antibody secretion by individual cells within a cell population. Biotechnology (NY). 1990;8:333-337. [PubMed] [DOI] |
| 7. | Alvin K, Ye J. Generation of cell lines for monoclonal antibody production. Methods Mol Biol. 2014;1131:263-271. [PubMed] [DOI] |
| 8. | Manz R, Assenmacher M, Pflüger E, Miltenyi S, Radbruch A. Analysis and sorting of live cells according to secreted molecules, relocated to a cell-surface affinity matrix. Proc Natl Acad Sci USA. 1995;92:1921-1925. [PubMed] [DOI] |
| 9. | Holmes P, Al-Rubeai M. Improved cell line development by a high throughput affinity capture surface display technique to select for high secretors. J Immunol Methods. 1999;230:141-147. [PubMed] [DOI] |
| 10. | Dukic AR, McClymont DW, Taskén K. A Cell-Based High-Throughput Assay for Gap Junction Communication Suitable for Assessing Connexin 43-Ezrin Interaction Disruptors Using IncuCyte ZOOM. J Biomol Screen. 2016; Sep 14. [Epub ahead of print]. [PubMed] [DOI] |
| 11. | Johnston ST, Shah ET, Chopin LK, Sean McElwain DL, Simpson MJ. Estimating cell diffusivity and cell proliferation rate by interpreting IncuCyte ZOOM™ assay data using the Fisher-Kolmogorov model. BMC Syst Biol. 2015;9:38. [PubMed] [DOI] |
| 12. | Artymovich K, Appledorn DM. A multiplexed method for kinetic measurements of apoptosis and proliferation using live-content imaging. Methods Mol Biol. 2015;1219:35-42. [PubMed] [DOI] |
| 13. | Kapellos TS, Taylor L, Lee H, Cowley SA, James WS, Iqbal AJ, Greaves DR. A novel real time imaging platform to quantify macrophage phagocytosis. Biochem Pharmacol. 2016;116:107-119. [PubMed] [DOI] |
| 14. | Stewart H, Bartlett C, Ross-Thriepland D, Shaw J, Griffin S, Harris M. A novel method for the measurement of hepatitis C virus infectious titres using the IncuCyte ZOOM and its application to antiviral screening. J Virol Methods. 2015;218:59-65. [PubMed] [DOI] |
| 15. | Widmeier E, Tan W, Airik M, Hildebrandt F. A small molecule screening to detect potential therapeutic targets in human podocytes. Am J Physiol Renal Physiol. 2017;312:F157-F171. [PubMed] [DOI] |
| 16. | Zhao Z, Ma X, Hsiao TH, Lin G, Kosti A, Yu X, Suresh U, Chen Y, Tomlinson GE, Pertsemlidis A. A high-content morphological screen identifies novel microRNAs that regulate neuroblastoma cell differentiation. Oncotarget. 2014;5:2499-2512. [PubMed] [DOI] |
| 18. | Helgason CD, Miller CL. Basic Cell Culture Protocols. Humana Press. 2013;17:371-381. [DOI] |
| 19. | Puck TT, Marcus PI. Rapid Method For Viable Cell Titration And Clone Production With Hela Cells In Tissue Culture: The Use Of X-Irradiated Cells To Supply Conditioning Factors. Proc Natl Acad Sci USA. 1955;41:432-437. [PubMed] [DOI] |
| 21. | Yanıkkaya Demirel G, Budak-Alpdoğan T, Aktaş S, Bayık M. Long-term culture-initiating cells (LTC-IC) produced from CD34+ cord blood cells with limiting dilution method. Turk J Haematol. 2010;27:234-241. [PubMed] [DOI] |
| 22. | Zhu L, Gao D, Yang J, Li M. Characterization of the phenotype of high collagen-producing fibroblast clones in systemic sclerosis, using a new modified limiting-dilution method. Clin Exp Dermatol. 2012;37:395-403. [PubMed] [DOI] |
| 24. | Hou JJ, Hughes BS, Smede M, Leung KM, Levine K, Rigby S, Gray PP, Munro TP. High-throughput ClonePix FL analysis of mAb-expressing clones using the UCOE expression system. N Biotechnol. 2014;31:214-220. [PubMed] [DOI] |
| 25. | Choi JH, Ogunniyi AO, Du M, Du M, Kretschmann M, Eberhardt J, Love JC. Development and optimization of a process for automated recovery of single cells identified by microengraving. Biotechnol Prog. 2010;26:888-895. [PubMed] [DOI] |
| 26. | Bahnassy AA, Fawzy M, El-Wakil M, Zekri AR, Abdel-Sayed A, Sheta M. Aberrant expression of cancer stem cell markers (CD44, CD90, and CD133) contributes to disease progression and reduced survival in hepatoblastoma patients: 4-year survival data. Transl Res. 2015;165:396-406. [PubMed] [DOI] |
| 27. | Hu Y, Fu L. Targeting cancer stem cells: a new therapy to cure cancer patients. Am J Cancer Res. 2012;2:340-356. [PubMed] |
| 28. | Eyler CE, Rich JN. Survival of the fittest: cancer stem cells in therapeutic resistance and angiogenesis. J Clin Oncol. 2008;26:2839-2845. [PubMed] [DOI] |