修回日期: 2016-01-12
接受日期: 2016-01-19
在线出版日期: 2016-02-28
目的: 应用全基因组表达谱芯片和microRNA芯片检测人结肠癌细胞DLD1在敲低FOXQ1基因前后mRNA和microRNA的表达改变.
方法: 抽提两组细胞的RNA, 分别与Whole Human Genome Oligo Microarray(4×44 K, Agilent Technologies)全基因组表达谱芯片和miRCURYTM LNA Array(v.18.0, Agilent Technologies)microRNA芯片进行杂交, 应用genespring、genepix软件对芯片结果进行统计, 对全基因组表达谱芯片进行GO、pathway分析, 并应用qRT-PCR对部分结果进行验证. 应用Microsoft Excel和SPSS17.0软件进行统计学分析.
结果: 全基因组表达谱的检测结果表明, 敲低FOXQ1基因后, 在芯片所涵盖的41093个基因中, 共有255个基因上调, 176个基因下调. qRT-PCR的验证结果与全基因组表达谱芯片的结果基本相符. MicroRNA芯片的检测结果表明, 敲低FOXQ1基因后, 共有31个microRNA上调, 12个microRNA下调.
结论: 本研究了解了FOXQ1基因敲低前后mRNA和microRNA的表达改变, 为后续FOXQ1在结肠癌的发生发展中的作用机制研究提供了重要的线索和实验依据.
核心提示: 首次研究了FOXQ1(homo sapiens fork head box Ql)敲低前后, 全基因组水平上的蛋白转录和microRNA表达水平的改变, 为探究FOXQ1在促进大肠癌发生发展中的分子生物学机制和后续临床实验指明了实验方向.
引文著录: 郑极, 唐慧, 白璇, 岳柯琳, 郭强. 利用微阵列芯片技术探究基因FOXQ1与大肠癌的关系. 世界华人消化杂志 2016; 24(6): 915-922
Revised: January 12, 2016
Accepted: January 19, 2016
Published online: February 28, 2016
AIM: To compare the difference in gene expression profiles in human colorectal cancer cell line DLD1 before and after FOXQ1 knockdown using the microarray technology.
METHODS: RNA was extracted from DLD1 cells and then hybridized with the probes on Whole Human Genome Oligo Microarray (4 × 44 K, Agilent Technologies) and miRCURY LNA Array (v.18.0, Agilent Technologies). Multiple target genes were then analyzed with Genespring and Genepix software. Quantitative real-time PCR was applied for the validation of microarray results.
RESULTS: After FOXQ1 knockdown, of 41093 tested genes, 255 were up-regulated and 176 down-regulated. This result was partially validated by qRT-PCR. After FOXQ1 knockdown, of 2075 tested microRNAs, 31 were up-regulated and 12 down-regulated.
CONCLUSION: The whole genome microarray provides clues and evidence for the function of FOXQ1 in the development and progression of colorectal cancer.
- Citation: Zheng J, Tang H, Bai X, Yue KL, Guo Q. Exploring relationship between FOXQ1 gene and colorectal cancer using microarray technology. Shijie Huaren Xiaohua Zazhi 2016; 24(6): 915-922
- URL: https://www.wjgnet.com/1009-3079/full/v24/i6/915.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i6.915
结肠癌是常见的恶性肿瘤. 在欧美等西方国家, 结直肠癌占恶性肿瘤死因的第2位. 据2014年的统计数据, 结直肠癌在我国恶性肿瘤发病率中居第6位、致死率居第5位. 由于结肠癌的病因为高蛋白、高脂类、低纤维饮食, 吸烟等不健康的生活方式疾病, 故随着人们的生活水平的改善, 结直肠癌发病率在我国呈逐年上升的趋势. 基因芯片技术是通过将大量的基因特异的探针或其cDNA片段固定在一块固相载体上, 与待测样品杂交后用激光共聚焦荧光检测系统进行扫描、检测, 获得大量有价值的信息. 微阵列基因芯片具有高密度、高灵敏度和高灵活性的特点, 为现代医学大规模研究人类基因的功能, 特别是研究新发现的疑似致病基因和功能已知基因间的相互作用和调控关系提供了有力的工具. 全基因组表达谱芯片, 覆盖基因面大, 分类全面详细, 十分适合对单一基因的功能和机制进行的全面了解.
癌基因FOXQ1是一属于叉头框(forkhead, FOX)蛋白家族的转录因子, FOXQ1基因定位于第6号常染色体短臂25.3区(6p25.3), 全长2319 bp, 仅含1个外显子, 编码有403个氨基酸的FOXQ1蛋白. 具有一个高度保守的叉头DNA结合域[1]. FOXQ1在多种组织中发现其广泛表达, 早期研究发现和毛发的分化有关[2]. 近年来发现, FOXQ1参与了多种癌症如乳腺癌、肝癌、胃癌[3]、非小细胞肺癌等的侵袭和转移进程, 功能涉及上皮间质转化(epithelial-mesenchymal transition, EMT)和癌细胞凋亡抑制等[4,5].
本研究组的前期研究[6]表明, FOXQ1 mRNA和蛋白质的表达水平随大肠腺瘤→大肠腺癌这一病理发展过程逐步升高. FOXQ1是结肠癌发生发展中重要的癌基因, 但具体的致癌机制研究尚未明确, 本实验拟通过检测人结肠癌细胞DLD1在敲低FOXQ1基因前后mRNA和microRNA的表达谱改变, 为FOXQ1在结肠癌中致癌机制的研究进行初步探索, 为后续分子机制研究奠定了基础, 并为在以后的临床中基因诊断及可能针对FOXQ1为靶点的基因治疗中, 发挥作用.
结肠癌细胞株DLD1细胞购自中国科学院昆明动物研究所, 慢病毒干扰FOXQ1正常表达的DLD1-shFOXQ1细胞和对应的慢病毒感染阴性对照细胞DLD1-shControl均由本实验室构建并长期保存[6]. 所用全基因组表达谱芯片为Whole Human Genome Oligo Microarray(4×44 K, Agilent Technologies)、所用microRNA芯片为miRCURYTM LNA Array(v.18.0, Agilent Technologies)microRNA芯片. TRIzol(Invitrogen); GoScriptTM Reverse Transcription System(Promega); SYBR® Premix Ex TaqTM(Takara); miRCURYTM Hy3TM/Hy5TM Power labeling kit(Exiqon).
1.2.1 总RNA提取: 细胞培养条件为含10%胎牛血清、1%青链霉素双抗的1640培养基(Hyclone), 37 ℃、50 mL/L CO2, 用TRIzol分别提取DLD1-shFOXQ1和DLD1-shControl细胞总RNA, 每组细胞2个生物学重复. 用Bioanalyzer 2100(Agilent)定量检测, 以评估总RNA的浓度、纯度、完整性和A260/280.
1.2.2 芯片杂交和检测: 全基因组表达谱基因芯片和microRNA芯片的检测和数据分析均由上海康成生物公司完成.
1.2.3 芯片检测数据分析: 采用GeneSpring GX v11.5.1(Aligent)获得表达谱芯片的校准数据, 按照DLD1-shFOXQ1/DLD1-shContro进行差异基因筛选, 以Fold change≥2作为基因表达具有显著性差异的阈值. 然后对表达差异基因结合DAVID进行GO和pathway分析. 使用GenePix Pro 6.0(Axon)对microRNA实验数据进行表达量差异分析. qRT-PCR引物由大连宝生物设计并合成, qRT-PCR使用Roche Lightcycler 480操作完成, 结果计算采用2-△△Ct法.
统计学处理 应用Microsoft Excel和SPSS17.0软件处理数据, 使用倍性变化(fold change, FC)进行基因表达量差异的计算. 并以|FC|≥2为差异有统计学意义.
人全基因组表达谱芯片的检测结果发现, 在芯片所涵盖的41093个基因中, 有255个基因上调, 176个基因下调. 芯片结果已提交GEO数据库(GSE74223). 部分结果如表1, 2.
| 基因名称 | 倍性变化 | mRNA编号 | 描述 |
| GPA33 | 6.626402 | NM_005814 | Homo sapiens glycoprotein A33 (transmembrane) (GPA33) |
| DNMBP-AS1 | 6.349088 | NR_024130 | Homo sapiens DNMBP antisense RNA 1 (non-protein coding) (DNMBP-AS1), non-coding RNA [NR_024130] |
| ANGPTL4 | 5.696401 | NM_139314 | Homo sapiens angiopoietin-like 4 (ANGPTL4) |
| DUOX2 | 5.223868 | NM_014080 | Homo sapiens dual oxidase 2 (DUOX2) |
| IL1B | 4.755472 | NM_000576 | Homo sapiens interleukin 1, beta (IL1B) |
| BLNK | 4.511446 | NM_013314 | Homo sapiens B-cell linker (BLNK) |
| VTCN1 | 4.171324 | NM_024626 | Homo sapiens V-set domain containing T cell activation inhibitor 1 (VTCN1) |
| SFTPB | 4.070699 | NM_000542 | Homo sapiens surfactant protein B (SFTPB) |
| SHE | 4.028845 | NM_001010846 | Homo sapiens Src homology 2 domain containing E (SHE) |
| - | 3.957717 | DB335107 | DB335107 SYNOV4 Homo sapiens cDNA clone SYNOV4009599 3', mRNA sequence [DB335107] |
| LOC100131581 | 3.913005 | AK092544 | Homo sapiens cDNA FLJ35225 fis, clone PROST2001116. [AK092544] |
| REPS2 | 3.894184 | NM_004726 | Homo sapiens RALBP1 associated Eps domain containing 2 (REPS2) |
| C7orf60 | 3.657602 | NM_152556 | Homo sapiens chromosome 7 open reading frame 60 (C7orf60) |
| DUOXA2 | 3.601023 | NM_207581 | Homo sapiens dual oxidase maturation factor 2 (DUOXA2) |
| C5orf4 | 3.594347 | NM_032385 | Homo sapiens chromosome 5 open reading frame 4 (C5orf4) |
| LOC283050 | 3.530351 | NR_024431 | Homo sapiens uncharacterized LOC283050 (LOC283050), transcript variant 1, non-coding RNA [NR_024431] |
| LOC100132354 | 3.422083 | NR_024478 | Homo sapiens uncharacterized LOC100132354 (LOC100132354), non-coding RNA [NR_024478] |
| ITM2B | 3.385245 | NM_021999 | Homo sapiens integral membrane protein 2B (ITM2B) |
| ALDOC | 3.37901 | NM_005165 | Homo sapiens aldolase C, fructose-bisphosphate (ALDOC) |
| C15orf48 | 3.370889 | NM_032413 | Homo sapiens chromosome 15 open reading frame 48 (C15orf48) |
| 基因名称 | 倍性变化 | mRNA编号 | 描述 |
| TCF24 | -7.699125 | NM_001193502 | Homo sapiens transcription factor 24 (TCF24) |
| MORN5 | -6.097624 | NM_198469 | Homo sapiens MORN repeat containing 5 (MORN5) |
| GRK1 | -5.3753 | NM_002929 | Homo sapiens G protein-coupled receptor kinase 1 (GRK1) |
| SPOCD1 | -4.863297 | NM_144569 | Homo sapiens SPOC domain containing 1 (SPOCD1) |
| GDF6 | -4.438107 | NM_001001557 | Homo sapiens growth differentiation factor 6 (GDF6) |
| SLC39A8 | -4.372665 | NM_001135147 | Homo sapiens solute carrier family 39 (zinc transporter), member 8 (SLC39A8) |
| FAM48B2 | -4.206 | NM_001136233 | Homo sapiens family with sequence similarity 48, member B2 (FAM48B2) |
| SERPINI1 | -3.898295 | NM_005025 | Homo sapiens serpin peptidase inhibitor, clade I (neuroserpin), member 1 (SERPINI1) |
| MYBPH | -3.67658 | NM_004997 | Homo sapiens myosin binding protein H (MYBPH) |
| VPS53 | -3.553322 | NM_001128159 | Homo sapiens vacuolar protein sorting 53 homolog (S. cerevisiae) (VPS53) |
| C3 | -3.442721 | NM_000064 | Homo sapiens complement component 3 (C3) |
| - | -3.427454 | - | HSU45880 X-linked inhibitor of apotosis protein {Homo sapiens} (exp = -1; wgp = 0; cg = 0), partial (29%) [THC2703908] |
| ZNF879 | -3.426777 | NM_001136116 | Homo sapiens zinc finger protein 879 (ZNF879) |
| CCL20 | -3.145884 | NM_004591 | Homo sapiens chemokine (C-C motif) ligand 20 (CCL20) |
| FMR1NB | -3.13995 | NM_152578 | Homo sapiens fragile X mental retardation 1 neighbor (FMR1NB) |
| GNN | -3.113418 | NR_027249 | Homo sapiens Grp94 neighboring nucleotidase pseudogene (GNN), non-coding RNA [NR_027249] |
| LOC400568 | -3.076233 | BC043554 | Homo sapiens cDNA clone IMAGE:5176687. [BC043554] |
| DGCR14 | -3.033284 | NM_022719 | Homo sapiens DiGeorge syndrome critical region gene 14 (DGCR14) |
| S100A8 | -2.988219 | NM_002964 | Homo sapiens S100 calcium binding protein A8 (S100A8) |
| RN28S1 | -2.977916 | NR_003287 | Homo sapiens RNA, 28S ribosomal 1 (RN28S1), ribosomal RNA [NR_003287] |
使用genespring软件, 以|FC|≥2作为基因表达量具有显著差异的依据, 对伴随着FOXQ1敲低上调255个基因进行GO分析, 共计参与155个BP分类(分子功能), 如氧含量反应、一元羧酸代谢、缺氧反应等; 31个CC分类(细胞组分), 如胞质囊泡、细胞质膜界囊泡、细胞膜结合囊泡等; 28个MF分类(生物学过程)如铁离子结合、氧化还原反应、双加氧酶活性等. 对伴随着FOXQ1敲低的176个基因, 共计参与326个BP分类, 如胚胎发育、外界刺激应答、机体调节控制; 15个CC分类, 如细胞外、细胞外基质、蛋白包涵体等; 28个MF分类, 如细胞因子、趋化因子、G蛋白结合受体等.
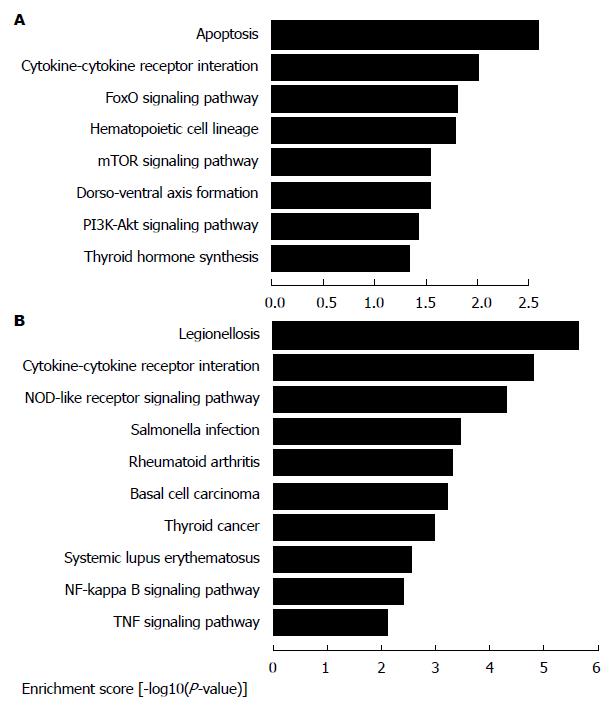
使用genespring软件和KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库, 对结果进行Pathway分析(表3, 图1). 伴随着FOXQ1的下调, 上调基因群组分别涉及为凋亡、细胞因子-细胞因子受体、FoxO信号通路、造血细胞谱系、mTOR信号通路、胚胎背腹形成、PI3K-Akt信号通路、甲状腺激素合成等. 随着FOXQ1的下调, 下调基因群组分别涉及细胞因子-细胞因子受体、NOD模式受体信号通路、基底细胞瘤、核因子κB信号通路、TNF信号通路、癌症相关转录失调、趋化因子信号通路、补体途径、凋亡、癌症信号通路等. 据已知FOXQ1功能报道, FOXQ1和细胞因子-细胞因子受体相关, 而细胞因子-细胞因子受体在本芯片中, 变化极为明显, 证实了芯片的可靠性.
| 通路 | 基因 | 富集评分 | 伪发现率 |
| 上调通路 | |||
| 凋亡通路 | CASP10/IL1B/IL1R1/IL3RA/PRKACB | 2.5925 | 0.7182 |
| 细胞因子-细胞因子受体 | ACVR2B/EGFR/IL1B/IL1R1/IL2RB/IL3RA/IL4/PDGFRB | 2.0017 | 1.0000 |
| FoxO通路 | CCNG2/EGFR/FBXO32/S1PR4/SGK2 | 1.8017 | 1.0000 |
| 造血相关 | IL1B/IL1R1/IL3RA/IL4 | 1.7900 | 1.0000 |
| mTOR通路 | DDIT4/RICTOR/TSC1 | 1.5428 | 1.0000 |
| 背腹轴形成相关 | EGFR/PIWIL3 | 1.5423 | 1.0000 |
| PI3K-Akt通路 | DDIT4/EGFR/IL2RB/IL3RA/IL4/PDGFRB/SGK2/TSC1 | 1.4303 | 1.0000 |
| 甲状腺激素合成相关 | DUOX2/DUOXA2/PRKACB | 1.3422 | 1.0000 |
| 下调通路 | |||
| 细胞因子-细胞因子受体 | CCL20/CXCL1/CXCL2/GDF6/IL17RA/IL8/LTB/TNF/TNFRSF11B/TPO | 4.8088 | 0.0022 |
| 点头样受体通路 | CARD9/CXCL1/CXCL2/IL8/TNF | 4.3323 | 0.0044 |
| 基底细胞瘤相关 | CTNNB1/GLI1/TP53/WNT16 | 3.2310 | 0.0275 |
| 核因子B通路 | CXCL2/IL8/LTB/TNF | 2.4180 | 0.1192 |
| 肿瘤坏死因子通路 | CCL20/CXCL1/CXCL2/TNF | 2.1262 | 0.2101 |
| 癌症转录失调相关 | HIST1H3H/IL8/NR4A3/TP53/WNT16 | 2.0744 | 0.2152 |
| 趋化因子相关 | CCL20/CXCL1/CXCL2/GRK1/IL8 | 1.9780 | 0.2463 |
| 补体、凝血相关 | C3/PROC/SERPIND1 | 1.8955 | 0.2749 |
| 凋亡通路 | PRKAR1B/TNF/TP53 | 1.6411 | 0.4281 |
| 肿瘤相关 | CTNNB1/GLI1/IL8/TP53/TPM3/WNT16 | 1.5656 | 0.4776 |
根据pathway和GO分析, 选取与FOXQ1正相关7个基因EGFR、IL1B、IL1R1、Bag4、SULT1C、DDIT4、CCNG2, FOXQ1负相关6个基因, CXCL1、CXCL2、PFAS、HRASLS5、TNFSF11b、TNF. 以GADPH为内参进行qRT-PCR实验. 结果如表4. 在验证的13个基因中, 有11和芯片结果相吻合, 表明芯片结果可信度较高.
经miRCURYTM Hy3TM/Hy5TM Power labeling kit(Exiqon)芯片检测DLD1-shcontrol和DLD1-shFOXQ1, 共计2075个microRNA, 并做4个重复, 使用Genepix统计结果. 伴随着FOXQ1的下调, 以|FC|≥2作为基因表达具有显著差异标准, 共计31个. microRNA上调, 12个microRNA下调部分结果如表5, 6.
| microRNA名称 | 倍比变化 |
| hsa-miR-642b-3p | 9.262590109 |
| hsa-miR-550a-3-5p/ hsa-miR-550a-5p | 9.084437272 |
| hsa-miR-320d | 6.849982996 |
| hsa-miR-195-5p | 6.109836399 |
| hsa-miR-4524b-5p | 5.078783166 |
| hsa-miR-4506 | 4.961078779 |
| hsa-miR-4419a | 4.827465547 |
| hsa-miR-3175 | 4.816025478 |
| hsa-miR-4784 | 4.408422585 |
| hsa-miR-548ap-5p/ hsa-miR-548j-5p | 3.97387298 |
| microRNA名称 | 倍比变化 |
| hsa-miR-5000-3p | -5.260356627 |
| hsa-miR-206 | -2.877979004 |
| hsa-miR-145-3p | -2.773082367 |
| hsa-miR-4796-5p | -2.745003957 |
| hsv1-miR-H8-5p | -2.598690795 |
| hsa-miR-19a-5p | -2.425592891 |
| hsa-miR-302e | -2.311195074 |
| hsa-let-7i-3p | -2.296028082 |
| hsa-miR-491-3p | -2.265283812 |
| hsa-miR-339-5p | -2.161523974 |
本实验所选用的表达谱芯片为Whole Human Genome Oligo Microarray(4×44 K, Agilent Technologies)为美国安捷伦公司采用喷墨打印化学原位合成技术生产. 长寡核苷酸探针和非接触实验原理的应用, 大大地提高了检测的灵敏度. 此款芯片汇总目前所了解的人类全基因组序列信息, 代表了超过41000人类基因和转录本. 设计该产品所用的序列信息源于对RefSeq、Goldenpath、Ensembl和Unigene等知名数据库的深入研究, 并与人类基因组拼接进行比对绝大多数探针经过Agilent专利的实验验证程序的检验和优化. MicroRNA芯片为miRCURYTM Hy3TM/Hy5TM Power labeling kit(Exiqon)基于LNATM(锁核酸)专利技术, 具有特异性高, 灵敏度高, 无偏好序列的特点. 此两款芯片与常用的功能分类芯片相比, 涉及基因与microRNA更多, 更有助于全面解析FOXQ1基因的功能.
肿瘤的发生和发展, 主要涉及细胞的异常增殖、肿瘤促血管生成、基底膜突破形成转移灶等. 这些过程, 均和细胞因子及其受体的异常激活和自活化有关[7]. 本实验成功对FOXQ1相关的受体信号转导和转录调控网络进行了描绘. 经前人的研究发现FOXQ1对P21[8], E-cadherin[9]的调控; 本次实验中, 如FOXQ1对于炎症反应和前炎性凋亡有关的IL1b[9]的负向调控; FOXQ1对于参与促血管生成过程, 促进结肠癌、胃癌和前列腺癌的侵袭和转移能力的CXCL1基因的正向调控[10]. 证实了FOXQ1在多种癌症的发生发展尤其是在癌细胞的增殖、凋亡和EMT过程中的重要作用[11].
MicroRNA是一类细胞内源性的非编码小RNA, 大约由21-25个核苷酸组成, 在人体组织中广泛表达. 作为机体内固有的一种生理调控机制, microRNA通过负性调节靶基因的表达, 参与对增殖、分化、死亡等基本细胞过程的调控[12]. 本实验中, 伴随着FOXQ1下调而上调的hsa-miR-195-5p能够抑制Fra-1基因的表达. 而Fra-1基因与前列腺癌和胃癌, 结肠癌等密切相关[13]. 伴随着FOXQ1的下调而下调的hsa-miR-145-3p基因已经被证实, 和结肠癌的死亡率成负相关[14]. 更加佐证了FOXQ1在多种癌症尤其是结肠癌中的重要地位.
FOXQ1作为一类转录因子, 其作为抗肿瘤治疗靶点的优越性显而易见. 转录因子是一类维持细胞生命活动的关键调控因子[15], 通过识别并结合其下游靶基因的启动子区, 实现对下游众多靶基因表达的正向或负向调节. 因此, 将这些表达较为稳定、对诸多下游效应分子有共同调节作用的转录因子作为治疗靶点, 较之于抑制单一的效应分子或信号通路更为合理可行. 如作为乳腺癌治疗药物的紫杉醇[16], 白血病治疗药物的KP372-1[17]和格列卫[18], 骨肉瘤的盐屋霉素A、乳腺癌、前列腺癌、肾癌、卵巢癌和非小细胞肺癌等的治疗药物曲妥珠单抗、西妥昔单抗、拉帕替尼[19]和用于骨肉瘤的吉非替尼[20]等均是以FOX家族中的转录因子作为直接或间接的药物作用靶点. 因此对于FOXQ1等转录因子的机制研究, 不但在解释相关肿瘤的发生发展的基础研究中具有重要作用. 在临床相关应用上, 也具有重要意义.
FOXQ1基因从属于转录因子, 在多种癌症中高表达. 已经证实在上皮间质转化(epithelial tomesenchymal transition, EMT)和细胞周期等与癌症密切相关的生物学过程中, 发挥了重要的作用.
王成锋, 主任医师, 中国医学科学院肿瘤医院腹部肿瘤外科
近年来, 关于FOXQ1在肿瘤中的重要作用引发了广泛关注, 本实验组的前期研究证实FOXQ1与结肠癌的Dukes分期有密切关系, 但对于癌基因FOXQ1在癌症发生发展中的分子机制研究较少. 本实验的研究有助于为FOXQ1的致癌分子机制进行探究, 并结肠癌的基因靶向治疗提供新靶点、新思路.
Esquela-Kerscher等经试验验证, FOXQ1直接激活了p21这个和结肠癌预后密切相关的基因.
本实验利用全基因组芯片, 首次研究了FOXQ1敲低前后, 全基因组水平上的蛋白转录本和microRNA表达水平的改变.
本文成功实施并验证了高通量表达谱芯片, 为后续针对FOXQ1的下游靶基因及FOXQ1在影响结肠癌发生发展的分子机制指明了方向.
基因芯片: 一种以基因碱基互补配对为基础, 利用固相载体技术等将大量特异性的探针固定在载体上, 然后与样本进行分子杂交, 对样本内大量基因表达差异进行检测的一种技术.
本实验组的前期工作证明, FOXQ1是结肠癌发生发展中重要的癌基因, 但FOXQ1导致结肠癌发病的具体分子机制研究较少, 本文为后续的分子机制和临床试验指明了方向.
编辑: 郭鹏 电编:闫晋利
| 1. | Katoh M, Katoh M. Human FOX gene family (Review). Int J Oncol. 2004;25:1495-1500. [PubMed] |
| 2. | Hannenhalli S, Kaestner KH. The evolution of Fox genes and their role in development and disease. Nat Rev Genet. 2009;10:233-240. [PubMed] [DOI] |
| 3. | Liang SH, Yan XZ, Wang BL, Jin HF, Yao LP, Li YN, Chen M, Nie YZ, Wang X, Guo XG. Increased expression of FOXQ1 is a prognostic marker for patients with gastric cancer. Tumour Biol. 2013;34:2605-2609. [PubMed] [DOI] |
| 4. | Sehrawat A, Kim SH, Vogt A, Singh SV. Suppression of FOXQ1 in benzyl isothiocyanate-mediated inhibition of epithelial-mesenchymal transition in human breast cancer cells. Carcinogenesis. 2013;34:864-873. [PubMed] [DOI] |
| 5. | Zhang H, Meng F, Liu G, Zhang B, Zhu J, Wu F, Ethier SP, Miller F, Wu G. Forkhead transcription factor foxq1 promotes epithelial-mesenchymal transition and breast cancer metastasis. Cancer Res. 2011;71:1292-1301. [PubMed] [DOI] |
| 8. | Esquela-Kerscher A, Slack FJ. Oncomirs-microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] [DOI] |
| 9. | Kaneda H, Arao T, Tanaka K, Tamura D, Aomatsu K, Kudo K, Sakai K, De Velasco MA, Matsumoto K, Fujita Y. FOXQ1 is overexpressed in colorectal cancer and enhances tumorigenicity and tumor growth. Cancer Res. 2010;70:2053-2063. [PubMed] [DOI] |
| 10. | Xia L, Huang W, Tian D, Zhang L, Qi X, Chen Z, Shang X, Nie Y, Wu K. Forkhead box Q1 promotes hepatocellular carcinoma metastasis by transactivating ZEB2 and VersicanV1 expression. Hepatology. 2014;59:958-973. [PubMed] [DOI] |
| 12. | 陈 伟, 何 裕隆. 趋化因子CXCL1对肿瘤的作用及其在胃癌中的研究进展. 消化肿瘤杂志(电子版). 2012;4:55-58. |
| 13. | Chen K, Rajewsky N. The evolution of gene regulation by transcription factors and microRNAs. Nat Rev Genet. 2007;8:93-103. [PubMed] [DOI] |
| 14. | Wu J, Ji A, Wang X, Zhu Y, Yu Y, Lin Y, Liu Y, Li S, Liang Z, Xu X. MicroRNA-195-5p, a new regulator of Fra-1, suppresses the migration and invasion of prostate cancer cells. J Transl Med. 2015;13:289. [PubMed] [DOI] |
| 15. | Slattery ML, Herrick JS, Mullany LE, Valeri N, Stevens J, Caan BJ, Samowitz W, Wolff RK. An evaluation and replication of miRNAs with disease stage and colorectal cancer-specific mortality. Int J Cancer. 2015;137:428-438. [PubMed] [DOI] |
| 16. | Sunters A, Fernández de Mattos S, Stahl M, Brosens JJ, Zoumpoulidou G, Saunders CA, Coffer PJ, Medema RH, Coombes RC, Lam EW. FoxO3a transcriptional regulation of Bim controls apoptosis in paclitaxel-treated breast cancer cell lines. J Biol Chem. 2003;278:49795-49805. [PubMed] [DOI] |
| 17. | Zeng Z, Samudio IJ, Zhang W, Estrov Z, Pelicano H, Harris D, Frolova O, Hail N, Chen W, Kornblau SM. Simultaneous inhibition of PDK1/AKT and Fms-like tyrosine kinase 3 signaling by a small-molecule KP372-1 induces mitochondrial dysfunction and apoptosis in acute myelogenous leukemia. Cancer Res. 2006;66:3737-3746. [PubMed] [DOI] |
| 18. | Essafi A, Fernández de Mattos S, Hassen YA, Soeiro I, Mufti GJ, Thomas NS, Medema RH, Lam EW. Direct transcriptional regulation of Bim by FoxO3a mediates STI571-induced apoptosis in Bcr-Abl-expressing cells. Oncogene. 2005;24:2317-2329. [PubMed] [DOI] |
| 19. | Real PJ, Benito A, Cuevas J, Berciano MT, de Juan A, Coffer P, Gomez-Roman J, Lafarga M, Lopez-Vega JM, Fernandez-Luna JL. Blockade of epidermal growth factor receptors chemosensitizes breast cancer cells through up-regulation of Bnip3L. Cancer Res. 2005;65:8151-8157. [PubMed] [DOI] |
| 20. | Yang JY, Xia W, Hu MC. Ionizing radiation activates expression of FOXO3a, Fas ligand, and Bim, and induces cell apoptosis. Int J Oncol. 2006;29:643-648. [PubMed] [DOI] |