修回日期: 2016-05-19
接受日期: 2016-05-31
在线出版日期: 2016-10-08
蛋白泛素化是一种重要的转录后修饰过程, 可与蛋白酶体一起识别并降解未折叠蛋白及多余蛋白. E3泛素连接酶Arkadia由RNF-111基因所编码, 决定了靶蛋白底物的特异性, 是泛素化过程中起决定作用的关键酶. 近年来研究发现, Arkadia可以通过泛素化降解细胞内蛋白而发挥增强转化生长因子β信号通路的作用. 本文回顾了Arkadia的最新研究进展, 对其基本特征、生物学功能及其在疾病发生发展中的作用综述如下.
核心提要: E3泛素连接酶Arkadia在泛素化降解过程发挥着重要的作用. Arkadia可以通过泛素化降解细胞内蛋白而发挥增强转化生长因子β信号通路的作用, 在多种疾病的发生发展过程中都发挥着重要的作用.
引文著录: 侯斐, 刘瑞霞, 阴赪宏. E3泛素连接酶Arkadia的结构特征、生物学功能及其在疾病发生发展中的作用. 世界华人消化杂志 2016; 24(28): 3963-3969
Revised: May 19, 2016
Accepted: May 31, 2016
Published online: October 8, 2016
Ubiquitination of proteins is a post-translational modification that involves targeting and degrading misfolded or unwanted proteins by the proteasome. Arkadia, a RING-type E3 ubiquitin ligase also known as RNF111, confers the substrate specificity for ubiquitination and has a pivotal role in catalyzing the degradation of key signaling molecules. Recent research reveals that Arkadia plays a pivotal role in the transforming growth factor-β1 signaling pathway by catalyzing the degradation of key signaling molecules. In this review, we highlight the recent progress in understanding the characteristics, function and the role of Arkadia in the development of human diseases.
- Citation: Hou F, Liu RX, Yin CH. Arkadia: Characteristics, function and role in development of human diseases. Shijie Huaren Xiaohua Zazhi 2016; 24(28): 3963-3969
- URL: https://www.wjgnet.com/1009-3079/full/v24/i28/3963.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i28.3963
泛素化降解是真核细胞内蛋白质降解的主要途径, 可调控细胞凋亡、转录调控、细胞周期[1]、DNA损伤修复[2]以及免疫应答等多种细胞活动[3-6], 该途径具有依赖ATP、高效、高度选择性的特点. 其中, E3泛素连接酶Arkadia是泛素化降解过程的关键酶. 目前已知的研究报道, Arkadia可以特异性泛素化降解细胞内蛋白, 在纤维化疾病、肿瘤的发生发展及转移、气管重塑等多种疾病发病过程中都发挥着重要的作用. 本文就Arkadia研究进展综述如下.
泛素最早是在1975年被发现的, 他是一种分子量仅为8.5 kDa的小分子调节蛋白, 由76个氨基酸组成. 其广泛表达于哺乳动物细胞内, 同时在细菌、酵母及高等植物体内也广泛表达, 在酵母和人类之间仅有3个氨基酸的差异, 因而具有高度的保守性. 泛素可以单体形式存在, 也可与靶蛋白形成复合物[7,8]
直到1982年, Ciechanover等对泛素化进行了更为具体的描述. 泛素化是指泛素分子在一系列酶作用下, 对靶蛋白进行特异性修饰的3步酶学反应过程[9-13]. 首先, E1泛素激活酶通过水解ATP获得能量, 激活泛素分子, 进而实现泛素分子与E1泛素激活酶的共价结合; 然后在E2泛素结合酶的协助下, 泛素分子由E1泛素激活酶传递给E2结合酶; E3泛素连接酶再将与E2泛素结合酶共价结合的泛素分子传递给靶蛋白[14,15], 靶蛋白经过多次泛素化过程, 被多个泛素分子标记, 形成多聚泛素链[16]; 最终, 被多聚泛素链所标记的靶蛋白被26S蛋白酶体降解为小分子的多肽片段, 同时释放游离的可重新利用的泛素分子[17-19]. 在体内, 泛素化为一动态过程, 与其他转录后修饰过程一样, 可影响细胞内的多个信号传导过程. 泛素化与去泛素化能否保持一定的平衡对于维持机体正常的生理状态具有非常重大的意义[20-22].
E3泛素连接酶决定了靶蛋白底物的特异性, 是泛素化过程中起决定作用的关键酶[23]. 目前, 已知的E3泛素连接酶有600多种[24], 根据其结构和功能的不同, 可以分为4类: HECT结构域E3酶、RING结构域E3酶、N末端规则E3酶、及环指相关E3酶[25-27]. Arkadia是一种环形E3泛素连接酶[28], 广泛表达于成年哺乳动物组织中, 如包括睾丸、脾脏、胰腺、肺脏、肾脏、肝脏及心脏等[29]. 2001年Niederländer等[30]及其同事首先对Arkadia进行了报道, 他们的研究发现Arkadia可增强Nodal信号通路, 诱导Node分子形成, 这在胚胎早期发生过程中发挥着非常重要的作用.
Arkadia是由994个氨基酸开放阅读框架构成的蛋白, 编码基因为RNF111; 他具有泛素结合酶的RING结构域及靶蛋白识别结构[31], 其N-末端时多个核定位信号, C-末端即是具有特征性结构-RING finger结构域[32,33]. RING-finger结构域是指70个氨基酸中的8个氨基酸(半胱氨酸和组氨酸)通过与锌离子螯合作用形成C3H2C3(RING-H2)或C3HIC4(RING-HC)构型. 其氨基酸序列为: Cys-X2-Cys-Xn(9-39)-Cys-Xl~3-His-X2~3-Cys-X2-Cys-Xn(4-48)-Cys-X2-Cys(C2HC4)(X可以是任何氨基酸残基, 但在特定的位置还有特定的氨基酸残基), Cys和His分别代表锌结合残基半胱氨酸和组氨酸, 并且每一环指结构域连有两个锌离子[34,35].
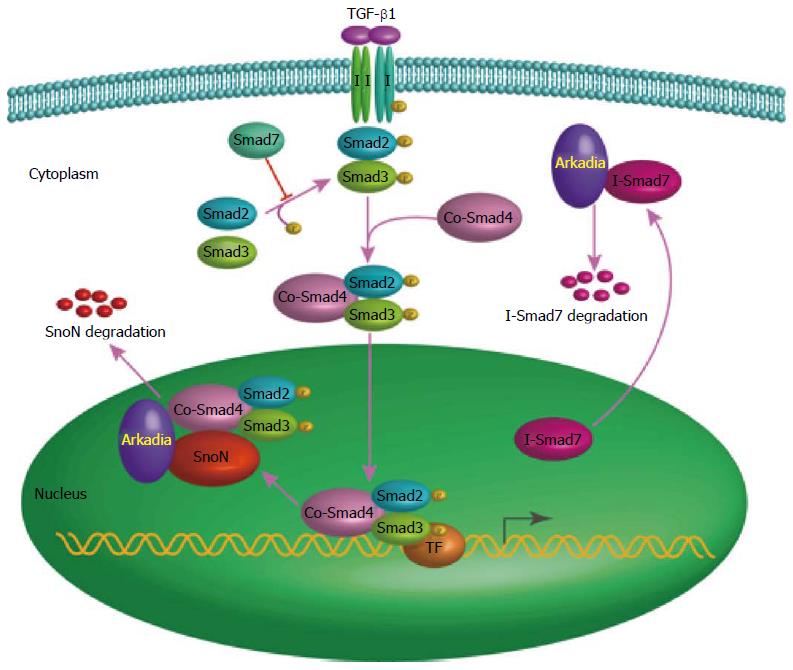
Arkadia是一种细胞内蛋白, 可以通过泛素化降解细胞内蛋白而发挥增强转化生长因子β(transforming growth factor-β, TGF-β)信号通路的作用. 与其他E3泛素连接酶所不同的是, Arkadia与Smad1、Smad2、Smad4、Smad5、Smad8分子不存在相互作用, 仅与Smad6、Smad7具有较强的相互作用; 而Smad6分子主要在BMP信号通路发挥重要作用. Smad7分子是TGF-β信号通路中的抑制性分子, 可以与TGF-β超家族的Ⅰ型受体相结合, 抑制Smad2、Smad3的活化, 从而抑制TGF-β、activin、nodal信号通路的激活. 因此, Arkadia增强TGF-β信号通路的转录活性主要是通过泛素化降解Smad7分子而实现的. 除Smad7分子以外, Arkadia还可以通过降解P-Smad2/3分子发挥增强Nodal 信号通路的作用[36].
另外, Arkadia还可以降解c-Ski/SnoN, 去除非活化状态的R-Smad复合体, 使得活化状态的R-Smad复合体与靶基因相结合, 进而促进TGF-β信号通路下游靶基因的转录. 例如, 在胚胎细胞中, Arkadia的表达缺失虽可募集P-Smad2/3分子, 但却不能启动TGF-β信号通路下游基因的转录[37].
因此, Arkadia参与调节TGF-β/Smad信号通路, 主要是通过泛素化降解TGF-β通路的抑制因子, 如Smad7、C-ski及SnoN, 来放大TGF-β信号[38-40](图1).
BMPs是TGF-β超家族的成员之一, 在细胞增殖及成骨细胞分化的过程中都发挥着非常重要的作用[41-43]. 其中, Smads蛋白是骨形态发生蛋白-7(bone morphogenetic protein-7, BMP-7)/Smads信号通路下游重要的调节因子. 首先, BMP-7蛋白与丝氨酸/苏氨酸激酶Ⅰ型和Ⅱ型受体结合形成多聚复合物, 而后结合Smads蛋白(如Smad1/5/8)发挥信号传导作用[44-47]. Smad6及Smad7是BMP-7/Smads信号通路中重要的抑制分子, 构成一个重要的负反馈的环路; 其中Smad7既是BMP-7/Smads信号通路的抑制分子, 又是TGF-β信号通路的抑制分子. 但Smad6对于TGF-β信号通路的抑制作用相对较弱, 却可以有效地阻断BMP-7/Smads信号通路. Tsubakihara等[48]的研究揭示了在BMP诱导成骨细胞分化过程中, Arkadia的缺失会导致成骨细胞分化明显减少. 他们的研究表明Arkadia主要通过降解Smad6来发挥正向调控BMP-7/Smads信号通路的作用.
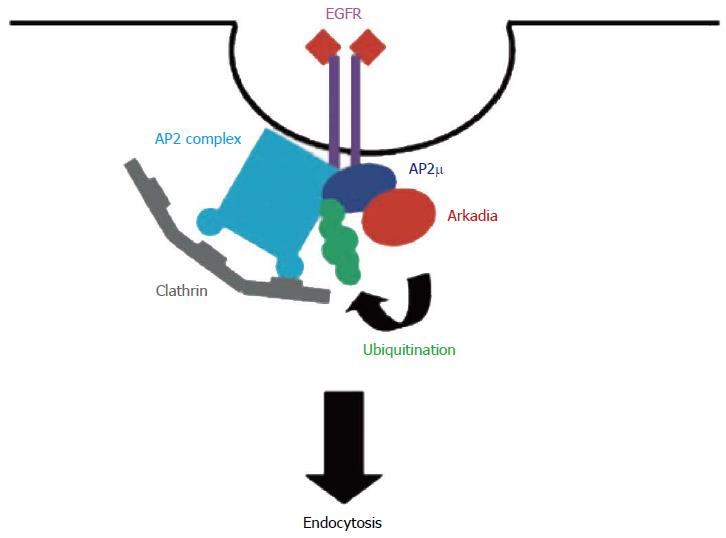
Arkadia不仅仅可以调控TGF-β信号通路, 在表皮生长因子(epidermal growth factor, EGF)信号通路中也发挥着重要的作用[49]. Mizutani等[50]研究发现, Arkadia可以与AP2复合体u2亚基的C-末端相结合, 促进u2亚基的泛素化, 进而可以调节内皮生长因子受体(epidermal growth factor receptor, EGFR)的介导的内吞作用. 因此, Arkadia可以通过调节EGFR的内吞作用来发挥调控EGF信号通路的作用(图2).
Arkadia能有效地增强TGF-β/Smads信号通路, 故Arkadia在与TGF-β信号通路相关疾病的发生发展过程中发挥着重要的作用. TGF-β是纤维化发生过程中的关键因子, 因此, Arkadia可能会参与纤维化发生发展的过程. Liu等[51,52]的研究指出, 人的正常肾小管上皮细胞在TGF-β刺激之后, Arkadia的mRNA表达水平明显升高, 而且用小干扰RNA干扰掉Arkadia之后, 肾脏α-平滑肌肌动蛋白(α-smooth muscle actin, α-SMA)的表达水平出现明显的降低, 因此Arkadia在肾小管上皮细胞间质转化过程中有着非常重要的作用; 他们的另一项研究发现: 在肾小管间质纤维化的大鼠模型中, Smad7的mRNA水平升高, 但蛋白水平却降低, 他们认为Arkadia可以通过降解Smad7促进肾小管上皮细胞向间质细胞转化; Gai等[53]的研究也进一步证实了Arkadia可以促进间质上皮转化这一观点.
4.2.1 Arkadia与消化系肿瘤: Arkadia在消化系肿瘤的发生发展过程中发挥着重要的作用. Sharma等[54]研究发现Arkadia在鼠的结肠上皮细胞中表达量较高, 且在Arkadia缺失的小鼠患结直肠癌的风险远远大于野生型小鼠; 因此Arkadia作为一种肿瘤抑制因子可以有效的抑制结直肠癌的发生发展. 同时, 他们认为Arkadia这一抑制结直肠癌发生发展的作用主要是通过募集SnoN分子抑制TGF-β信号通路而实现的. 此外, 在20种人类肿瘤细胞中也检测到Arkadia的泛素化表达[55], 因此认为Arkadia或许参与弥漫性胃癌的发展过程.
4.2.2 Arkadia与肺部肿瘤: 体外研究[56]发现, Arkadia在肺肿瘤细胞NCI-H460中稳定表达, 且可明显地抑制该细胞的生长; 但在体外实验中, Arkadia却不能抑制小鼠肺部肿瘤的发展. 另有研究报道Arkadia可以加速乳腺癌及肺癌鼠的肿瘤转移, 其主要机制是因为Arkadia可以降解c-Ski, 从而发挥促进间质上皮转移的作用[40]; Briones-Orta等[57]利用3种不同的肿瘤细胞进行了研究, 研究发现Arkadia被干扰之后, 肿瘤细胞在肺内的定植明显受到抑制, 他们得出这样的结论: Arkadia虽不能调控肿瘤的生长, 但在肿瘤细胞的定植及早期转移过程中却有着举足轻重的作用.
哮喘是一种气道慢性炎性疾病, 反复发作的炎症反应致使气管重塑, 主要表现为上皮下纤维化形成、肌细胞增生、上皮损伤、杯状细胞化生、水肿及新生血管增加. TGF-β作为一种致纤维化因子, 而Arkadia作为TGF-β信号通路的调控因子, 在气管重塑过程中发挥着重要的作用. Li等[58]发现Arkadia通过降低Smad7、SnoN/Ski蛋白表达可以显著增强TGF-β信号通路, 进而促进气管重塑. 他们的研究结果预示着Arkadia或许可以作为气管重塑治疗的新的切入点, 但是具体的机制还有待于更深入的研究.
Arkadia具有增强TGF-β信号通路转录活性, 继而放大TGF-β信号通路的作用; 还可以通过降解Smad6分子起到增强BMPs/Smad信号通路的作用, 同时也可以发挥调控EGF信号通路的作用. 此外, Arkadia在人类多种疾病的发生发展过程中都发挥着举足轻重的作用. 以上结果预示着Arkadia或许可以作为疾病治疗的新靶点, 但是其在疾病发生发展中的具体作用还有待于进一步的深入研究.
目前的研究报道, Arkadia可以特异性泛素化降解细胞内蛋白, 可调控细胞凋亡、转录调控、细胞周期、DNA损伤修复以及免疫应答等多种细胞活动.
魏继福, 研究员, 江苏省人民医院
Arkadia在纤维化疾病、肿瘤的发生发展及转移、气管重塑等多种疾病发病过程中都发挥着重要的作用, 或许可以作为疾病治疗的新靶点.
本文系统阐释了Arkadia的基本特征、生物学功能及其在维化疾病、肿瘤的发生发展及转移、气管重塑等发病过程中的作用.
Arkadia在人类多种疾病的发生发展过程中都发挥着举足轻重的作用, 预示着Arkadia或许可以作为疾病治疗的新靶点.
HECT: E6AP羧基末端的同源物, E6AP可以间接和肿瘤抑制因子p53结合, 导致p53蛋白的泛素化;
RING-finger结构域: 是一种N-末端规则酶E3α, 这类酶通常与N-末端具有某些碱性氨基酸残基(如精氨酸、赖氨酸)或与N-末端具有大量的疏水氨基酸残基(如苯丙氨基酸、亮氨酸)的蛋白质结合.
本文对环状泛素连接酶Arkadia的结构功能及其在疾病发展中的作用进行了研究, 这将有助于研发能辅助治疗相关疾病的药物, 尤其是对辅助治疗恶性肿瘤有重要意义.
手稿来源: 邀请约稿
学科分类: 胃肠病学和肝病学
手稿来源地: 北京市
同行评议报告分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): 0
D级 (一般): C
E级 (差): 0
编辑: 于明茜 电编:胡珊
| 1. | Zheng N, Wang Z, Wei W. Ubiquitination-mediated degradation of cell cycle-related proteins by F-box proteins. Int J Biochem Cell Biol. 2016;73:99-110. [PubMed] [DOI] |
| 2. | Poulsen SL, Hansen RK, Wagner SA, van Cuijk L, van Belle GJ, Streicher W, Wikström M, Choudhary C, Houtsmuller AB, Marteijn JA. RNF111/Arkadia is a SUMO-targeted ubiquitin ligase that facilitates the DNA damage response. J Cell Biol. 2013;201:797-807. [PubMed] [DOI] |
| 3. | Eldridge AG, O'Brien T. Therapeutic strategies within the ubiquitin proteasome system. Cell Death Differ. 2010;17:4-13. [PubMed] [DOI] |
| 4. | Husnjak K, Dikic I. Ubiquitin-binding proteins: decoders of ubiquitin-mediated cellular functions. Annu Rev Biochem. 2012;81:291-322. [PubMed] [DOI] |
| 5. | Lecker SH, Goldberg AL, Mitch WE. Protein degradation by the ubiquitin-proteasome pathway in normal and disease states. J Am Soc Nephrol. 2006;17:1807-1819. [PubMed] [DOI] |
| 6. | Popovic D, Vucic D, Dikic I. Ubiquitination in disease pathogenesis and treatment. Nat Med. 2014;20:1242-1253. [PubMed] [DOI] |
| 7. | Pickart CM, Eddins MJ. Ubiquitin: structures, functions, mechanisms. Biochim Biophys Acta. 2004;1695:55-72. [PubMed] [DOI] |
| 8. | Komander D. The emerging complexity of protein ubiquitination. Biochem Soc Trans. 2009;37:937-953. [PubMed] [DOI] |
| 10. | Pickart CM. Mechanisms underlying ubiquitination. Annu Rev Biochem. 2001;70:503-533. [PubMed] [DOI] |
| 11. | Hershko A, Ciechanover A. The ubiquitin system for protein degradation. Annu Rev Biochem. 1992;61:761-807. [PubMed] [DOI] |
| 12. | Varshavsky A. The ubiquitin system, an immense realm. Annu Rev Biochem. 2012;81:167-176. [PubMed] [DOI] |
| 13. | Hershko A, Ciechanover A. The ubiquitin system. Annu Rev Biochem. 1998;67:425-479. [PubMed] [DOI] |
| 14. | Deshaies RJ, Joazeiro CA. RING domain E3 ubiquitin ligases. Annu Rev Biochem. 2009;78:399-434. [PubMed] [DOI] |
| 15. | Berndsen CE, Wolberger C. New insights into ubiquitin E3 ligase mechanism. Nat Struct Mol Biol. 2014;21:301-307. [PubMed] [DOI] |
| 16. | Komander D, Rape M. The ubiquitin code. Annu Rev Biochem. 2012;81:203-229. [PubMed] [DOI] |
| 17. | Grice GL, Nathan JA. The recognition of ubiquitinated proteins by the proteasome. Cell Mol Life Sci. 2016;73:3497-3506. [PubMed] [DOI] |
| 18. | Peth A, Uchiki T, Goldberg AL. ATP-dependent steps in the binding of ubiquitin conjugates to the 26S proteasome that commit to degradation. Mol Cell. 2010;40:671-681. [PubMed] [DOI] |
| 19. | Ciechanover A. The ubiquitin-proteasome pathway: on protein death and cell life. EMBO J. 1998;17:7151-7160. [PubMed] [DOI] |
| 20. | Suresh B, Lee J, Kim KS, Ramakrishna S. The Importance of Ubiquitination and Deubiquitination in Cellular Reprogramming. Stem Cells Int. 2016;2016:1-14. [PubMed] [DOI] |
| 21. | Sun SC. Deubiquitylation and regulation of the immune response. Nat Rev Immunol. 2008;8:501-511. [PubMed] [DOI] |
| 22. | Heride C, Urbé S, Clague MJ. Ubiquitin code assembly and disassembly. Curr Biol. 2014;24:R215-R220. [PubMed] [DOI] |
| 23. | Goru SK, Pandey A, Gaikwad AB. E3 ubiquitin ligases as novel targets for inflammatory diseases. Pharmacol Res. 2016;106:1-9. [PubMed] [DOI] |
| 24. | Li W, Bengtson MH, Ulbrich A, Matsuda A, Reddy VA, Orth A, Chanda SK, Batalov S, Joazeiro CA. Genome-wide and functional annotation of human E3 ubiquitin ligases identifies MULAN, a mitochondrial E3 that regulates the organelle's dynamics and signaling. PLoS One. 2008;3:e1487. [PubMed] [DOI] |
| 25. | Metzger MB, Hristova VA, Weissman AM. HECT and RING finger families of E3 ubiquitin ligases at a glance. J Cell Sci. 2012;125:531-537. [PubMed] [DOI] |
| 26. | Ardley HC, Robinson PA. E3 ubiquitin ligases. Essays Biochem. 2005;41:15-30. [PubMed] [DOI] |
| 27. | Nakayama KI, Nakayama K. Ubiquitin ligases: cell-cycle control and cancer. Nat Rev Cancer. 2006;6:369-381. [PubMed] [DOI] |
| 28. | Erker Y, Neyret-Kahn H, Seeler JS, Dejean A, Atfi A, Levy L. Arkadia, a novel SUMO-targeted ubiquitin ligase involved in PML degradation. Mol Cell Biol. 2013;33:2163-2177. [PubMed] [DOI] |
| 29. | Koinuma D, Shinozaki M, Komuro A, Goto K, Saitoh M, Hanyu A, Ebina M, Nukiwa T, Miyazawa K, Imamura T. Arkadia amplifies TGF-beta superfamily signalling through degradation of Smad7. EMBO J. 2003;22:6458-6470. [PubMed] [DOI] |
| 30. | Niederländer C, Walsh JJ, Episkopou V, Jones CM. Arkadia enhances nodal-related signalling to induce mesendoderm. Nature. 2001;410:830-834. [PubMed] [DOI] |
| 31. | Metzger MB, Pruneda JN, Klevit RE, Weissman AM. RING-type E3 ligases: master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination. Biochim Biophys Acta. 2014;1843:47-60. [PubMed] [DOI] |
| 32. | Budhidarmo R, Nakatani Y, Day CL. RINGs hold the key to ubiquitin transfer. Trends Biochem Sci. 2012;37:58-65. [PubMed] [DOI] |
| 33. | Wright JD, Mace PD, Day CL. Secondary ubiquitin-RING docking enhances Arkadia and Ark2C E3 ligase activity. Nat Struct Mol Biol. 2016;23:45-52. [PubMed] [DOI] |
| 34. | Chasapis CT, Loutsidou AK, Orkoula MG, Spyroulias GA. Zinc Binding Properties of Engineered RING Finger Domain of Arkadia E3 Ubiquitin Ligase. Bioinorg Chem Appl. 2010;pii:323152. [PubMed] [DOI] |
| 35. | Capili AD, Edghill EL, Wu K, Borden KL. Structure of the C-terminal RING finger from a RING-IBR-RING/TRIAD motif reveals a novel zinc-binding domain distinct from a RING. J Mol Biol. 2004;340:1117-1129. [PubMed] [DOI] |
| 36. | Episkopou V, Arkell R, Timmons PM, Walsh JJ, Andrew RL, Swan D. Induction of the mammalian node requires Arkadia function in the extraembryonic lineages. Nature. 2001;410:825-830. [PubMed] [DOI] |
| 37. | Mavrakis KJ, Andrew RL, Lee KL, Petropoulou C, Dixon JE, Navaratnam N, Norris DP, Episkopou V. Arkadia enhances Nodal/TGF-beta signaling by coupling phospho-Smad2/3 activity and turnover. PLoS Biol. 2007;5:e67. [PubMed] [DOI] |
| 38. | Nagano Y, Mavrakis KJ, Lee KL, Fujii T, Koinuma D, Sase H, Yuki K, Isogaya K, Saitoh M, Imamura T. Arkadia induces degradation of SnoN and c-Ski to enhance transforming growth factor-beta signaling. J Biol Chem. 2007;282:20492-20501. [PubMed] [DOI] |
| 39. | Levy L, Howell M, Das D, Harkin S, Episkopou V, Hill CS. Arkadia activates Smad3/Smad4-dependent transcription by triggering signal-induced SnoN degradation. Mol Cell Biol. 2007;27:6068-6083. [PubMed] [DOI] |
| 40. | Le Scolan E, Zhu Q, Wang L, Bandyopadhyay A, Javelaud D, Mauviel A, Sun L, Luo K. Transforming growth factor-beta suppresses the ability of Ski to inhibit tumor metastasis by inducing its degradation. Cancer Res. 2008;68:3277-3285. [PubMed] [DOI] |
| 41. | Wozney JM. The bone morphogenetic protein family and osteogenesis. Mol Reprod Dev. 1992;32:160-167. [PubMed] [DOI] |
| 43. | Weiskirchen R, Meurer SK. BMP-7 counteracting TGF-beta1 activities in organ fibrosis. Front Biosci (Landmark Ed). 2013;18:1407-1434. [PubMed] [DOI] |
| 44. | Miyazono K, Kamiya Y, Morikawa M. Bone morphogenetic protein receptors and signal transduction. J Biochem. 2010;147:35-51. [PubMed] [DOI] |
| 45. | Miyazono K. Signal transduction by bone morphogenetic protein receptors: functional roles of Smad proteins. Bone. 1999;25:91-93. [PubMed] [DOI] |
| 46. | Nohe A, Keating E, Knaus P, Petersen NO. Signal transduction of bone morphogenetic protein receptors. Cell Signal. 2004;16:291-299. [PubMed] [DOI] |
| 47. | Yamashita H, Miyazono K. [Bone morphogenetic protein (BMP) receptors and signal transduction]. Nihon Rinsho. 1999;57:220-226. [PubMed] |
| 48. | Tsubakihara Y, Hikita A, Yamamoto S, Matsushita S, Matsushita N, Oshima Y, Miyazawa K, Imamura T. Arkadia enhances BMP signalling through ubiquitylation and degradation of Smad6. J Biochem. 2015;158:61-71. [PubMed] [DOI] |
| 49. | Miyazono K, Koinuma D. Arkadia--beyond the TGF-β pathway. J Biochem. 2011;149:1-3. [PubMed] [DOI] |
| 50. | Mizutani A, Saitoh M, Imamura T, Miyazawa K, Miyazono K. Arkadia complexes with clathrin adaptor AP2 and regulates EGF signalling. J Biochem. 2010;148:733-741. [PubMed] [DOI] |
| 51. | Liu FY, Li XZ, Peng YM, Liu H, Liu YH. Arkadia regulates TGF-beta signaling during renal tubular epithelial to mesenchymal cell transition. Kidney Int. 2008;73:588-594. [PubMed] [DOI] |
| 52. | Liu FY, Li XZ, Peng YM, Liu H, Liu YH. Arkadia-Smad7-mediated positive regulation of TGF-beta signaling in a rat model of tubulointerstitial fibrosis. Am J Nephrol. 2007;27:176-183. [PubMed] |
| 53. | Gai Z, Zhou G, Gui T, Itoh S, Oikawa K, Uetani K, Muragaki Y. Trps1 haploinsufficiency promotes renal fibrosis by increasing Arkadia expression. J Am Soc Nephrol. 2010;21:1468-1476. [PubMed] [DOI] |
| 54. | Sharma V, Antonacopoulou AG, Tanaka S, Panoutsopoulos AA, Bravou V, Kalofonos HP, Episkopou V. Enhancement of TGF-β signaling responses by the E3 ubiquitin ligase Arkadia provides tumor suppression in colorectal cancer. Cancer Res. 2011;71:6438-6449. [PubMed] [DOI] |
| 55. | Nagano Y, Koinuma D, Miyazawa K, Miyazono K. Context-dependent regulation of the expression of c-Ski protein by Arkadia in human cancer cells. J Biochem. 2010;147:545-554. [PubMed] [DOI] |
| 56. | Chen H, Yang T, Lei Z, Wang L, Yang H, Tong X, Yang WT, Zhao J, Gu Y, Chen Y. RNF111/Arkadia is regulated by DNA methylation and affects TGF-β/Smad signaling associated invasion in NSCLC cells. Lung Cancer. 2015;90:32-40. [PubMed] [DOI] |
| 57. | Briones-Orta MA, Levy L, Madsen CD, Das D, Erker Y, Sahai E, Hill CS. Arkadia regulates tumor metastasis by modulation of the TGF-β pathway. Cancer Res. 2013;73:1800-1810. [PubMed] [DOI] |
| 58. | Li XZ, Feng JT, Hu CP, Chen ZQ, Gu QH, Nie HP. Effects of Arkadia on airway remodeling through enhancing TGF-beta signaling in allergic rats. Lab Invest. 2010;90:997-1003. [PubMed] [DOI] |