修回日期: 2016-04-19
接受日期: 2016-04-23
在线出版日期: 2016-06-08
目的: 研究慢性乙型肝炎发病的分子机制.
方法: 通过基因表达谱芯片, 运用GeneSpring软件筛选慢性乙型肝炎患者的差异表达基因; 运用GeneTrail软件, 对差异表达基因进行信号通路富集分析.
结果: 发现417个差异表达基因, 其中表达上调的基因数目为205个, 表达下调的基因数目为212个; 下调基因的显著性信号通路为ErbB、非小细胞肺癌、mTOR、RNA降解、T细胞受体、慢性粒细胞白血病、肾细胞癌信号通路, 上调基因的显著性信号通路为趋化因子、溶酶体、霍乱弧菌感染、IgG Fc受体介导的吞噬作用信号通路.
结论: PI3K/AKT下调可能是乙型肝炎患者持续感染的主要分子机制之一.
核心提示: 使用基因芯片及生物信息学分析等方法研究慢性乙型肝炎的发病分子机制, 提出PI3K/AKT下调可能是乙型肝炎病毒患者持续感染的主要分子机制之一. 差异表达基因PTPRC与T细胞功能是乙型肝炎患者分子免疫机制研究的新方向之一.
引文著录: 宁文锋, 王芳, 邓宏军, 陈宏辉. 慢性乙型肝炎患者的差异表达基因筛选及相关信号通路预测. 世界华人消化杂志 2016; 24(16): 2485-2491
Revised: April 19, 2016
Accepted: April 23, 2016
Published online: June 8, 2016
AIM: To study the molecular mechanism of pathogenesis of chronic hepatitis B.
METHODS: Based on microarray experiment, GeneSpring software was used to screen differentially expressed genes in chronic hepatitis B patients, and GeneTrail software was used to perform enrichment analysis of related biological pathways.
RESULTS: A total of 417 differentially expressed genes were identified, of which 205 were upregulated and 212 downregulated. Significant pathways to which downregulated genes belong include ErbB, non-small cell lung cancer, mTOR, RNA degradation, T cell receptor, chronic myeloid leukemia, and renal cell carcinoma pathways. Significant pathways to which upregulated genes belong include chemokine, lysosomes, Vibrio cholerae infection, and IgG Fc receptor-mediated phagocytosis pathways.
CONCLUSION: PI3K/AKT downregulation is likely a major molecular mechanism of persistent hepatitis B.
- Citation: Ning WF, Wang F, Deng HJ, Chen HH. Screening of differentially expressed genes in chronic hepatitis B patients and prediction of related biological pathways. Shijie Huaren Xiaohua Zazhi 2016; 24(16): 2485-2491
- URL: https://www.wjgnet.com/1009-3079/full/v24/i16/2485.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v24.i16.2485
乙型肝炎病毒(hepatitis B virus, HBV)是一种嗜肝DNA病毒, 基因组长约3.2 kb, 为部分双链环状DNA[1]. HBV感染了约20亿人口, 约5%的成年人和95%的新生儿感染HBV后发展为慢性乙型肝炎(chronic hepatitis B, CHB)[2,3], WHO报告全球现有3.5亿CHB患者, 约25%-35%的CHB患者死于肝癌和肝硬化[4,5]. 目前临床治疗乙型肝炎的药物干扰素及核苷类似物主要作用为抑制HBV, 不能清除cccDNA, 造成HBV感染的持续存在, HBV的慢性化机制是目前的研究热点之一[6].
乙型肝炎的慢性化与人的遗传易感性、HBV的复制、免疫功能缺陷等多种因素有关. 主要组织相容性复合体Ⅱ(major histocompatibility complex Ⅱ, MHC Ⅱ)与细胞因子受体的基因多态性与乙型肝炎的持续感染有关; HBV特异性的复制机制及HBV DNA聚合酶纠错功能差容易产生变异的HBV都有利于逃避机体的免疫监视、免疫应答, 造成HBV的持续感染; T淋巴细胞持续暴露于高浓度的HBV抗原, 乙型肝炎特异性的CD8+ T淋巴细胞功能缺陷是乙型肝炎持续感染的主要免疫因素, 其分子机制与细胞膜高表达PD-1、LAG-3、Tim-3等抑制性分子有关[7,8]. HBV本身不引起肝细胞病变, 乙型肝炎患者的临床表现是HBV与宿主免疫系统相互作用的结果, 宿主的免疫及炎症反应是清除HBV和介导肝炎、肝硬化及肝癌发生和发展的主要病理生理机制[9]. 病毒性乙型肝炎研究由于HBV的易变异性、宿主免疫状态的动态变化, 以及HBV种属特异性原因导致研究模型限制等的原因, 目前CHB的发病分子机制研究仍有一定的局限性[10].
基因表达谱芯片能同时检测上万个基因mRNA的表达种类及丰度, 高通量分析基因之间变化的相互关系, 能够在基因组的层面对基因表达谱进行研究, 研究结果有一定的代表性和普遍性[11]. 但如何解读基因芯片实验结果产生的成千上万个基因的表达信息, 将无机的信息数据与有机的生命活动联系起来, 阐释生命特征和规律以及基因的功能, 是生物学研究难点之一[12,13]. 本研究通过基因芯片研究, 筛选CHB患者的差异表达基因, 并通过生物信息学软件对差异表达基因进行信号通路的富集分析, 从而探讨CHB的主要发病分子机制.
选择南华大学附属第二医院2010-08/2013-07门诊CHB患者, 共收集9例CHB患者, CHB患者组男性5例, 女性4例, 平均年龄32.3岁±10.7岁, 平均总胆红素(total bilirubin, TBIL)57.2 μmol/L(41-67 μmol/L), 平均丙氨酸氨基转移酶(alanine transaminase, ALT)92 IU/mL(45-221 IU/mL). 纳入标准: 确诊为CHB患者. 排除标准: 所有患者均排除合并肝癌、肝硬化、肝功能衰竭, 排除其他肝炎病毒混合感染、无自身免疫性疾病、无肝毒性药物使用史、未使用核苷类抗HBV药物和干扰素等免疫调节剂. 各型肝病的诊断符合中华医学会制订的诊断标准[14,15]. 另选取南华大学附属第二医院同期5例健康体检者为对照组, 男性3例, 女性2例, 平均年龄32.6岁±10.3岁, 健康对照组均无明确的甲型、乙型、丙型、丁型、戊型肝炎病毒以及巨细胞病毒、EB病毒等感染史. 两组在性别、年龄等一般资料均无统计学差异(P>0.05). RNeasy mini试剂盒、RNeasy Total RNA Isolation kit购自德国QIAGEN公司; GeneChip Scanner 3000、基因芯片购自美国Affymetrix公司; cDNA Synthesis Kit购自美国Promega公司.
1.2.1 细胞RNA的提取: 采集9例CHB患者及5例正常对照静脉血5 mL, 分离外周血单核细胞. TRIzol一步法提取细胞中的总RNA, 异丙醇沉淀法浓缩RNA, 采用RNeasy mini试剂盒对总RNA进行过柱纯化, 分光光度计定量, 甲醛变性胶电泳质检.
1.2.2 样品RNA标记及芯片杂交: 取5 μg总RNA, cDNA Synthesis Kit合成双链cDNA并进行纯化. 用Genechip IVT Labeling kit合成生物素标记的cRNA, 并按RNeasy Total RNA Isolation kit提供试剂盒操作手册对生物素标记cRNA进行纯化. cRNA片段化后与Affymetrix U133 Plus 2.0表达谱芯片在Hybridization Oven 640中45 ℃杂交16 h.
1.2.3 芯片图像扫描与数据分析: 芯片结果采用GeneChip Scanner 3000进行扫描, Expression Console Version 1.1读取原始数据, 质控合格的数据采用GeneSpring Software 11进行归一化处理及统计分析. 采用单因素方差分析法对样本不同分组的表达信号值进行统计学分析, 组间差异倍数>2且P<0.05定义为差异表达基因.
1.2.4 生物学通路的富集分析: 将上述差异表达基因, 导入GeneTrail软件, 在KEGG数据库进行信号通路富集分析, 显著性筛选的标准为P<0.05.
1.2.5 实时荧光定量PCR: 将实验组、阴性对照组和空白对照组细胞提取总RNA并逆转录为cDNA, 以β-actin为内参进行qRT-PCR, 引物如表1. 扩增条件为: 95 ℃预变性2 min, 95 ℃变性15 s, 60 ℃退火1 min, 70 ℃延伸40 s, 共40个循环. 结果采用2-△△Ct法计算相对表达量.
| 基因名称 | 引物序列 |
| PRKCD | 上游: 5'-CCTGACTATATCGCCCCTGA-3' |
| 下游: 5'-GTCCTTGGACTCCTTGGTGA-3' | |
| PIK3R1 | 上游: 5'-AGCATTGGGACCTCACATTACACA -3' |
| 下游: 5'-ACTGGAAACACAGTCCATGCACATA-3' | |
| PTPRC | 上游: 5'-AGCACCCTGAGACCCTACC-3' |
| 下游: 5'-TGAAATGCCTTAATCCCCTT-3' | |
| FOXO3 | 上游: 5'-GCAAAGCAGACCCTCAAACTG-3' |
| 下游: 5'-TGAGAGCAGATTTGGCAAAGG-3' | |
| STAT3 | 上游: 5'-ACCTGCAGCAATACCATTGAC-3' |
| 下游: 5'-AAGGTGAGGGACTCAAACTGC-3' | |
| GNAI2 | 上游: 5'-GCCTACTACCTGAACGACCT-3' |
| 下游: 5'-ATGATGGACGTGTCTGTGAACC-3' |
统计学处理 采用SPSS19.0统计软件进行统计学分析, 各组数据以mean±SD表示. 两组数据间比较采用独立样本t检验, 以P<0.05为差异有统计学意义.
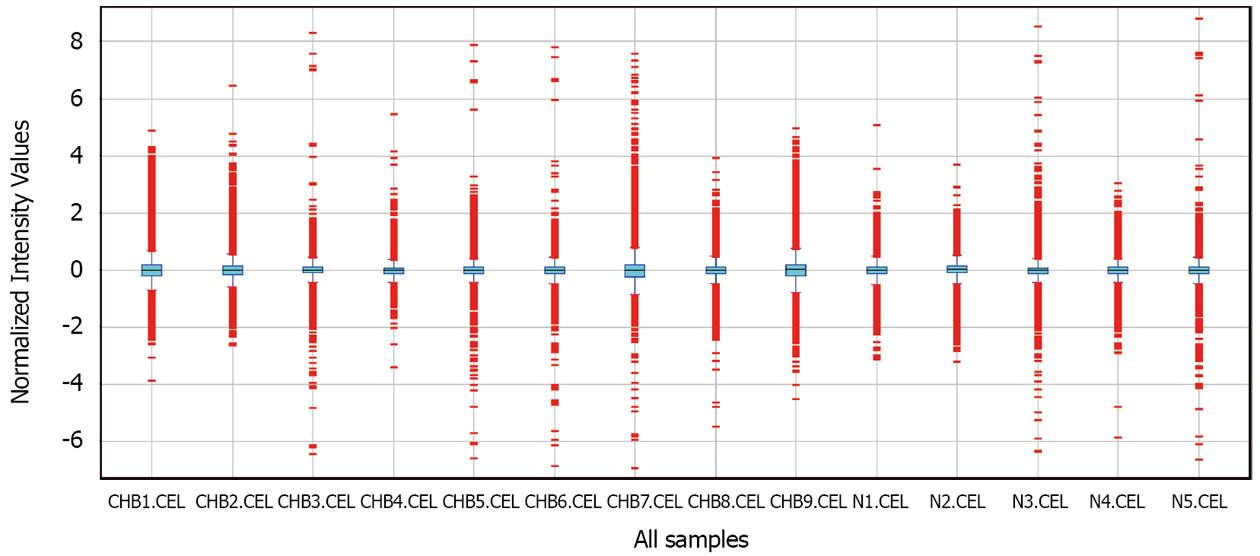
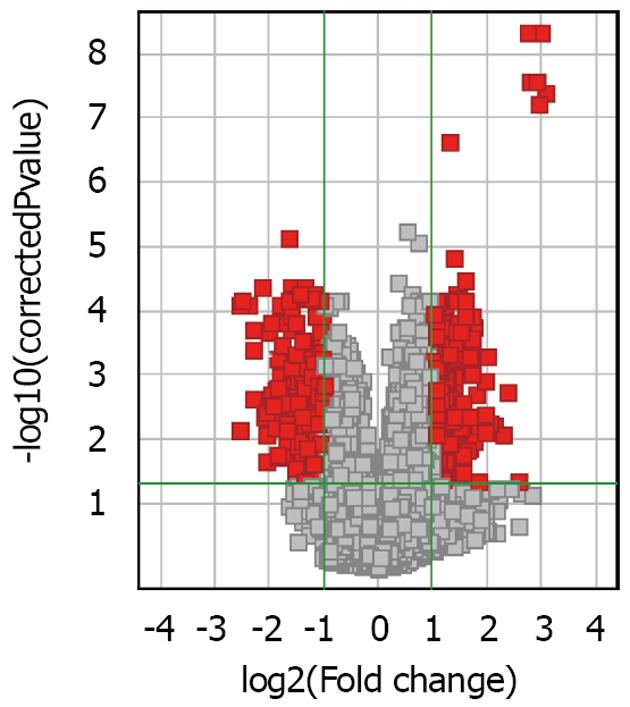
基因芯片数据归一化处理结果如图2, 基因芯片之间基因表达强度的中位数相近, 表示数据具有可比性. 差异表达基因筛选的标准为: 倍数(Fold Chang)>2或<-2、P<0.05, 显示基因的差异显著性及倍数见 "火山图"(图3).
GeneTrail软件对差异表达基因信号通路富集分析结果如表2, 3, 表的左侧为信号通路在KEGG数据库的"注释名称", 表的中间为信号通路相关的具体差异表达基因. 下调的差异表达基因被富集到7条信号通路(表2), 上调的差异表达基因被富集到4条信号通路(表3).
| 信号通路 | 基因 | P值 |
| ErbB通路 | EREG、PIK3R1、SOS2、AKT3、CBL | 0.029282 |
| 非小细胞肺癌通路 | STK4、PIK3R1、SOS2、AKT3 | 0.029282 |
| mTOR通路 | PIK3R1、AKT3、RHEB、RICTOR | 0.029282 |
| RNA降解通路 | DDX6、PAPOLA、DIS3、TTC37 | 0.030507 |
| T细胞受体信号通路 | PIK3R1、SOS2、AKT3、CBL、PTPRC | 0.037193 |
| 慢性白血病信号通路 | PIK3R1、SOS2、AKT3、CBL | 0.037765 |
| 肾细胞癌信号通路 | PIK3R1、SOS2、AKT3、ETS1 | 0.037765 |
| 信号通路 | 基因 | P值 |
| 趋化因子信号通路 | STAT1、GNB2、GNAI2、GRK6 NCF1、HCK、STAT3、FOXO3、PRKCD、ARRB1、ADCY3、JAK3 | 0.000616 |
| 溶酶体信号通路 | CTSA、CTSD、PSAP、AP3D1 ATP6V0D1、CTSB、AP1M1 | 0.030655 |
| 霍乱弧菌感染信号通路 | ACTB、ARF1、ADCY3、ATP6V0D1、ATP6V0E1 | 0.030655 |
| IgG Fc受体介导的吞噬作用通路 | PRKCD、INPP5D、NCF1、HCK ARPC4、SPHK1 | 0.032924 |
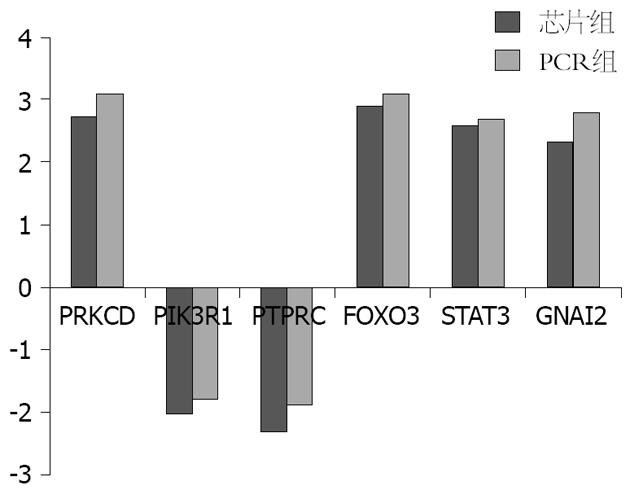
运用Real-time PCR测得验证基因的表达强度, 经过与内参的比较及转换, 得到验证基因的差异倍数, 从中可以看出PCR验证结果与芯片分析结果基本一致(图4).
基因芯片能够在基因组的层面整体而全面地分析生物学过程的改变. 本研究选用了美国Affymetrix公司生产的Human Genome U133 Plus 2.0寡核苷酸芯片, 该芯片包含了>22000个探针组, 代表14500个已知功能的人类基因[16].
GeneSpring是一套非常实用的生物芯片数据分析软件, 软件预设了基因芯片数据分析流程及参数, 其流程包括数据归一化处理、数据分组、芯片数据质量控制、探针数据质量控制、显著性差异分析、差异性倍数选择等[17]. 本研究GeneSpring软件对基因芯片数据进行归一化处理后得到约54680个转录本, 通过统计学分析, 差异有显著性的基因数目约为5420个, 差异大于2倍定义为差异表达基因[18,19], 差异表达基因数目为417个, 其中表达上调的基因数目为205个, 表达下调的基因数目为212个.
基因功能富集分析是目前分析基因芯片数据生物功能的基本方法之一, 基因功能富集分析又称功能聚类分析[20,21]. GeneTrail是一个常用且开放的基因富集分析软件, 整合了KEGG、TRANSPATH、TRANSFAC等信号通路数据库[22], 本研究在"KEGG PATHWAY"数据库中, 运用GeneTrail软件对差异表达基因进行信号通路的富集分析, KEGG PATHWAY数据库包括细胞生化过程如代谢、膜转运、信号传递、细胞周期等信号通路信息[23]. GeneTrail软件在下调的212个差异表达基因中富集到7条信号通路(表2), 具体为: ErbB、非小细胞肺癌、mTOR、RNA降解、T细胞受体、慢性粒细胞白血病、肾细胞癌等信号通路有显著性差异.
从表2中各信号通路及相关的差异基因可发现除"RNA降解"信号通路以外, 其他信号通路都与PI3K/AKT信号通路相关, 可见PI3K/AKT信号通路是下调差异表达基因的共同信号通路, 提示PI3K/AKT信号通路可能是CHB发病机制的中心环节之一.
PI3K/AKT是一经典的促癌信号通路, 与人体多种肿瘤的发生发展密切相关, 同时也与病毒感染有关[24,25], 近期研究发现: 抑制PI3K/AKT信号通路, 可以促进HBV的转录与复制, 该机制与AKT磷酸化核转录因子HNF4α后减少HNF4α核定位和与HBV启动子区域结合等密切相关[26]. 同时Li等[27]研究发现PI3K/AKT下调可促进CHB患者T细胞PD-1分子的表达, PD-1的表达是功能缺陷性T细胞的标志性分子.
本研究的差异表达基因分析显示PI3K、AKT在CHB患者中下调, 基因功能富集分析提示PI3K/AKT信号通路可能是CHB发病机制的中心环节之一, 结合现有的研究发现PI3K/AKT的抑制病毒表达、抑制T细胞免疫等作用, 提示PI3K/AKT下调可能是乙型肝炎患者持续感染的主要环节之一.
下调的差异表达基因信号通路也提示"T细胞受体信号通路的下调", 其中的差异表达基因PTPRC是T细胞活化的协同刺激信号分子, 是T淋巴细胞发育、活化的重要调节分子, PTPRC的多态性与乙型肝炎、丙肝的易感性相关, 但具体的分子机制不明确[28], PTPRC可能是乙型肝炎免疫抑制机制研究的新位点.
对上调的差异表达基因分析结果显示(表3): 趋化因子、溶酶体、霍乱弧菌感染、IgG Fc受体介导的吞噬作用信号通路具有显著性差异. 趋化因子信号通路与炎性细胞募集有关[29], 溶酶体、IgG Fc受体介导的吞噬作用信号通路与大分子物质降解、清除病毒、抗原递呈等固有免疫反应相关[30], 上述结果提示上调差异表达基因的信号通路与机体的固有免疫为主, 提示CHB患者的固有免疫反应增强.
本研究由于样本量的原因未对CHB患者进行分组对照研究, 未考虑到乙型肝炎患者免疫状况及肝功能不同对研究结果的影响, 故不能对CHB患者的分子免疫机制有更深入的了解.
从本研究也可以发现, 在信号通路数据库内进行差异基因的生物学功能分析, 与信号通路相关的基因数量不到差异基因总数的20%(表2, 3), 遗漏了一部分有生物学意义的差异表达基因, 其原因与信号通路数据库的"注释"有关, 同时这些差异基因也可能是实验的误差所致.
通过CHB患者基因表达谱芯片及差异表达基因的生物学功能等的研究, 对CHB的发病分子机制有了进一步的了解, PI3K/AKT信号通路对于乙型肝炎的药物治疗提供了新的思路, 差异基因PTPRC与T细胞功能是乙型肝炎患者分子免疫机制研究的新方向之一.
慢性乙型肝炎(chronic hepatitis B, CHB)治疗效果不理想与CHB的发病机制研究相关, 研究受限于乙型肝炎病毒(hepatitis B virus, HBV)的易变异性、宿主免疫状态的变化、研究模型限制等因素. 基因芯片产生海量的生物学信息, 如何客观、真实、全面地反映其生物学意义是目前后基因组时代研究的难点和重点.
朱新宇, 教授, 山西医科大学第一医院传染病科
目前乙型肝炎研究的热点及重点为: 乙型肝炎致肝癌的分子机制、抗病毒药物的临床应用、乙型肝炎的免疫分子机制研究、HBV转录表达的分子机制、新的抗HBV药物靶点的选择. 开发能清除共价闭合环状DNA(covalently closed circular DNA, cccDNA)的治疗乙型肝炎药物是研究的共同方向.
Liu等报道免疫调节分子Tim-3在CHB患者免疫细胞中表达上调, 从而降低免疫细胞清除HBV的能力. Witt-Kehati等最近提出联合应用免疫调节剂、HBV抑制剂才有可能根除HBV. 路东方等提出在后基因组时代, 单一的统计模式逐渐被智能化与综合分析相结合的方式所取代.
通过生物信息学软件来预测差异表达基因的生物学功能是基因芯片研究的常用方法, 但多数研究仅仅是罗列生物信息学软件的预测结果. 本研究以"富集"到的差异表达基因为基础, 研究分析"富集"到的信号通路之间的联系, 再结合文献对"富集"到差异表达基因的生物学功能进行"二次挖掘", 发现信号通路之间的内在联系及关键的"子信号通路".
本研究对乙型肝炎的发病分子机制研究、新的药物靶点选择和开发有一定的指导作用. 也对于基因芯片数据的生物信息学分析提供了一定的经验.
归一化: 归一化(normalization)的目的是使各次/组测量或各种实验条件下的测量可以相互比较, 消除测量间的非实验差异;
信号通路富集分析: 又称功能聚类分析, 将研究的差异表达基因群"投射"到已知的生物学信息数据库, 利用分析工具进行统计分析, 每个有差异表达基因存在的信号通路返回一个p值, 小的p值表示差异表达基因在该信号通路中出现了富集.
本文研究较重要, 对乙型肝炎的研究有一定的参考价值.
编辑: 郭鹏 电编:闫晋利
| 1. | Yoshio S, Kanto T. Host-virus interactions in hepatitis B and hepatitis C infection. J Gastroenterol. 2016;51:409-420. [PubMed] [DOI] |
| 2. | Wang L, Wang K, Zou ZQ. Crosstalk between innate and adaptive immunity in hepatitis B virus infection. World J Hepatol. 2015;7:2980-2991. [PubMed] [DOI] |
| 3. | Bertoletti A, Ferrari C. Adaptive immunity in HBV infection. J Hepatol. 2016;64:S71-S83. [PubMed] [DOI] |
| 4. | Yi P, Chen R, Huang Y, Zhou RR, Fan XG. Management of mother-to-child transmission of hepatitis B virus: Propositions and challenges. J Clin Virol. 2016;77:32-39. [PubMed] [DOI] |
| 5. | Mazzanti R, Arena U, Tassi R. Hepatocellular carcinoma: Where are we? World J Exp Med. 2016;6:21-36. [PubMed] [DOI] |
| 6. | Nassal M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B. Gut. 2015;64:1972-1984. [PubMed] [DOI] |
| 7. | Liu Y, Gao LF, Liang XH, Ma CH. Role of Tim-3 in hepatitis B virus infection: An overview. World J Gastroenterol. 2016;22:2294-2303. [PubMed] [DOI] |
| 8. | Wang XL, Ren JP, Wang XQ, Wang XH, Yang SF, Xiong Y. Mutations in pre-core and basic core promoter regions of hepatitis B virus in chronic hepatitis B patients. World J Gastroenterol. 2016;22:3268-3274. [PubMed] [DOI] |
| 9. | Said ZN, Abdelwahab KS. Induced immunity against hepatitis B virus. World J Hepatol. 2015;7:1660-1670. [PubMed] [DOI] |
| 10. | Witt-Kehati D, Bitton Alaluf M, Shlomai A. Advances and Challenges in Studying Hepatitis B Virus In Vitro. Viruses. 2016;8:pii E21. [PubMed] [DOI] |
| 11. | Braun R. Systems analysis of high-throughput data. Adv Exp Med Biol. 2014;844:153-187. [PubMed] [DOI] |
| 13. | Mantione KJ, Kream RM, Kuzelova H, Ptacek R, Raboch J, Samuel JM, Stefano GB. Comparing bioinformatic gene expression profiling methods: microarray and RNA-Seq. Med Sci Monit Basic Res. 2014;20:138-142. [PubMed] [DOI] |
| 15. | 中华医学会感染病学分会肝衰竭与人工肝学组中华医学会肝病学分会重型肝病与人工肝学组, 中华医学会肝病学分会重型肝病与人工肝学组. 肝衰竭诊治指南(2012年版). 中华肝脏病杂志. 2013;21:177-183. |
| 16. | Vathipadiekal V, Wang V, Wei W, Waldron L, Drapkin R, Gillette M, Skates S, Birrer M. Creation of a Human Secretome: A Novel Composite Library of Human Secreted Proteins: Validation Using Ovarian Cancer Gene Expression Data and a Virtual Secretome Array. Clin Cancer Res. 2015;21:4960-4969. [PubMed] [DOI] |
| 17. | Dresen IM, Hüsing J, Kruse E, Boes T, Jöckel KH. Software packages for quantitative microarray-based gene expression analysis. Curr Pharm Biotechnol. 2003;4:417-437. [PubMed] [DOI] |
| 18. | Zhou W, Zhang K, Chen D, Gao P, Wang Q. Gene microarray analyses for potential biomarkers of single and recurrent venous thromboembolism. Mol Med Rep. 2015;12:7358-7366. [PubMed] [DOI] |
| 19. | He H, Cao S, Niu T, Zhou Y, Zhang L, Zeng Y, Zhu W, Wang YP, Deng HW. Network-Based Meta-Analyses of Associations of Multiple Gene Expression Profiles with Bone Mineral Density Variations in Women. PLoS One. 2016;11:e0147475. [PubMed] [DOI] |
| 20. | Curtis RK, Oresic M, Vidal-Puig A. Pathways to the analysis of microarray data. Trends Biotechnol. 2005;23:429-435. [PubMed] [DOI] |
| 21. | Bessarabova M, Ishkin A, JeBailey L, Nikolskaya T, Nikolsky Y. Knowledge-based analysis of proteomics data. BMC Bioinformatics. 2012;13 Suppl 16:S13. [PubMed] [DOI] |
| 22. | Backes C, Keller A, Kuentzer J, Kneissl B, Comtesse N, Elnakady YA, Müller R, Meese E, Lenhof HP. GeneTrail--advanced gene set enrichment analysis. Nucleic Acids Res. 2007;35:W186-W192. [PubMed] [DOI] |
| 23. | Kanehisa M, Goto S, Hattori M, Aoki-Kinoshita KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M. From genomics to chemical genomics: new developments in KEGG. Nucleic Acids Res. 2006;34:D354-D357. [PubMed] [DOI] |
| 24. | Diehl N, Schaal H. Make yourself at home: viral hijacking of the PI3K/Akt signaling pathway. Viruses. 2013;5:3192-3212. [PubMed] [DOI] |
| 25. | Zhang J, Yu XH, Yan YG, Wang C, Wang WJ. PI3K/Akt signaling in osteosarcoma. Clin Chim Acta. 2015;444:182-192. [PubMed] [DOI] |
| 26. | Rawat S, Bouchard MJ. The hepatitis B virus (HBV) HBx protein activates AKT to simultaneously regulate HBV replication and hepatocyte survival. J Virol. 2015;89:999-1012. [PubMed] [DOI] |
| 27. | Li M, Sun XH, Zhu XJ, Jin SG, Zeng ZJ, Zhou ZH, Yu Z, Gao YQ. HBcAg induces PD-1 upregulation on CD4+T cells through activation of JNK, ERK and PI3K/AKT pathways in chronic hepatitis-B-infected patients. Lab Invest. 2012;92:295-304. [PubMed] [DOI] |
| 28. | Thiel N, Zischke J, Elbasani E, Kay-Fedorov P, Messerle M. Viral interference with functions of the cellular receptor tyrosine phosphatase CD45. Viruses. 2015;7:1540-1557. [PubMed] [DOI] |
| 29. | Marra F, Tacke F. Roles for chemokines in liver disease. Gastroenterology. 2014;147:577-594.e1. [PubMed] [DOI] |
| 30. | Bournazos S, Ravetch JV. Fcγ receptor pathways during active and passive immunization. Immunol Rev. 2015;268:88-103. [PubMed] [DOI] |