修回日期: 2015-11-12
接受日期: 2015-11-17
在线出版日期: 2015-12-18
目的: 检测组蛋白去乙酰化酶3(histone deacetylases 3, HDAC3)的缺失对部分肝切除(partial hepatectomy, PH)后肝细胞的增殖、细胞周期和凋亡的影响.
方法: 通过HDAC3flox/flox小鼠和他莫昔芬(tamoxifen, TAM)诱导的ALB-CREERT工具鼠杂交, 获得TAM诱导肝脏中HDAC3敲除小鼠(ALB-CREERTHDAC3flox/flox mice). Western blot和组织学分析分别检测TAM诱导的ALB-CREERTHDAC3flox/flox小鼠肝脏中HDAC3的敲除效率以及组织学改变. 通过70% PH构建肝再生模型, 并在相应时间点取材. 溴脱氧尿苷(5-Bromo-2-deoxyuridine, BrdU)掺入实验和Ki67免疫组织化学染色评估肝细胞增殖情况. Western blot检测细胞周期蛋白(cyclins)和细胞周期蛋白依赖性激酶(cyclin-dependent-kinases, CDKs)的表达水平. 原位缺口末端标记法(terminal deoxynucleotidyl transferase-mediated dUTP nick-end labeling, TUNEL)和凋亡标志物Cleaved Caspase3蛋白表达水平反映肝细胞的凋亡.
结果: TAM诱导的ALB-CREERTHDAC3flox/flox小鼠(HDAC3△小鼠)肝脏中HDAC3表达水平显著降低, 但其肝脏结构和代谢并不发生明显改变. BrdU和Ki67免疫组织化学染色结果显示70% PH后36 h和48 h肝细胞增殖率明显降低. Western blot显示HDAC3△小鼠cyclin B和cyclin D表达水平显著降低而CDK2和CDK4的表达水平无显著差异, 组织学及Western blot均提示肝再生期间肝细胞凋亡增多.
结论: HDAC3在小鼠肝再生中起至关重要的作用, HDAC3的缺失抑制肝再生并引起肝细胞凋亡.
核心提示: 本研究通过构建组蛋白去乙酰化酶3(histone deacetylases 3, HDAC3)可诱导条件性敲除小鼠, 采用70%肝切除模型研究肝再生, 检测HDAC3缺失与细胞周期及细胞凋亡的关系, 从而研究HDAC3缺失对小鼠肝再生和肝细胞凋亡的作用.
引文著录: 张蕾, 卢徐锋, 纪洪杰, 周永杰, 武振汝, 李丽, 陈菲, 步宏, 石毓君. HDAC3缺失抑制小鼠肝再生并引起肝细胞凋亡. 世界华人消化杂志 2015; 23(35): 5597-5605
Revised: November 12, 2015
Accepted: November 17, 2015
Published online: December 18, 2015
AIM: To investigate the role of histone deacetylase 3 (HDAC3) in liver regeneration followed by partial hepatectomy (PH).
METHODS: HDAC3 floxed mice were crossed with a tamoxifen-inducible ALB-CREERT allele to generate inducible tamoxifen-dependent liver-specific HDAC3 knock-out mice (ALB-CREERTHDAC3flox/flox mice). The efficiency of HDAC3 recombination and histological changes after tamoxifen treatment in adult ALB-CREERTHDAC3flox/flox mice were determined. Liver regeneration was induced by a 70% PH and mice were sacrificed at various time points after surgery. 5-Bromo-2-deoxyuridine (BrdU) incorporation and Ki67 immunohistochemistry were performed to observe the mitotic progression. The expression of mitotic markers cyclins and cyclin-dependent-kinases (CDKs) was detected by Western blot. Apoptosis of hepatocytes was analyzed by terminal deoxynucleotidyl transferase-mediated dUTP nick-end labeling (TUNEL) assay and immunoblotting of cleaved Caspase3.
RESULTS: HDAC3 in tamoxifen-treated ALB-CREERTHDAC3flox/flox mice (HDAC3△ mice) was efficiently deleted. HDAC3△ mice did not show any apparent changes in liver histology or metabolism compared to control mice without TAM injeciton. The restoration of remnant liver mass was significantly reduced in HDAC3△ mice. Dramatic lower hepatocyte mitotic figures in HDAC3△ mice determined were observed at 36-48 h after PH as indicated by BrdU and Ki67 immunohistochemistry staining. Western blot analysis confirmed that deletion of HDAC3 resulted in poor expression of cyclin B and cyclin D, whereas CDK2 and CDK4 were kept similar both in control and HDAC3△ mice at 36 h and 48 h after PH. In addition, noticeably increased apoptotic hepatocytes were found in HDAC3△ livers.
CONCLUSION: HDAC3 plays crucial roles in the regulation of liver regeneration, and loss of HDAC3 inhibits liver regeneration and induces apoptosis.
- Citation: Zhang L, Lu XF, Ji HJ, Zhou YJ, Wu ZR, Li L, Chen F, Bu H, Shi YJ. Ablation of histone deacetylase 3 in hepatocytes inhibits liver regeneration and induces apoptosis in mice. Shijie Huaren Xiaohua Zazhi 2015; 23(35): 5597-5605
- URL: https://www.wjgnet.com/1009-3079/full/v23/i35/5597.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i35.5597
组蛋白去乙酰化酶3(histone deacetylases 3, HDAC3)是HDACⅠ类家族成员, 普遍表达于生物体各组织细胞[1,2]. HDAC3通过去乙酰化作用移除核心组蛋白氨基酸末端赖氨酸残基上的乙酰基, 与基因转录活化密切相关[3,4]. HDAC3还与核激素抑制因子NCoR和SMRT形成转录抑制复合物, 与启动子的特异性位点相结合抑制基因的表达[2,5,6]. HDAC3在肝脏中能够抑制酯类合成、促进糖异生作用, 肝脏特异性敲除HDAC3严重破坏肝细胞代谢平衡并对肝细胞造成严重损伤, 最终自发形成肝细胞肝癌[7-10]. 小鼠部分肝切除(partial hepatectomy, PH)是目前研究肝再生最常用的手段[11,12], 小鼠70% PH后, 剩余的肝细胞能很快从分裂间期(G0期)进入细胞增殖周期[13,14]. 由于肝脏具有很强的再生能力, PH后48 h肝细胞增殖达到高峰, 168 h左右就能恢复到原来的肝质量[15]. 近年来, 研究[9,16]发现HDAC3对细胞周期的进程转换起至关重要的作用, 通过组蛋白的乙酰化作用改变染色质结构, 进而影响细胞周期的转换. 然而, HDAC3对小鼠肝再生过程中肝细胞周期的作用还没有相关报道. 此前报道中采用的HDAC3肝细胞特异基因敲除小鼠, 在小鼠出生后逐渐出现严重的肝细胞病变, 不适合研究其对肝细胞再生的影响. 本研究首次建立了HDAC3可诱导条件性敲除小鼠, 在成年小鼠体内敲除其肝细胞HDAC3基因, 观察HDAC3缺失对PH后肝细胞相关增殖指标、周期相关蛋白和细胞凋亡的影响.
ALB-CREERT转基因工具鼠购自上海南方模式动物研究所, HDAC3flox/flox小鼠胚胎购自奥地利维也纳兽医大学, 严格按照SPF级饲养条件进行操作. HDAC3抗体(Novus Biologicals, USA); GAPDH抗体(康成生物工程有限公司, 上海); 他莫昔芬(Sigma-Aldrich, USA); Ki67抗体、BrdU抗体(Thermo Fisher, USA); Glutamine Synthetase抗体、CyclinD抗体、cyclinB抗体、CDK2抗体、CDK4抗体(Abcam, USA); Cleaved Caspase3抗体(Cell Signaling Technology, USA); HRP标记二抗(中杉金桥生物技术有限公司, 北京); HE染色试剂盒、PAS染色试剂盒、ORO染色试剂盒(贝索生物技术有限公司, 珠海); 原位缺口末端标记法(terminal deoxynucleotidyl transferase-mediated dUTP nick-end labeling, TUNEL)凋亡检测试剂盒(Roche, 瑞士); DAB显色试剂盒(Dako, 丹麦); RIPA强裂解液(碧云天生物技术有限公司, 上海); BCA试剂盒(康为世纪生物科技有限公司, 北京); ECL发光试剂盒(PerkinElmer, USA).
1.2.1 他莫昔芬诱导肝脏中HDAC3敲除小鼠的建立: 将HDAC3flox/flox小鼠和ALB-CREERT转基因小鼠杂交, 获得他莫昔芬(tamoxifen, TAM)诱导肝脏中HDAC3敲除小鼠(ALB-CREERTHDAC3flox/flox小鼠). 通过PCR检测小鼠尾巴组织, 以确定小鼠的基因型. 取2月龄ALB-CREERTHDAC3flox/flox小鼠, 将TAM溶于玉米油中, 浓度为20 mg/mL, 每只1 mg/d, 腹腔注射, 连续给药5 d, 停药5 d待TAM被清除后, 建立肝脏特异性敲除HDAC3的小鼠(HDAC3△小鼠)用于后续实验. 对照组小鼠则给予等体积玉米油.
1.2.2 70% PH模型的建立: HDAC3△小鼠和对照组小鼠进行70% PH. 小鼠术前禁食过夜, 术中乙醚行半开放吸入持续麻醉, 采用经典的Higgins-Anderson法进行手术[6], 术后注意小鼠的保温和避免感染.
1.2.3 标本收集: 在PH后0、36、48、72、120、168 h取材. 在取材前1 h腹腔注射BrdU(浓度为10 mg/mL, 按小鼠体质量的0.1 mg/g给药). 到达相应时间点称量小鼠体质量后将其处死, 取出完整肝脏并称残肝质量. 取约0.7 cm×0.7 cm的小鼠肝组织浸泡于中性甲醛溶液中固定36 h左右, 用于组织学检测及免疫组织化学染色; 剩余肝组织部分用于冰冻切片, 其余置于液氮保存用于提取蛋白和核酸.
1.2.4 组织学分析: 将各时间点固定好的小鼠肝组织行石蜡包埋, 切成厚度为4 μm的切片, 进行HE染色和PAS染色. 对于油红O染色, 取新鲜肝组织切成6 μm厚的冰冻切片, 油红O染液中室温浸泡20 min, 苏木素复染细胞核.
1.2.5 免疫组织化学染色: 采用Envision二步法(Dako)检测肝组织石蜡切片中BrdU和Ki67的表达. 一抗浓度均为1:100, 用TBS代替一抗做为空白对照. 细胞核呈棕黄色着色者为阳性细胞. 在高倍视野(10×40)下, 肝脏汇管区周围随机选取5个视野, 计数每个视野下阳性细胞, 取其均数代表该样本的阳性细胞数n1, 总细胞数为n, 阳性细胞率 = n1/n×100%.
1.2.6 TUNEL染色: 将石蜡切片脱蜡至水化, 采用Roche公司的In-Situ Cell Death Detection Kit, AP试剂盒, 按说明书进行操作. 水溶性封片剂封片后, 在光学显微镜下采图并计数.
1.2.7 Western blot: 称取PH后各时间点肝组织, 加入RIPA强裂解液进行组织匀浆, 冰上静置1 h后离心取上清液, 用BCA法测定蛋白浓度, 取蛋白上清液加上样缓冲液(用之前加入β巯基乙醇)沸水浴5-10 min, 用12%SDS-PAGE进行电泳分离, 转膜至PVDF膜. 用含5%脱脂奶粉的TBST封闭液室温封闭60 min, 加入相应浓度的一抗, 4 ℃冰箱孵育过夜. TBST缓冲液清洗5次后加入相应的二抗, 室温孵育60 min, TBST缓冲液清洗5次后, 用胶片曝光显影, 用Image J软件对Western blot条带进行灰度值分析.
统计学处理 采用GraphPad Prism5.0软件进行统计学分析和作图, 实验数据用mean±SD表示, 两组间比较采用非配对t检验, P<0.05为差异具有统计学意义.
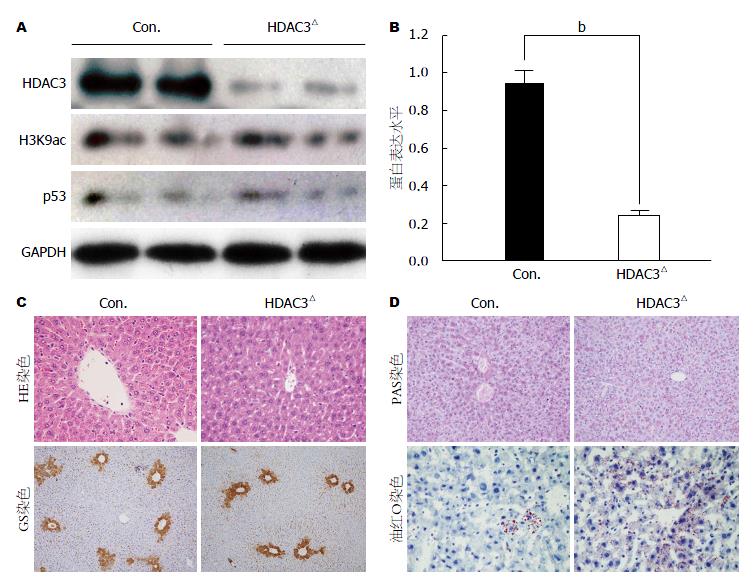
在TAM腹腔连续给药后第5天, 2月龄HDAC3△小鼠肝组织中HDAC3的蛋白表达水平降低80%(图1A, B), 说明肝细胞中的HDAC3被有效地敲除了. 而反映组蛋白乙酰化水平的H3K9ac和DNA损伤标志物p53无明显的变化(图1A), 表明HDAC3的敲除至少在短期内不影响肝细胞异染色质水平并导致DNA损伤.
已报道肝脏中特异性敲除HDAC3会严重改变肝脏糖脂代谢平衡[17], 我们进一步分析了可诱导敲除小鼠肝脏中的糖脂代谢水平. HE染色显示, HDAC3△小鼠具有正常的肝小叶结构; 同时, 谷氨酰氨合成酶(glutamine synthetase, GS)免疫组织化学显示, GS在HDAC3△小鼠肝中央静脉周围表达无区域性差异(图1C). PAS染色和油红O染色显示, HDAC3△小鼠肝脏内的糖脂代谢和对照小鼠相比并无明显改变(图1D).
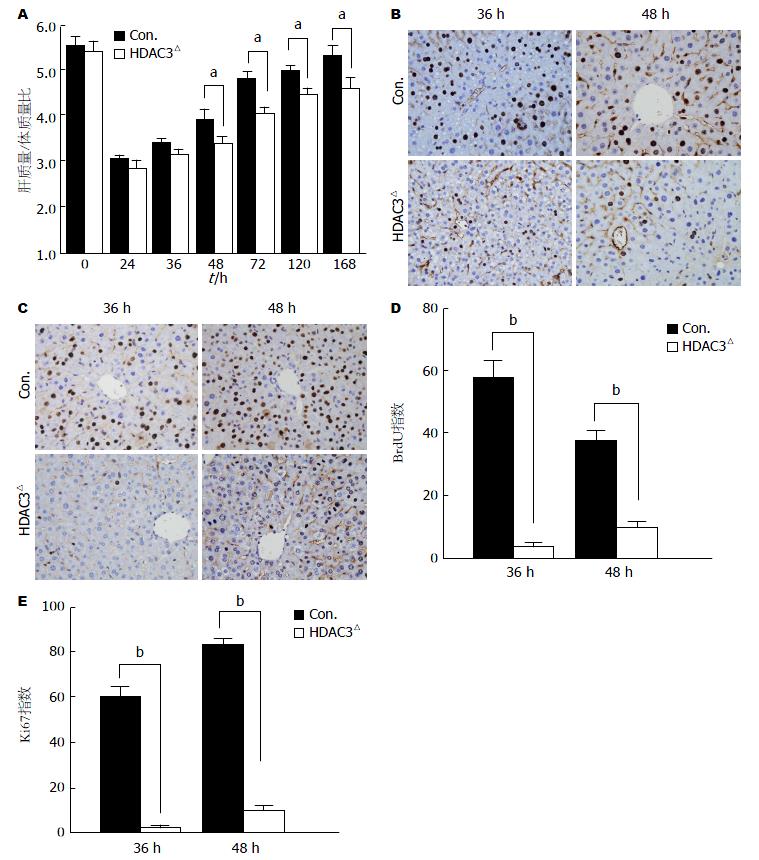
肝质量/体质量比测量显示, 在PH后48-120 h, HDAC3△小鼠肝质量/体质量比恢复水平较对照小鼠低25%左右(图2A); 在肝切后第168 h, 对照小鼠的肝脏质量已完全恢复, 而HDAC3△小鼠的肝脏质量只恢复到原来水平的80%(图2A).
PH后36 h和48 h, 即小鼠肝细胞增殖的第一个高峰期[18], BrdU和Ki67免疫组织化学染色显示HDAC3△小鼠阳性肝细胞仅分别为5%和10%, 显著低于对照小鼠, 说明HDAC3△小鼠肝再生被明显抑制(图2B-E).
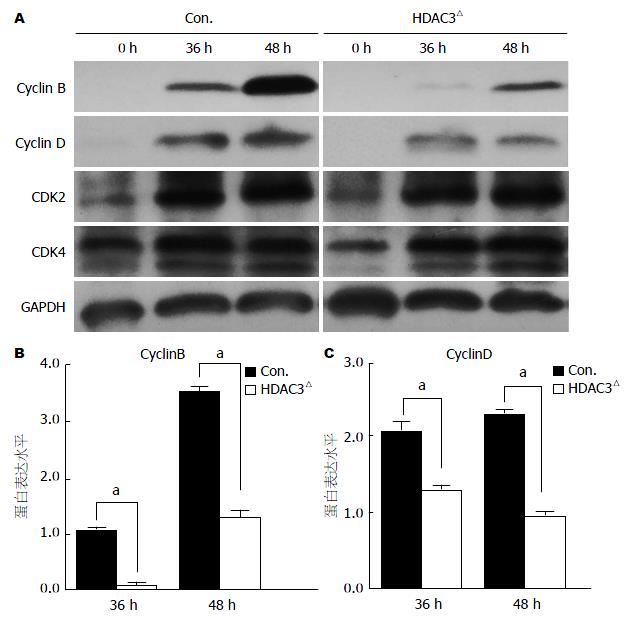
肝细胞周期相关蛋白的Western blot检测结果显示在PH后36 h和48 h, HDAC3△小鼠的G1期标志物cyclinD和G2/M期的标志物cyclinB表达水平明显下调(图3A-C), 而CDK2和CDK4的表达水平无明显差异(图3A).
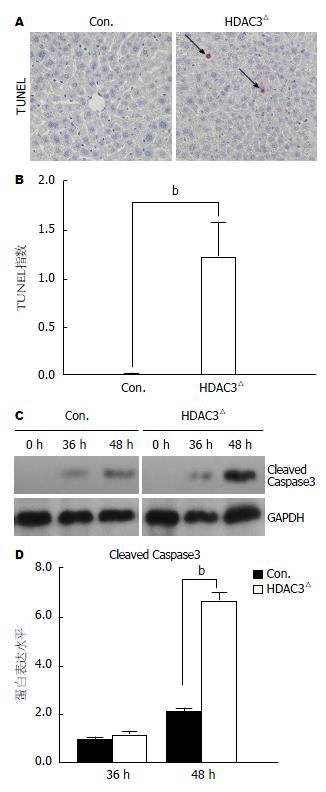
对PH 48 h后肝脏组织进行TUNEL染色, 并对阳性细胞数进行统计, 结果显示, 在对照小鼠肝脏中, 几乎看不到凋亡的肝细胞; 而HDAC3△小鼠肝脏中, 约1%的肝细胞呈TUNEL阳性(图4A, B). Western blot检测显示, HDAC3△小鼠PH 48 h后肝脏中凋亡相关蛋白Cleaved Caspase3的表达水平增加了3倍左右(图4C, D).
HDAC3在小鼠的个体发育过程中必不可少, HDAC3全身性敲除的小鼠在胚胎期9.5 d将由于原肠胚发育缺陷而死亡[7,19]. 在肝脏和心脏中, HDAC3基因的敲除引起脂类代谢水平的显著变化[10,17]. HDAC3还参与了肝脏巨噬细胞的两极化及小鼠纤维母细胞的细胞周期进程[16]. 最新的研究[9,20,21]报道显示, HDAC3可能通过调节染色体组蛋白的乙酰化水平, 参与肝细胞肝癌的形成. HDAC3低水平表达还与人肝癌恶性程度及预后密切相关[20,22,23]. 但HDAC3在肝再生中的作用还未见报道. 本研究首次构建了可诱导条件性敲除肝脏中HDAC3基因的小鼠, 在正常发育至成熟阶段的小鼠实施肝细胞特异性HDAC3基因敲除, 探讨了HDAC3缺失对肝细胞增殖、细胞周期及凋亡的影响.
此前研究中发现, 采用常规ALB-CRE小鼠与HDAC3flox/flox小鼠杂交, 其子代鼠肝细胞的HDAC3基因在出生前即被敲除, 出生后将逐渐出现严重的脂质和糖类代谢失衡[7]. 同时, 肝细胞出现DNA断裂损伤, 细胞中异染色质明显减少, 基因组稳定性减弱; 在10月龄时小鼠往往自发形成肝细胞肝癌[9,16]. 因此, 该小鼠本身存在严重而复杂的肝细胞病变, 不适于进行肝再生研究. 本研究通过TAM可诱导条件性敲除小鼠肝脏中的HDAC3, HDAC3的敲除效率高达80%, 而且该过程并不引起H3K9ac和p53表达水平的改变. 同时, 组织学检测结果显示, HDAC3可诱导性敲除也不显著影响HDAC3△小鼠肝脏的正常结构和糖脂代谢, 能更精确地研究HDAC3对肝再生的调控作用.
在多种细胞中已明确HDAC3参与了细胞周期的调控. 在小鼠胚胎纤维母细胞中, HDAC3缺失引起G0期DNA损伤, 进而抑制S期的进程[16]. 在HeLa细胞中, HDAC3的沉默会导致G1/S期转换障碍[24]. HDAC3缺失在造血干细胞中造成S期的阻断[25]. HDAC3在人类的结肠癌细胞中表达水平异常, 会导致细胞S期和G2/M期停滞[21]. 肝再生模型是研究体内细胞增殖再生的理想模型[26], 但HDAC3对肝再生的影响还未见报道. 我们的研究结果显示, PH后HDAC3△小鼠肝质量/体质量比的恢复水平显著低于对照小鼠. 在肝细胞增殖的高峰期, 进入分裂期的HDAC3△小鼠肝细胞远低于对照小鼠. 而细胞凋亡的检测结果表明再生肝脏中出现一些凋亡细胞. Ki67作为增殖标志物, 在细胞进入G1中后期开始微量表达, S期后半期表达显著增高, 持续至有丝分裂结束[27]. 而BrdU作为核苷类似物, 在S期被掺入到新合成的DNA链中[28], 因此只有进入S期的细胞才会呈现BrdU免疫组织化学染色阳性[29]. 本研究结果提示, HDAC3的缺失很可能是通过强烈抑制肝细胞S期DNA的合成, 进而减少肝细胞的增殖以及阻碍肝再生进程. 最近研究[24,30]发现, HDAC3可通过调控S/G2期转换的关键周期蛋白cyclinA乙酰化水平以及G2/M期CDK1的稳定性进而调控细胞周期转换. 在HDAC3△小鼠肝脏中, PH 36 h和48 h后, G1期中后期的cyclinD和G2/M期的cyclinB表达水平较对照小鼠明显下调, 也提示肝细胞发生S期的周期阻滞. 而CDK2和CDK4的表达水平并无显著改变, 因此HDAC3在肝再生过程中如何调控肝细胞的S期, 是否还存在新的作用位点, 还有待深入研究. 我们还观察到HDAC3△小鼠再生肝脏中出现较多的凋亡细胞. 我们此前在肝再生研究[29]中发现, 与HDAC3同家族的成员HDAC1和HDAC2缺失后, 通过抑制Ki67的表达, 导致肝细胞有丝分裂M期缺陷, 进而肝细胞发生凋亡. HDAC3敲除后发生的再生肝细胞凋亡可能也与此类似, 尚有待证实.
总之, HDAC3在小鼠的肝再生中起至关重要的作用, HDAC3缺失抑制肝再生并引起肝细胞凋亡, 而HDAC3对肝再生细胞周期的调控机制还有待于进一步的研究.
组蛋白去乙酰化酶3(histone deacetylases 3, HDAC3)在肝脏的代谢平衡和肝癌的形成中发挥着重要的作用, HDAC3能够抑制酯类合成、促进糖异生作用, HDAC3敲除会对肝细胞造成严重损伤, 最终自发形成肝细胞肝癌.
高国全, 教授, 中山大学中山医学院生物化学教研室
肝脏具有强大的再生能力, 小鼠部分肝切除是目前研究体内肝再生的理想模型. HDAC3对细胞周期的进程转换起至关重要的作用, 然而, HDAC3对小鼠肝再生过程中肝细胞周期的作用还没有相关报道.
HDAC3在细胞周期的调控起举足轻重的作用. 在小鼠胚胎纤维母细胞中, HDAC3缺失引起G0期DNA损伤, 进而抑制S期的进程. 在HeLa细胞中, HDAC3的沉默会导致G1/S期转换障碍. HDAC3缺失在造血干细胞中造成S期的阻断. HDAC3在人类的结肠癌细胞中表达水平异常, 会导致细胞S期和G2/M期停滞.
HDAC1和HDAC2在肝细胞中敲除后, 通过抑制Ki67的表达, 导致肝细胞有丝分裂M期缺陷, 进而肝细胞发生凋亡. 而作为同一家族成员的HDAC3敲除后在肝再生中的作用, 尚有待证实.
ALB-CRE; HDAC3flox/flox小鼠肝脏会出现严重糖脂代谢失衡, DNA断裂损伤以及自发形成肝癌. 由于该小鼠本身存在严重而复杂的肝细胞病变, 不适于进行肝再生研究. 本研究通过TAM可诱导条件性敲除小鼠肝脏中的HDAC3, 不影响肝脏的正常结构和糖脂代谢, 从而能更精确地研究HDAC3在肝再生中的作用.
肝再生: 肝细胞是终末分化的细胞, 绝大多数的肝细胞处于G0期, 同时, 肝脏又保持着强大的再生能力, 人以及啮齿类动物在部分肝切除后, 残留肝细胞会快速进入细胞周期, 肝实质能在短时间内完全恢复到原来的细胞数量和功能.
本文探讨了HDAC3对肝脏再生的影响, 具有一定的临床意义, 同时首次构建了可诱导条件性敲除肝脏中HDAC3基因的动物模型, 模型较为新颖, 能够为后续研究HDAC3影响肝脏再生奠定基础,从增殖、凋亡、细胞周期等方面探讨HDAC3对肝细胞的影响.
编辑: 于明茜 电编:都珍珍
| 1. | de Ruijter AJ, van Gennip AH, Caron HN, Kemp S, van Kuilenburg AB. Histone deacetylases (HDACs): characterization of the classical HDAC family. Biochem J. 2003;370:737-749. [PubMed] [DOI] |
| 2. | Moser MA, Hagelkruys A, Seiser C. Transcription and beyond: the role of mammalian class I lysine deacetylases. Chromosoma. 2014;123:67-78. [PubMed] [DOI] |
| 3. | Fischle W, Dequiedt F, Hendzel MJ, Guenther MG, Lazar MA, Voelter W, Verdin E. Enzymatic activity associated with class II HDACs is dependent on a multiprotein complex containing HDAC3 and SMRT/N-CoR. Mol Cell. 2002;9:45-57. [PubMed] [DOI] |
| 4. | Watson PJ, Fairall L, Santos GM, Schwabe JW. Structure of HDAC3 bound to co-repressor and inositol tetraphosphate. Nature. 2012;481:335-340. [PubMed] [DOI] |
| 5. | Zhang J, Kalkum M, Chait BT, Roeder RG. The N-CoR-HDAC3 nuclear receptor corepressor complex inhibits the JNK pathway through the integral subunit GPS2. Mol Cell. 2002;9:611-623. [PubMed] [DOI] |
| 6. | Boyce S, Harrison D. A detailed methodology of partial hepatectomy in the mouse. Lab Anim (NY). 2008;37:529-532. [PubMed] [DOI] |
| 7. | Knutson SK, Chyla BJ, Amann JM, Bhaskara S, Huppert SS, Hiebert SW. Liver-specific deletion of histone deacetylase 3 disrupts metabolic transcriptional networks. EMBO J. 2008;27:1017-1028. [PubMed] [DOI] |
| 8. | Montgomery RL, Potthoff MJ, Haberland M, Qi X, Matsuzaki S, Humphries KM, Richardson JA, Bassel-Duby R, Olson EN. Maintenance of cardiac energy metabolism by histone deacetylase 3 in mice. J Clin Invest. 2008;118:3588-3597. [PubMed] [DOI] |
| 9. | Bhaskara S, Knutson SK, Jiang G, Chandrasekharan MB, Wilson AJ, Zheng S, Yenamandra A, Locke K, Yuan JL, Bonine-Summers AR. Hdac3 is essential for the maintenance of chromatin structure and genome stability. Cancer Cell. 2010;18:436-447. [PubMed] [DOI] |
| 10. | Feng D, Liu T, Sun Z, Bugge A, Mullican SE, Alenghat T, Liu XS, Lazar MA. A circadian rhythm orchestrated by histone deacetylase 3 controls hepatic lipid metabolism. Science. 2011;331:1315-1319. [PubMed] [DOI] |
| 11. | Fausto N, Campbell JS, Riehle KJ. Liver regeneration. J Hepatol. 2012;57:692-694. [PubMed] [DOI] |
| 12. | Hashemi Goradel N, Darabi M, Shamsasenjan K, Ejtehadifar M, Zahedi S. Methods of Liver Stem Cell Therapy in Rodents as Models of Human Liver Regeneration in Hepatic Failure. Adv Pharm Bull. 2015;5:293-298. [PubMed] [DOI] |
| 13. | Fausto N, Campbell JS, Riehle KJ. Liver regeneration. Hepatology. 2006;43:S45-S53. [PubMed] [DOI] |
| 14. | Taub R. Liver regeneration: from myth to mechanism. Nat Rev Mol Cell Biol. 2004;5:836-847. [PubMed] [DOI] |
| 15. | Michalopoulos GK, Khan Z. Liver regeneration, growth factors, and amphiregulin. Gastroenterology. 2005;128:503-506. [PubMed] [DOI] |
| 16. | Bhaskara S, Chyla BJ, Amann JM, Knutson SK, Cortez D, Sun ZW, Hiebert SW. Deletion of histone deacetylase 3 reveals critical roles in S phase progression and DNA damage control. Mol Cell. 2008;30:61-72. [PubMed] [DOI] |
| 17. | Sun Z, Miller RA, Patel RT, Chen J, Dhir R, Wang H, Zhang D, Graham MJ, Unterman TG, Shulman GI. Hepatic Hdac3 promotes gluconeogenesis by repressing lipid synthesis and sequestration. Nat Med. 2012;18:934-942. [PubMed] [DOI] |
| 18. | Behrens A, Sibilia M, David JP, Möhle-Steinlein U, Tronche F, Schütz G, Wagner EF. Impaired postnatal hepatocyte proliferation and liver regeneration in mice lacking c-jun in the liver. EMBO J. 2002;21:1782-1790. [PubMed] [DOI] |
| 19. | Haberland M, Montgomery RL, Olson EN. The many roles of histone deacetylases in development and physiology: implications for disease and therapy. Nat Rev Genet. 2009;10:32-42. [PubMed] [DOI] |
| 20. | Wu LM, Yang Z, Zhou L, Zhang F, Xie HY, Feng XW, Wu J, Zheng SS. Identification of histone deacetylase 3 as a biomarker for tumor recurrence following liver transplantation in HBV-associated hepatocellular carcinoma. PLoS One. 2010;5:e14460. [PubMed] [DOI] |
| 21. | Wilson AJ, Byun DS, Popova N, Murray LB, L'Italien K, Sowa Y, Arango D, Velcich A, Augenlicht LH, Mariadason JM. Histone deacetylase 3 (HDAC3) and other class I HDACs regulate colon cell maturation and p21 expression and are deregulated in human colon cancer. J Biol Chem. 2006;281:13548-13558. [PubMed] [DOI] |
| 22. | Yang Z, Zhou L, Wu LM, Xie HY, Zhang F, Zheng SS. Combination of polymorphisms within the HDAC1 and HDAC3 gene predict tumor recurrence in hepatocellular carcinoma patients that have undergone transplant therapy. Clin Chem Lab Med. 2010;48:1785-1791. [PubMed] [DOI] |
| 23. | Quint K, Agaimy A, Di Fazio P, Montalbano R, Steindorf C, Jung R, Hellerbrand C, Hartmann A, Sitter H, Neureiter D. Clinical significance of histone deacetylases 1, 2, 3, and 7: HDAC2 is an independent predictor of survival in HCC. Virchows Arch. 2011;459:129-139. [PubMed] [DOI] |
| 24. | Vidal-Laliena M, Gallastegui E, Mateo F, Martínez-Balbás M, Pujol MJ, Bachs O. Histone deacetylase 3 regulates cyclin A stability. J Biol Chem. 2013;288:21096-21104. [PubMed] [DOI] |
| 25. | Summers AR, Fischer MA, Stengel KR, Zhao Y, Kaiser JF, Wells CE, Hunt A, Bhaskara S, Luzwick JW, Sampathi S. HDAC3 is essential for DNA replication in hematopoietic progenitor cells. J Clin Invest. 2013;123:3112-3123. [PubMed] [DOI] |
| 26. | Kang LI, Mars WM, Michalopoulos GK. Signals and cells involved in regulating liver regeneration. Cells. 2012;1:1261-1292. [PubMed] [DOI] |
| 27. | Gerlach C, Sakkab DY, Scholzen T, Dassler R, Alison MR, Gerdes J. Ki-67 expression during rat liver regeneration after partial hepatectomy. Hepatology. 1997;26:573-578. [PubMed] [DOI] |
| 28. | Muskhelishvili L, Latendresse JR, Kodell RL, Henderson EB. Evaluation of cell proliferation in rat tissues with BrdU, PCNA, Ki-67(MIB-5) immunohistochemistry and in situ hybridization for histone mRNA. J Histochem Cytochem. 2003;51:1681-1688. [PubMed] [DOI] |
| 29. | Xia J, Zhou Y, Ji H, Wang Y, Wu Q, Bao J, Ye F, Shi Y, Bu H. Loss of histone deacetylases 1 and 2 in hepatocytes impairs murine liver regeneration through Ki67 depletion. Hepatology. 2013;58:2089-2098. [PubMed] [DOI] |
| 30. | Jiang Y, Hsieh J. HDAC3 controls gap 2/mitosis progression in adult neural stem/progenitor cells by regulating CDK1 levels. Proc Natl Acad Sci U S A. 2014;111:13541-13546. [PubMed] [DOI] |