修回日期: 2015-10-30
接受日期: 2015-11-09
在线出版日期: 2015-12-08
目的: 本研究通过检测消化不良患者胃黏膜白色念珠菌多位点序列分型(multilocus sequence typing, MLST)状况, 探寻是否有胃黏膜炎症损伤密切相关的白色念珠菌基因型, 为研究胃黏膜炎症损伤的提供微生物学基础.
方法: 用胃镜钳取胃炎、胃溃疡患者的病损区胃黏膜标本102例, 并采集口腔黏膜拭子样本111例, 为患者组. 采集健康志愿者口腔黏膜拭子样本162例, 为正常对照组. 样本用念珠菌显示培养基进行分离培养, 初步菌种鉴定. 分离培养的念珠菌菌株样本提取DNA, ITS1-5.8S-ITS2序列检测, 进一步菌种鉴定. 将白色念珠菌菌株样本进行MLST检测, 并向MLST数据库申报ST序列号, 用UPGMA法构建进化树, 分析进化分支方向与胃黏膜炎症损伤的关系.
结果: 对照组和患者组的口腔黏膜拭子样本, 念珠菌阳性率分别为29.6%和36.0%, 白色念珠菌构成比分别为64.6%和95.0%. 患者组的胃黏膜样本中, 念珠菌阳性率为41.4%, 白色念珠菌构成比为97.8%. 患者组的念珠菌阳性率及白色念珠菌构成比均高于正常对照组(χ2 = 4.071, P<0.01; χ2 = 7.650, P = 0.006). 白色念珠菌MLST检测, ST1593基因型在患者组中检出率高于对照组(60.0% vs 14.8%; χ2 = 12.815, P<0.001); 菌株所在的不同进化分支与胃黏膜炎症损伤关系密切(Kendall's tau-b r = 0.591, P<0.001).
结论: 在消化不良患者中, 可以检出与胃黏膜炎症损伤密切相关的白色念珠菌基因型.
核心提示: 本研究通过检测胃炎、胃溃疡患者胃黏膜和口腔黏膜的白色念珠菌多位点序列分型(multilocus sequence typing)状况, 发现ST1593基因型是胃黏膜炎症损伤密切相关的白色念珠菌基因型, 这为研究胃黏膜炎症损伤的研究提供微生物学基础.
引文著录: 齐贺, 宫雁冰, 黄竹青, 刘颖, 郑剑玲, 张秀英, 车颖, 赵同香, 张蓉. 消化不良患者口腔和胃黏膜白色念珠菌多位点序列分型状况. 世界华人消化杂志 2015; 23(34): 5443-5451
Revised: October 30, 2015
Accepted: November 9, 2015
Published online: December 8, 2015
AIM: To detect the genetic profiles of Candida albicans (C. albicans) strains in the digestive tract of dyspeptic patients by multilocus sequence typing (MLST), and to explore whether lesion of the gastric mucosa is closely related to the genotype of C. albicans.
METHODS: A total of 111 oral swab samples and 102 gastric mucosa samples were collected from patients with gastritis or gastric ulcer. In addition, 162 oral swab samples collected from healthy volunteers were used as a control group. Candida species isolates from separate samples were identified by amplifying the ITS1-5.8S-ITS2 region sequence. C. albicans isolates were characterized and analyzed by multilocus sequence typing, and submitted to the C. albicans MLST database. The phylogenetic tree was constructed by the method of unweighted-pair group method using average linkages (UPGMA) to analyze the relationship between the evolutionary clades and gastric mucosal inflammation lesion.
RESULTS: In the oral mucosa swab samples of the control group and patient group, the positive rates of Candida spp. were 29.6% vs 36.0%, and the constitute ratios of C. albicans were 64.6% vs 95%, respectively. In the gastric samples of the patient group, the positive rate of Candida spp. was 41.4%, and the constituent ratio of C. albicans was 97.8%. Both the positive rate and constituent ratio of C. albicans in the patient group were significantly higher than those in the control group (χ2 = 4.071, P < 0.01; χ2 = 7.650, P = 0.006). In C. albicans MLST detection, the positive rate of genotype ST1593 was significantly higher in the patient group than in the control group (60% vs 14.8%; χ2 = 12.815, P < 0.001). The different evolutionary clades of C. albicans strains were closely related to the inflammatory lesion of the gastric mucosa (Kendall's tau-b r = 0.591, P < 0.001).
CONCLUSION: C. albicans could be detected in the gastric mucosa of patients with dyspepsia, and its special genotype is closely related to the inflammatory lesion of the gastric mucosa.
- Citation: Qi H, Gong YB, Huang ZQ, Liu Y, Zheng JL, Zhang XY, Che Y, Zhao TX, Zhang R. Multilocus sequence typing of Candida albicans isolates from oral and gastric mucosa of dyspeptic patients. Shijie Huaren Xiaohua Zazhi 2015; 23(34): 5443-5451
- URL: https://www.wjgnet.com/1009-3079/full/v23/i34/5443.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i34.5443
念珠菌被认为是人类的常见菌群, 但越来越多的报道发现念珠菌与慢性胃炎和胃溃疡有密切的关系[1-3]. 在胃溃疡活动期, 白色念珠菌的检出率达30%-50%, 并使溃疡症状加重, 并延长愈合时间[4-9]. 常有报道单一的抗幽门螺杆菌(Helicobacter pylori, H. pylori)治疗不能愈合的胃溃疡, 需经抗真菌治疗后, 溃疡才能愈合[10-12]. 甚至, 有真菌炎症性肉芽肿被误诊为胃癌的报道[13-15]. 由此, 本研究采集分析了消化不良患者口腔和胃黏膜的白色念珠菌多位点序列分型(multilocus sequence typing, MLST)状况, 探索是否有与胃黏膜炎症损伤密切相关的基因型, 及其在病理损伤过程中的变异和进化方向, 以便了解其流行病学和致病性, 为消化不良的诊断、治疗和预防研究, 提供微生物学基础.
在2008-05-01/10-29在辽宁中医药大学的师生中采集健康志愿者的口腔黏膜拭子样本, 作为正常对照组. 得到162例口腔黏膜拭子样本, 包括92例女性, 70例男性, 年龄范围4-81岁(平均年龄39.55岁±19.16岁). 在2008-07-10/09-12在辽宁中医药大学附属第一医院消化科, 采集消化不良患者及其家属的样本. 得到111例消化不良患者的口腔黏膜拭子样本(n = 111)和胃黏膜样本(n = 102), 其中39例女性, 72例男性, 年龄范围18-80岁(平均年龄47.59岁±14.94岁). 包括77例慢性胃炎, 29例消化性溃疡, 5例胃癌患者. 用胃镜采集胃黏膜样本用于病理检查、H. pylori和真菌检查. 检查前进行过化疗、糖皮质激素、抗生素治疗的患者被排除. 液体沙氏培养基、念珠菌显色培养基, 北京奥博星生物技术有限责任公司; DNA提取试剂盒, 北京艾比根生物技术有限公司; PCR Super Mix试剂盒, 北京全式金生物技术有限公司; 测序, 北京擎科新业生物技术有限公司.
1.2.1 念珠菌分离培养: 口腔拭子样本接种于液体沙氏培养基中, 37 ℃ 150 r/min振荡培养24 h, 菌液浑浊者为真菌培养阳性. 将菌液接种于念珠菌显色培养基中, 30 ℃培养48 h, 根据菌落颜色进行菌种初步鉴定, 绿色菌落为白色念珠菌[16]. 每个平皿挑取2个菌落, 增菌保存.
1.2.2 菌种鉴别: 每个菌株用3 mL液体沙氏培养基, 37 ℃ 150 r/min振荡培养24 h增菌, 根据厂家说明书提取DNA. 进行ITS1-5.8S-ITS2序列PCR扩增[17], 用PCR SuperMix试剂盒进行扩增, 引物序列(ITS1: TCCGTAGGTGAACCTGCGG; ITS4: TCCTCCGCTTATTGATATGC). PCR产物进行测序. ITS1-5.8S-ITS2序列检测结果提交到GenBank网站, 注册序列号, 做菌种鉴定.
1.2.3 MLST检测及进化树构建: 鉴定为白色念珠菌的菌株DNA, 进行MLST检测, 方法参照文献[18]. DNA序列检测结果提交到MLST database 网站(http://pubmlst.org/calbicans/), 注册基因型(sequence type, ST)序列号[19]. 用MLSTest软件(http://www.ipe.unsa.edu.ar/software)构建unweighted-pair group method using average linkages(UPGMA)进化树[20], 分析白色念珠菌菌株的进化分支关系.
统计学处理 用SPSS11.5软件进行χ2检验和双因素相关分析, P<0.05为有统计学意义.
正常对照组中的口腔黏膜拭子样本中, 29.6%(48/162)为念珠菌阳性. 在念珠菌阳性者中, 白色念珠菌构成比为64.6%(31/48), 近平滑念珠菌(Candida parapsilosis)为20.8%(10/48), 热带念珠菌(Candida tropicalis)为10.4%(5/48), 季也蒙毕赤念珠菌(Meyerozyma guilliermondii)为6.3%(3/48), 其他念珠菌菌种为6.3%(包括C. metapsilosis, Pichia kudriavzevii, C. ethanolica).
消化不良患者的口腔黏膜拭子样本中, 36.0%(40/111)为念珠菌阳性. 在念珠菌阳性者中, 白色念珠菌的构成比为95%(38/40), 近平滑念珠菌为5%(2/40). 正常对照组与患者组口腔黏膜样本中, 念珠菌阳性率和白色念珠菌阳性率情况如表1.
| 分组 | n | 念珠菌阳性率n(%) | 白色念珠菌阳性率n(%) | 白色念珠菌构成比(%) |
| 患者组 | 111 | 40(36.0) | 38(34.2) | 95.0 |
| 正常对照组 | 162 | 48(29.6) | 31(19.1) | 64.6 |
| 总计 | 273 | 88(32.2) | 69(25.3) | 78.4 |
消化不良患者的胃黏膜样本中, 41.4%(46/111)为念珠菌阳性. 在念珠菌阳性者中, 白色念珠菌构成比为97.8%(45/46), 光滑念珠菌(Candida glabrata)为2.2%(1/46). 消化不良患者的念珠菌阳性率, 高于正常对照组(χ2 = 4.071, P<0.01). 消化不良患者口腔拭子样本的白色念珠菌阳性率及构成比, 均高于正常对照组(χ2 = 11.206, P<0.01; χ2 = 7.650, P = 0.006).
27例消化不良患者的胃黏膜样本, 65例口腔黏膜拭子样本(38例消化不良患者, 27份健康志愿者)分离培养出白色念珠菌菌株, 进行MLST检测. 各ST基因型在正常对照组和消化不良患者组检出数量情况(表2). 检出44个ST基因型, 其中有8个ST基因型在多个样本中检出(ST142、ST367、ST601、ST656、ST1593、ST1594、ST1779、ST1971).
| Clade | ST | 检出数量 | 总计 | ||
| 正常对照组口腔黏膜 | 患者组口腔黏膜 | 患者组胃黏膜 | |||
| 1 | 367 | 2 | 0 | 0 | 2 |
| 1 | 766 | 1 | 0 | 0 | 1 |
| 1 | 1963 | 1 | 0 | 0 | 1 |
| 3 | 1961 | 1 | 0 | 0 | 1 |
| 3 | 1965 | 1 | 0 | 0 | 1 |
| 4 | 95 | 1 | 0 | 0 | 1 |
| 4 | 142 | 6 | 1 | 0 | 7 |
| 4 | 144 | 1 | 0 | 0 | 1 |
| 4 | 605 | 0 | 1 | 0 | 1 |
| 4 | 622 | 1 | 0 | 0 | 1 |
| 4 | 656 | 3 | 0 | 0 | 3 |
| 4 | 659 | 1 | 0 | 0 | 1 |
| 4 | 1061 | 1 | 0 | 0 | 1 |
| 4 | 1605 | 1 | 0 | 0 | 1 |
| 4 | 1609 | 1 | 0 | 0 | 1 |
| 4 | 1962 | 1 | 0 | 0 | 1 |
| 4 | 1973 | 0 | 1 | 0 | 1 |
| 4 | 1976 | 0 | 1 | 0 | 1 |
| 4 | 1977 | 0 | 1 | 0 | 1 |
| 5 | 1960 | 0 | 1 | 0 | 1 |
| 5 | 1964 | 1 | 0 | 0 | 1 |
| 6 | 1779 | 1 | 0 | 2 | 3 |
| 6 | 1957 | 0 | 1 | 0 | 1 |
| 6 | 1966 | 1 | 0 | 0 | 1 |
| 6 | 1967 | 1 | 0 | 0 | 1 |
| 7 | 1972 | 0 | 1 | 0 | 1 |
| 7 | 1975 | 0 | 1 | 0 | 1 |
| 8 | 365 | 0 | 1 | 0 | 1 |
| 9 | 918 | 0 | 1 | 0 | 1 |
| 12 | 601 | 0 | 1 | 1 | 2 |
| 14 | 677 | 0 | 1 | 0 | 1 |
| 14 | 747 | 0 | 1 | 0 | 1 |
| 14 | 1933 | 0 | 1 | 0 | 1 |
| 14 | 1959 | 0 | 1 | 0 | 1 |
| 14 | 1968 | 0 | 1 | 0 | 1 |
| 14 | 1974 | 0 | 1 | 0 | 1 |
| 15 | 1865 | 0 | 1 | 0 | 1 |
| 15 | 1956 | 1 | 0 | 0 | 1 |
| 16 | 1978 | 0 | 1 | 0 | 1 |
| 18 | 1593 | 4 | 17 | 21 | 42 |
| 18 | 1594 | 0 | 0 | 2 | 2 |
| 18 | 1958 | 0 | 1 | 0 | 1 |
| 18 | 1970 | 0 | 1 | 0 | 1 |
| 18 | 1971 | 0 | 2 | 3 | 5 |
| 总计 | 32 | 41 | 29 | 102 | |
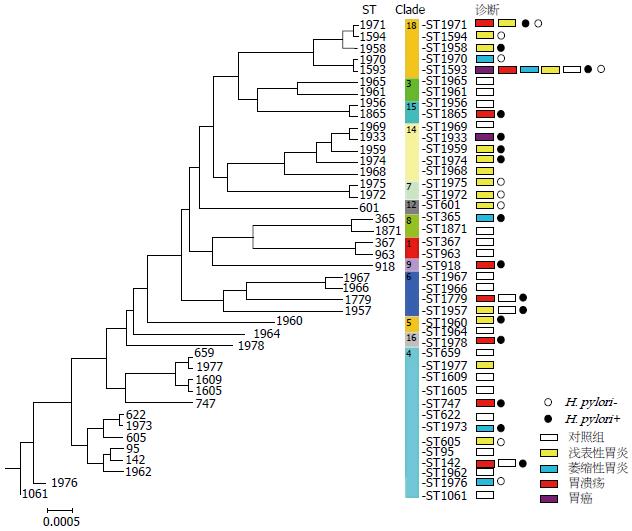
建立UPGMA进化树, 分析白色念珠菌的ST进化分支(Clade)情况(图1). 在UPGMA进化树中, ST基因型被分为18个大进化分支[21]. 本研究中, 最常见的白色念珠菌进化分支是Clade 18, 可在35个人中检出(46.1%), 其次是Clade 4(25%). 正常对照组的大部分菌株位于Clade 4(48.1%), 消化不良患者组的大部分菌株位于Clade 18(66.7%). 在消化不良患者组中未检出Clade 1和Clade 3的菌株; 在正常对照组中未检出Clade 7、Clade 8、Clade 9、Clade 12、Clade 14、Clade 16分支的菌株. 菌株所在的不同进化分支Clade与胃黏膜炎症损伤关系密切(Kendall's tau-b r = 0.591, P<0.001).
在进化分支Clade 18中的基因型ST1593, 是消化不良患者组的最主要基因型, 在26例患者中检出(57.8%, 26/45); 而在正常对照组中, ST1593只在4个样本中检出(14.8%, 4/27), 两组间差异有统计学意义(χ2 = 12.815, P<0.001). Clade 18的其他ST基因型(ST1594、ST1958、ST1970、ST1971) 均来自消化不良患者组. 位于Clade 4的ST142基因型, 是第2检出率最高的基因型, 也是正常对照组中的最常见基因型, 可见于6名健康志愿者, 1例消化不良患者.
本研究目的是检测消化不良患者的口腔和胃黏膜中, 白色念珠菌的阳性率和基因型状况. 经过对111例消化不良患者和162名健康志愿者的检查发现, 消化不良患者携带的念珠菌菌种非常单一, 白色念珠菌是主要菌种, 在念珠菌阳性者中占97.8%; 而健康志愿者的菌种则更加多样性, 白色念珠菌在念珠菌阳性者中仅占56.3%. 在正常对照组与消化不良患者组间, 这种念珠菌菌种分布的差异, 可能与宿主不同的内环境相关. 白色念珠菌为胃黏膜的主要菌种, 可能受胃内较低的pH值影响, 因白色念珠菌可以在pH = 2的环境下生长良好, 而其他菌种不能[22,23]. 但在pH值接近中性的口腔黏膜中, 消化不良患者组中白色念珠菌仍占绝对优势地位, 这值得我们进一步研究, 消化不良患者的黏膜上, 是否存在白色念珠菌更易黏附的黏附分子.
对白色念珠菌菌株进行MLST检测, 发现二组间白色念珠菌ST基因型分布, 也有较大差异. 消化不良患者组的念珠菌阳性者中, 基因型ST1593占57.8%, 来自Clade 18的菌株为优势菌株(66.7%); 正常对照组中, 来自Clade18的菌株仅占14.8%, 而来自Clade 4的菌株是优势菌株(48.1%). 在本研究相关操作者中未能检出ST1593, 排除该基因型来自院内感染的可能. 本研究显示宿主不同的内环境影响念珠菌菌种多样性的分布, 及白色念珠菌基因型多态性的分布.
Sampaio等[24]的研究认为白色念珠菌会根据宿主内环境的变化而发生基因变异, 以适应宿主的环境. 白色念珠菌的黏附素能否黏附于宿主的上皮细胞黏附分子上[25], 以及是否能被宿主的免疫系统相容[26], 是决定白色念珠菌是否能定植的关键因素. 亦有研究认为白色念珠菌有可能作为自身免疫或炎症反应的诱发因子, 参与胃炎、胃溃疡的炎症过程[27]. 同时, 白色念珠菌与胃黏膜菌群有复杂的共生关系, 甚至在白色念珠菌中可以检出H. pylori的毒性因子(如空泡毒素VacA、尿素酶UreA)[28-31]. 由图1可见, 患者组多见的Clade 18和Clade 14分支的菌株, 多伴生H. pylori; 正常组多见的Clade 1、Clade 3、Clade 4分支的菌株, 较少伴生H. pylori. 由此推测, Clade18分支的ST1593基因型菌株可能是最适合于黏附在消化不良患者的黏膜上皮细胞上, 能被消化不良患者的免疫系统相容, 甚至与H. pylori协同参与胃黏膜的炎症损伤过程. 由此, 白色念珠菌基因型ST1593可以成为消化不良易感者的信号指征.
Wrobel等[32]认为的传播主要是人间传播, 由环境到人的传播极少见. 随着经济全球化, 人类旅行的日益方便和频繁, 白色念珠菌分化分支的地域差异会逐渐缩小[33], 白色念珠菌分布的地域、种族和民族差异还是要被考虑的[34-37]. 在白色念珠菌MLST数据库中显示, 第2、6、10分支在大中国区未见报道. 本研究中, 健康志愿者的71.9%菌株来自Clade 1、Clade 3、Clade 4分支, 这几个分支分布缺乏明显的种族和地域差异. 而消化不良患者组的78.6%菌株来自Clade 14和Clade 18, 这2个分支主要被报道分布在东亚地区(中国、日本、韩国). 似乎种族和地域的差异, 不影响白色念珠菌基因型在健康志愿者中的分布; 而在消化不良患者中, 却成为重要的影响因素. 目前尚未见其他地区消化不良患者的白色念珠菌MLST基因型的报道, ST1593基因型是仅分布于亚裔的消化不良患者中, 还是在其他地区和种族的患者也有分布, 尚需更多的研究报道.
目前针对某种疾病与某些进化分支Clade关系的报道较少, 尚不能明确疾病、种族、地域对Clade分布的影响[38]. Bougnoux等[39]在比利时采集分析溃疡性结肠炎(Crohn's disease)患者及家属粪便的白色念珠菌, 发现Clade 4为主要分支, 并存在家庭传播. 同样, 他们的研究也缺乏该病种在其他地区的研究报道, 难以明确疾病、种族、地域对基因型的影响. McManus等[40]采集分析爱尔兰自体免疫病自身免疫性多内分泌病-念珠菌病-外胚层营养不良(autoimmune polyendocrinopathy-candidiasis-ectodermal dystrophy, APECED)患者口腔黏膜的白色念珠菌, 发现Clade 1是主要分支(35.7%), 其次是Clade 4(21.4%)和Clade 15(14.3%), 因其缺乏正常对照组, 难以判断影响因素. Da Matta等[41]在巴西采集分析血液感染的白色念珠菌, 发现Clade 1为主要分支, 但其没有区分病种, 只是判断地域可能是其主要影响因素. 同在巴西, McManus等[42]采集分析口腔白色念珠菌对牙周炎的影响, 发现牙周炎患者中Clade 1是主要分支(67.7%), 而健康对照组中Clade 2是主要分支(31.3%), 其次是Clade 1和4(25%). Jacobsen等[43]在英国采集分析普通志愿者的口腔白色念珠菌, 发现Clade 1为主要分支, 其次是Clade 2. 由此看来, Clade 1的分布似乎更多与高加索人种相关, 而Clade 1中是否存在与某种疾病相关的基因型, 目前缺乏报道.
Shin等[44]在韩国采集分析血液感染的白色念珠菌, 发现Clade 18为主要分支(18.6%), 其次是Clade 4(15.4%)、Clade 1(14.7%)、Clade 12(11.5%). 他们没有区分病种, 因其地理位置及种族都与本研究相似, 且都是Clade 18为主要分支, 可能疾病、种族、地域对本研究中的Clade 18都有重要影响. Wu等[45]在上海采集分析痰和尿液的白色念珠菌, 发现 Clade 6(37.5%)为主要分支, 其次是Clade 17(15.0%) 和Clade 1(15.0%). 他们也没有区分病种, 但Clade分布与Shin等[44]的报道差异较大, 可能是地域影响. 与本研究相似的是, 他们也发现Clade 18目前只在东亚地区有报道, 只是在他们的研究中检出率很低(5.0%). Ge等[46]采集分析中国多个城市的阴道和龟头黏膜的白色念珠菌, 发现Clade 1在阴道炎(71.6%)和龟头炎(92.3%)患者中为主要分支, 而在健康自愿者中较少检出(40.6%的阴道黏膜, 7.8%口腔). 在健康自愿者口腔中Clade 1检出率较低, 这与我们的研究结果是一致的. 而且, Wu等[45]与Ge等[46]的报道说明Clade 18并非在东亚地区人群口腔中, 检出率普遍高, Clade 18的分布是有疾病特异性的. 只是缺乏其他地区对消化不良患者白色念珠菌的研究报道, 目前不清楚在其他地区胃黏膜炎症是否也与Clade 18相关.
总之, 本研究发现消化不良患者与正常对照组间, 念珠菌菌种多样性和白色念珠菌基因型多态性分布, 都有较大的差异. 白色念珠菌Clade18分支的ST1593基因型与消化不良有密切关系, 可以作为消化不良易感者的候选指征, 做进一步研究.
志谢: 感谢沈阳市口腔医院口腔内科赵英姿与梁宝刚医生; 辽宁中医药大学附属第一医院消化科王垂杰与傅纪婷医生; 辽宁中医药大学职业技术学院师生, 在本实验中给予的大力帮助.
念珠菌被认为是人类的常见菌群, 但越来越多的报道发现念珠菌与慢性胃炎和胃溃疡有密切的关系. 因白色念珠菌具有高度的遗传多样性, 所以必须深入了解其基因型及变异和进化方向, 以便更精确了解其流行病学意义.
牛英才, 研究员, 齐齐哈尔医学院医药科学研究所; 彭宗根, 研究员, 博士生导师, 中国医学科学院医药生物技术研究所病毒室
现今对病原菌进化分支研究多采用多位点序列分型(multilocus sequence typing, MLST)方法, 用于研究细菌的流行病学、群体生物学、致病性和进化, 为传染病的临床诊断和流行病学监测提供依据, 对临床和公共卫生有重要意义.
在胃溃疡活动期, 白色念珠菌的检出率达30%-50%, 并使溃疡症状加重, 并延长愈合时间. 常有报道单一的抗幽门螺杆菌(Helicobacter pylori, H. pylori)治疗不能愈合的胃溃疡, 需经抗真菌治疗后, 溃疡才能愈合. 甚至, 有真菌炎症性肉芽肿被误诊为胃癌的报道.
已有大量研究报道白色念珠菌与胃炎、胃溃疡密切相关, 但不能解释白色念珠菌作为正常菌群成员, 广泛存在于消化系中. 本研究发现ST1593基因型是胃黏膜炎症损伤密切相关的白色念珠菌基因型, 这明确了致病性白色念珠菌与正常菌群白色念珠菌的区别.
本研究发现的ST1593基因型是胃黏膜炎症损伤密切相关的白色念珠菌基因型, 可以作为胃炎、胃溃疡预防诊断的指征, 也可用于相关特异性药物研发.
本文研究结果对基础和临床深入研究具有重要的参考意义. 研究方法先进, 讨论条理分明, 实验设计合理, 研究结论可信, 研究具有一定的科学价值.
编辑: 郭鹏 电编: 闫晋利
| 2. | Gupta N. A rare cause of gastric perforation-Candida infection: a case report and review of the literature. J Clin Diagn Res. 2012;6:1564-1565. [PubMed] [DOI] |
| 3. | Joo MK, Park JJ, Lee BJ, Kim JH, Yeon JE, Kim JS, Byun KS, Bak YT. A case of stenotic change from gastric candidiasis managed with temporary stent insertion. Gut Liver. 2011;5:238-241. [PubMed] [DOI] |
| 7. | Zwolińska-Wcisło M, Budak A, Bogdał J, Trojanowska D, Stachura J. Effect of fungal colonization of gastric mucosa on the course of gastric ulcers healing. Med Sci Monit. 2001;7:266-275. [PubMed] |
| 8. | Kalogeropoulos NK, Whitehead R. Campylobacter-like organisms and Candida in peptic ulcers and similar lesions of the upper gastrointestinal tract: a study of 247 cases. J Clin Pathol. 1988;41:1093-1098. [PubMed] |
| 9. | Hirasaki S, Koide N, Ogawa H, Tsuji T. Benign gastric ulcer associated with Canidida infection in a healthy adult. J Gastroenterol. 1999;34:688-693. [PubMed] |
| 10. | Sasaki K. Candida-associated gastric ulcer relapsing in a different position with a different appearance. World J Gastroenterol. 2012;18:4450-4453. [PubMed] [DOI] |
| 11. | Karczewska E, Wojtas I, Sito E, Trojanowska D, Budak A, Zwolinska-Wcislo M, Wilk A. Assessment of co-existence of Helicobacter pylori and Candida fungi in diseases of the upper gastrointestinal tract. J Physiol Pharmacol. 2009;60 Suppl 6:33-39. [PubMed] |
| 12. | Zwolińska-Wcisło M, Brzozowski T, Mach T, Budak A, Trojanowska D, Konturek PC, Pajdo R, Drozdowicz D, Kwiecień S. Are probiotics effective in the treatment of fungal colonization of the gastrointestinal tract? Experimental and clinical studies. J Physiol Pharmacol. 2006;57 Suppl 9:35-49. [PubMed] |
| 14. | Rajablou M, Ganz RA, Batts KP. Candida infection presenting as multiple ulcerated masses. Gastrointest Endosc. 2007;65:164-166. [PubMed] [DOI] |
| 15. | Mansueto P, Pisciotta G, Tomasello G, Cabibi D, Seidita A, D'Alcamo A, Patti AM, Sprini D, Carroccio A, Rini GB. Malignant tumor-like gastric lesion due to Candida albicans in a diabetic patient treated with cyclosporin: a case report and review of the literature. Clin Exp Med. 2012;12:201-205. [PubMed] [DOI] |
| 16. | Odds FC, Bernaerts R. CHROMagar Candida, a new differential isolation medium for presumptive identification of clinically important Candida species. J Clin Microbiol. 1994;32:1923-1929. [PubMed] |
| 17. | Schoch CL, Seifert KA, Huhndorf S, Robert V, Spouge JL, Levesque CA, Chen W. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proc Natl Acad Sci U S A. 2012;109:6241-6246. [PubMed] [DOI] |
| 18. | Bougnoux ME, Tavanti A, Bouchier C, Gow NA, Magnier A, Davidson AD, Maiden MC, D'Enfert C, Odds FC. Collaborative consensus for optimized multilocus sequence typing of Candida albicans. J Clin Microbiol. 2003;41:5265-5266. [PubMed] [DOI] |
| 19. | Gong YB, Zheng JL, Jin B, Zhuo DX, Huang ZQ, Qi H, Zhang W, Duan W, Fu JT, Wang CJ. Particular Candida albicans strains in the digestive tract of dyspeptic patients, identified by multilocus sequence typing. PLoS One. 2012;7:e35311. [PubMed] [DOI] |
| 20. | Tomasini N, Lauthier JJ, Llewellyn MS, Diosque P. MLSTest: novel software for multi-locus sequence data analysis in eukaryotic organisms. Infect Genet Evol. 2013;20:188-196. [PubMed] [DOI] |
| 21. | McManus BA, Coleman DC. Molecular epidemiology, phylogeny and evolution of Candida albicans. Infect Genet Evol. 2014;21:166-178. [PubMed] [DOI] |
| 22. | Zwolinska-Wcisło M, Budak A, Bogdał J, Trojanowska D, Stachura J. Fungal colonization of gastric mucosa and its clinical relevance. Med Sci Monit. 2001;7:982-988. [PubMed] [DOI] |
| 23. | O'May GA, Reynolds N, Macfarlane GT. Effect of pH on an in vitro model of gastric microbiota in enteral nutrition patients. Appl Environ Microbiol. 2005;71:4777-4783. [PubMed] [DOI] |
| 24. | Sampaio P, Santos M, Correia A, Amaral FE, Chavéz-Galarza J, Costa-de-Oliveira S, Castro AG, Pedrosa J, Pais C. Virulence attenuation of Candida albicans genetic variants isolated from a patient with a recurrent bloodstream infection. PLoS One. 2010;5:e10155. [PubMed] [DOI] |
| 25. | Moreno-Ruiz E, Galán-Díez M, Zhu W, Fernández-Ruiz E, d'Enfert C, Filler SG, Cossart P, Veiga E. Candida albicans internalization by host cells is mediated by a clathrin-dependent mechanism. Cell Microbiol. 2009;11:1179-1189. [PubMed] [DOI] |
| 27. | Kumamoto CA. Inflammation and gastrointestinal Candida colonization. Curr Opin Microbiol. 2011;14:386-391. [PubMed] [DOI] |
| 28. | Siavoshi F, Saniee P. Vacuoles of Candida yeast as a specialized niche for Helicobacter pylori. World J Gastroenterol. 2014;20:5263-5273. [PubMed] [DOI] |
| 29. | Salmanian AH, Siavoshi F, Akbari F, Afshari A, Malekzadeh R. Yeast of the oral cavity is the reservoir of Heliobacter pylori. J Oral Pathol Med. 2008;37:324-328. [PubMed] [DOI] |
| 30. | Mason KL, Erb Downward JR, Falkowski NR, Young VB, Kao JY, Huffnagle GB. Interplay between the gastric bacterial microbiota and Candida albicans during postantibiotic recolonization and gastritis. Infect Immun. 2012;80:150-158. [PubMed] [DOI] |
| 31. | Saniee P, Siavoshi F, Nikbakht Broujeni G, Khormali M, Sarrafnejad A, Malekzadeh R. Immunodetection of Helicobacter pylori-specific proteins in oral and gastric Candida yeasts. Arch Iran Med. 2013;16:624-630. [PubMed] [DOI] |
| 32. | Wrobel L, Whittington JK, Pujol C, Oh SH, Ruiz MO, Pfaller MA, Diekema DJ, Soll DR, Hoyer LL. Molecular phylogenetic analysis of a geographically and temporally matched set of Candida albicans isolates from humans and nonmigratory wildlife in central Illinois. Eukaryot Cell. 2008;7:1475-1486. [PubMed] [DOI] |
| 33. | Takakura S, Ichiyama S, Bain JM, Davidson AD, Jacobsen MD, Shaw DJ, Gow NA, Odds FC. Comparison of Candida albicans strain types among isolates from three countries. Int J Med Microbiol. 2008;298:663-668. [PubMed] [DOI] |
| 34. | MacCallum DM, Castillo L, Nather K, Munro CA, Brown AJ, Gow NA, Odds FC. Property differences among the four major Candida albicans strain clades. Eukaryot Cell. 2009;8:373-387. [PubMed] [DOI] |
| 35. | Chen KW, Chen YC, Lo HJ, Odds FC, Wang TH, Lin CY, Li SY. Multilocus sequence typing for analyses of clonality of Candida albicans strains in Taiwan. J Clin Microbiol. 2006;44:2172-2178. [PubMed] [DOI] |
| 36. | Tavanti A, Davidson AD, Fordyce MJ, Gow NA, Maiden MC, Odds FC. Population structure and properties of Candida albicans, as determined by multilocus sequence typing. J Clin Microbiol. 2005;43:5601-5613. [PubMed] [DOI] |
| 37. | Chávez-Galarza J, Pais C, Sampaio P. Microsatellite typing identifies the major clades of the human pathogen Candida albicans. Infect Genet Evol. 2010;10:697-702. [PubMed] [DOI] |
| 38. | Bai FY. Association of genotypes with infection types and antifungal susceptibilities in Candida albicans as revealed by recent molecular typing strategies. Mycology. 2014;5:1-9. [PubMed] [DOI] |
| 39. | Bougnoux ME, Diogo D, François N, Sendid B, Veirmeire S, Colombel JF, Bouchier C, Van Kruiningen H, d'Enfert C, Poulain D. Multilocus sequence typing reveals intrafamilial transmission and microevolutions of Candida albicans isolates from the human digestive tract. J Clin Microbiol. 2006;44:1810-1820. [PubMed] [DOI] |
| 40. | McManus BA, McGovern E, Moran GP, Healy CM, Nunn J, Fleming P, Costigan C, Sullivan DJ, Coleman DC. Microbiological screening of Irish patients with autoimmune polyendocrinopathy-candidiasis-ectodermal dystrophy reveals persistence of Candida albicans strains, gradual reduction in susceptibility to azoles, and incidences of clinical signs of oral candidiasis without culture evidence. J Clin Microbiol. 2011;49:1879-1889. [PubMed] [DOI] |
| 41. | Da Matta DA, Melo AS, Colombo AL, Frade JP, Nucci M, Lott TJ. Candidemia surveillance in Brazil: evidence for a geographical boundary defining an area exhibiting an abatement of infections by Candida albicans group 2 strains. J Clin Microbiol. 2010;48:3062-3067. [PubMed] [DOI] |
| 42. | McManus BA, Maguire R, Cashin PJ, Claffey N, Flint S, Abdulrahim MH, Coleman DC. Enrichment of multilocus sequence typing clade 1 with oral Candida albicans isolates in patients with untreated periodontitis. J Clin Microbiol. 2012;50:3335-3344. [PubMed] [DOI] |
| 43. | Jacobsen MD, Duncan AD, Bain J, Johnson EM, Naglik JR, Shaw DJ, Gow NA, Odds FC. Mixed Candida albicans strain populations in colonized and infected mucosal tissues. FEMS Yeast Res. 2008;8:1334-1338. [PubMed] [DOI] |
| 44. | Shin JH, Bougnoux ME, d'Enfert C, Kim SH, Moon CJ, Joo MY, Lee K, Kim MN, Lee HS, Shin MG. Genetic diversity among Korean Candida albicans bloodstream isolates: assessment by multilocus sequence typing and restriction endonuclease analysis of genomic DNA by use of BssHII. J Clin Microbiol. 2011;49:2572-2577. [PubMed] [DOI] |
| 45. | Wu K, Luo T, Li L, Zhang Q, Zhu J, Gao Q, Chen M, Zhu M. Multilocus Sequence Typing of Pathogenic Candida albicans Isolates Collected from a Teaching Hospital in Shanghai, China: A Molecular Epidemiology Study. PLoS One. 2015;10:e0125245. [PubMed] [DOI] |
| 46. | Ge SH, Xie J, Xu J, Li J, Li DM, Zong LL, Zheng YC, Bai FY. Prevalence of specific and phylogenetically closely related genotypes in the population of Candida albicans associated with genital candidiasis in China. Fungal Genet Biol. 2012;49:86-93. [PubMed] [DOI] |