修回日期: 2015-04-28
接受日期: 2015-05-07
在线出版日期: 2015-06-28
目的: 了解贵州省黔南州幽门螺杆菌(Helicobacter pylori, H. pylori)克拉霉素耐药相关基因突变情况.
方法: 采用改良琼脂稀释法检测分离自黔南州人民医院的74株H. pylori对克拉霉素的敏感性, 选取H. pylori克拉霉素耐药菌株22株、敏感株10株, 以及NCTC11637进行23S rRNA基因功能区V区片段的PCR扩增和测序, 与GenBank中收录的H. pylori敏感菌株 U27270相关序列进行比对和分析.
结果: 74株H. pylori克拉霉素耐药率为29.7%(22/74). 共发现10种突变类型, 其中在耐药菌株及敏感菌株23S rRNA V区均存在的基因突变包括: T2183C、T2245C和T2321C; 仅在耐药株(22株)中检测到的突变包括: A2144G(4/22)、A2224G(4/22)、C2196T(1/22)、C2289T(1/22)、A2435G(1/22)、C2695A(1/22)、T2712C(1/22).
结论: 黔南州医院分离的H. pylori克拉霉素耐药率较高; 与耐药性相关的23S rRNA基因突变主要为A2144G和A2224G; 本院菌株存在A2435G、C2695A、T2712C 3种尚未见报道的突变.
核心提示: 本文表明分离自黔南州人民医院的幽门螺杆菌(Helicobacter pylori)临床菌株克拉霉素耐药率已高达29%; 本地耐药菌株的23S rRNA基因突变具有复杂的多态性, 与耐药性相关的突变主要为A2144G和A2224G; 且存在不同于其他地区的新型的突变类型.
引文著录: 綦廷娜, 潘科, 王菲, 魏刚, 蒋勇, 张人弘, 郑娟, 张丹丹, 陈峥宏. 贵州省黔南州幽门螺杆菌克拉霉素的耐药性及23S rRNA基因突变的特征. 世界华人消化杂志 2015; 23(18): 2901-2906
Revised: April 28, 2015
Accepted: May 7, 2015
Published online: June 28, 2015
AIM: To investigate the antibiotic sensitivity of Helicobacter pylori (H. pylori) isolates from patients in the People's Hospital of Qiannan Autonomous Prefecture and to analyze mutations in the domain V of 23S rRNA gene that are related with clarithromycin resistance.
METHODS: Clarithromycin susceptibility tests of 74 H. pylori isolates were performed by the modified agar dilution method (breakpoint susceptibility testing). Domain V of 23S rRNA gene of 22 clarithromycin resistant strains, 10 clarithromycin susceptible strains and a control stain (NCTC11637) were amplified and sequenced. Nucleotide homology of these sequences were compared to H. pylori U27270 (GenBank accession number: U27270) which is a susceptible strain.
RESULTS: Clarithromycin resistance rate of 74 H. pylori isolates was 29.7% (22/74). Ten types of mutations in domain V of 23S rRNA gene were found in this study. T2183C, T2245C and T2321C mutations were found in both clarithromycin resistant strains and susceptible strains. Mutations only found in resistant strains included A2144G(4/22), A2224G(4/22), C2196T(1/22), C2289T(1/22), A2435G(1/22), C2695A(1/22) and T2712C(1/22).
CONCLUSION: Clarithromycin resistance rate of H. pylori isolates from the People's Hospital of Qiannan Autonomous Prefecture was high. A2144G and A2224G mutations were the most prevalent mutation types related with clarithromycin resistance and there were three novel mutation types (A2435G, C2695 and T2712C).
- Citation: Qi TN, Pan K, Wang F, Wei G, Jiang Y, Zhang RH, Zheng J, Zhang DD, Chen ZH. Clarithromycin resistance and 23S rRNA gene mutation characteristics of Helicobacter pylori isolates from patients in Qiannan Autonomous Prefecture. Shijie Huaren Xiaohua Zazhi 2015; 23(18): 2901-2906
- URL: https://www.wjgnet.com/1009-3079/full/v23/i18/2901.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i18.2901
幽门螺杆菌(Helicobacter pylori, H. pylori)在人群中的感染非常普遍, 是急慢性胃炎、消化性胃溃疡的重要致病因子, 并且与胃腺癌和胃黏膜相关B细胞淋巴瘤的发生密切相关[1]. 克拉霉素是应用于根除H. pylori治疗方案中常用抗生素之一. 近年来国内外资料报道, H. pylori对克拉霉素的耐药率有逐年增高趋势, 克拉霉素耐药性的产生与H. pylori 23S rRNA V区上的点突变有关, 且不同国家或地区的耐药基因突变位点呈现不同特征[2]. 了解地区性H. pylori克拉霉素耐药情况及耐药基因突变特征, 有助于指导临床合理将克拉霉素应用于H. pylori感染所致疾病的治疗, 并为进一步完善快速检测H. pylori克拉霉素耐药性的基因诊断方法提供实验依据. 我们检测了黔南州人民医院临床分离H. pylori菌株对克拉霉素的敏感性及H. pylori 23S rRNA基因的突变特征, 现报告如下.
H. pylori临床菌株来源于黔南州人民医院消化科胃镜室行胃镜检查知情患者(慢性胃炎、胃溃疡、十二指肠球部溃疡、胃癌患者). 质控菌株: H. pylori菌株NCTC l l637, 获赠于中国疾病预防控制中心传染病预防控制所, 贵阳医学院微生物学教研室保存. 引物: 采用文献[3]的方法, 由生工生物工程(上海)股份有限公司合成H. pylori 23S rRNA基因功能区V区(2055-2889)片段PCR特异性引物F: 5'-TGGCGTAACGAGATGGGAGCTGT-3'和R: 5'-AGTCTTTGTGCGAGCTTAGGGA-3'. 哥伦比亚培养基(青岛高科园海博生物技术有限公司); MH培养基(杭州天和微生物试剂有限公司); 脱纤维绵羊血(采自贵阳医学院实验动物中心); H. pylori添加剂(英国, OXOID); 微需氧产气袋(日本, 三菱化学株式会社); 厌氧罐(日本, 三菱化学株式会社); H. pylori转送液(100 g/L蔗糖+50%小牛血清); 细菌基因组DNA提取试剂盒(北京三博远志生物技术有限公司); 克拉霉素(中国食品药品检定研究所130558-200902).
1.2.1 H. pylori的分离培养和鉴定: 无菌操作, 将胃镜下采集的胃底和胃体部胃黏膜组织充分剪碎后接种于含有H. pylori选择添加剂的哥伦比亚血琼脂培养基中, 37 ℃微需氧培养5 d, 将疑似菌株经革兰染色初步鉴定后划线分离培养, 挑取单菌落传代培养并经革兰染色、快速尿素酶试验和H. pylori 16S rDNA特异性PCR扩增进行鉴定[4]. 将鉴定阳性菌株保存于脱纤维绵羊血中, -80 ℃保存备用.
1.2.2 H. pylori的克拉霉素敏感性检测: 参照美国临床实验标准化委员会推荐的改良琼脂稀释法[5]检测H. pylori对克拉霉素的敏感性. 取2 µL待检H. pylori菌液点种在含有终浓度为1 µg/mL克拉霉素的MH血琼脂平板中微需氧培养96 h后观察, 若接种点有细菌生长判为耐药. 实验均重复2次, 质控株选用NCTC 11637.
1.2.3 H. pylori 23S rRNA基因片段的PCR扩增和测序: 按细菌基因组DNA提取试剂盒说明书方法分别提取22株克拉霉素耐药株、10株敏感菌株和NCTC11637基因组DNA. 扩增其23S rRNA基因V区(2055-2899)片段. PCR产物经1.0%琼脂糖凝胶电泳观察, 送生工生物工程(上海)股份有限公司进行双向序列测定.
1.2.4 H. pylori 23S rRNA序列分析: 采用ClustalW2软件对32株测序结果与文献[6]的敏感菌株H. pylori U27270序列进行比对和分析, 寻找与克拉霉素耐药性相关的突变类型, 并与文献报道的宁波[7]、青岛[8]和贵阳[3]耐药菌株的突变类型进行比较.
280例临床标本经分离培养, 得到微需氧生长、革兰染色阴性螺杆菌、快速尿素酶试验阳性及16S rDNA鉴定阳性、可用于药敏试验的H. pylori纯培养物共74例.
74株H. pylori中, 52株为克拉霉素敏感株, 22株为克拉霉素耐药株, 耐药率为29.7%(22/74).
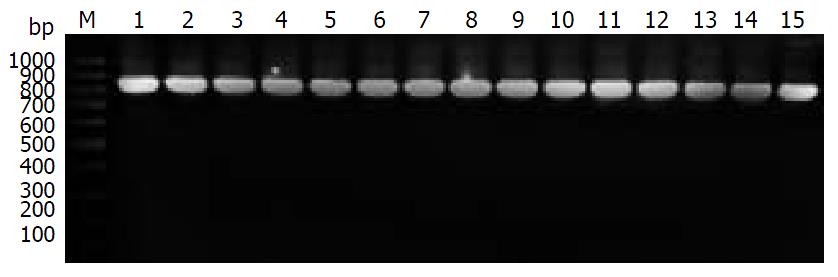
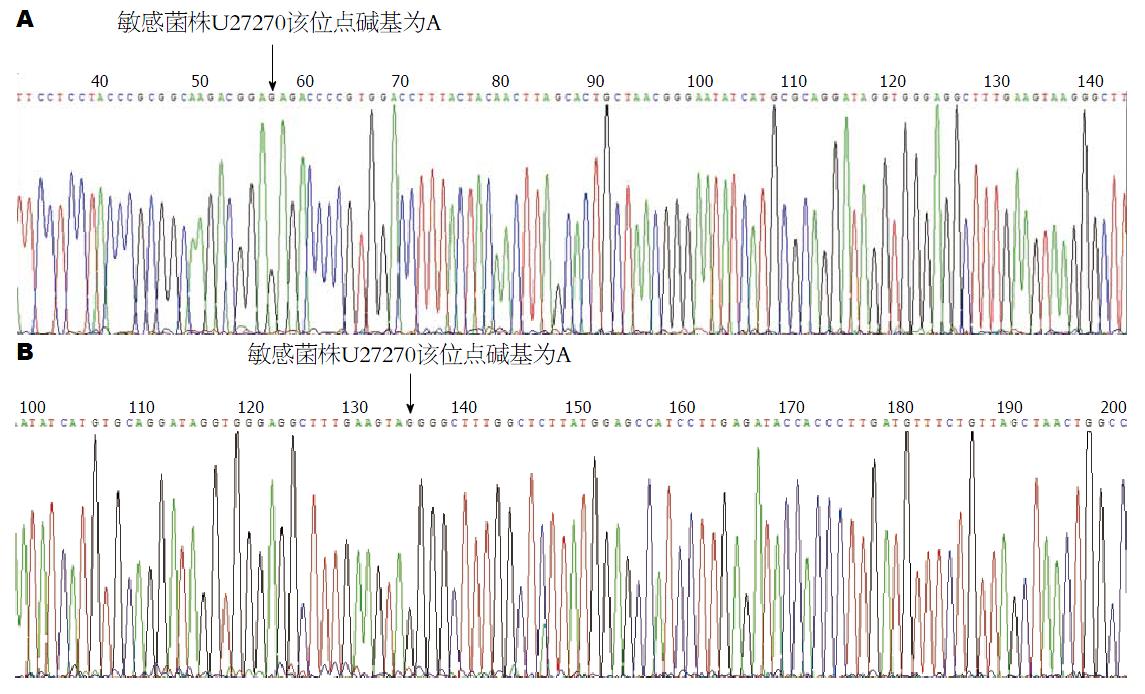
NCTC11637及22株H. pylori克拉霉素耐药株、10株敏感株的23S rRNA基因片段PCR产物, 经1%琼脂糖凝胶电泳, 紫外检测仪下观察, 约850 bp处均见明显目的条带(图1). 经测序结果和与U27270序列比对, 克拉霉素耐药株中, 2144和2224位点有18株为A, 4株为G; 2183位点有9株为T, 13株为C; 2196和2289位点有21株为C, 1株为T; 2245位点有2株为T, 20株为C; 2321位点有19株为T, 3株为C; 2435位点有21株为A, 1株为G; 2695点有21株为C, 1株为A; 2712点有21株为T, 1株为C敏感株中, 2183位点有4株为T, 6株为C; 2245位点10株均为C; 2321位点有9株为T, 1株为C; 其他位点与参考株H. pylori U27270一致. A2144G和A2224G突变的测序峰图如图2, 各突变位点的碱基分布如表1. 宁波、青岛、贵阳和本地突变的比较如表2.
| 突变位点 | U27270 | NCTC11637 | 10株敏感株 | 22株耐药株 |
| (核苷酸/菌株数) | (核苷酸/菌株数) | |||
| 2144 | A | A | A/10 | A/18, G/4 |
| 2183 | T | T | T/6, C/4 | T/9, C/13 |
| 2196 | C | C | C/10 | C/21, T/1 |
| 2224 | A | A | A/10 | A/18, G/4 |
| 2245 | T | C | C/10 | T/2, C/20 |
| 2289 | C | C | C/10 | C/21, T/1 |
| 2321 | T | T | T/9, C/1 | T/19, C/3 |
| 2435 | A | A | A/10 | A/21, G/1 |
| 2695 | C | C | C/10 | C/21, A/1 |
| 2712 | T | T | T/10 | T/21, C/1 |
| 地区 | 耐药菌株优势突变类型 | 发现的其他突变类型 |
| 宁波 | A2143G、A2142G、A2097G | T2182C、 A2097T、A2097C、A2116G、A2116T、A2099G、A2099C、A2223G、T2090G、T2095A |
| 青岛 | A2143G | T2182C、 A2223G、T2190C、C2195T |
| 贵阳 | A2144G | T2183C、C2916T、A2224G、T2245C |
| 黔南 | A2144G、A2224G | T2183C、T2245C、T2321C、C2196T、C2289T、A2435G、C2695A、T2712C |
区测序图; B: 发生A2224G突变的耐药菌株23S rRNA基因V区测序图.
克拉霉素属大环内酯类抗生素, 具有耐酸、能溶解于低pH胃液中的特性, 口服后生物利用度好, 不良反应少, 被认为是H. pylori根除治疗方案中最有效的抗生素[9]. 其抗菌机制为: 与细菌核糖体紧密结合后作用于细菌23S rRNA V区的多肽转移酶环, 抑制肽酰基转移酶活性, 阻止肽链延长, 抑制细菌蛋白质的合成而发挥抗菌作用. 然而, 随着克拉霉素的长期应用, 国内外报道H. pylori对克拉霉素耐药率呈逐年增高趋势[10-12]. 1996-2008年, 日本[13]菌株克拉霉素耐药率分别从0%上升至24.1%, 根治率却从70%下降到60%. 本文结果显示黔南州医院分离到的H. pylori菌株对克拉霉素的耐药率达到了29.7%, 高于原素梅等[14]报道的7.4%及杜军等[15]报道的9.17%. 根据国内外共识报告意见[16-18], 当克拉霉素耐药率高于15%-20%时, 则不宜再选用含有甲硝唑或克拉霉素的标准三联疗法作为根除H. pylori的一线方案, 应引起临床的重视.
H. pylori 23S rRNA基因V区上的点突变与克拉霉素的耐药性的产生有关[19]. 突变菌株核糖体和克拉霉素亲合力下降, 药物失去抑制细菌蛋白质合成的作用, 因而产生耐药性. 各国研究者[20,21]对H. pylori 23S rRNA V区的研究发现, 与克拉霉素耐药相关的突变主要包括: A2142G、A2143G/C、A2144G/C/T、T2183C、A2224G等. 不同国家和地区报道的突变位点碱基分布不尽相同. 西班牙[22]克拉霉素耐药株中85.3%(29/34)存在A2143G突变, 17.6%(6/34)存在T2182C突变, 8.8%(3/34)存在A2142G突变. 法国[23]克拉霉素耐药株中有81.9%(113/138)存在A2144G突变, 14.5%(20/138)存在A2143G突变, 仅3.6%(5/138)存在A2143C突变. 亚洲国家菌株耐药突变以A2143G为主[24-26]. 但孟加拉国[27]克拉霉素耐药菌株以T2182C突变为主, 而未发现A2143G和A2144G突变. 国内淮南、宁波、重庆及青岛等多地耐药菌株与亚洲国家突变情况大致相符, 以A2143G突变为主[7,8,28,29]. 而司书梅等[3]报道的贵阳地区耐药株主要存在A2144G突变. 以H. pylori U27270序列为参考, 本次黔南州医院临床分离的菌株中, 未发现文献报道的与耐药性相关的A2142G、A2143G/C、A2144C/T等突变; 耐药菌株及敏感菌株均发现有T2183C(13/22, 4/10)、T2245C(20/22, 10/10)和T2321C(3/22, 1/10)突变, 提示这几个位点的突变与本院分离的H. pylori克拉霉素耐药性关系不大; 仅在耐药株中检测到A2144G(4/22)、A2224G(4/22)、C2196T(1/22)、C2289T(1/22)、A2435G(1/22)、C2695A(1/22)、T2712C(1/22)突变, 其中以A2144G和A2224G突变率高于其他类型突变率, 提示其为本次分离临床菌株克拉霉素耐药性相关的主要突变类型; C2196T及C2289T突变在文献[20,30,31]中亦有报道, 本文进一步证实了这两个位点突变与H. pylori克拉霉素耐药的相关性. 有意义的是尚未见其他地区报道耐药株中有A2435G、C2695A、T2712C位点的突变, 提示本地区菌株23S rRNA V区基因碱基分布具有较为复杂的多态性, 有待进一步研究其与H. pylori克拉霉素耐药的相关性.
H. pylori分离培养困难, 且易发生耐药性变异, 因此若充分了解本地菌株耐药变异的机制和规律, 则有望通过分子生物学手段快速检测耐药性变异以选择敏感抗生素. 本文团队长期致力于临床菌株的分离和耐药性突变的研究.
李瑜元, 教授, 广州市第一人民医院内科
H. pylori的耐药性变异及感染复发的机制是本领域的研究热点和重点之一, 该菌的耐药变异是治疗失败的主要原因. 该菌培养困难, 难以普遍开展常规药敏检测, 其耐药变异的机制和规律及检测方法的建立是亟待研究的问题.
国内外有文献报道当地菌株的克拉霉素耐药相关基因突变, 各地的突变类型不尽相同. 本文作者之前也对贵阳菌株的突变特征进行了分析, 与本省贵阳相比, 黔南菌株的优势突变除A2144G外, 尚有A2224G突变.
如同基因芯片法在结核分枝杆菌耐药性检测中的开发和应用, 随着对H. pylori耐药相关基因突变的了解, 将有望研发快速诊断该菌耐药性的技术. 本文为该菌克拉霉素耐药相关基因突变规律的揭示提供了实验数据.
23S rRNA基因V区: 23S rRNA是细菌核糖体大亚基的组成部分, 是核糖体催化功能的核心, 其结构域V区具有肽酰转移酶活性, 负责转移肽酰基并催化肽键形成, 使肽链延长. 克拉霉素能与H. pylori核糖体23S rRNA V区紧密结合, 抑制肽酰转移酶活性, 影响核糖体的移位过程, 阻止肽链延长, 从而抑制细菌蛋白质的合成, 达到杀菌目的.
近年国内外已有较多对23S rRNA基因功能区用PCR测序的报告, 但各地区菌种不同. 本文报告贵州省黔南州的资料, 有一定价值.
编辑: 郭鹏 电编:闫晋利
| 1. | Lahner E, Virili C, Santaguida MG, Annibale B, Centanni M. Helicobacter pylori infection and drugs malabsorption. World J Gastroenterol. 2014;20:10331-10337. [PubMed] [DOI] |
| 3. | 司 书梅, 张 涛, 陈 峥宏, 王 菲, 吴 晓娟, 綦 廷娜, 杨 廷秀. 幽门螺杆菌临床菌株克拉霉素耐药性及耐药基因突变位点分析. 中国人兽共患病学报. 2012;28:1117-1119. |
| 5. | Mégraud F, Lehours P. Helicobacter pylori detection and antimicrobial susceptibility testing. Clin Microbiol Rev. 2007;20:280-322. [PubMed] |
| 6. | Barile KA, Silva AL, Xavier JN, Assumpção MB, Corvelo TC. Characterization of 23S rRNA domain V mutations in gastric biopsy patients from the eastern Amazon. Mem Inst Oswaldo Cruz. 2010;105:314-317. [PubMed] |
| 7. | 温 晋锋, 陈 韶华, 陈 春晓. 幽门螺杆菌对克拉霉素的耐药性与23S rRNA基因突变相关性分析. 中国中西医结合消化杂志. 2014;22:69-73. |
| 10. | Horiki N, Omata F, Uemura M, Suzuki S, Ishii N, Iizuka Y, Fukuda K, Fujita Y, Katsurahara M, Ito T. Annual change of primary resistance to clarithromycin among Helicobacter pylori isolates from 1996 through 2008 in Japan. Helicobacter. 2009;14:86-90. [PubMed] [DOI] |
| 11. | Glupczynski Y, Mégraud F, Lopez-Brea M, Andersen LP. European multicentre survey of in vitro antimicrobial resistance in Helicobacter pylori. Eur J Clin Microbiol Infect Dis. 2001;20:820-823. [PubMed] |
| 13. | Tokunaga K, Tanaka A, Sugano H, Takahashi S. [The present status and problems of Helicobacter pylori first-line eradication therapy]. Nihon Rinsho. 2009;67:2291-2296. [PubMed] |
| 16. | 中华医学会消化病学分会幽门螺杆菌学组/全国幽门螺杆菌研究协作组, 刘文忠, 谢勇, 成虹, 吕农华, 胡伏莲, 张万岱, 周丽雅, 陈烨, 曾志荣, 王崇文, 萧树东, 潘国宗, 胡品津. 第四次全国处理共识报告. 中华内科杂志. 2012;51:832-837. |
| 18. | Malfertheiner P, Megraud F, O'Morain CA, Atherton J, Axon AT, Bazzoli F, Gensini GF, Gisbert JP, Graham DY, Rokkas T. Management of Helicobacter pylori infection--the Maastricht IV/ Florence Consensus Report. Gut. 2012;61:646-664. [PubMed] [DOI] |
| 19. | Ribeiro ML, Vitiello L, Miranda MC, Benvengo YH, Godoy AP, Mendonca S, Pedrazzoli J. Mutations in the 23S rRNA gene are associated with clarithromycin resistance in Helicobacter pylori isolates in Brazil. Ann Clin Microbiol Antimicrob. 2003;2:11. [PubMed] [DOI] |
| 20. | Kim JM, Kim JS, Kim N, Kim YJ, Kim IY, Chee YJ, Lee CH, Jung HC. Gene mutations of 23S rRNA associated with clarithromycin resistance in Helicobacter pylori strains isolated from Korean patients. J Microbiol Biotechnol. 2008;18:1584-1589. [PubMed] |
| 21. | Sakinc T, Baars B, Wüppenhorst N, Kist M, Huebner J, Opferkuch W. Influence of a 23S ribosomal RNA mutation in Helicobacter pylori strains on the in vitro synergistic effect of clarithromycin and amoxicillin. BMC Res Notes. 2012;5:603. [PubMed] [DOI] |
| 22. | Agudo S, Pérez-Pérez G, Alarcón T, López-Brea M. Rapid detection of clarithromycin resistant Helicobacter pylori strains in Spanish patients by polymerase chain reaction-restriction fragment length polymorphism. Rev Esp Quimioter. 2011;24:32-36. [PubMed] |
| 23. | Raymond J, Lamarque D, Kalach N, Chaussade S, Burucoa C. High level of antimicrobial resistance in French Helicobacter pylori isolates. Helicobacter. 2010;15:21-27. [PubMed] [DOI] |
| 24. | Maeda S, Yoshida H, Matsunaga H, Ogura K, Kawamata O, Shiratori Y, Omata M. Detection of clarithromycin-resistant helicobacter pylori strains by a preferential homoduplex formation assay. J Clin Microbiol. 2000;38:210-214. [PubMed] |
| 25. | Kato S, Fujimura S, Udagawa H, Shimizu T, Maisawa S, Ozawa K, Iinuma K. Antibiotic resistance of Helicobacter pylori strains in Japanese children. J Clin Microbiol. 2002;40:649-653. [PubMed] |
| 26. | Pan ZJ, Su WW, Tytgat GN, Dankert J, van der Ende A. Assessment of clarithromycin-resistant Helicobacter pylori among patients in Shanghai and Guangzhou, China, by primer-mismatch PCR. J Clin Microbiol. 2002;40:259-261. [PubMed] |
| 27. | Khan R, Nahar S, Sultana J, Ahmad MM, Rahman M. T2182C mutation in 23S rRNA is associated with clarithromycin resistance in Helicobacter pylori isolates obtained in Bangladesh. Antimicrob Agents Chemother. 2004;48:3567-3569. [PubMed] [DOI] |
| 30. | Francesco VD, Zullo A, Hassan C, Giorgio F, Rosania R, Ierardi E. Mechanisms of Helicobacter pylori antibiotic resistance: An updated appraisal. World J Gastrointest Pathophysiol. 2011;2:35-41. [PubMed] [DOI] |
| 31. | Hao Q, Li Y, Zhang ZJ, Liu Y, Gao H. New mutation points in 23S rRNA gene associated with Helicobacter pylori resistance to clarithromycin in northeast China. World J Gastroenterol. 2004;10:1075-1077. [PubMed] |