修回日期: 2015-03-06
接受日期: 2015-03-18
在线出版日期: 2015-05-08
目的: 通过高通量转录组测序和相对定量血清蛋白组学技术筛选肝癌转移相关的关键基因.
方法: 利用Ion Proton™测序仪比较人肝癌细胞株(Smmc-7721)和正常人肝细胞株(L-02)的差异表达基因, 对差异表达基因进行聚类分析、GO功能注释和富集分析, 获得肝癌细胞转移相关基因; 收集10例肝细胞癌(hepatocellular carcinoma, HCC)患者血清及匹配10例正常人血清, 通过相对和绝对定量的同位素标记(isobaric tags for relative and absolute quantitation, iTRAQ)联合基质辅助激光解吸电离串联飞行时间质谱(matrix-assisted laser desorption/ionization tandem time of flight mass spectrometry, MALDI-TOF/MS)检测肝癌与正常对照组血清差异表达蛋白; 将两组学结果进行交集分析, 确定肝癌转移关键基因, 并在76例HCC和癌旁组织中进行免疫组织化学验证.
结果: 对肝癌细胞及正常肝细胞进行转录组测序分析, 共得到肝癌细胞差异表达基因618个, 生物信息学分析差异表达基因主要聚集在转移相关、转录因子相关、氧化还原过程等14个分子功能, 其中转移相关功能基因所占比例最大, 达15.05%(93/618); 血清蛋白组学分析共得到69个肝癌血清差异表达蛋白, 其中在肝癌组上调33个, 下调36个; 将转录组测序筛选的与转移功能相关基因与蛋白质组学进行交集分析, 发现有3个共同交集的差异因子, 其中差异最明显的是肝癌中表达上调的热休克蛋白90 AA1(heat shock protein 90 AA1, HSP90AA1); 免疫组织化学验证结果显示, HSP90α表达强阳性在门脉转移和无门脉转移HCC组织中的比例分别为66.7%(16/24)和25%(13/52)(P<0.005), HSP90α在有门脉转移的肝癌组织表达明显增高.
结论: 利用转录组结合血清蛋白组策略, 发现HSP90AA1可能是在肝癌转移重要基因.
核心提示: 转录组测序是发现标志物的可行方法. 但转录组测序数据庞大, 本研究将转录组学与血清蛋白质组学结合, 发现热休克蛋白90 AA1(heat shock protein 90 AA1)是肝癌转移相关的重要基因. HSP90α已被证明是全新的肿瘤标志物, 其抑制剂成为抗癌药研究热点.
引文著录: 秦荔荣, 周怡, 邓小芳, 李洪涛, 臧宁, 何敏. 转录组测序结合蛋白组学技术筛选肝癌转移基因. 世界华人消化杂志 2015; 23(13): 2050-2057
Revised: March 6, 2015
Accepted: March 18, 2015
Published online: May 8, 2015
AIM: To screening key genes related to hepatocellular carcinoma (HCC) metastasis by high-throughput transcriptomics sequencing and serum proteomics.
METHODS: Differentially expressed genes between liver cancer cells Smmc-7721 and normal liver cells L-02 were analyzed by Ion Proton™ high-throughput sequencing. Bioinformatics methods were used to perform GO annotation, clustering and enrichment analysis. Ten serum samples from HCC patients and 10 normal serum samples were recruited to detect the differential protein expression by isobaric tags for relative and absolute quantitation (iTRAQ) and matrix-assisted laser desorption/ionization tandem time of flight mass spectrometry (MALDI-TOF/MS). The transcriptomics data and serum proteomics data were analyzed together to screen key genes related to HCC metastasis. Then, a screened key gene was verified by immunohistochemistry in 76 HCC and adjacent tissues.
RESULTS: A total of 618 differentially expressed genes (DEGs) in liver cancer cells were identified by transcriptome sequencing, and the gene functions were enriched in 14 terms, including metastasis process, transcription and REDOX process, among which metastasis process owned the most DEGs [15.05% (93/618)]. The proteomics data showed that a total of 69 differentially expressed proteins in HCC were detected, including 33 up-regulated and 36 down-regulated ones. Combination analysis found three common factors in transcriptomics and proteomics, among which heat shock protein 90 AA1 (HSP90AA1) was up-regulated in HCC and presented the most significant ratio. According to the immunohistochemical results, the strongly positive rates of HSP90α in HCC with portal vein metastasis and without were 66.7% (16/24) and 25% (13/52), respectively (P < 0.005). HSP90α was overexpressed in HCC with portal vein metastasis.
CONCLUSION: Transcriptomics and proteomics analysis revealed that HSP90AA1 might be a key gene related to HCC metastasis.
- Citation: Qin LR, Zhou Y, Deng XF, Li HT, Zang N, He M. Identification of genes related to hepatocellular carcinoma metastasis by a combined transcriptomics and proteomics approach. Shijie Huaren Xiaohua Zazhi 2015; 23(13): 2050-2057
- URL: https://www.wjgnet.com/1009-3079/full/v23/i13/2050.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i13.2050
肝癌的转移与复发是导致肝癌具有较高病死率的原因, 至今对肝癌转移的机制不清[1,2]. 转录组测序是发现功能基因和标志物的一种可行方法[3]. 20世纪70年代诞生第一代化学降解法测序, 到二代测序技术已经具备高通量、高度并行等特点, 但昂贵的费用成本成为其大范围应用的绊脚石, 新发展起来的第三代测序技术以较低成本、长度长及高碱基识别率为测序技术发展带来广阔的前景, 已经广泛应用于包括结肠癌、前列腺癌等肿瘤标志物研究[4,5]. 由于转录组测序的结果数据庞大, 如何从中挖掘出有价值的关键基因, 除经典的差异表达基因聚类分析、GO功能注释、富集分析, Kegg Pathway功能富集分析, COG功能注释分析等, 本研究尝试利用多组学交集分析策略, 为肝癌转移相关重要基因和标志物研究提供有价值的线索.
正常人肝细胞株(L-02)、人肝癌细胞株(Smmc-7721)均购于中国科学院细胞库; RNA纯化免疫磁珠试剂盒(Life Technologies, USA); 一抗[Anti-热休克蛋白90α(heat shock protein 90α, HSP90α)antibody2G5.G3, abcam, USA]; 相对和绝对定量的同位素标记(isobaric tags for relative and absolute quantitation, iTRAQ)试剂盒(AB, USA). 10例肝细胞癌(hepatocellular carcinoma, HCC)患者血清, 来自广西医科大学附属肿瘤医院的住院患者, 10例健康对照者血清来自广西医科大学第一附属医院体检中心. 76例肝癌组织及癌旁组织标本收集于2011-11/2013-09广西医科大学第一附属医院肝胆外科.
1.2.1 转录组测序分析肝癌差异表达基因: (1)转录组学测序检测: 正常人肝细胞株(L-02)、人肝癌细胞株(Smmc-7721)均购于中国科学院细胞库. 提取总RNA, NanoDrop分光光度计测定RNA样品纯度和浓度, 2%琼脂糖凝胶电泳检测RNA完整性. 使样本的RNA含量均在100 µg以上, RNA纯化免疫磁珠试剂盒分离纯化mRNA, 使其含量达5-400 ng. 纯化的mRNA片段化处理后, 反转录反应合成cDNA, 再用聚合酶扩增构建成功的测序文库. 利用Ion Proton™测序仪对样品进行高通量测序; (2)生物信息学分析: Proton™ Torrent服务器预加载了Torrent Suite软件, 测序分析获得的数据自动传至Proton™ Torrent服务器, 对转录组测序数据进行数据覆盖度、对比信息统计, 作覆盖分析、作图、变异识别等检测, FPKM法计算样本的表达量, Fold-change法分析基因的差异表达, 发现新基因或新转录本Ion Reporter™软件进行生物学功能注释和常见变异体的鉴定, 对差异表达进行聚类分析、GO功能注释富集分析, Kegg Pathway功能富集分析. 基因表达水平在肝癌细胞和正常肝细胞分别用A和B表示, 计算每个基因的log2(A/B), log2(A/B)≥3或log2(A/B)≤-3为明显表达差异基因.
1.2.2 iTRAQ联合基质辅助激光解吸电离串联飞行时间质谱(matrix-assisted laser desorption/ionization tandem time of flight mass spectrometry, MALDI-TOF/MS)筛选和鉴定肝癌血清差异表达蛋白: (1)相对定量蛋白组学检测: 10例HCC和10例健康对照血清, 均为清晨空腹采集, 3000 g离心10 min, -80 ℃保存. 血清经除高丰度蛋白, 蛋白酶解, iTRAQ标记(HCC标记116, 对照标记113), SCX强阳离子交换柱分离蛋白与反相色谱分离与点靶, 5800 MALDI质谱仪检测. 详细操作参照文献[6]; (2)血清差异表达蛋白筛选和鉴定: 所得的数据用IPI数据库(ipi.HUMAN.V3.62.fasta)进行蛋白搜库检索, 鉴定报告的蛋白至少有一个95%以上置信度的肽段, 116:113>1.2或<0.8, P<0.05. 同时, 经含正反两种人类蛋白获得模式的数据库评估错检率(false discovery rate, FDR), 评估值低于5.0%.
1.2.3 免疫组织化学验证: (1)样本: 采集76例肝癌组织及癌旁组织, 患者术前证实未接受过化疗和介入治疗并经病理检查证实为肝癌患者, 术前签署知情同意书; (2)免疫组织化学染色SP法: 按照北京中杉金桥生物技术公司的试剂说明书操作, 一抗浓度1:400; 4 ℃冰箱孵育过夜. 染色, 细胞核复染用苏木精染色40 s, 盐酸乙醇分化后充分洗涤、晾干、封片镜检; (3)结果判定: 免疫组织化学组织切片以胞浆内出现棕黄色颗粒为目标蛋白阳性细胞, 采用半定量方法分析表达水平, 参照Liu等[7]的研究. 200倍显微镜下观察切片, 随机观察每个视野内的细胞, 染色强度记分: 无染色为0分, 浅黄色为1分, 棕黄色为2分, 棕褐色为3分. 染色阳性细胞占比例: <5%为0分; 5%-25%为1分; 26%-50%为2分; 51%-75%为3分; 76%-100%为4分. 蛋白表达为两种评分之和, 表达强度分三个等级, 0分: "0"; 1-2分: "1+"; 3-5分: "2+"; 6-7分: "3+"; "3+"为强阳性.
统计学处理 采用SPSS19.0统计学软件进行非参数检验分析, P<0.05为差异有统计学意义.
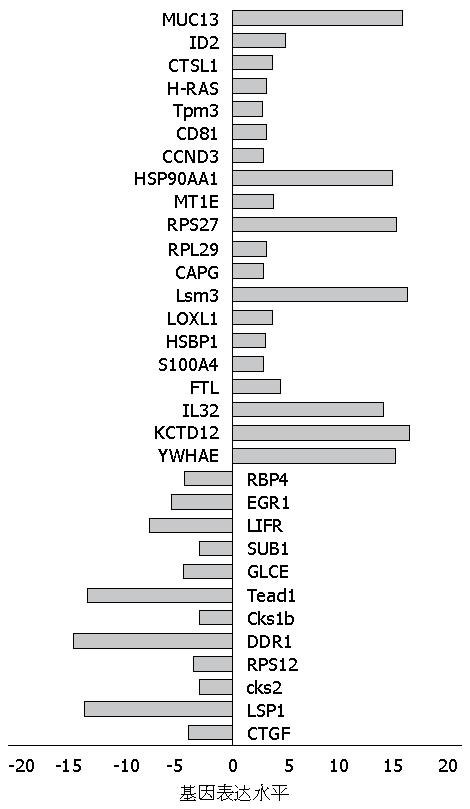
测序原始数据经质量评估和过滤之后, 肝癌组与正常对照组各得到10.5 G和8.2 G可用的数据产量, 平均读长在110 bp左右, 可信读段百分比分别为65%和59%. FPKM法计算基因的表达量, 绝对表达值倍数变化(Fold-change)法比较两组的差异表达, 共得出差异基因618个(Fold-change≥1.0或Fold-change≤-1.0). 对618个差异表达基因GO功能分类, 包括binding, cellular process, biological regulation在内的63个功能组, 这些差异表达基因主要聚集在转移相关、转录因子相关、氧化还原过程等14个分子功能. 其中转移相关功能基因所占比例最大, 达15.05%(93/618), 其次为转录因子相关基因8.09%(50/618), 氧化还原过程相关基因6.63%(41/618). 本研究将93个转移相关功能基因作为主要的关注对象, 其中在肝癌细胞明显表达增高基因20个, 明显表达水平降低的基因12个, 如图1所示.
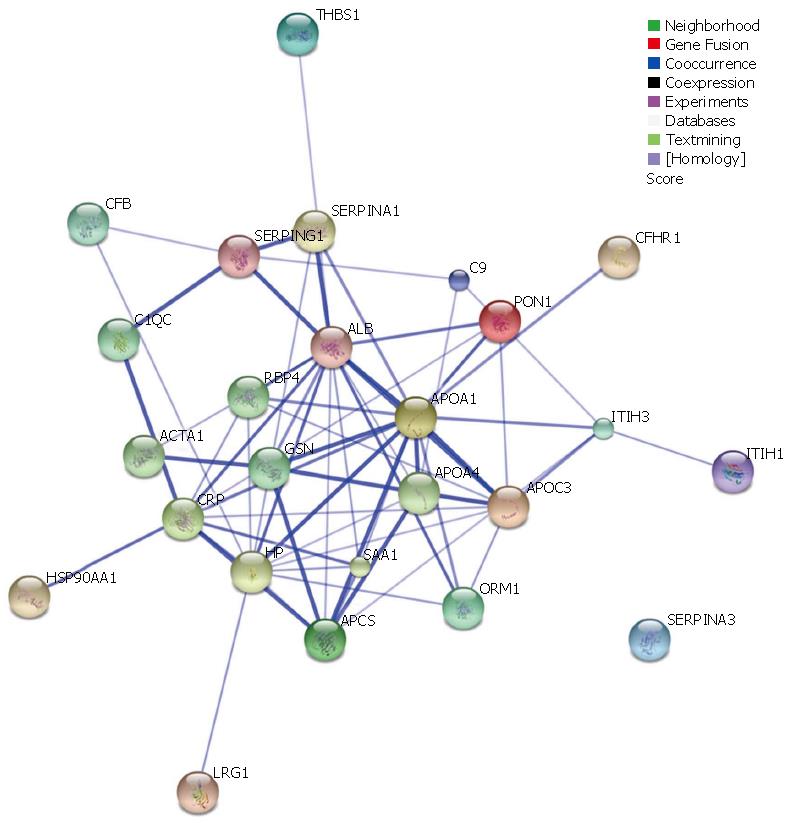
质谱检测结果经IPI数据库检索鉴定, 识别的蛋白共189个, 按照差异蛋白的纳入标准筛选, 共得到69个差异蛋白, 其中在肝癌患者血清中上调33个, 下调36个. 对报告的69个蛋白作PANTHER蛋白分类分析, 发现大部分蛋白属于防御/免疫蛋白类(20.5%)、酶调节类(20.5%)及转移/载体蛋白类(16.4%). 69个差异蛋白确认的相互作用图如图2所示.
血清差异蛋白结合转录组测序筛选的与转移功能相关基因分析, 发现蛋白质组学与转录组学可得到3个共同的交集差异因子如图3所示, 其中肝癌中表达上调的HSP90AA1基因, 在蛋白质组学和转录组学均显示差异表达变化最明显如表1所示, 因此, 将该基因作为验证的目标基因.
| 编号 | 基因 | 表达水平 | log2(A/B) | log2(116/113) | P值 |
| ENSG00000173432 | SAA1 | 上调 | 1.8194 | 1.970412 | <0.05 |
| ENSG00000080824 | HSP90AA1 | 上调 | 4.9306 | 4.929744 | <0.05 |
| ENSG00000138207 | RBP4 | 下调 | -4.1720 | -1.522430 | <0.05 |
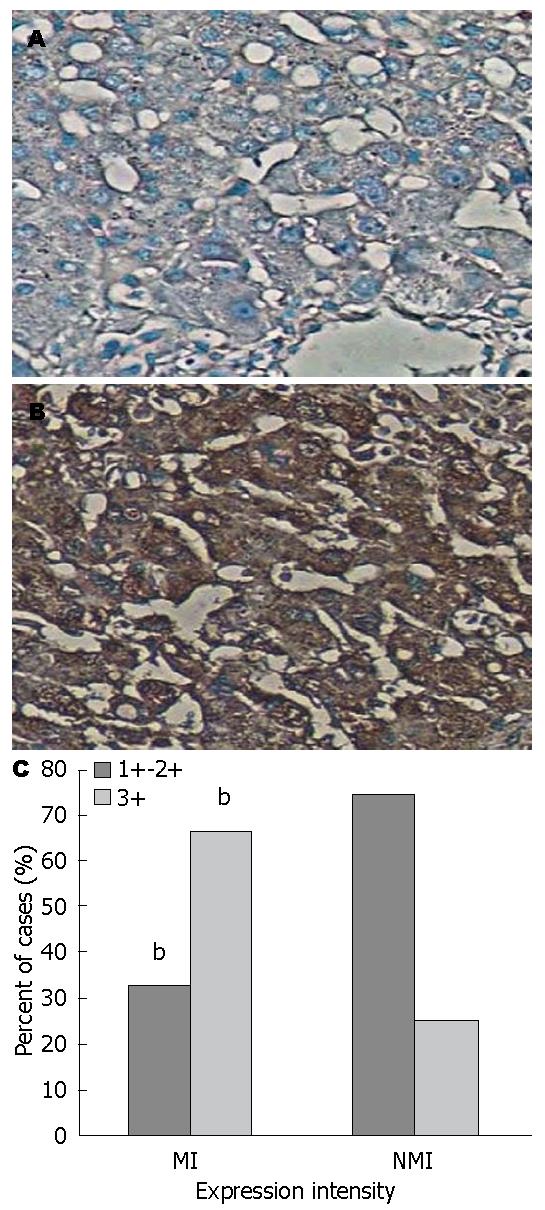
经过RT-PCR验证HSP90AA1在肝癌细胞和肝癌组织中表达水平明显增高, 免疫组织化学结果显示, HSP90AA1编码的蛋白HSP90α强阳性病例的比例, 在门脉转移和无门脉转移HCC患者组织中分别为66.7%(16/24)和25%(13/52)(P<0.001), HSP90α在有门脉转移的肝癌组织表达明显增高, 如图4所示.
比较基因组学研究为探索肝癌致病机制带来全新思路[8,9], 他的主要目标是研究肝癌与对照的基因差异表达, 进而筛选肝癌相关生物标志物. 直接测序技术即第三代测序, 包括Heliscope测序技术、纳米孔单分子测序技术、Ion Torrent测序技术等[10-12]. Ion Torrent测序技术使用一种布满小孔的高密度半导体芯片, 一个小孔就是一个测序反应池. 当DNA聚合酶把核苷酸聚合到延伸中的DNA链上时, 释放出一个氢离子, 反应池中的PH发生改变, 位于池下的离子感受器感受到信号, 把化学信号直接转化为数字信号. 该技术平台测序分析获得的数据均可以自动传至其服务器预加载的Torrent Suite软件, 将原始的半导体测序数据直接识别为DNA碱基序列. 本研究利用Ion Proton™测序仪对肝癌细胞Smmc-7721及对照组正常肝细胞L-02进行转录组测序, 筛选肝癌与正常对照组的差异表达基因, 两组样本均得到了较高质量的数据结果, 通过FPKM法计算基因表达水平及Fold-change法比较两组样本的差异表达分析, 共得到618个肝癌与正常对照组的差异表达基因. 利用生物信息学GO功能分类分析发现差异表达基因中, 转移相关功能基因所占百分比最大, 共93个(15.05%), 其次为转录因子相关基因50个(11.14%), 氧化还原过程相关基因41个(9.13%), 因而与转移相关的基因值得关注.
在转录组学这些相对海量的改变中, 既含有肿瘤形成所必需的关键性改变, 也含有仅仅因肿瘤基因组的不稳定性而产生的伴随性改变. 因此, 如何将那些影响肿瘤发生发展的关键性分子改变从伴随性改变中识别出来, 即如何从93个肝癌转移相关的基因中挖掘出关键基因, 本研究采用转录组与血清蛋白组联合分析的策略, 这主要出于两种考虑: (1)近年来许多疾病标志物研究采用转录组结合蛋白组的策略取得新进展[13,14]; (2)对于其他的临床标本, 人类血清具有含量相当丰富、收集安全、保存方便等优点, 分析外周血中蛋白质图谱的变化可以反映病变组织中蛋白质图谱的改变, 如果通过转录组发现关键基因其转录翻译的蛋白同时在血清中检测到趋势一致的表达水平, 其实用性和应用价值将大大提高. 因此, 本研究将转录组测序结果与同位素相对标记与绝对定量技术筛选肝癌血清差异蛋白相结合, 筛选到3个共同出现在蛋白质组学与基因组学差异因子, 其中在肝癌中表达上调的HSP90AA1在蛋白质组学和转录组学差异表达值均最大, 已有研究表明HSP90AA1编码的蛋白HSP90α在前列腺癌血清呈上调表达[15], 而其他两个因子与肿瘤转移的关系研究则刚刚开始起步, 为此确定HSP90AA1为进一步验证的关键基因.
HSP90α是一个分子伴侣蛋白, 属于人类HSP90的4个亚型之一HSP90AA1编码的蛋白质, 目前认为HSP90 4个亚型的作用方式几乎一致, 主要是促进目标蛋白的成熟与结构维护, 参与ATP酶活性有关的功能循环, 维持蛋白的正常功能发挥[16]. 早在1992年, 苯醌安莎霉素类(benzoquinone ansamycins)抗生素被发现有强大的抗肿瘤活性而成为肿瘤抑制剂, 该类抗生素的结合蛋白就是HSP90[17]. 近年来HSP90α陆续被发现在多种肿瘤呈上调表达, 包括肺癌、胃癌、乳腺癌、卵巢癌、恶性胶质瘤等[18-22], 被证明为一个全新的肿瘤标志物. HSP90α在恶性肿瘤的转移过程中的作用也倍受关注, 作为分子伴侣, HSP90α可以与基质金属蛋白酶基质金属蛋白酶2(matrix metallopeptidase 2, MMP2)、MMP9等共同作用影响肿瘤细胞运动能力和侵袭能力, 过表达的HSP90α可以增强细胞运动和侵袭能力, 反之则产生抑制作用[23-25]. 肿瘤细胞内HSP90α与细胞增殖、凋亡功能密切相关, Chu等[22]研究发现卵巢癌细胞SKOV3内HSP90AA1的RNAi干扰抑制了细胞的增殖, 并使凋亡增加; 乳腺癌细胞株经HSP90-shRNA处理使HSP90活性被抑制后, 细胞停留在G1/S期[26]. 因此, 目前HSP90抑制剂已成为抗癌药物研究热点.
虽然目前有关HSP90AA1与肝癌的关系研究很少, 本课题利用转录组学测序的方法发现转移相关基因HSP90AA1在肝癌细胞中高表达, 利用血清蛋白质组学发现该基因编码的蛋白HSP90α在肝癌患者血清中表达水平也增高, HSP90α在肝癌组织中表达增高与肝癌门静脉转移相关, 提示HSP90AA1是肝癌转移相关的重要基因.
肝癌的转移与复发是导致肝癌具有较高病死率的原因, 至今对肝癌转移的机制不清. 蛋白组学、转录组学、代谢组学等新技术都是发现功能基因和标志物的新方法, 各种组学技术的有机结合将为肝癌转移相关重要基因和标志物研究提供有价值的线索.
王蒙, 副教授, 中国人民解放军第二军医大学附属东方肝胆外科医院肝外综合治疗一科
转录组学是发现功能基因和标志物的一种可行方法. 由于转录组测序的结果数据庞大, 如何从中挖掘出有价值的关键基因, 除经典的差异表达基因聚类分析、GO功能注释、富集分析, Kegg Pathway功能富集分析, COG功能注释分析等, 本研究尝试将转录组学与血清蛋白质组学技术结合, 发现热休克蛋白90 AA1(heat shock protein 90 AA1, HSP90AA1)是肝癌转移相关的重要基因.
现有研究发现: HSP90α是HSP90AA1编码的蛋白质, 作为一个分子伴侣蛋白, 主要是促进目标蛋白的成熟与结构维护, 参与ATP酶活性有关的功能循环, 维持蛋白的正常功能发挥; HSP90α被发现在多种肿瘤呈上调表达, 被证明为一个全新的肿瘤标志物; HSP90α在恶性肿瘤的转移过程中可以与基质金属蛋白酶基质金属蛋白酶基质金属蛋白酶2(matrix metallopeptidase 2, MMP2)、MMP9等共同作用影响肿瘤细胞运动能力和侵袭能力; 目前HSP90抑制剂已成为抗癌药物研究热点.
本课题首先利用新一代转录组学测序的方法筛选肝癌转移相关基因; 其次利用转录组与血清蛋白质组结合的策略, 快速确定肝癌转移相关的关键基因; 最后经验证首次报道HSP90AA1是肝癌转移相关的重要基因.
(1)利用转录组与血清蛋白质组结合的策略, 不仅可以快速确定肝癌转移相关的关键基因, 也可用于快速确定转录因子、氧化还原、凋亡等相关基因; (2)首次报道肝癌转移相关的重要基因HSP90AA1, 其编码蛋白在血清和组织有一致表达水平, 是具有可操作性和应用价值的潜在标志物.
高通量测序技术: 是对传统测序一次革命性的改变, 一次对几十万到几百万条DNA分子进行序列测定, 因此在有些文献中称其为下一代测序技术, 足见其划时代的改变, 同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能, 所以又被称为深度测序.
本文研究方法创新性好, 内容新颖, 结果有新意, 具有较好的科学意义.
编辑: 郭鹏 电编: 都珍珍
| 1. | Shen H, Zhong F, Zhang Y, Yu H, Liu Y, Qin L, He F, Tang Z, Yang P. Transcriptome and proteome of HCC reveal shared metastatic pathways with significant genes. Proteomics. 2015; Feb 4. [Epub ahead of print]. [PubMed] [DOI] |
| 2. | Chen CY, Zhong JH, Liu JL. Retrobulbar metastasis and intracranial invasion from postoperative hepatocellular carcinoma: A case report and review of the literature. Oncol Lett. 2015;9:721-726. [PubMed] [DOI] |
| 3. | Lindskog C, Fagerberg L, Hallström B, Edlund K, Hellwig B, Rahnenführer J, Kampf C, Uhlén M, Pontén F, Micke P. The lung-specific proteome defined by integration of transcriptomics and antibody-based profiling. FASEB J. 2014;28:5184-5196. [PubMed] [DOI] |
| 4. | Løvf M, Nome T, Bruun J, Eknaes M, Bakken AC, Mpindi JP, Kilpinen S, Rognum TO, Nesbakken A, Kallioniemi O. A novel transcript, VNN1-AB, as a biomarker for colorectal cancer. Int J Cancer. 2014;135:2077-2084. [PubMed] [DOI] |
| 5. | Long Q, Xu J, Osunkoya AO, Sannigrahi S, Johnson BA, Zhou W, Gillespie T, Park JY, Nam RK, Sugar L. Global transcriptome analysis of formalin-fixed prostate cancer specimens identifies biomarkers of disease recurrence. Cancer Res. 2014;74:3228-3237. [PubMed] [DOI] |
| 6. | He X, Wang Y, Zhang W, Li H, Luo R, Zhou Y, Liao CL, Huang H, Lv X, Xie Z. Screening differential expression of serum proteins in AFP-negative HBV-related hepatocellular carcinoma using iTRAQ -MALDI-MS/MS. Neoplasma. 2014;61:17-26. [PubMed] [DOI] |
| 7. | Liu XK, Zhang XR, Zhong Q, Li MZ, Liu ZM, Lin ZR, Wu D, Zeng MS. Low expression of PTK6/Brk predicts poor prognosis in patients with laryngeal squamous cell carcinoma. J Transl Med. 2013;11:59. [PubMed] [DOI] |
| 8. | Na K, Jeong SK, Lee MJ, Cho SY, Kim SA, Lee MJ, Song SY, Kim H, Kim KS, Lee HW. Human liver carboxylesterase 1 outperforms alpha-fetoprotein as biomarker to discriminate hepatocellular carcinoma from other liver diseases in Korean patients. Int J Cancer. 2013;133:408-415. [PubMed] [DOI] |
| 9. | Zhong DN, Ning QY, Wu JZ, Zang N, Wu JL, Hu DF, Luo SY, Huang AC, Li LL, Li GJ. Comparative proteomic profiles indicating genetic factors may involve in hepatocellular carcinoma familial aggregation. Cancer Sci. 2012;103:1833-1838. [PubMed] [DOI] |
| 10. | Harris TD, Buzby PR, Babcock H, Beer E, Bowers J, Braslavsky I, Causey M, Colonell J, Dimeo J, Efcavitch JW. Single-molecule DNA sequencing of a viral genome. Science. 2008;320:106-109. [PubMed] [DOI] |
| 11. | Clarke J, Wu HC, Jayasinghe L, Patel A, Reid S, Bayley H. Continuous base identification for single-molecule nanopore DNA sequencing. Nat Nanotechnol. 2009;4:265-270. [PubMed] [DOI] |
| 12. | Sandberg J, Werne B, Dessing M, Lundeberg J. Rapid flow-sorting to simultaneously resolve multiplex massively parallel sequencing products. Sci Rep. 2011;1:108. [PubMed] [DOI] |
| 13. | Lin LL, Hsia CR, Hsu CL, Huang HC, Juan HF. Integrating transcriptomics and proteomics to show that tanshinone IIA suppresses cell growth by blocking glucose metabolism in gastric cancer cells. BMC Genomics. 2015;16:41. [PubMed] [DOI] |
| 14. | Azimzadeh O, Sievert W, Sarioglu H, Merl-Pham J, Yentrapalli R, Bakshi MV, Janik D, Ueffing M, Atkinson MJ, Multhoff G. Integrative proteomics and targeted transcriptomics analyses in cardiac endothelial cells unravel mechanisms of long-term radiation-induced vascular dysfunction. J Proteome Res. 2015;14:1203-1219. [PubMed] [DOI] |
| 15. | Burgess EF, Ham AJ, Tabb DL, Billheimer D, Roth BJ, Chang SS, Cookson MS, Hinton TJ, Cheek KL, Hill S. Prostate cancer serum biomarker discovery through proteomic analysis of alpha-2 macroglobulin protein complexes. Proteomics Clin Appl. 2008;2:1223. [PubMed] [DOI] |
| 16. | Martínez-Ruiz A, Villanueva L, González de Orduña C, López-Ferrer D, Higueras MA, Tarín C, Rodríguez-Crespo I, Vázquez J, Lamas S. S-nitrosylation of Hsp90 promotes the inhibition of its ATPase and endothelial nitric oxide synthase regulatory activities. Proc Natl Acad Sci U S A. 2005;102:8525-8530. [PubMed] [DOI] |
| 17. | Xie Q, Wondergem R, Shen Y, Cavey G, Ke J, Thompson R, Bradley R, Daugherty-Holtrop J, Xu Y, Chen E. Benzoquinone ansamycin 17AAG binds to mitochondrial voltage-dependent anion channel and inhibits cell invasion. Proc Natl Acad Sci U S A. 2011;108:4105-4110. [PubMed] [DOI] |
| 18. | Senju M, Sueoka N, Sato A, Iwanaga K, Sakao Y, Tomimitsu S, Tominaga M, Irie K, Hayashi S, Sueoka E. Hsp90 inhibitors cause G2/M arrest associated with the reduction of Cdc25C and Cdc2 in lung cancer cell lines. J Cancer Res Clin Oncol. 2006;132:150-158. [PubMed] [DOI] |
| 19. | Chang W, Ma L, Lin L, Gu L, Liu X, Cai H, Yu Y, Tan X, Zhai Y, Xu X. Identification of novel hub genes associated with. liver metastasis of gastric cancer. Int J Cancer. 2009;125:2844-2853. [PubMed] [DOI] |
| 20. | Cheng Q, Chang JT, Geradts J, Neckers LM, Haystead T, Spector NL, Lyerly HK. Amplification and high-level expression of heat shock protein 90 marks aggressive phenotypes of human epidermal growth factor receptor 2 negative breast cancer. Breast Cancer Res. 2012;14:R62. [PubMed] [DOI] |
| 21. | Com E, Clavreul A, Lagarrigue M, Michalak S, Menei P, Pineau C. Quantitative proteomic Isotope-Coded Protein Label (ICPL) analysis reveals alteration of several functional processes in the glioblastoma. J Proteomics. 2012;75:3898-3913. [PubMed] [DOI] |
| 22. | Chu SH, Liu YW, Zhang L, Liu B, Li L, Shi JZ, Li L. Regulation of survival and chemoresistance by HSP90AA1 in ovarian cancer SKOV3 cells. Mol Biol Rep. 2013;40:1-6. [PubMed] [DOI] |
| 23. | Eustace BK, Sakurai T, Stewart JK, Yimlamai D, Unger C, Zehetmeier C, Lain B, Torella C, Henning SW, Beste G. Functional proteomic screens reveal an essential extracellular role for hsp90 alpha in cancer cell invasiveness. Nat Cell Biol. 2004;6:507-514. [PubMed] [DOI] |
| 24. | Eustace BK, Jay DG. Extracellular roles for the molecular chaperone, hsp90. Cell Cycle. 2004;3:1098-1100. [PubMed] [DOI] |
| 25. | Stellas D, El Hamidieh A, Patsavoudi E. Monoclonal antibody 4C5 prevents activation of MMP2 and MMP9 by disrupting their interaction with extracellular HSP90 and inhibits formation of metastatic breast cancer cell deposits. BMC Cell Biol. 2010;11:51. [PubMed] [DOI] |
| 26. | Zuo K, Li D, Pulli B, Yu F, Cai H, Yuan X, Zhang X, Lv Z. Short-hairpin RNA-mediated Heat shock protein 90 gene silencing inhibits human breast cancer cell growth in vitro and in vivo. Biochem Biophys Res Commun. 2012;421:396-402. [PubMed] [DOI] |