修回日期: 2014-11-03
接受日期: 2014-11-12
在线出版日期: 2014-12-28
目的: 富含嘌呤元件结合蛋白α(purine-rich element binding protein alpha, PURα)是调控细胞周期和恶性转化的关键因子. 本研究旨在探讨在食管鳞状细胞癌中转录因子PURα蛋白的多种功能及其相关机制.
方法: 构建过表达PURα蛋白的食管癌细胞KYSE510-PURα和pCMV6空载体转染的对照食管癌细胞KYSE 510-pCMV6. 用蛋白免疫印迹和细胞免疫荧光分析检测过表达PURα蛋白的食管癌细胞和对照食管癌细胞中上皮间叶转变相关蛋白的表达差异, 如E-钙黏蛋白和波形蛋白等; 采用细胞划痕实验和Transwell小室检测食管癌细胞的侵袭转移能力.
结果: 成功构建过表达PURα的食管癌细胞. 蛋白免疫印迹和免疫荧光分析表明, 与对照细胞相比, 过表达PURα的食管癌细胞中E-钙黏蛋白的表达减少, N-钙黏蛋白、波形蛋白和Snail蛋白表达升高. 引人注目的是, 过表达PURα的食管癌细胞的划痕愈合能力增强, 侵袭性显著增加. 与此同时, 细胞形态发生了纤维样改变, 呈现上皮间叶转化表型.
结论: PURα能通过调控食管鳞癌细胞上皮间叶转化而促进肿瘤细胞的侵袭转移能力.
核心提示: 过表达富含嘌呤元件结合蛋白α(purinerich element binding protein alpha, PURα)的食管癌细胞划痕愈合能力增强, 侵袭性显著增加. 同时, 细胞形态发生了纤维样改变, 呈现上皮间叶转化表型. 说明PURα具有调控食管癌细胞运动和上皮间叶转变的作用, 并间接影响食管癌的侵袭和转移.
引文著录: 郭志敏, 李薇, 赵晓航. PURα通过上皮间叶转化促进食管癌细胞KYSE 510的侵袭和转移. 世界华人消化杂志 2014; 22(36): 5579-5586
Revised: November 3, 2014
Accepted: November 12, 2014
Published online: December 28, 2014
AIM: To investigate the role of purine-rich element binding protein alpha (PURα) in the invasion and migration of esophageal squamous cell carcinoma (ESCC) KYSE 510 cells and the underlying mechanisms.
METHODS: An ESCC cell line overexpressing PURα (KYSE 510-PURα) was established, and the expression levels of epithelial-mesenchymal transition associated proteins were determined by comparing with control cells transfected with an empty vector of pCMV6 (KYSE 510-pCMV6). The expression of E-cadherin and vimentin was analyzed by Western blot and immunofluorescent staining. The capabilities of invasion and migration of cancer cells were assessed via transwell and wound healing assays.
RESULTS: PURα was overexpressed in KYSE 510 cells transfected with the pCMV6- PURα vector. The expression of E-cadherin was reduced, and that of Vimentin, N-cadherin and Snail was increased in KYSE 510-PURα cells. Most strikingly, the cell morphology was changed as fibroblasts and the abilities of migration and invasion were altered.
CONCLUSION: Our data suggest that regulation of PURα expression in ESCC cells may induce esophageal epithelial-mesenchymal transition.
- Citation: Guo ZM, Li W, Zhao XH. Purine-rich element binding protein alpha promotes invasion and migration of esophageal squamous cell carcinoma KYSE 510 cells by inducing epithelial-mesenchymal transition. Shijie Huaren Xiaohua Zazhi 2014; 22(36): 5579-5586
- URL: https://www.wjgnet.com/1009-3079/full/v22/i36/5579.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i36.5579
食管癌是我国常见恶性肿瘤之一, 其发病率居我国恶性肿瘤第4位[1]. 食管癌的主要组织学类型包括食管鳞状细胞癌(esophageal squamous cell carcinoma, ESCC)和食管腺癌(esophageal adenocarcinoma, EA)两种, ESCC是我国食管癌的主要组织类型[2]. 食管癌进展较快, 易发生淋巴结转移, 预后较差, 5年生存率<10%[3-5]. 侵袭和转移是与食管癌预后差密切相关的重要生物学特性. 因此, 探讨食管癌细胞侵袭转移的分子机制对认识食管癌癌变机制, 寻找新的治疗方法具有重要意义.
细胞上皮间叶改变(epithelial-mesenchymal transition, EMT)是胚胎发育、组织重建和伤口修复的基础过程, 同时也是肿瘤细胞突破基质束缚, 进入循环系统发生远处转移的重要机制[6]. 在这一过程中, 上皮细胞由于受到某种外界信号的刺激和内源转录因子的影响, 获得了间叶样的表型, 由多角形叠瓦状排列转变为梭形不规则排列, 同型黏附能力减弱, 异型黏附能力和运动能力增强. 生长因子信号在大多数情况下诱导许多转录因子表达, 而转录因子对细胞上皮间叶转变转变具有关键作用, 如Snail-1/2、Zeb-1/2和Twist等为强有力的抑制性转录因子, 可抑制维持上皮极性和组织结构的基因表达[7,8]. 其中Snail作为参与上皮间叶转变过程重要的转录因子, 通过直接抑制E-cadherin转录和间接促进间叶表型分子如Vimentin、N-cadherin的转录从而促使细胞上皮间叶转变转化[9]. 许多转录因子在细胞EMT转变的不同阶段发挥调控作用[10].
富含嘌呤元件结合蛋白α(purine-rich element binding protein alpha, PURα)是PUR蛋白家族成员之一, 能结合单链或双链DNA以及RNA, 从而在DNA复制和RNA转录与翻译过程中发挥重要作用[11]. 研究[12]表明, 转录因子PURα与多种肿瘤的形成和脑肿瘤的发育密切相关. PURα在DNA复制中双链DNA的解旋过程中起着重要的作用, 同时能与单链DNA或RNA结合, 促进后者的不稳定性[13]. 我们前期工作[14]发现, PURα转录因子可通过与E2F1和PolⅡ相互作用而结合到CASP8的第二个启动子, 影响CASP8基因G亚型的表达. 本研究对比食管癌KYSE 510细胞过表达PURα前后, 食管癌细胞划痕愈合、迁移和上皮间叶转变相关表型改变, 发现PURα具有调控食管癌细胞运动和上皮间叶转变的作用.
人食管癌细胞KYSE 510由日本兵库县医学院Shimada Y教授惠赠. RPMI 1640和Opti-MEM培养液、LipofectamineTM 2000购自Gibco/BRL公司(Grand land, NY, USA)公司; 胎牛血清购自PAA公司(Pasching, Austria); 兔抗人Snail和PURα多克隆抗体购自Abcam(Cambridge, UK)公司; 小鼠抗人E-cadherin单克隆抗体购自BD Transduction Lab(Lexington, KY, USA); 小鼠抗人β-actin单克隆抗体购自Sigma-Aldrich(St. Louis, MO, USA); 小鼠抗人Flag单克隆抗体和pCMV6-Entry-PURα真核基因表达质粒购自Origene公司(Rockville, MD, USA); Alexa 488标记荧光二抗购自Invitrogen(Carslbad, CA, USA); 辣根过氧化物酶标记山羊抗兔和抗鼠二抗购自中杉金桥生物技术公司(北京, 中国); DH5α大肠杆菌感受态细胞购自北京天根生物技术公司; pCMV6-Entry-PURα质粒购自Origene(Rockville, USA); 倒置相差和Nikon E400荧光显微镜为Nikon(Japan)产品; TCS SP2共聚焦显微镜为Leica产品; LAS4000图像分析仪为GE公司产品; 细胞培养箱购自 NAPCO(Winchester, VA, USA).
1.2.1 过表达PURα食管癌稳定细胞株构建与筛选: pCMV6-Entry-PURα为SV40启动子驱动的, 带有青霉素和新霉素抗性基因的PURα真核基因表达载体, 其重组PURα蛋白C端带有Myc和Flag标签. 将处于对数生长期的KYSE 510细胞接种于6孔板, 细胞密度为80%汇合度. 分别用150 µL Opti-MEM培养基稀释1 µg质粒DNA和4 µL LipofectamineTM 2000试剂, 将二者混合后室温放置45 min; 细胞用PBS洗2遍, 每孔加入Opti-MEM无血清培养基1 mL并逐滴加入转染试剂, 转染6 h后每孔加入1 mL含20%胎牛血清的RPMI 1640培养基, 24 h后加入800 µg/mL的G418, 筛选和克隆化, 获得稳定表达细胞株.
1.2.2 蛋白提取和Western blot分析: 细胞经预冷的1×PBS洗涤3次, 加入含有蛋白酶抑制剂的RIPA buffer(50 mmol/L Tris-HCl, pH 7.4; 150 mmol/L NaCl; 1 mmol/L EDTA; 1%NP-40; 0.1%SDS)裂解细胞并收集裂解液至1.5 mL离心管中; 冰浴超声并于12000 g, 4 ℃离心15 min后取上清. 考马斯亮蓝法测定蛋白质浓度, 10%SDS-PAGE电泳, 110 V恒压转膜, 10%脱脂牛奶室温封闭3 h, 加入稀释抗体于4 ℃摇床上孵育过夜, 经TBST洗膜后加入二抗室温孵育1 h, 加入ECL化学发光试剂显色并在LAS4000图像分析仪中曝光和采集图像.
1.2.3 细胞爬片制备和免疫荧光分析: 将无菌盖玻片置于6孔细胞培养板中, 表面经0.01%多聚赖氨酸处理30 min; 将消化后的细胞悬液滴于爬片上, 37 ℃孵箱孵育1 h后每孔加入完全培养液1 mL继续培养至30%-50%细胞汇合. 细胞爬片经4 ℃预冷的1×PBS洗涤3次, 4%多聚甲醛室温固定30 min; 以1×PBS洗涤后加入0.1% Triton X-100处理10 min, 1×PBS洗涤3次; 2% BSA室温封闭30 min; 加入1:50稀释Vimentin或E-cadherin抗体湿盒内4 ℃孵育过夜; 1×PBS漂洗后, 加入1:400稀释的Alexa 488标记二抗, 室温孵育1 h; DAPI染色, 50%甘油-PBS封片, 荧光显微镜下观察.
1.2.4 细胞Transwell侵袭实验: 密度为1×106/L细胞悬液100 µL接种于Fibronectin包被的Corning 24孔板Transwell小室, 下室加入600 µL完全培养基后于37 ℃孵箱孵育36 h, 取出上室, 细胞经20%甲醇固定15 min后用结晶紫染色. 倒置显微镜下计数穿膜到下层的细胞, 每张膜分别计数4个随机视野并取平均值.
1.2.5 细胞划痕实验和划痕创伤愈合修复分析: 处于对数生长期的食管癌细胞经2.5 g/L的胰酶消化后, 与含100 mL/L胎牛血清的RPMI 1640培养基混合, 制成单细胞悬液, 按每孔约3×105细胞密度均匀接种于预先放置有0.01%多聚赖氨酸包被的无菌盖玻片的6孔细胞培养板中, 37 ℃培养至细胞融合率达到60%-70%, 用无菌200 μL枪头沿细胞层纵轴作两道平行的划痕, 造成单层融合细胞划痕创伤模型. 沿划痕边缘等间距作4个标记作为观察点, 倒置显微镜下拍照, 此时划痕宽度作为划痕初始宽度(时间记为0 h). 观察细胞迁移能力的影响: 根据不同处理因素随机分为对照组和实验组, 根据不同处理时间, 每组又进一步分为48 h和72 h组, 各组均设3个平行对照孔. 37 ℃孵育相应时间后, 在上述标记的4个相同观察点拍照, 然后1×PBS轻轻洗3次, 更换2 mL含100 mL/L胎牛血清的DMEM培养液继续培养. 每隔4 h拍照1次, 直至划痕愈合或基本愈合, 结束观察. 各处理组划痕的愈合速度即反映各组细胞的迁移能力[15,16].
统计学处理 计量资料以mean±SD表示, 用t检验比较PURα转染前后KYSE 510细胞的侵袭和迁移能力的差异, P<0.05为差异具有统计学意义.
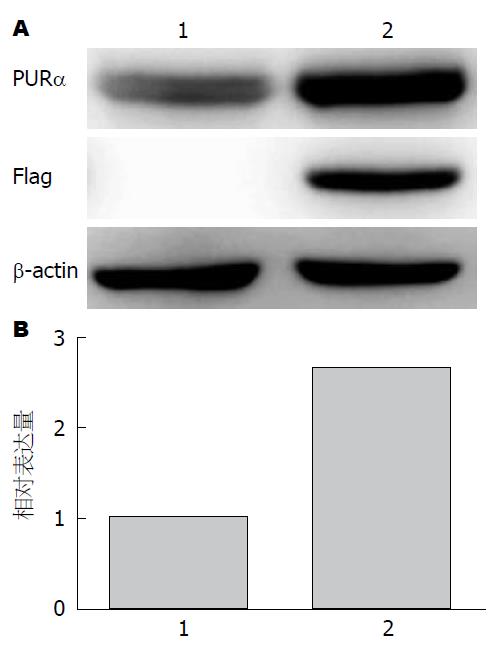
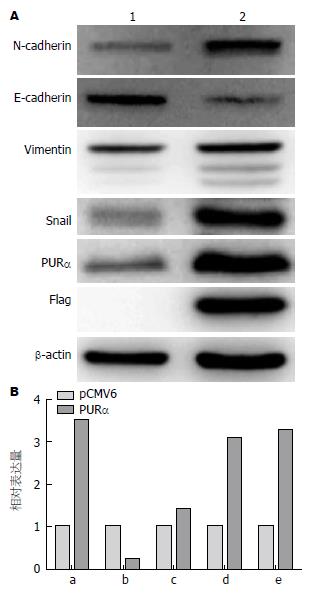
为研究PURα过表达对细胞的影响, 首先构建PURα过表达食管癌KYSE 510细胞系. 采用外源表达PURα的pCMV6-PURα载体介导转染食管鳞癌细胞KYSE 510, 经800 µg/mL G418筛选和克隆化, 获得稳定表达PURα的单克隆食管癌细胞KYSE 510-PURα和多克隆空载体做对照细胞KYSE 510-pCMV6. 分别扩大培养后, 提取细胞总蛋白进行Western blot分析, 鉴定PURα的表达水平. 结果显示, 在KYSE 510-PURα中鉴定到外源表达PURα的标签蛋白Flag, 且分子量与PURα大小相同. 鉴定到PURα蛋白的过表达, 说明稳定过表达PURα的食管癌细胞系构建成功(图1).
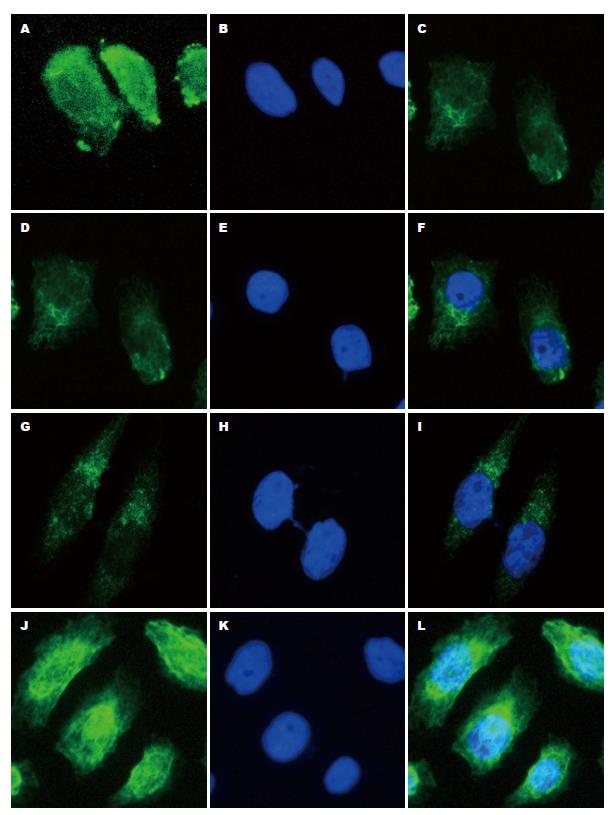
为检测过表达PURα食管癌KYSE 510细胞的上皮间叶转化, 采用免疫荧光法观察食管癌细胞上皮间叶转化重要标志分子E-cadherin和Vimentin的表达、定位和细胞形态改变. 结果发现与转染空载体对照细胞KYSE 510-pCMV6相比, 过表达PURα食管癌细胞KYSE 510-PURα膜蛋白E-cadherin的表达明显降低, 而骨架蛋白Vimentin的表达明显升高. 细胞形态由上皮样趋于纤维样改变(图2).
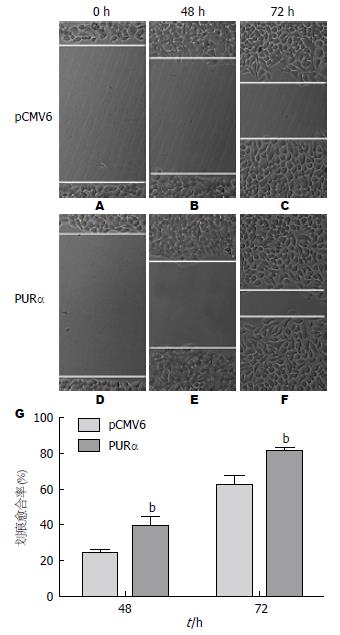
采用细胞划痕试验检测食管癌细胞迁移能力, 分别在培养的KYSE 510-PURα和对照KYSE 510-pCMV6食管癌细胞爬片上划痕, 并于划痕后继续培养48 h和72 h测量划痕区域愈合度. 与转染空载体对照细胞KYSE 510-pCMV6相比, PURα过表达的KYSE 510食管癌细胞划痕区域细胞移行速度和划痕愈合率明显加快, 具有显著差异(P<0.001). 结果提示PURα过表达增强了食管癌KYSE 510细胞的迁移能力(图3).
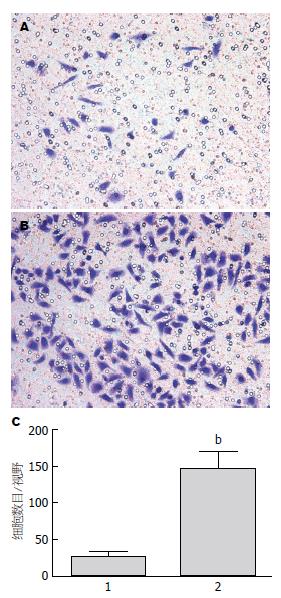
采用底膜带孔并涂胶的培养小室接种癌细胞, 观察迁移到底膜另一面的癌细胞数目, 以此来观察癌细胞的侵袭力的变化. 细胞经Transwell小室培养模型培养后, 每个小室随机抽取4个视野, 计算每个视野的细胞总数. 结果发现, 经PURα基因转染后KYSE 510细胞穿过微孔膜的细胞数明显高于空载质粒转染组, 提示在PURα过表达后KYSE 510细胞的体外迁移能力有显著增强, 具有显著差异(P<0.001)(图4).
用Western blot比较过表达PURα食管癌细胞和转染空载体对照细胞上皮间叶改变相关蛋白标志分子的表达变化, 结果显示KYSE 510-PURα细胞E-cadherin表达明显降低, 而N-cadherin、Vimentin和Snail表达明显增加, 提示PURα过表达促进了食管癌KYSE 510细胞上皮间叶转化(图5).
PUR蛋白家族包含PURα、PURβ和PURγ三个亚型, 在进化过程中高度保守. 其中PURα基因定位于人类染色体5q31[17], 编码322个氨基酸, 表达一个亲单双链DNA结合蛋白, 功能多样[18,19]. 其结构由一个DNA结合结构域, 富含甘氨酸的氨基端结构域和富含谷氨酰胺与谷氨酸盐的羧基端结构域组成. 研究[20-22]发现, PURα蛋白与c-MYC基因上游的DNA复制起始区相结合, 该区域是一段约1.6 kb富含嘌呤的序列. 在肿瘤发生机制的相关研究中发现PURα蛋白能与Pol Ⅱ、E2F1等蛋白相互作用[23], 从而影响肿瘤细胞的发生发展. 另有研究[24]发现, PURα蛋白参与某些化疗药物诱导的DNA损伤修复过程. PURα蛋白在细胞生长过程中扮演着多种角色, 通过与DNA和RNA相互作用, 并招募调节蛋白到特定的核酸序列[25], 从而参与DNA复制与基因转录; 在胞质中参与RNA转运以及细胞周期调节和致癌性转化等多种生理功能[26,27].
在食管鳞癌中E-cadherin表达水平降低与低分化肿瘤及其侵袭和转移表型密切相关[28]. E-cadherin是跨膜糖蛋白, 以钙离子依赖的模式介导特异的细胞间黏附, 其表达水平降低与肿瘤进展和转移相关[29]. Vimentin是细胞骨架的重要成分之一, 主要在间叶组织来源的细胞及某些未分化细胞中表达, 在多种发生上皮间叶转变的肿瘤细胞中, E-cadherin表达下降或缺失, 而Vimentin表达异常增高. 本研究通过在食管鳞癌KYSE 510细胞中过表达PURα, 促进肿瘤细胞中E-cadherin表达降低和Vimentin的表达增高. PURα过表达后, KYSE 510细胞形态趋于长梭形, 具有明显的上皮间叶转变特征. 通过细胞划痕实验发现, 与对照细胞相比, 48 h和72 h后转染PURα的食管癌KYSE 510细胞迁移能力明显增强. Transwell实验也表明, 在铺有基质胶的Transwell小室中培养36 h后, 过表达PURα的食管癌KYSE 510细胞的侵袭转移能力显著增加(P<0.001). 我们进一步发现, 过表达PURα蛋白的食管癌细胞, 上皮间叶转变相关蛋白标志E-cadherin、N-cadherin、Vimentin和Snail等蛋白表达变化, 除E-cadherin表达降低外, 其他几个标志蛋白均表达升高, 说明该细胞的侵袭能力增加. PURα可能是作为转录因子, 通过结合到Snail基因转录调控区, 上调Snail的表达, 而Snail是参与上皮间叶转变调节的重要转录因子, 可以通过直接抑制E-cadherin的转录和间接促进间叶表型分子如Vimentin、N-cadherin的转录促使细胞发生上皮间叶转变[30-33], 进而促进细胞的侵袭和转移.
本研究发现转录因子PURα通过诱导食管癌细胞上皮间叶转变而参与食管癌细胞侵袭转移过程, 对认识食管癌转移复发分子机制及其治疗措施积累了科学数据, 具有重要意义. 以往研究, 尽管已经发现PURα可以作为DNA/RNA结合蛋白和转录因子, 调节肿瘤细胞的发生发展以及对化疗药物的耐药, 但未能阐明PURα与肿瘤细胞侵袭转移表型间的关系. 本研究发现PURα这一单双链DNA/RNA结合蛋白同样参与对细胞侵袭转移的调节. 通过影响对该目标基因的转录和翻译, 有可能阻止上皮细胞的间叶转变, 使肿瘤细胞恢复上皮细胞特性, 就有可能逆转上皮间叶转变, 阻碍肿瘤细胞的侵袭转移能力, 从而为降低食管癌的复发转移措施提供新思路. 另外, 通过检测食管癌组织中PURα蛋白的表达水平, 可以对食管癌患者可能的转移进行预测, 而对临床治疗有一定的辅助意义.
食管癌是我国常见恶性肿瘤之一, 具有进展较快, 易发生淋巴结转移, 预后较差的特点, 而他的侵袭和转移是与食管癌预后差密切相关的重要生物学特性. 细胞上皮间叶改变对于肿瘤细胞的侵袭有重要的影响.
李苏宜, 教授, 主任医师, 安徽省肿瘤医院肿瘤内科
在肿瘤的侵袭转移过程中细胞上皮间叶转化发具有重要作用, 这一过程受多种转录因子的调节. 富含嘌呤元件结合蛋白α(purine-rich element binding protein alpha, PURα)在细胞生长过程中扮演着多种角色, 研究PURα作为转录因子对阐明食管癌细胞侵袭转移机制有重要意义.
我们前期工作发现, PURα作为转录因子可通过与E2F1和PolⅡ相互作用而影响Caspases8不同异构体的表达. 同样有研究发现PURα与多种肿瘤的形成和发育密切相关, 还在胞质中参与RNA转运以及细胞致癌性转化等多种生理功能.
本研究发现了转录因子PURα对细胞上皮间叶转变的影响, 转录因子PURα抑制E-cadherin表达, 间接促进Vimentin和N-cadherin等间叶表型分子的表达, 使细胞发生上皮间叶转变, 进而促进肿瘤细胞的侵袭和转移.
本研究发现PURα过表达显著增强食管鳞癌细胞发生细胞上皮间叶改变(epithelial-mesenchymal transition, EMT), 使食管鳞癌细胞运动能力和侵袭转移能力增强, 为认识食管癌侵袭转移机制积累了新的数据.
上皮间叶转化: 在上皮细胞与周围间质的相互作用过程中由于受到某种外界信号的刺激和内源转录因子的影响, 获得间叶样表型, 由多角形叠瓦状排列转变为梭形不规则排列, 同型黏附能力减弱, 异型黏附能力和运动能力增强等.
本文就食管鳞癌的转移机制展开研究. 创新性较为突出. 发现转录因子PURα通过诱导食管癌细胞EMT而参与食管癌细胞侵袭转移过程, 对认识食管癌转移复发分子机制具有重要意义. 实验设计严谨, 结论可信程度高.
编辑: 郭鹏 电编:闫晋利
| 1. | Jemal A, Bray F, Center MM, Ferlay J, Ward E, Forman D. Global cancer statistics. CA Cancer J Clin. 2011;61:69-90. [PubMed] [DOI] |
| 2. | Lambert R, Hainaut P. Esophageal cancer: cases and causes (part I). Endoscopy. 2007;39:550-555. [PubMed] |
| 3. | Weir HK, Thun MJ, Hankey BF, Ries LA, Howe HL, Wingo PA, Jemal A, Ward E, Anderson RN, Edwards BK. Annual report to the nation on the status of cancer, 1975-2000, featuring the uses of surveillance data for cancer prevention and control. J Natl Cancer Inst. 2003;95:1276-1299. [PubMed] |
| 5. | Wang LD, Yang HH, Fan ZM, Lü XD, Wang JK, Liu XL, Sun Z, Jiang YN, He X, Zhou Q. Cytological screening and 15 years' follow-up (1986-2001) for early esophageal squamous cell carcinoma and precancerous lesions in a high-risk population in Anyang County, Henan Province, Northern China. Cancer Detect Prev. 2005;29:317-322. [PubMed] |
| 6. | Shook D, Keller R. Mechanisms, mechanics and function of epithelial-mesenchymal transitions in early development. Mech Dev. 2003;120:1351-1383. [PubMed] |
| 7. | Peinado H, Olmeda D, Cano A. Snail, Zeb and bHLH factors in tumour progression: an alliance against the epithelial phenotype? Nat Rev Cancer. 2007;7:415-428. [PubMed] |
| 8. | Zhang S, Schafer-Hales K, Khuri FR, Zhou W, Vertino PM, Marcus AI. The tumor suppressor LKB1 regulates lung cancer cell polarity by mediating cdc42 recruitment and activity. Cancer Res. 2008;68:740-748. [PubMed] [DOI] |
| 9. | Zheng H, Shen M, Zha YL, Li W, Wei Y, Blanco MA, Ren G, Zhou T, Storz P, Wang HY. PKD1 phosphorylation-dependent degradation of SNAIL by SCF-FBXO11 regulates epithelial-mesenchymal transition and metastasis. Cancer Cell. 2014;26:358-373. [PubMed] [DOI] |
| 10. | Wyckoff JB, Wang Y, Lin EY, Li JF, Goswami S, Stanley ER, Segall JE, Pollard JW, Condeelis J. Direct visualization of macrophage-assisted tumor cell intravasation in mammary tumors. Cancer Res. 2007;67:2649-2656. [PubMed] |
| 11. | Johnson EM. The Pur protein family: clues to function from recent studies on cancer and AIDS. Anticancer Res. 2003;23:2093-2100. [PubMed] |
| 12. | Johnson EM, Daniel DC, Gordon J. The pur protein family: genetic and structural features in development and disease. J Cell Physiol. 2013;228:930-937. [PubMed] [DOI] |
| 13. | Darbinian N, Gallia GL, Khalili K. Helix-destabilizing properties of the human single-stranded DNA- and RNA-binding protein Puralpha. J Cell Biochem. 2001;80:589-595. [PubMed] |
| 14. | Lin Z, Guo Z, Xu Y, Zhao X. Identification of a secondary promoter of CASP8 and its related transcription factor PURα. Int J Oncol. 2014;45:57-66. [PubMed] [DOI] |
| 15. | Zhang S, Yang X, Shi H, Li M, Xue Q, Ren H, Yao L, Chen X, Zhang J, Wang H. Overexpression of leucine aminopeptidase 3 contributes to malignant development of human esophageal squamous cell carcinoma. J Mol Histol. 2014;45:283-292. [PubMed] [DOI] |
| 16. | Han P, Fu Y, Luo M, He J, Liu J, Liao J, Tian D, Yan W. BVES inhibition triggers epithelial-mesenchymal transition in human hepatocellular carcinoma. Dig Dis Sci. 2014;59:992-1000. [PubMed] [DOI] |
| 17. | 熊 鸣, 曲 佳, 周 兰萍, 张 达矜, 乔 媛媛, 胡 明, 赵 晓航. 海水浸泡降低血管内皮细胞迁移能力并抑制血管生成. 世界华人消化杂志. 2009;17:2152-2158. [DOI] |
| 18. | Gallia GL, Johnson EM, Khalili K. Puralpha: a multifunctional single-stranded DNA- and RNA-binding protein. Nucleic Acids Res. 2000;28:3197-3205. [PubMed] |
| 19. | Ma ZW, Pejovic T, Najfeld V, Ward DC, Johnson EM. Localization of PURA, the gene encoding the sequence-specific single-stranded-DNA-binding protein Pur alpha, to chromosome band 5q31. Cytogenet Cell Genet. 1995;71:64-67. [PubMed] |
| 20. | Ma ZW, Bergemann AD, Johnson EM. Conservation in human and mouse Pur alpha of a motif common to several proteins involved in initiation of DNA replication. Gene. 1994;149:311-314. [PubMed] |
| 21. | Muralidharan V, Cort L, Meier E, Blankenhorn EP, Khalili K. Molecular characterization and chromosomal localization of mouse Puralpha gene. J Cell Biochem. 2000;77:1-5. [PubMed] |
| 22. | Bergemann AD, Ma ZW, Johnson EM. Sequence of cDNA comprising the human pur gene and sequence-specific single-stranded-DNA-binding properties of the encoded protein. Mol Cell Biol. 1992;12:5673-5682. [PubMed] |
| 23. | Bergemann AD, Johnson EM. The HeLa Pur factor binds single-stranded DNA at a specific element conserved in gene flanking regions and origins of DNA replication. Mol Cell Biol. 1992;12:1257-1265. [PubMed] |
| 24. | Kaminski R, Darbinyan A, Merabova N, Deshmane SL, White MK, Khalili K. Protective role of Puralpha to cisplatin. Cancer Biol Ther. 2008;7:1926-1935. [PubMed] |
| 25. | Wang H, Wang M, Reiss K, Darbinian-Sarkissian N, Johnson EM, Iliakis G, Amini S, Khalili K, Rappaport J. Evidence for the involvement of Puralpha in response to DNA replication stress. Cancer Biol Ther. 2007;6:596-602. [PubMed] |
| 26. | van Roy F, Berx G. The cell-cell adhesion molecule E-cadherin. Cell Mol Life Sci. 2008;65:3756-3788. [PubMed] [DOI] |
| 27. | Zhang T, Zhang H, Wang Y, McGown LB. Capture and identification of proteins that bind to a GGA-rich sequence from the ERBB2 gene promoter region. Anal Bioanal Chem. 2012;404:1867-1876. [PubMed] [DOI] |
| 28. | Darbinian N, White MK, Gallia GL, Amini S, Rappaport J, Khalili K. Interaction between the pura and E2F-1 transcription factors. Anticancer Res. 2004;24:2585-2594. [PubMed] |
| 29. | König K, Meder L, Kröger C, Diehl L, Florin A, Rommerscheidt-Fuss U, Kahl P, Wardelmann E, Magin TM, Buettner R. Loss of the keratin cytoskeleton is not sufficient to induce epithelial mesenchymal transition in a novel KRAS driven sporadic lung cancer mouse model. PLoS One. 2013;8:e57996. [PubMed] [DOI] |
| 30. | White MK, Johnson EM, Khalili K. Multiple roles for Puralpha in cellular and viral regulation. Cell Cycle. 2009;8:1-7. [PubMed] |
| 31. | Thiery JP, Sleeman JP. Complex networks orchestrate epithelial-mesenchymal transitions. Nat Rev Mol Cell Biol. 2006;7:131-142. [PubMed] |