修回日期: 2014-07-03
接受日期: 2014-09-21
在线出版日期: 2014-11-18
DNA甲基化是影响表观遗传的重要机制之一, 其在基因表观遗传调控中起重要作用. 研究表明, DNA甲基化在食管癌的发生发展中起重要作用, 食管癌患者血浆和癌组织中基因甲基化水平具有良好一致性, 血浆是研究甲基化的可靠资源. 血浆DNA启动子异常甲基化的检测对食管癌的早期诊断、病情和治疗效果的监测、预后评估等具有良好的应用价值.
核心提示: 研究表明, DNA甲基化与食管癌的发生、发展密切相关, 食管癌患者的血浆游离DNA与组织中基因的甲基化状态具有高度一致性, 是研究甲基化的可靠资源. 本文综述了血浆游离DNA的甲基化在食管癌中的研究进展.
引文著录: 李莹, 黄晓俊. 血浆游离DNA的甲基化与食管癌的关系. 世界华人消化杂志 2014; 22(32): 4915-4920
Revised: July 3, 2014
Accepted: September 21, 2014
Published online: November 18, 2014
DNA methylation is one of the important epigenetic mechanisms, playing an important role in epigenetic gene regulation. Studies have shown that DNA methylation has an important role in the occurrence and development of esophageal cancer. The levels of gene methylation in plasma and tissues in patients with esophageal cancer have a good consistency, so plasma is a reliable resource for the study of methylation. The detection of DNA hypermethylation in plasma has good value for early diagnosis and monitoring of therapeutic effect and prognosis of esophageal cancer.
- Citation: Li Y, Huang XJ. DNA methylation in plasma and esophageal cancer. Shijie Huaren Xiaohua Zazhi 2014; 22(32): 4915-4920
- URL: https://www.wjgnet.com/1009-3079/full/v22/i32/4915.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i32.4915
食管癌是常见的消化系恶性肿瘤之一, 其发生是一个涉及多因素、多阶段、多基因变异积累及相互作用的复杂过程, 这个过程可能发生在基因组DNA水平上, 也可能在mRNA或蛋白质水平上. 表观遗传学指DNA序列不发生变化但基因表达却发生了可遗传的改变, 包括DNA甲基化、组蛋白修饰、染色质重塑、非编码RNA调控等形式. DNA甲基化是影响表观遗传的重要机制之一, 与细胞增殖、凋亡、分化有关的基因表达大多受甲基化的影响. 研究显示食管癌患者的血浆游离DNA与组织中基因的甲基化状态具有高度一致性[1], 血浆游离DNA的甲基化检测有助于食管癌的早期诊断、治疗效果监测及预后评估[2-4].
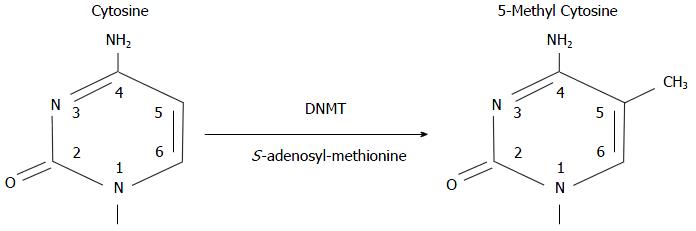
DNA甲基化是指生物体在DNA甲基转移酶(DNA methyltransferase, DNMT)的催化下, 以S-腺苷甲硫氨酸(S-adenosyl-methionine, SAM)为甲基供体, 将甲基转移到特定的碱基上的过程. 在哺乳动物中, DNA的甲基化主要发生在5'-CpG-3'序列的胞嘧啶的C-5位上, 生成5-甲基胞嘧啶(5mC)(图1).
DNA甲基化模式的改变包括整体基因组的低甲基化和启动子的高甲基化, 目前备受关注的是启动子区域CpG岛的高甲基化导致的抑癌基因转录沉默. 多数CpG岛位于基因启动子区域, 也可延伸到基因外显子区域, 这些区域的CpG岛正常情况下处于非甲基化状态, 这对维持机体的功能是必需的, 如基因印记、X染色体失活、细胞分化、胚胎发育等[5]; 而当启动子区域CpG岛发生异常甲基化时, 则可引发人类各种疾病甚至肿瘤的发生, 异常CpG的重新甲基化通常被认为是人类癌症发生的一个早期特征[6].
人类DNA的甲基化有4个特点: (1)基因组内甲基化多数发生在与鸟嘌呤相连的胞嘧啶上, 形成mCpG, 基因组中有3%的胞嘧啶是甲基化的, 而CpG中70%的胞嘧啶是甲基化的; (2)G+C丰富区域的CpG岛是非甲基化的; (3)启动子区的CpG被甲基化时转录受到抑制, 基因下游即非岛区的CpG甲基化不抑制基因的转录; (4)启动子区CpG甲基化的密度与转录的抑制程度有关, 弱的启动子能被密度较低的甲基化完全抑制, 当启动子被增强子增强时, 恢复转录功能; 如果甲基化的密度进一步增加, 转录也进一步被完全抑制[7].
目前, 可以分辨出甲基化修饰的方法有以下几种[8]: (1)甲基化敏感性内切酶; (2)亚硫酸氢盐的修饰; (3)特异性识别甲基化修饰DNA的抗体或蛋白; (4)质谱或色谱等精密检测手段. 由于仪器等条件的制约, 目前应用比较广泛的是前三种方法.
甲基化敏感的限制性内切酶方法是经典的甲基化分析方法, 优点是相对简单、成本低廉、甲基化位点明确、实验结果易解释. 缺点是容易忽略非CCGG序列中的CG, 方法较复杂, 需要的样本量大, 存在着酶不完全消化引起的假阳性问题, 不适用于混合样本.
NaHSO3法(Sodium bisulfite法)属于位点特异性DNA甲基化检测技术. 包括BSP测序法(BSP)、PCR(COBRA、MSP等)、芯片杂交技术(microarray)等. 其中, MSP是目前应用最广泛的CpG岛甲基化检测方法, 可检出比例为千分之一的甲基化片段, 敏感性、特异性高, 且可用于全血、血浆及石蜡包埋样本的检测, 但是只能用于定性研究.
芯片技术方法主要包括以下3种: 甲基化高密度芯片(CpG islands microarr- ay)、差异甲基化杂交和甲基化特异性寡核苷酸芯片(MSO microarray). 该法适于临床样本, 可用于多样本、多位点甲基化的检测, 样本需要量少, 是一种有效检测特定基因的不同甲基化位点的高通量、高灵敏度的甲基化检测方法.
研究表明[9-13], DNA甲基化是食管癌发生中的一种早期、普遍事件. 不同作用的基因在食管癌的不同病理类型甚至癌前病变中的甲基化频率不尽相同. Kaz等[14]在对与食管癌表观遗传生物标志物相关的研究文献进行总结时发现, 发生异常甲基化的各个基因在正常食管与食管癌、食管腺癌(esophageal adenocarcinoma, EAC)与食管鳞癌(esophageal squamous cell carcinoma, ESCC)以及他们各自的癌前病变组织中的甲基化频率不尽相同, 如p16基因在EAC及其癌前病变中的甲基化频率为: Barrett食管3%-77%, 不确定的异常增生60%, 低度异型增生20%-56%, 高度异型增生60%-75%, EAC 39%-85%; 在ESCC及其癌前病变中的甲基化频率为: 低度异型增生31%, 中度异型增生42%, 高度异型增生33%, ESCC 40%-62%. 相关研究亦证明, DNA的异常甲基化与食管癌患者对放-化疗治疗的敏感性[15,16]、治疗后生存期的长短[17,18]、肿瘤复发及预后[19-22]等密切相关.
游离DNA是存在于血液中游离于细胞外的DNA, 血清和血浆中均含有, 最初由Mandel等[23]于1948年报道. 游离DNA在健康人血清中的含量为0-100 ng/mL, 平均为30 ng/mL, 而在肿瘤患者血清中, 血清游离DNA的含量在个体之间的差异比较大, 为0-1000 ng/mL, 平均约为180 ng/mL. 在肿瘤研究中, Gormally等[24]研究显示1个有质量为100 g肿瘤的患者, 每天约有3.3%以上的肿瘤DNA释放入循环中, 而对游离DNA的稳定性研究未见报道. 关于血清游离DNA来源的确切机制还不清楚, 目前认为, 主要存在以下两种机制: (1)细胞分离和外渗到血液中, 随后发生裂解释放出DNA; (2)细胞发生调亡/坏死后DNA释放入血循环中[25]. 也有研究指出, 当对循环游离DNA(circulating free DNA, cfDNA)的长度进行测量时, 经典的梯度模式经常被假定在180个碱基对的整数倍上[26], 这是凋亡的过程所特有的[25,27,28]; 而事实上, 大部分cfDNA的片段在180-200个碱基对之间, 提示大多数cfDNA很可能是由细胞凋亡产生的[29-32]. 由细胞凋亡或坏死被动释放于血液中的cfDNA依赖于肿瘤的位置、大小及血管供应, 这可能是cfDNA水平变异性的原因[33], 而从反方面来看, 对cfDNA的实时监测将有助于动态的了解和监测肿瘤的发展变化.
目前, 食管癌的诊断仍然依赖于内镜检查及组织活检, 辅助以影像学检查技术. 作为食管癌临床诊断的金标准, 组织活检在采集和实用性方面仍存在着许多障碍[33]: (1)不利于患者的行程安排; (2)属于有创检查, 给患者带来了一定的痛苦, 增加了患者临床护理等方面的费用; (3)也是最重要的, 活检过程中存在着一定的风险及并发症. 美国德克萨斯大学MD安德森癌症中心的一项关于活检的调查研究显示[34], 胸部活检和腹部/盆腔取样的不良事件比率分别为17.1%和1.6%. 因此, 发展能够捕捉到肿瘤的异质性和分子变化的微创技术显得十分必要. 血浆游离DNA在原则上可以提供相同的遗传信息, 而且获得的血液有着十分明显的优势[33]: 血液是新鲜DNA的来源, 不受防腐剂的影响; 血液通过细针抽取获得, 创伤性小, 避免了组织活检的危险; 血液在治疗过程中的任何时间都可以采集, 能够动态的检测肿瘤中的分子变化而不是依赖于某一个静态的时间点. Ghorbian等[35]认为循环游离DNA作为生物标志物的应用, 为多种癌症早期检测的非侵入性测试的构建, 恶性肿瘤预后和治疗之后的癌症患者的管理提供了一个最佳地机会. 相对于组织生物标志物, 循环生物标志物由于易于测试、成本低更适合于大规模人群的筛查策略[36]. 因此, 血浆游离DNA因其方便、经济、快捷及微创的特点有望在食管癌高发区人群筛查、早期诊断、治疗后监测及预后评估等方面发挥潜在的生物学指标作用.
p16基因又称多瘤抑制基因, 位于人类第9号染色体短臂2区1带(9p21), 全长8.5 kb. 他直接参与细胞周期调控, 在细胞周期G1/S限制点起关键负调控作用, 调节细胞的增殖. Wang等[1]应用实时甲基化特异性PCR(methylation-specific PCR, MSP)的方法对76例ESCC组织、癌旁正常组织及外周血中的p16基因甲基化情况进行检测, 发现p16基因在癌组织[86.8%(66/76)]及外周血液[71.1%(54/76)]中的甲基化情况具有良好的一致性; p16基因的异常甲基化与食管癌的病理分期、淋巴结转移及神经血管侵犯密切相关(P<0.05); DNA在外周血中的甲基化率先是增加, 然后在术前、术中、术后阶段逐渐降低. 因此, 血浆是研究甲基化的可靠资源; ESCC患者血浆p16基因的甲基化检测可以反映身体的荷瘤状态, 并有助于手术完整切除程度的判断.
FHIT基因定位于染色体3p14.2, 是近年来发现的一种新型候选抑癌基因. 该基因跨越了人类基因组中最常见的脆性部位, 故被命名为FHIT. 郭晓青等[2]对10例慢性食管炎、44例食管癌前病变(轻度不典型增生22例, 中度不典型增生13例, 重度不典型增生9例)、14例食管鳞状细胞原位癌及37例浸润性鳞癌患者血浆中p16及FHIT基因的甲基化情况进行检测, 结果14例食管病变患者血浆中出现p16基因甲基化(2例为原位癌, 12例为浸润性鳞癌), 16例患者血浆中出现FHIT基因甲基化(2例为原位癌, 14例为浸润性鳞癌); 在51例食管原位癌及浸润性鳞癌患者中, 24例血浆中出现p16和/或FHIT基因甲基化, 即两基因同时检测, 甲基化频率达47.06%. 因此, 血浆FHIT基因的甲基化检测可以将食管癌的筛查提前到原位癌阶段, 为食管癌的早期发现提供帮助; 而血浆游离DNA基因的联合检测能进一步提高甲基化的检出率, 有助于临床食管癌患者的筛查.
APC基因为家族性腺瘤样结肠息肉病的易感基因, 位于染色体5q21. APC蛋白与β连环蛋白(β-catenin)(一种转录因子)形成复合物, 导致β-catenin降解. 如果APC基因表达异常, 就会使游离β-catenin增多, 引起基因的不正常表达, 导致细胞黏附、生长、分化等方面的重要改变, 使细胞发生癌变. 程蕾等[37]对182例食管癌及癌旁组织中APC启动子区5'-CpG岛的甲基化情况进行了检测, 结果在182例食管癌组织中APC启动子区5'-CpG岛的甲基化率为54.40%(99/182), 相应癌旁组织中的甲基化率为9.90%(18/182), 差异有统计学意义(P<0.05). APC启动子区5'-CpG岛甲基化分别与淋巴结转移、肿瘤远处转移、临床分期及不良预后相关, 差异有统计学意义(P = 0.000). 单因素结果提示, APC启动子区5'-CpG岛甲基化与食管癌患者的生存期相关(P<0.05). Cox多因素分析提示, APC启动子区5'-CpG岛甲基化是独立的预后因素(P<0.05). Hoffmann等[20]对59例食管癌患者(其中腺癌35例, 鳞癌24例)术前、术后血浆中DAPK、APC启动子的甲基化情况进行检测, 发现术前血浆游离DAPK、APC启动子甲基化的联合检测有助于更好地估计食管癌患者尤其是腺癌患者术后的生存概率, 其中血浆APC甲基化可能作为术后检测肿瘤是否明显残留的标志物. 但由于该研究以EAC为主, 其结论尚需进一步研究来印证.
RASSF1A基因位于3p21.3, 是一种新的抑癌基因. 该基因调控的靶基因涉及Ras信号转导、细胞周期、基因转录、细胞凋亡等方面, 但具体作用机制尚不完全清楚. 秦豫培等[3]应用MSP方法对食管癌高发区30例食管癌患者的血浆、肿瘤组织及癌旁正常组织中RASSF1A基因甲基化情况进行检测, 结果RASSF1A基因在食管癌组织中的甲基化率为40%(12/30), 在12例癌组织甲基化阳性的患者中, 有7例血浆甲基化阳性, 癌组织与血浆RASSF1A甲基化阳性一致率为58%(7/12), 18例癌组织甲基化阴性的患者其血浆也均为阴性, 阴性一致率为100%(18/18). 癌旁正常食管组织甲基化率为13%(4/30), 明显低于癌组织(40%, 12/30)(P<0.05). 7例血浆甲基化阳性的患者中, 淋巴结转移阳性5例(55%, 5/9), 阴性2例(10%, 2/21), 差异有统计学意义(P<0.05). 低分化鳞癌的RASSF1A甲基化阳性率(83%, 5/6)明显高于中分化鳞癌(24%, 5/21)(P<0.05). 因此, 血浆RASSF1A基因甲基化可以反映同一个体ESCC组织中RASSF1A基因的甲基化状态, 可能是高危人群筛查的重要候选分子标志之一. RASSF1A基因可能是食管癌发生的重要抑癌基因, 而且RASSF1A基因甲基化与ESCC患者的预后相关.
溶质载体家族5成员8(solute carrier family 5 member 8, SLC5A8)基因位于人类染色体的12q13, 是一种新的候选抑癌基因. 该基因属Na+/葡萄糖共转运蛋白家族成员, 是由Na+偶联的转运短链脂肪酸(short-chain fatty acids, SCFAs)和其他单羧酸(如乳酸盐、丙酮酸盐)的载体[38]. 目前有关控制SLC5A8基因表达的转录机制还不是很清楚, 为了更好地了解调节SLC5A8基因表达的分子机制, Zhang等[39]通过质粒转染、PCR放大等方法对SLC5A8基因5'-调节区及外显子1的一部分进行了研究, 结果SLC5A8启动子区缺失突变体的荧光素酶报告基因分析证实, 一个295 bp的区域对于维持SLC5A8基因的基本启动子活性是必需的. 进一步的分析表明, CCAAT盒和GC盒参与了SLC5A8基因的正调控. CCAAT/增强子结合蛋白β(C/EBPβ)和特异转录因子1(specific transcription factor 1, SP1)的过表达对于人SLC5A8基因启动子活性和蛋白表达的上调作用, 提示C/EBPβ和SP1在SLC5A8的转录方面可能发挥作用. 雷霆等[4]采用MSP法对45例食管癌、癌旁、切缘组织及30例食管癌术前、术后血浆中SLC5A8基因的甲基化状态进行了检测, 结果45例食管癌、癌旁及切缘组织中SLC5A8基因的甲基化率分别为68.8%、11.1%和4.4%, 癌组织的甲基化阳性率显著高于癌旁及切缘组织(P<0.01). 30例术前血浆、术后血浆中SLC5A8基因的甲基化率分别为43.3%和16.7%, 差异有统计学意义(P<0.05). 食管癌组织中SLC5A8基因甲基化阴性的患者其术前、术后血浆中均未检测到该基因的甲基化. 因此, 血浆中SLC5A8基因的甲基化状态与肿瘤组织的甲基化状态密切相关, SLC5A8基因甲基化是食管癌发生的重要早期分子事件, 血浆中SLC5A8基因甲基化检测可作为术前诊断的重要参考指标, 并有可能成为监测治疗反应与判断预后的一项生物学指标.
DNA甲基化与食管癌的发生发展密切相关, 血浆游离DNA启动子的甲基化检测具有微创、经济、方便、快捷的特点, 有望成为食管癌筛查不可或缺的辅助检查手段. 但是目前与食管癌有关的基因甲基化的研究大部分仍以组织标本为主, 有关血浆DNA甲基化与食管癌发生发展关系的研究还很少. 由于检测手段及方法的限制, 食管癌血浆DNA的甲基化检测尚处于"有"或"无"的阶段, 对于基因启动子甲基化程度的判断缺乏一个明确的判定标准; 血浆DNA甲基化检测的灵敏度及特异性还不是很高. 这些都阻碍了血浆DNA的甲基化检测在食管癌中的应用和发展, 进一步的大样本临床实验研究仍然十分必要.
DNA甲基化是影响表观遗传的重要机制之一, 与细胞增殖、凋亡、分化有关的基因表达大多受甲基化的影响. 研究表明, 食管癌患者的血浆游离DNA与组织中基因的甲基化状态具有高度一致性, 血浆游离DNA的甲基化检测有助于食管癌的早期诊断、治疗效果监测及预后评估.
高英堂, 研究员, 天津市第三中心医院
DNA甲基化与食管癌的发生、发展密切相关, 有助于食管癌的早期诊断、治疗效果监测及预后评估. 近年来研究发现, 血浆DNA与组织中基因的甲基化状态具有高度一致性, 血浆DNA因为取材方便、微创等优点有望成为研究甲基化的可靠资源.
研究发现, 血浆DNA的甲基化不仅与食管癌的发生、发展密切相关, 还与肝癌、大肠癌、胰腺癌、肺癌、乳腺癌、宫颈癌、白血病、脑胶质瘤等肿瘤密切相关.
血浆游离DNA作为循环生物标志物具有易取材、微创等特点, 本文综述了血浆游离DNA的特点及其与组织生物标志物相比的优点, 并详细综述了其在食管癌的早期诊断、治疗效果监测及预后评估等方面的潜在应用价值.
本文综述了血浆DNA的甲基化在食管癌发生、发展中的作用, 可能在食管癌的早期诊断、治疗效果检测及预后评估等方面发挥重要作用.
本文综述了血浆游离DNA启动子异常甲基化对食管癌的早期诊断、疗效监测和预后评估等方面的诊断价值, 具有一定的指导意义.
编辑: 田滢 电编:闫晋利
| 1. | Wang CC, Mao WM, Ling ZQ. [DNA methylation status of RARβ2 and p16(INK4α) in peripheral blood and tumor tissue in patients with esophageal squamous cell carcinoma]. Zhonghua Zhong Liu Za Zhi. 2012;34:441-445. [PubMed] |
| 5. | Robertson KD. DNA methylation and human disease. Nat Rev Genet. 2005;6:597-610. [PubMed] [DOI] |
| 6. | Wilson AS, Power BE, Molloy PL. DNA hypomethylation and human diseases. Biochim Biophys Acta. 2007;1775:138-162. [PubMed] |
| 7. | Robertson KD, Jones PA. DNA methylation: past, present and future directions. Carcinogenesis. 2000;21:461-467. [PubMed] [DOI] |
| 9. | Wang F, Xie XJ, Piao YS, Liu B, Wang LD. [Methylation of p16 and hMLH1 genes in esophageal squamous cell carcinoma and reflux esophagitis]. Zhonghua Bing Li Xue Za Zhi. 2011;40:537-541. [PubMed] |
| 10. | Jin Z, Zhao Z, Cheng Y, Dong M, Zhang X, Wang L, Fan X, Feng X, Mori Y, Meltzer SJ. Endoglin promoter hypermethylation identifies a field defect in human primary esophageal cancer. Cancer. 2013;119:3604-3609. [PubMed] [DOI] |
| 11. | Guo M, Ren J, Brock MV, Herman JG, Carraway HE. Promoter methylation of HIN-1 in the progression to esophageal squamous cancer. Epigenetics. 2008;3:336-341. [PubMed] |
| 12. | Jia Y, Yang Y, Zhan Q, Brock MV, Zheng X, Yu Y, Herman JG, Guo M. Inhibition of SOX17 by microRNA 141 and methylation activates the WNT signaling pathway in esophageal cancer. J Mol Diagn. 2012;14:577-585. [PubMed] [DOI] |
| 13. | Jin Z, Olaru A, Yang J, Sato F, Cheng Y, Kan T, Mori Y, Mantzur C, Paun B, Hamilton JP. Hypermethylation of tachykinin-1 is a potential biomarker in human esophageal cancer. Clin Cancer Res. 2007;13:6293-6300. [PubMed] |
| 14. | Kaz AM, Grady WM. Epigenetic biomarkers in esophageal cancer. Cancer Lett. 2014;342:193-199. [PubMed] [DOI] |
| 15. | Hamilton JP, Sato F, Greenwald BD, Suntharalingam M, Krasna MJ, Edelman MJ, Doyle A, Berki AT, Abraham JM, Mori Y. Promoter methylation and response to chemotherapy and radiation in esophageal cancer. Clin Gastroenterol Hepatol. 2006;4:701-708. [PubMed] [DOI] |
| 16. | Hamilton JP, Sato F, Jin Z, Greenwald BD, Ito T, Mori Y, Paun BC, Kan T, Cheng Y, Wang S. Reprimo methylation is a potential biomarker of Barrett's-Associated esophageal neoplastic progression. Clin Cancer Res. 2006;12:6637-6642. [PubMed] [DOI] |
| 17. | Ling ZQ, Li P, Ge MH, Zhao X, Hu FJ, Fang XH, Dong ZM, Mao WM. Hypermethylation-modulated down-regulation of CDH1 expression contributes to the progression of esophageal cancer. Int J Mol Med. 2011;27:625-635. [PubMed] [DOI] |
| 18. | Zare M, Jazii FR, Alivand MR, Nasseri NK, Malekzadeh R, Yazdanbod M. Qualitative analysis of Adenomatous Polyposis Coli promoter: hypermethylation, engagement and effects on survival of patients with esophageal cancer in a high risk region of the world, a potential molecular marker. BMC Cancer. 2009;9:24. [PubMed] [DOI] |
| 19. | Liu JB, Qiang FL, Dong J, Cai J, Zhou SH, Shi MX, Chen KP, Hu ZB. Plasma DNA methylation of Wnt antagonists predicts recurrence of esophageal squamous cell carcinoma. World J Gastroenterol. 2011;17:4917-4921. [PubMed] [DOI] |
| 20. | Hoffmann AC, Vallböhmer D, Prenzel K, Metzger R, Heitmann M, Neiss S, Ling F, Hölscher AH, Schneider PM, Brabender J. Methylated DAPK and APC promoter DNA detection in peripheral blood is significantly associated with apparent residual tumor and outcome. J Cancer Res Clin Oncol. 2009;135:1231-1237. [PubMed] [DOI] |
| 22. | Hibino S, Kanda M, Oya H, Takami H, Shimizu D, Nomoto S, Hishida M, Niwa Y, Koike M, Yamada S. Reduced expression of DENND2D through promoter hypermethylation is an adverse prognostic factor in squamous cell carcinoma of the esophagus. Oncol Rep. 2014;31:693-700. [PubMed] [DOI] |
| 23. | Mandel P, Metais P. Les acides nucleiques du plasma sanguin chez 1'Homme. C R Seances Soc Biol Fil. 1948;142:241-243. [PubMed] |
| 24. | Gormally E, Caboux E, Vineis P, Hainaut P. Circulating free DNA in plasma or serum as biomarker of carcinogenesis: practical aspects and biological significance. Mutat Res. 2007;635:105-117. [PubMed] [DOI] |
| 25. | Jahr S, Hentze H, Englisch S, Hardt D, Fackelmayer FO, Hesch RD, Knippers R. DNA fragments in the blood plasma of cancer patients: quantitations and evidence for their origin from apoptotic and necrotic cells. Cancer Res. 2001;61:1659-1665. [PubMed] |
| 26. | Wyllie AH. Glucocorticoid-induced thymocyte apoptosis is associated with endogenous endonuclease activation. Nature. 1980;284:555-556. [PubMed] [DOI] |
| 27. | Mouliere F, Robert B, Arnau Peyrotte E, Del Rio M, Ychou M, Molina F, Gongora C, Thierry AR. High fragmentation characterizes tumour-derived circulating DNA. PLoS One. 2011;6:e23418. [PubMed] [DOI] |
| 28. | Chan KC, Zhang J, Hui AB, Wong N, Lau TK, Leung TN, Lo KW, Huang DW, Lo YM. Size distributions of maternal and fetal DNA in maternal plasma. Clin Chem. 2004;50:88-92. [PubMed] |
| 29. | Fan HC, Blumenfeld YJ, Chitkara U, Hudgins L, Quake SR. Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood. Proc Natl Acad Sci U S A. 2008;105:16266-16271. [PubMed] [DOI] |
| 30. | Diehl F, Schmidt K, Choti MA, Romans K, Goodman S, Li M, Thornton K, Agrawal N, Sokoll L, Szabo SA. Circulating mutant DNA to assess tumor dynamics. Nat Med. 2008;14:985-990. [PubMed] [DOI] |
| 31. | Diehl F, Li M, Dressman D, He Y, Shen D, Szabo S, Diaz LA, Goodman SN, David KA, Juhl H. Detection and quantification of mutations in the plasma of patients with colorectal tumors. Proc Natl Acad Sci U S A. 2005;102:16368-16373. [PubMed] |
| 32. | Liu Q, Sommer SS. Pyrophosphorolysis-activated polymerization (PAP): application to allele-specific amplification. Biotechniques. 2000;29:1072-106, 1078, 1080 passim. [PubMed] |
| 33. | Diaz LA, Bardelli A. Liquid biopsies: genotyping circulating tumor DNA. J Clin Oncol. 2014;32:579-586. [PubMed] [DOI] |
| 34. | Overman MJ, Modak J, Kopetz S, Murthy R, Yao JC, Hicks ME, Abbruzzese JL, Tam AL. Use of research biopsies in clinical trials: are risks and benefits adequately discussed? J Clin Oncol. 2013;31:17-22. [PubMed] [DOI] |
| 35. | Ghorbian S, Ardekani AM. Non-Invasive Detection of Esophageal Cancer using Genetic Changes in Circulating Cell-Free DNA. Avicenna J Med Biotechnol. 2012;4:3-13. [PubMed] |
| 36. | Shah AK, Saunders NA, Barbour AP, Hill MM. Early diagnostic biomarkers for esophageal adenocarcinoma--the current state of play. Cancer Epidemiol Biomarkers Prev. 2013;22:1185-1209. [PubMed] [DOI] |
| 38. | Miyauchi S, Gopal E, Fei YJ, Ganapathy V. Functional identification of SLC5A8, a tumor suppressor down-regulated in colon cancer, as a Na(+)-coupled transporter for short-chain fatty acids. J Biol Chem. 2004;279:13293-13296. [PubMed] [DOI] |
| 39. | Zhang Y, Bao YL, Wu Y, Yu CL, Sun Y, Li YX. Identification and characterization of the human SLC5A8 gene promoter. Cancer Genet Cytogenet. 2010;196:124-132. [PubMed] [DOI] |