修回日期: 2014-09-19
接受日期: 2014-10-15
在线出版日期: 2014-11-18
目的: 探讨PIK3CA在食管鳞癌细胞系Eca109中的作用.
方法: 用脂质体介导siRNA瞬时转染Ecal09细胞, 转染细胞分为3组: 空白对照组(正常培养的Eca109细胞)、阴性对照组(转染随机序列的siRNA)及实验组(转染PIK3CA-siRNA). 运用Western blot技术检测PIK3CA基因干扰后蛋白水平表达的变化; 四甲基偶氮唑盐微量酶反应比色法(MTT法)和细胞划痕实验检测PIK3CA干扰后Eca109细胞增殖和迁移能力的改变; 流式细胞仪检测PIK3CA干扰后细胞周期和凋亡率的变化.
结果: 干扰PIK3CA基因表达后, 其蛋白水平表达较空白对照组及阴性对照组显著性降低(P<0.05). MTT实验和划痕实验显示干扰PIK3CA的表达后, Eca109细胞增殖受到显著性抑制(P<0.05), 迁移能力显著性降低(P<0.05). 流式细胞仪检测下调PIK3CA的表达后细胞周期阻滞在细胞分裂的S期(P<0.05), 且使细胞凋亡率显著性增加(P<0.05).
结论: PIK3CA基因能够促进Eca109细胞的增殖和迁移能力, 并具有抗凋亡作用. PIK3CA基因有望成为食管鳞癌治疗的潜在靶点.
核心提示:PIK3CA基因能够促进Eca109细胞的增殖和迁移能力, 并具有抗凋亡作用. PIK3CA基因有望成为抑制食管鳞癌转移的一个潜在治疗靶点.
引文著录: 鲁芒, 郑树涛, 刘清, 刘涛, 高向朋, 伊力亚尔·夏合丁, 卢晓梅. PIK3CA在食管鳞癌细胞系Eca109中的功能. 世界华人消化杂志 2014; 22(32): 4893-4900
Revised: September 19, 2014
Accepted: October 15, 2014
Published online: November 18, 2014
AIM: To explore the role of PIK3CA in cell proliferation, migration and apoptosis in esophageal squamous cell carcinoma (ESCC) cell line Eca109.
METHODS: Eca109 cells were divided into three groups: a blank control group (non-transfected cells), a negative control group (transfected with a scramble siRNA) and an experimental group (transfected with a specific siRNA against PIK3CA). After transfection using Lipofectamin 2000, the protein expression of PIK3CA was detected by Western blot. Cell proliferation and migration were examined by MTT assay and wound healing assay, respectively. Cell apoptosis and cell cycle progression were analyzed by flow cytometry.
RESULTS: After transfection with the specific siRNA against PIK3CA, PIK3CA protein expression was significantly decreased compared with the blank control group and negative control group (P < 0.05). After successful knock-down of PIK3CA, both cell proliferation and migration were significantly reduced (P < 0.05), the cell cycle was arrested at S phase (P < 0.05), and the apoptosis rate was significantly increased (P < 0.05).
CONCLUSION: PIK3CA plays an important role in promoting cell proliferation and migration and suppressing apoptosis, which may become a new potential target in the therapy of metastasis of ESCC.
- Citation: Lu M, Zheng ST, Liu Q, Liu T, Gao XP, Ilyar∙Sheyhidin, Lu XM. Role of PIK3CA in cell proliferation, migration and apoptosis in human esophageal squamous cell carcinoma cell line Ecal09. Shijie Huaren Xiaohua Zazhi 2014; 22(32): 4893-4900
- URL: https://www.wjgnet.com/1009-3079/full/v22/i32/4893.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v22.i32.4893
食管癌(esophageal cancer, EC)是世界上常见的恶性消化系肿瘤, 对人类的健康危害极大, 5年生存率较低[1]. 在我国其主要的病理类型是鳞状细胞癌. 食管癌患者在就诊时多数已处于中晚期, 多数已发生了转移, 淋巴结转移是食管癌重要的转移途径之一, 也是食管癌复发、死亡最常见的原因之一[2]. 研究发现食管癌的转移是多因素相互作用、相互影响的一个复杂的过程, 但食管癌转移的分子机制尚未明了[3]. 因此, 阐明食管癌转移的分子机制, 为指导临床提高食管癌的治疗效果具有重要的意义.
本课题组在前期工作中, 采用比较基因组杂交(comparative genomic hybridization, CGH)技术发现PIK3CA基因在发生转移的食管癌患者的染色体中发生显著性扩增. PIK3CA基因编码Ⅰ型磷脂酰肌醇-3激酶(phosphatidylinositol 3-kinase, PI3K)的p110α催化亚基, 是由Volinia等[4]采用原位杂交技术检测出来的, 定位于3q26.3, 长34 kb, 包含20个外显子, 编码1068种氨基酸, 该组氨基酸产生一组长124 kDa的蛋白. 目前已发现PIK3CA在多种肿瘤中高度突变[5], 且PIK3CA基因的不同位点的突变在肿瘤的发生发展中起到了重要的作用[6], 但PIK3CA扩增在食管鳞癌中功能研究鲜有报道. 因此, 本研究采用脂质体介导的RNA干扰(RNA interference, RNAi)技术, 沉默PIK3CA的表达, 探讨PIK3CA在食管鳞癌细胞中对增殖、迁移、细胞周期及凋亡等的影响, 继而探讨PIK3CA基因能否作为一个抑制食管鳞癌转移的有效的治疗靶点.
食管鳞癌细胞系Ecal09购自武汉大学细胞保藏中心; 酶标仪(美国Bio-Rad公司); 流式细胞仪(BD FACSC alibur System); Quantity One软件(美国Bio-Rad公司); RPMI 1640培养基、胰蛋白酶、胎牛血清(FBS, 美国Gibco公司); DMSO、Opti-MEM、Lipofectamine 2000(美国Invitrogen公司); RIPA裂解液(美国Thermo公司); BCA试剂盒(美国Thermo公司); PIK3CA抗体、GAPDH抗体(美国Santa Cruz公司); Western blot二抗试剂盒(美国Invitrogen公司); 四甲基偶氮唑盐(MTT, 美国Invitrogen公司); TritonX-100(北京鼎国昌盛生物技术有限责任公司); RNA酶(10 mg/mL, 北京天根生化科技有限公司); 碘化丙啶(PI, Sigma公司); 细胞凋亡试剂盒(美国Invitrogen公司). PIK3CA siRNA及随机序列siRNA由上海吉玛制药技术有限公司合成. PIK3CA siRNA序列为: 5'-GGUGGUGCGAAAUUCUCACACUAUU-3'; 随机序列siRNA序列为: 5'-UAUAUGUACUGCGCGUGGAGA-3'.
1.2.1 细胞培养和转染: 食管鳞癌细胞系Eca109, 用含10%胎牛血清的RPMI 1640培养液在37 ℃、50 mL/L CO2恒温培养箱中培养. 转染细胞分为3组: 空白对照组(正常生长的Eca109细胞, 不做任何处理)、阴性对照组(转染随机序列的siRNA)及实验组(转染PIK3CA-siRNA), 用LipofectamineTM 2000试剂进行转染. 将细胞按约3×105/孔的密度接种于6孔板中, 次日待细胞达到70%-80%时, 按说明书转染, 4-6 h后换含10%胎牛血清的RPMI 1640培养液在37 ℃、50 mL/L CO2恒温培养箱中继续培养. 每组设3个复孔. 细胞总蛋白在转染后72 h提取, 用于Western blot实验.
1.2.2 Western blot实验: 用RIPA裂解液裂解提取细胞总蛋白, 采用BCA试剂盒测定蛋白浓度后, 以每孔80 µg上样, 经体积分数为10%SDS-PAGE进行分离后, 将蛋白转移至聚偏氟乙烯(PVDF)膜上, 将膜置于封闭液中封闭1 h, 加入1:200稀释的PIK3CA抗体, 1:800稀释的GAPDH抗体, 4 ℃孵育过夜, 之后用洗膜液洗3次每次5 min, 加入二抗室温孵育1.5 h, 洗膜液洗3次每次5 min, 显影液显色直到紫色条带显出后用蒸馏水终止显色, 观察结果. 以GAPDH为内参照, 用Quantity One软件计算灰度值, 计算各组PIK3CA蛋白的相对表达量.
1.2.3 四甲基偶氮唑盐微量酶反应比色法(MTT法): 细胞培养及RNA干扰方法同1.2.1, 按3×103/孔接种细胞于96孔板, 每组设4个复孔. 分别于转染前及转染后24、48、72、96 h后每孔加20 µL MTT(5 g/L), 37 ℃、50 mL/L CO2恒温培养箱孵育4 h, 弃去MTT, 每孔加二甲基亚砜(DMSO)150 µL, 用酶标仪检测各组细胞490 nm波长处的吸光度(A)值.
1.2.4 划痕实验: 按5×105/孔接种细胞于6孔板中, 用灭菌的10 µL枪头划痕, 再用磷酸盐缓冲液(PBS)洗2遍后进行转染, 每条划痕取3个不同的点, 分别在转染前及转染后24、48、72 h时拍照, 观察划痕愈合速度.
1.2.5 细胞周期检测: 按3×105/孔接种细胞于6孔板中, 分3组转染后继续培养, 待48 h收集细胞, PBS漂洗2次, 用125 µL PBS悬浮细胞, 缓慢加入-20 ℃预冷的375 µL无水乙醇中, 摇匀, 4 ℃固定过夜. 次日离心去掉乙醇, PBS漂洗2次, 加入500 µL含RNA酶(50 mg/L)及0.1%Triton X-100的PBS, 37 ℃孵育1 h后, 加入PI(1 g/L)染色液5 µL, 冰上避光孵育30 min, 200目钢筛过滤, 流式细胞仪检测细胞周期, 重复3次.
1.2.6 细胞凋亡检测: 按3×105/孔接种细胞于6孔板中, 分3组转染后继续培养48 h收集细胞, PBS漂洗2次, 每管加入100 µL Binding Buffer及Annexin-Ⅴ染色液5 µL, 5 min后加入PI(10 g/L)染色液0.4 µL, 冰上避光孵育5 min, 200目钢筛过滤, 于30 min之内上机检测细胞凋亡, 重复3次.
统计学处理 采用SPSS17.0统计软件进行分析, 计量资料采用mean±SD表示. 若为计量资料且服从正态分布, 多组之间采用单因素方差分析, 以P<0.05为差异具有统计学意义.
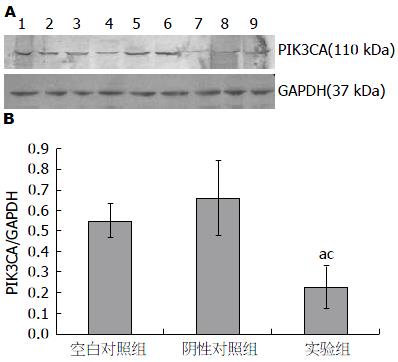
通过RNA转染干扰PIK3CA基因的表达, 72 h后, 提取细胞总蛋白做Western blot检测, PIK3CA蛋白条带在110 kDa处显色, GAPDH蛋白条带在37 kDa处显色(图1A). 实验组细胞PIK3CA蛋白的相对表达量(0.22±0.11)显著低于空白对照组(0.55±0.08)及阴性对照组细胞(0.66±0.19)PIK3CA蛋白的相对表达量, 且差异有统计学意义(P<0.05, 图1B). 结果说明, 通过脂质体介导的siRNA转染使PIK3CA蛋白的表达下降.
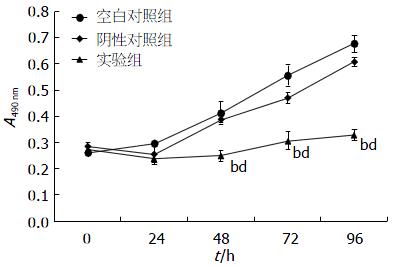
通过RNA转染干扰PIK3CA基因的表达后, 分别检测0、24、48、72、96 h细胞增殖活性(图2). 0 h各组490 nm波长处A值之间差异无统计学意义(P>0.05), 24 h实验组A值与阴性对照组之间差异无统计学意义(P>0.05). 48、72、96 h实验组A值明显低于其余组(P<0.01). 结果说明, PIK3CA基因表达下调后细胞的增殖被抑制.
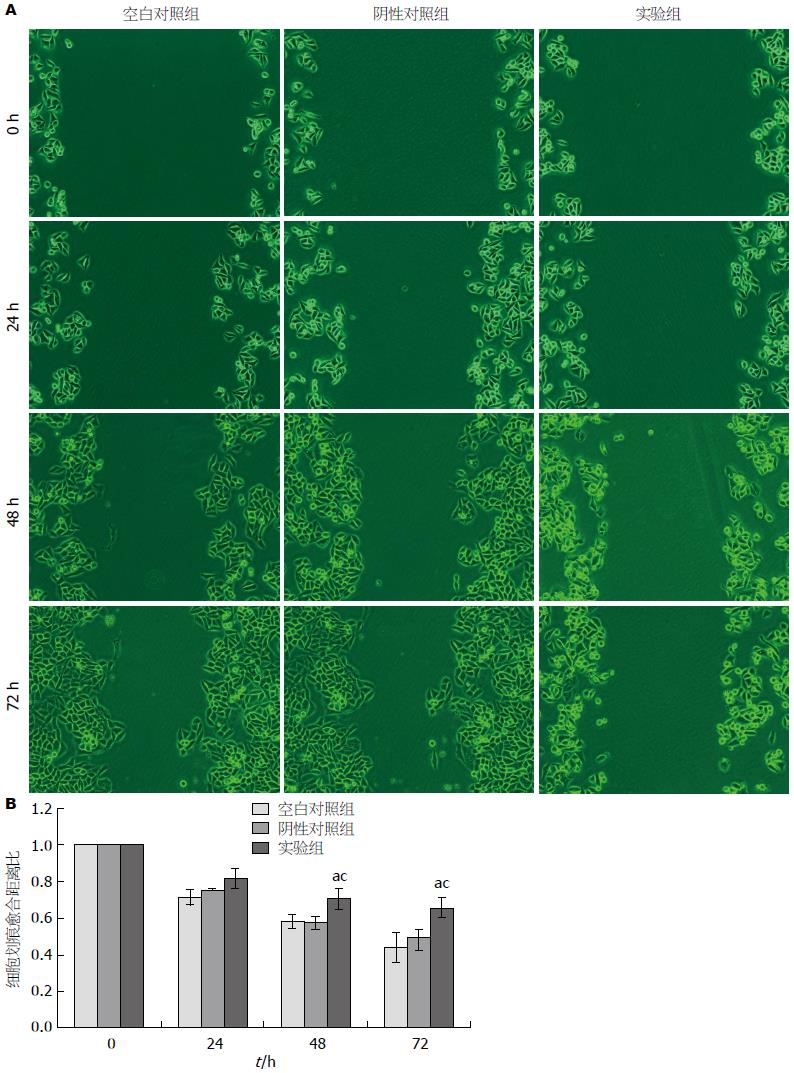
转染siRNA后, 细胞划痕, 检测对细胞迁移能力的影响(图3A). 可见0、24 h各组细胞划痕愈合距离比之间差异无统计学意义, 而转染后48、72 h实验组愈合距离明显宽于其他两组, 愈合距离比明显大于其他两组, 可见转染48、72 h后细胞迁移被抑制, 划痕愈合迟缓, 且差异均有统计学意义(P<0.05, 图3B). 结果显示, PIK3CA沉默后可抑制Eca109细胞的迁移能力.
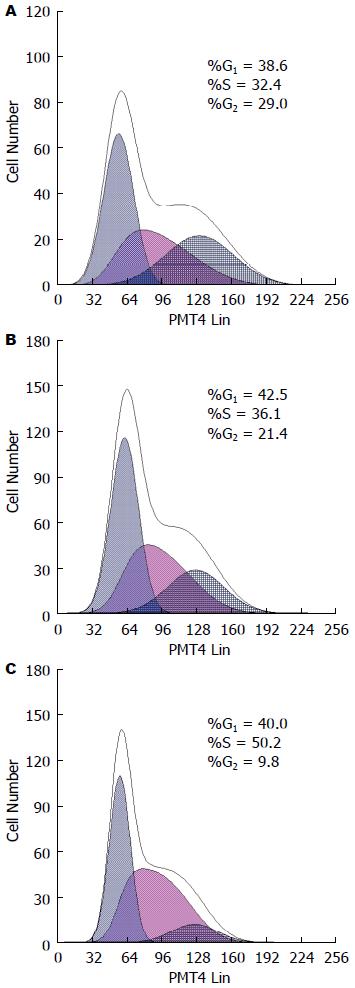
转染siRNA 48 h后, 流式细胞仪检测细胞周期变化(图4). 各组G2/M期与S期细胞百分比差异有统计学意义(P<0.05), 其中实验组G2/M期细胞百分比低于空白对照组及阴性对照组, 实验组S期细胞百分比高于空白对照组及阴性对照组, 差异均有统计学意义(P<0.05, 表1). 提示下调PIK3CA的表达使Eca109细胞阻滞在细胞周期的S期.
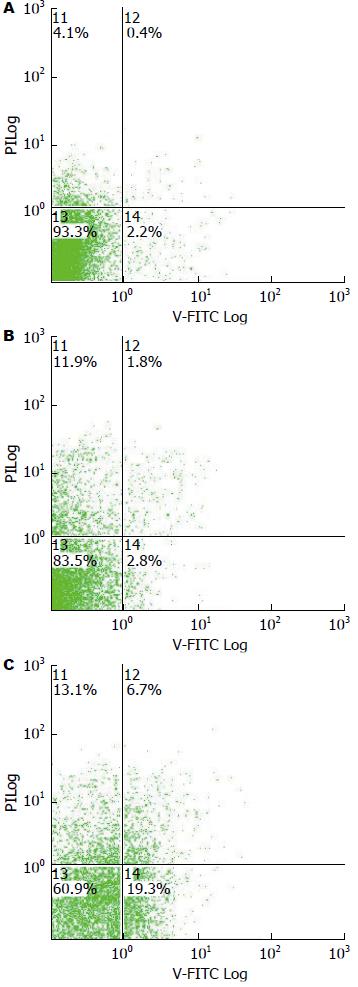
转染siRNA 48 h后, 用Annexin Ⅴ及PI对细胞进行标记, 用流式细胞仪检测细胞凋亡的改变(图5). 散点图上第四象限显示的是早期凋亡细胞, 可见实验组细胞凋亡率19.10%±1.31%明显高于空白对照组1.93%±0.78%及阴性对照组2.83%±1.00%, 且差异有统计学意义(P<0.05). 提示PIK3CA的表达下调能促进Eca109细胞的凋亡.
研究发现PI3K/Akt信号通路在细胞的增殖、凋亡、迁移、膜泡转运和细胞的恶性转化等病理生理过程中起重要的作用[7,8]. 而PIK3CA是PI3K/Akt通路中一个重要的组成部分[9], 具有调控肿瘤细胞增殖、分化、代谢、存活[10-13]等生理功能. 目前关于PIK3CA基因在肿瘤中的研究多集中在两方面: (1)是外显子突变研究; (2)是基因拷贝数变化. 研究[14-16]发现PIK3CA在胃腺癌、食管癌、垂体瘤等多种实体肿瘤中存在外显子突变; 而PIK3CA基因在宫颈癌[17]、卵巢癌[18]、乳腺癌[19]、淋巴瘤[20]、肺癌[21]及甲状腺癌[22]等多种肿瘤中存在基因拷贝数扩增. 但在食管鳞癌中, PIK3CA基因拷贝数变化的研究鲜有报道. 因此, 在前期研究中我们运用CGH技术发现PIK3CA基因在食管癌鳞癌组织中呈显著性扩增. Konopka等[23]研究发现在子宫内膜癌中PIK3CA的扩增率达12.2%, Shayesteh等[24]报道在约80%的早期卵巢癌和一些上皮性卵巢癌细胞中PIK3CA表达增高. PIK3CA在胃癌细胞系和原发性胃癌组织中的扩增率分别为60.0%和36.4%, 而癌旁组织中无扩增[25]. Akagi等[26]研究提出食管鳞癌中PIK3CA的mRNA水平是正常上皮的2.61倍. 这与我们前期CGH研究发现PIK3CA基因在食管鳞癌组织中显著性扩增这一现象是一致的.
研究[17]发现, 在宫颈组织中, PIK3CA基因的扩增随着肿瘤的恶性程度而增加. 在胃癌中, Zhou等[27]发现, 下调PIK3CA能显著性抑制胃癌细胞的增殖、迁移及侵袭能力; Shi等[28]研究发现PIK3CA基因的扩增与肿瘤的侵袭能力呈正相关. 在本研究中, 我们利用RNAi成功地将食管癌细胞系Eca109的PIK3CA基因沉默后, 与对照组相比, MTT实验发现在转染48 h后实验组细胞增殖显著性降低, 划痕实验显示转染48 h后实验组细胞伤痕愈合受到显著性抑制, 并且差异均具有统计学意义(P<0.05), 表明沉默PIK3CA能显著性抑制食管癌细胞的增殖及迁移能力, 这与Zhou等[28]在胃癌中报道的结果是一致的, 提示PIK3CA基因能促进食管癌细胞的增殖和迁移, 在食管鳞癌的发生发展中可能起着重要的作用.
Huang等[29]发现, 干扰PIK3CA基因的表达可使结肠癌细胞系SW948的细胞周期阻滞在G0/G1期, 且细胞凋亡增加; 而Zhang等[30]研究发现, 在卵巢癌细胞系SKOV3中下调PIK3CA表达后, 细胞周期阻滞在S期, 且细胞凋亡增加. 在本研究中, 与对照组细胞相比, 沉默PIK3CA基因后, 实验组细胞的S期显著性增高, G2/M期显著性降低(P<0.05), 即细胞周期阻滞在S期. 此外与对照组细胞相比, 实验组细胞凋亡率显著性增高(P<0.05), 可见沉默PIK3CA基因可促进Eca109细胞的凋亡. 这与Zhang等[30]的研究结果是一致的. 提示沉默PIK3CA基因后, 细胞有丝分裂周期被阻滞在S期, 抑制细胞进入有丝分裂后期, 从而抑制Eca109细胞的增殖.
总之, 在本研究中, 我们发现PIK3CA基因能够促进食管鳞癌细胞Eca109的增殖及迁移, 并且具有抗凋亡作用; 沉默PIK3CA基因后, 细胞周期阻滞在S期. 因此, PIK3CA基因在食管鳞癌的发生发展中起着重要的作用, 有望成为食管鳞癌治疗的新靶点. 在后续的研究中, 我们将对PIK3CA基因扩增的临床病理学意义及相关的信号通路进行研究.
研究发现PIK3CA基因的不同外显子的突变及基因扩增在肿瘤的发生发展中起到了重要的作用, 但PIK3CA基因在食管鳞癌中功能研究鲜有报道.
郭炜, 教授, 河北医科大学第四医院河北省肿瘤研究所病理研究室
PIK3CA基因能否成为抑制食管癌转移的一个潜在治疗靶点是目前研究的一个热点.
Wang等发现PIK3CA基因在中国人食管癌组织中显著性扩增, PIK3CA可能是食管癌个体分子治疗的一个潜在靶点.
国内外研究报道多是围绕PIK3CA外显子突变, 但关于PIK3CA扩增在食管癌中的作用鲜有报道. 本文围绕PIK3CA在食管癌中发生扩增这一现象展开.
本文研究其在食管癌细胞中的作用, 为探讨该基因能否作为一个抑制转移的有效的治疗靶点提供实验依据.
比较基因组杂交技术(CGH): 将消减杂交、荧光原位杂交相结合, 用于检测DNA序列的拷贝数变异并将其定位在染色体上的方法.
本文立题依据充分, 研究方法可靠, 数据详实, 有一定科学意义.
编辑: 郭鹏 电编:闫晋利
| 1. | Napier KJ, Scheerer M, Misra S. Esophageal cancer: A Review of epidemiology, pathogenesis, staging workup and treatment modalities. World J Gastrointest Oncol. 2014;6:112-120. [PubMed] [DOI] |
| 2. | Merkow RP, Bilimoria KY, Keswani RN, Chung J, Sherman KL, Knab LM, Posner MC, Bentrem DJ. Treatment trends, risk of lymph node metastasis, and outcomes for localized esophageal cancer. J Natl Cancer Inst. 2014;106:pii: dju133. [PubMed] [DOI] |
| 3. | Pech O. Nodes or no nodes? The lymph node metastasis risk of T1 esophageal cancer revisited. J Natl Cancer Inst. 2014;106:pii: dju174. [PubMed] [DOI] |
| 4. | Volinia S, Hiles I, Ormondroyd E, Nizetic D, Antonacci R, Rocchi M, Waterfield MD. Molecular cloning, cDNA sequence, and chromosomal localization of the human phosphatidylinositol 3-kinase p110 alpha (PIK3CA) gene. Genomics. 1994;24:472-477. [PubMed] |
| 5. | Rivière JB, Mirzaa GM, O'Roak BJ, Beddaoui M, Alcantara D, Conway RL, St-Onge J, Schwartzentruber JA, Gripp KW, Nikkel SM. De novo germline and postzygotic mutations in AKT3, PIK3R2 and PIK3CA cause a spectrum of related megalencephaly syndromes. Nat Genet. 2012;44:934-940. [PubMed] [DOI] |
| 6. | Walls M, Baxi SM, Mehta PP, Liu KK, Zhu J, Estrella H, Li C, Zientek M, Zong Q, Smeal T. Targeting small cell lung cancer harboring PIK3CA mutation with a selective oral PI3K inhibitor PF-4989216. Clin Cancer Res. 2014;20:631-643. [PubMed] [DOI] |
| 7. | Su JC, Lin KL, Chien CM, Tseng CH, Chen YL, Chang LS, Lin SR. Naphtho[1,2-b]furan-4,5-dione inactivates EGFR and PI3K/Akt signaling pathways in human lung adenocarcinoma A549 cells. Life Sci. 2010;86:207-213. [PubMed] [DOI] |
| 8. | Baryawno N, Sveinbjörnsson B, Eksborg S, Chen CS, Kogner P, Johnsen JI. Small-molecule inhibitors of phosphatidylinositol 3-kinase/Akt signaling inhibit Wnt/beta-catenin pathway cross-talk and suppress medulloblastoma growth. Cancer Res. 2010;70:266-276. [PubMed] [DOI] |
| 9. | Ye B, Jiang LL, Xu HT, Zhou DW, Li ZS. Expression of PI3K/AKT pathway in gastric cancer and its blockade suppresses tumor growth and metastasis. Int J Immunopathol Pharmacol. 2012;25:627-636. [PubMed] |
| 10. | Wu G, Xing M, Mambo E, Huang X, Liu J, Guo Z, Chatterjee A, Goldenberg D, Gollin SM, Sukumar S. Somatic mutation and gain of copy number of PIK3CA in human breast cancer. Breast Cancer Res. 2005;7:R609-R616. [PubMed] |
| 11. | Dumont AG, Dumont SN, Trent JC. The favorable impact of PIK3CA mutations on survival: an analysis of 2587 patients with breast cancer. Chin J Cancer. 2012;31:327-334. [PubMed] [DOI] |
| 12. | Zhu YF, Yu BH, Li DL, Ke HL, Guo XZ, Xiao XY. PI3K expression and PIK3CA mutations are related to colorectal cancer metastases. World J Gastroenterol. 2012;18:3745-3751. [PubMed] [DOI] |
| 13. | Broderick DK, Di C, Parrett TJ, Samuels YR, Cummins JM, McLendon RE, Fults DW, Velculescu VE, Bigner DD, Yan H. Mutations of PIK3CA in anaplastic oligodendrogliomas, high-grade astrocytomas, and medulloblastomas. Cancer Res. 2004;64:5048-5050. [PubMed] |
| 14. | Li VS, Wong CW, Chan TL, Chan AS, Zhao W, Chu KM, So S, Chen X, Yuen ST, Leung SY. Mutations of PIK3CA in gastric adenocarcinoma. BMC Cancer. 2005;5:29. [PubMed] |
| 15. | Wang L, Shan L, Zhang S, Ying J, Xue L, Yuan Y, Xie Y, Lu N. PIK3CA gene mutations and overexpression: implications for prognostic biomarker and therapeutic target in Chinese esophageal squamous cell carcinoma. PLoS One. 2014;9:e103021. [PubMed] [DOI] |
| 16. | Lin Y, Jiang X, Shen Y, Li M, Ma H, Xing M, Lu Y. Frequent mutations and amplifications of the PIK3CA gene in pituitary tumors. Endocr Relat Cancer. 2009;16:301-310. [PubMed] [DOI] |
| 17. | Ma YY, Wei SJ, Lin YC, Lung JC, Chang TC, Whang-Peng J, Liu JM, Yang DM, Yang WK, Shen CY. PIK3CA as an oncogene in cervical cancer. Oncogene. 2000;19:2739-2744. [PubMed] |
| 18. | Kolasa IK, Rembiszewska A, Felisiak A, Ziolkowska-Seta I, Murawska M, Moes J, Timorek A, Dansonka-Mieszkowska A, Kupryjanczyk J. PIK3CA amplification associates with resistance to chemotherapy in ovarian cancer patients. Cancer Biol Ther. 2009;8:21-26. [PubMed] |
| 19. | Firoozinia M, Zareian Jahromi M, Moghadamtousi SZ, Nikzad S, Abdul Kadir H. PIK3CA gene amplification and PI3K p110α protein expression in breast carcinoma. Int J Med Sci. 2014;11:620-625. [PubMed] [DOI] |
| 20. | Cui W, Cai Y, Wang W, Liu Z, Wei P, Bi R, Chen W, Sun M, Zhou X. Frequent copy number variations of PI3K/AKT pathway and aberrant protein expressions of PI3K subunits are associated with inferior survival in diffuse large B cell lymphoma. J Transl Med. 2014;12:10. [PubMed] [DOI] |
| 21. | Yu S, Zhang Z, Zhang B, Shu Y, Wu H, Huang X, Yu Q, Guo R. Clinical significance of PIK3CA and survivin in primary adenosquamous lung carcinoma. Med Oncol. 2014;31:983. [PubMed] [DOI] |
| 22. | Abubaker J, Jehan Z, Bavi P, Sultana M, Al-Harbi S, Ibrahim M, Al-Nuaim A, Ahmed M, Amin T, Al-Fehaily M. Clinicopathological analysis of papillary thyroid cancer with PIK3CA alterations in a Middle Eastern population. J Clin Endocrinol Metab. 2008;93:611-618. [PubMed] |
| 23. | Konopka B, Janiec-Jankowska A, Kwiatkowska E, Najmoła U, Bidziński M, Olszewski W, Goluda C. PIK3CA mutations and amplification in endometrioid endometrial carcinomas: relation to other genetic defects and clinicopathologic status of the tumors. Hum Pathol. 2011;42:1710-1719. [PubMed] [DOI] |
| 24. | Shayesteh L, Lu Y, Kuo WL, Baldocchi R, Godfrey T, Collins C, Pinkel D, Powell B, Mills GB, Gray JW. PIK3CA is implicated as an oncogene in ovarian cancer. Nat Genet. 1999;21:99-102. [PubMed] |
| 25. | Byun DS, Cho K, Ryu BK, Lee MG, Park JI, Chae KS, Kim HJ, Chi SG. Frequent monoallelic deletion of PTEN and its reciprocal associatioin with PIK3CA amplification in gastric carcinoma. Int J Cancer. 2003;104:318-327. [PubMed] |
| 26. | Akagi I, Miyashita M, Makino H, Nomura T, Hagiwara N, Takahashi K, Cho K, Mishima T, Ishibashi O, Ushijima T. Overexpression of PIK3CA is associated with lymph node metastasis in esophageal squamous cell carcinoma. Int J Oncol. 2009;34:767-775. [PubMed] |
| 27. | Zhou XK, Tang SS, Yi G, Hou M, Chen JH, Yang B, Liu JF, He ZM. RNAi knockdown of PIK3CA preferentially inhibits invasion of mutant PIK3CA cells. World J Gastroenterol. 2011;17:3700-3708. [PubMed] [DOI] |
| 28. | Shi J, Yao D, Liu W, Wang N, Lv H, Zhang G, Ji M, Xu L, He N, Shi B. Highly frequent PIK3CA amplification is associated with poor prognosis in gastric cancer. BMC Cancer. 2012;12:50. [PubMed] [DOI] |
| 29. | Huang WS, Wang TB, He Y, Chen YJ, Zhong SL, Tan M. Phosphoinositide-3-kinase, catalytic, alpha polypeptide RNA interference inhibits growth of colon cancer cell SW948. World J Gastroenterol. 2012;18:3458-3464. [PubMed] [DOI] |