修回日期: 2013-08-27
接受日期: 2013-09-09
在线出版日期: 2013-09-18
目的: 明确用基因表达水平的贝叶斯分析(Bayesian analysis of gene expression levels, BAGEL)所建立的胃癌弱差异基因表达谱(fold change<1.5)的特异性及生物学意义.
方法: Cluster3.0聚类分析基因DNA甲基转移酶3A[DNA(cytosine-5-)-methyltransferase 3 alpha, DNMT3A]、发育分化增强因子2 (development and differentiation-enhancing factor 2, DDEF2)、分化群59(cluster of differentiation 59, CD59)的表达; 机器学习和模式识别技术对弱差异基因表达谱(fold change<1.5, P<0.001)进行特征提取; GoMiner软件分析弱差异表达基因DNMT3A、DDEF2、CD59的生物学功能; RT-PCR在实验室验证弱差异表达基因DNMT3A、DDEF2、CD59在胃癌及癌旁配对组织中mRNA的表达水平.
结果: 从弱差异基因表达谱中得到能够识别样本类别的62个分类特征基因和4个分类能力较强的基因; GoMiner分析表明这组基因参与细胞黏附、细胞吞噬、免疫调节、基因甲基化、转录调控等重要生物学作用; RT-PCR确定基因表达变化与芯片中基因表达谱的数据一致, 证实了弱差异基因表达谱的可靠性和灵敏性.
结论: 通过BAGEL分析得到的fold change<1.5的弱差异基因表达谱灵敏可靠, 在肿瘤发生发展中关系密切.
核心提示: BAGEL统计学方法建立的弱差异表达基因谱灵敏可靠, 弱差异表达基因与胃癌关系密切, 为胃癌特征基因的筛选提供了新的思路.
引文著录: 孙伟, 高芳, 龙启福, 王晓龙, 朱德锐, 顾存林, 安娟, 党国全, 吴穹. 胃癌弱差异基因表达谱建立的生物学意义. 世界华人消化杂志 2013; 21(26): 2717-2723
Revised: August 27, 2013
Accepted: September 9, 2013
Published online: September 18, 2013
AIM: To clarify the specificity and biological significance of profiling of weakly differentially expressed genes (fold change < 1.5) in gastric cancer by Bayesian analysis of gene expression levels (BAGEL) analysis.
METHODS: The mRNA expression of DNA (cytosine-5-)-methyltransferase 3 alpha (DNMT3A), development and differentiation-enhancing factor 2 (DDEF2) and cluster of differentiation 59 (CD59) was clustered by Cluster 3.0. Machine study and pattern recognition analysis was used to study the feature of weakly differentially expressed genes. The functions of CD59, DDEF2 and DNMT3A were analyzed using GoMiner. The mRNA expression of CD59, DDEF2 and DNMT3A in tissues was detected by RT-PCR.
RESULTS: We have collected 62 classification characteristic genes from 64 weakly differentially expressed genes and obtained 4 strongest classification genes by machine study and pattern recognition analysis. GoMiner analysis showed that these weakly differentially expressed genes were associated with cell adhesion, phagocytosis, immunological regulation, gene methylation and transcription regulation. Furthermore, the differential expression of these genes in tissue samples was confirmed by RT-PCR.
CONCLUSION: Profiling of weakly differentially expressed genes by BAGEL analysis is specific and allows to identify genes that are closely related to tumorigenesis.
- Citation: Sun W, Gao F, Long QF, Wang XL, Zhu DR, Gu CL, An J, Dang GQ, Wu Q. Biological significance of profiling of weakly differentially expressed genes in gastric cancer. Shijie Huaren Xiaohua Zazhi 2013; 21(26): 2717-2723
- URL: https://www.wjgnet.com/1009-3079/full/v21/i26/2717.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i26.2717
基因芯片技术自上世纪90年代在美国率先发展起来后, 已被广泛应用于各种生物学研究领域. 其中最重要应用之一就是通过确定样本在不同条件下的差异表达基因, 来明确某种因素对基因表达的调控作用. 一般而言, 目前在生物学研究领域中, 往往利用芯片进行全基因组的扫描来初筛可能的差异表达基因, 然后再采用逆转录PCR反应(RT-PCR)或核酸印迹分析(Northern blot)等实验手段来对基因芯片筛选出的差异表达基因进行验证. 但由于基因芯片信号噪音偏大, 芯片数据的不同分析方法等主客观因素都会对结果产生显著的影响[1]. 所以在选择差异表达基因验证方法时, 往往选择基因变化倍数为2倍以上的基因作进一步分析, 而大量的变化倍数为2倍以下的基因被删除, 这些基因是否真的是无关基因并没有确切的证据. 而Yan等[2]通过对结肠腺瘤样息肉(adenomatosis polyposis coli, APC)基因的定量分析发现该基因表达的微弱减少与遗传的结直肠肿瘤发生有关, 提示弱差异表达基因可能在肿瘤的发生发展中有更重要的功能. 因此, 对弱差异表达基因的认识亟待深入认识.
Zang等[3]采用SAM[4]和BAGEL[5]两种统计学方法进行差异表达基因的统计学推断, 并利用基因表达水平的贝叶斯分析(Bayesian analysis of gene expression levels, BAGEL)建立了胃癌差异表达基因谱. Guo等对fold change>1.5的55个具有重要生物学功能的差异表达基因进行了验证, 获得了与芯片杂交数据一致的结果, 吻合率为98%(54/55). 为了继续明确基因表达谱中弱差异表达基因(fold change<1.5, P<0.001)的敏感性、特异性和生物学意义, 我们在Zang等[3]建立的弱差异表达基因的基础上进行了进一步生物信息学分析, 并选取部分基因进行了生物学功能分析及实验室验证, 探讨弱差异表达基因在胃癌发生发展过程中的生物学意义.
胃肿瘤组织标本来自青海大学附属医院于2005-05/2005-10收治的胃肿瘤患者20例(男13例, 女7例), 年龄51-87岁(中位年龄65岁). 所有标本获得患者知情同意, 均由胃癌组织及相距5 cm以上的癌旁正常组织配对组成, 标本手术切除后立即放于液氮, -80 ℃冻存备用.
1.2.1 聚类分析法: 聚类分析采用Cluster 3.0[6]芯片分析工具, 分析过程采用average linkage算法.
1.2.2 机器学习和模式识别法: 以每个基因所含的20对胃癌组织与癌旁正常配对组织的样本分类信息的指标作为尺度来过滤与样本分类无关的噪声基因, 然后采用支持向量机(support vector machine, SVM)作为分类器对特征基因集合的分类能力进行检验. 从而由原始基因集合中提取出最能反映样本分类的特征基因集合.
1.2.3 基因功能注释法: 功能注释分析采用GoMiner[7]软件, 该软件可从分子功能、细胞定位以及生物学过程三方面对基因进行系统分析, 并形成相应的定向无环图和树型结构. 点击树型结构中的基因可以直接链接到外挂的信息库: 如LocusLink、PubMed、MedMiner、GeneCards、NCBI's Structure Database、BioCarta、KEGG、CGAP等, 从而得到信息的整合.
1.2.4 逆转录聚合酶链反应法: 总RNA提取及逆转录聚合酶链反应法(reverse transcription-PCR, RT-PCR)参照文献[8]. PCR反应: 以cDNA为模板, β-actin作为内参, Taq酶购自Takara公司, 内参和目的基因的扩增在同一反应中扩增. PCR参数: 94 ℃ 5min; 94 ℃ 30 s, 53 ℃ 30 s, 72 ℃ 30 s, 分化群59(cluster of differentiation 59, CD59)、发育分化增强因子2(development and differentiation-enhancing factor 2, DDEF2)、DNA甲基转移酶3A[DNA(cytosine-5-)-methyltransferase 3 alpha, DNMT3A]循环次数分别为22、26、28; 72 ℃延伸5 min. 引物序列如表1所示. 扩增的PCR产物于20 g/L琼脂糖凝胶电泳, 电压100 V, 20 min, Alphalmager2200v5.5凝胶图像分析系统摄片并测定分析其光密度.
| 基因名称 | 引物序列 | 扩增长度(bp) |
| DDEF2 | 上游 5'-GGCGACTACTCCACGAAGACC-3' | 229 |
| 下游 5'-AGGAGGGCTCCGTAAGTCTCA-3' | ||
| CD59 | 上游 5'-TGCGTGTCTCATTACCAAA-3' | 258 |
| 下游 5'-GCTCTCCTGGTGTTGACTTAG-3' | ||
| DNMT3A | 上游 5'-GCACTGAAATGGAAAGGGTAT-3' | 196 |
| 下游 5'-ACTCCATCGCTGTGTTTCAAT-3' | ||
| β-actin-1 | 上游 5'-TTAGTTGCGTTACACCCTTTC-3' | 150 |
| 下游 5'-ACCTTCACCGTTCCAGTTT-3' | ||
| β-actin-2 | 上游 5'-TCACCCACACTGTGCCCATCTACGA-3' | 295 |
| 下游 5'-CAGCGGAACCGCTCATTGCCAATGG-3' |
统计学处理 各个样本基因表达水平表达量的相对值 = 扩增条带灰度值/β-actin扩增条带灰度值; 癌与癌旁表达差异用t检验及百分比同时进行统计分析. P<0.05为差异具有统计学意义.
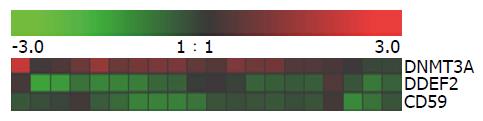
以BAGEL确定的弱差异基因表达谱为基础, 利用Cluster3.0软件采用average linkage算法分析比较了弱差异表达基因CD59、DDEF2、DNMT3A的聚类结果. 显示了这3个基因在胃癌组织相较于癌旁正常组织时的表达变化: DNMT3A在胃癌组织中为高表达, DDEF2、CD59在胃癌组织中为低表达(图1, 红色: 基因表达水平上调; 绿色: 基因表达水平下调; 黑色: 基因表达变化不明显).
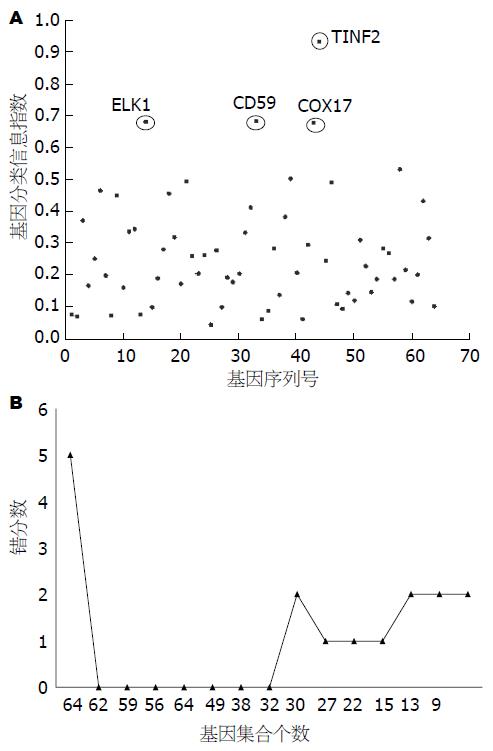
从20对胃癌及癌旁正常配对样本中各选取15对样本作为训练集, 其余5对样本作为测试集. 在训练样本集上计算每个基因的分类信息指数, 并作出了基因分类信息指数散点图(图2A). 分类信息指数越大这个基因的分类能力越强, 说明这个基因在两类样本中的特征越明显, 越有可能成为靶基因. 图中红色圆圈中的基因是信息指数比较大的4个基因: 分别是TINF2、ELK1、COX17、CD59.
从64个基因构成的特征集合F64开始到一系列具有不同维数的分类特征子集F64、F63、...F1. 当特征基因集合等于32个基因的时候, 基因集合的样本错分数为零, 直到集合中的基因为62个的时候, 其样本分类正确率达100%. 但是特征基因集合增加2个基因到64的时候分类正确率仅为50%, 这2个基因分别是BC017696基因和DGCR2基因, 说明这2个基因是冗余基因(图2B); 弱差异基因表达谱中有62个分类特征基因集合(62/64, 97%)能够有效识别胃癌样本类别.
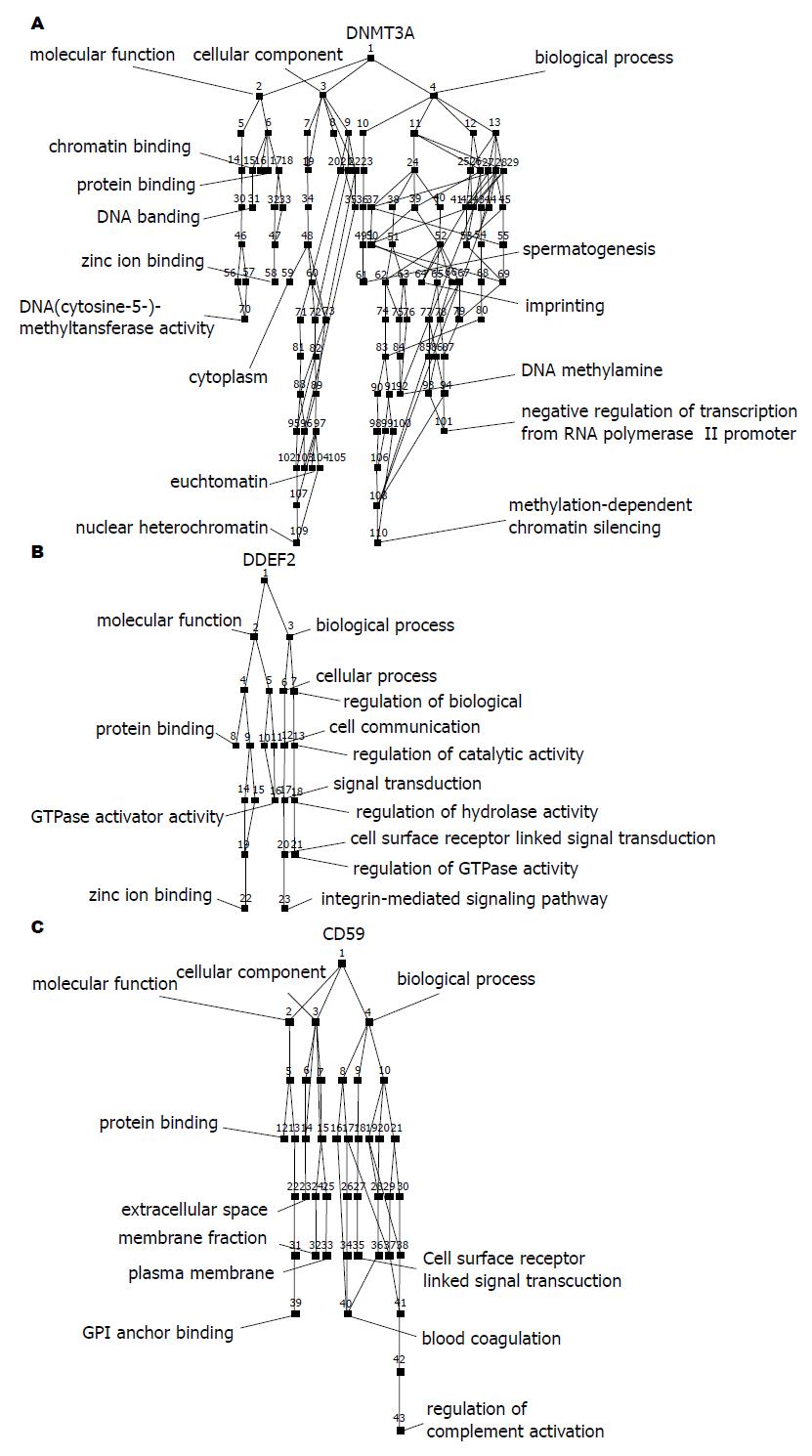
GoMiner软件注释三个弱差异表达候选基因. 结果表明DNMT3A主要具有甲基转移酶的活性, 在精子发生、胚胎发育与分化、DNA甲基化、甲基化依赖的染色质失活、基因转录抑制等方面具有重要作用(图3A); DDEF2主要具有蛋白结合、GTP酶激活活性, 在整联蛋白介导的信号途径、GTP酶活性调节等重要生物学途径中具有重要作用(图3B); CD59主要具有蛋白结合、糖基磷脂酰肌醇锚结合活性, 在补体活性的调节等重要生物学途径中具有重要调节作用(图3C).
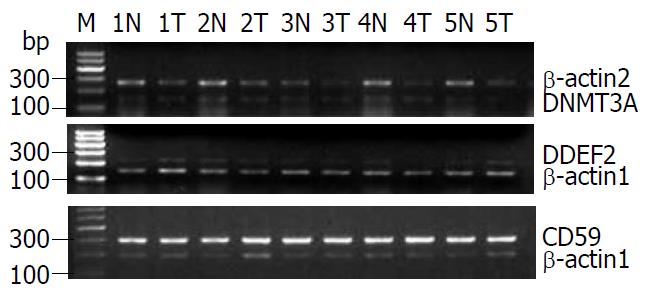
对20对胃癌/癌旁配对组织进行RT-PCR凝胶电泳成像及灰度值扫描计算表达相对量, 分析mRNA表达差异(图4, 表2). DDEF2与CD59基因在胃癌组织中低表达, 在16例胃癌组织中表达水平低于配对的癌旁正常组织(16/20, 80%, P<0.001); 而DNMT3A基因在肿瘤组织中高表达, 在17例胃癌组织中表达水平高于配对的癌旁正常组织(17/20, 85%, P<0.001). 基因表达灰度变化倍数为胃癌组织与癌旁正常组织中基因表达量相对值的平均值之比, 比较实验室灰度变化倍数与基因芯片变化倍数发现两者变化倍数一致, DNMT3A、DDEF2、CD59 mRNA表达变化倍数分别为1.13 vs1.10、1.06 vs 1.01、1.30 vs 1.40.
| 临床样本 | n | DNMT3A | DDEF2 | CD59 | ||||||
| 高表达 | 低表达 | P值 | 高表达 | 低表达 | P值 | 高表达 | 低表达 | P值 | ||
| 胃癌 | 20 | 17 | 3 | <0.001 | 4 | 16 | <0.001 | 4 | 16 | <0.001 |
| 癌旁 | 20 | 3 | 17 | 16 | 4 | 16 | 4 | |||
1990年基因芯片作为一种高新的生物学实验技术率先在美国发展起来, Schena等[9]首次报道利用基因芯片技术定量监测基因表达模式以来, 芯片技术不断地应用于各种生物学研究领域, 并迅速发展成为基因组学和系统生物学研究的重要工具. 随着芯片技术的不断完善, 其实验数据的可重复性和可靠性明显增加, 但选择不同的数据整理和分析方法对于结果的有显著的影响[10]. 强差异表达基因易于实验室验证, 但由于基因芯片噪音及统计学方法等不同因素所确立的弱差异表达基因(fold chang<1.5)一般难以验证, 其在胃癌中的生物学意义也亟待明确. 基于Zang等[3]建立的胃癌弱差异基因表达谱具有较高的敏感度和可靠性, 有助于进一步实验室验证及生物学功能研究.
机器学习和支持SVM模式识别技术主要用来进行肿瘤分类, 其分类能力也反应了表达谱的灵敏性. 因此, 我们对弱差异基因表达谱进行了进一步的生物信息学研究. 在基因表达谱数据的分析中, 该方法对弱差异基因表达谱的分析得到了能够100%识别胃癌和癌旁组织的62个分类特征基因(62/64, 97%)和4个分类能力较强的特征基因: 即TINF2、ELK1、COX7和CD59. 从另一个侧面理论证实了弱差异基因表达谱的可靠性和灵敏性. 在此基础上, 选择了具有强分类能力的特征基因CD59及另外两个弱差异表达基因DDEF2、DNMT3A进行了实验室验证, 证实这3个基因以及另一个弱差异表达基因TINF2[11]均与芯片数据保持一致, 一致率达到100%(4/4). 实验室验证的结果说明胃癌弱差异基因表达谱的可靠性和灵敏性, 利于进一步探讨其生物学意义.
我们综合应用生物信息学及文献检索对弱差异基因表达谱基因功能进行进一步的确定, 以明确弱差异表达基因并非肿瘤发生的无关基因, 甚至可能是肿瘤发生过程中的关键基因, 为肿瘤研究方向提供新的思考方向. 通过高通量GoMiner的信息整合, 我们发现这64个弱差异表达基因的重要的生物学功能主要表现在血管形成、免疫调节和基因转录调控等方面, 更多地与细胞营养, 不同基因的表达调控等代谢方面有更紧密地联系. 在实验室验证的3个基因均与肿瘤发生发展有密切联系. 研究发现DNMT3A与P53[12]、组蛋白甲基转移酶SETDB1[13]相互作用, 其与结直肠癌[14]、胰腺癌[15]、胃癌[16]等肿瘤相关. DDEF2是蛋白酪氨酸激酶2(PYK2)和SRC作用通路的底物, Matsui等[17]发现DDEF2能抑癌蛋白APC的特异结合; Coutinho-Camillo等[18]认为DDEF2在前列腺癌中的表达模式发生改变. CD59分子是人体内重要的同源限制因子, 他能阻止补体穿膜复合体MAC在膜上的组装而保护细胞不被自身补体溶解[19], 参与肿瘤细胞生长与凋亡[20], 在乳腺癌[21]、膀胱癌[22]、口腔癌[23]、前列腺癌[24]等多种肿瘤中表达升高.
来自不同层面的分析都表明弱差异基因表达谱具有较高的敏感性和特异性, 在肿瘤的发生发展中具有重要的生物学功能和临床意义. 我们看到这些弱差异表达基因的表达改变导致肿瘤细胞内环境的改变, 是参与胃癌发生发展的重要因素, 所以挖掘BEGEL建立的弱差异表达基因对胃癌发生发展的研究具有重要意义.
基因芯片技术在生物领域广泛应用, 但由于其自身的噪音及统计学方法的差异等因素使基因芯片结果的实验室验证更多集中在差异倍数大于2的基因. 本文针对利用BEGEL统计学方法建立的胃癌弱差异表达基因进行了实验室验证, 旨在探讨弱差异表达基因在胃癌发生发展中的生物学意义.
李淑德, 教授, 中国人民解放军第二军医大学长海医院消化内科
胃癌发生发展中的特征基因的探寻是胃癌诊疗中研究的热点及重点, 众多研究借助基因芯片技术进行了高通量的筛选以期获得目的基因, 但不同统计方法使结果迥异. 因此在当前基因芯片自身缺陷难以突破的状况下, 寻找适合的统计方法得到有意义的统计结果是当前亟待解决的关键问题之一.
人们很少对差异倍数小于2倍的弱差异表达基因基因芯片进行实验室验证, 但Yan等对结肠腺瘤样息肉(adenomatosis polyposis coli, APC)基因的定量分析发现该基因表达的微弱减少与遗传的结直肠肿瘤发生有关, 提示弱差异表达基因可能在肿瘤的发生发展中有更重要的功能. 因此, 对弱差异表达基因的认识亟待深入认识.
本文以很少被学者关注的胃癌弱差异基因谱为研究对象, 系统验证弱差异表达基因及其生物学功能, 进而分析胃癌弱差异表达基因的生物学意义, 为胃癌标志基因寻找新的切入点.
通过基因芯片技术筛选胃癌特征基因忽略了大量的弱差异表达基因, 但这些弱差异表达基因在胃癌的发生发展中可能具有极其重要的生物学功能. 因此本研究为阐明弱差异表达基因的筛选方法及其在胃癌中的角色具有重要意义, 为胃癌特征基因的筛选提供了新的思路.
弱差异表达基因: 差异基因谱中基因表达差异倍数一般介于0.5-2倍的基因; BAGEL: Bayesian analysis of gene expression levels, 简称BAGEL, 由Jeffrey Townsend研发. 是用于分析spotted microarray数据的统计学工具; DNMT3A: DNA甲基转移酶3A, 参与胚胎发育、遗传印迹及x染色体失活等现象中DNA CpG岛中胞嘧啶的从头甲基化过程; DDEF2: 发育分化增强因子2 , 该基因编码的蛋白定位于高尔基和质膜, 与蛋白络氨酸激酶2-β相互作用形成复合体并被激活的蛋白络氨酸激酶磷酸化. 可能与小泡的转运调节有关; CD59: 分化群59, 补体调节蛋白. 编码的蛋白位于细胞膜表明形成糖蛋白调节补体介导的溶细胞作用, 并与淋巴细胞的信号转导有关.
本文建立胃癌弱差异基因表达谱并探讨其生物学意义, 研究方法先进, 研究结论可靠, 讨论客观, 对阐明胃癌发生发展的分子机制, 有重要的理论指导意义.
编辑: 郭鹏 电编: 闫晋利
| 1. | Quackenbush J. Computational approaches to analysis of DNA microarray data. Yearb Med Inform. 2006;91-103. [PubMed] |
| 2. | Yan H, Dobbie Z, Gruber SB, Markowitz S, Romans K, Giardiello FM, Kinzler KW, Vogelstein B. Small changes in expression affect predisposition to tumorigenesis. Nat Genet. 2002;30:25-26. [PubMed] |
| 3. | Zang S, Guo R, Zhang L, Lu Y. Integration of statistical inference methods and a novel control measure to improve sensitivity and specificity of data analysis in expression profiling studies. J Biomed Inform. 2007;40:552-560. [PubMed] |
| 4. | Tusher VG, Tibshirani R, Chu G. Significance analysis of microarrays applied to the ionizing radiation response. Proc Natl Acad Sci USA. 2001;98:5116-5121. [PubMed] |
| 5. | Townsend JP. Resolution of large and small differences in gene expression using models for the Bayesian analysis of gene expression levels and spotted DNA microarrays. BMC Bioinformatics. 2004;5:54. [PubMed] |
| 6. | Eisen MB, Spellman PT, Brown PO, Botstein D. Cluster analysis and display of genome-wide expression patterns. Proc Natl Acad Sci USA. 1998;95:14863-14868. [PubMed] |
| 7. | Zeeberg BR, Qin H, Narasimhan S, Sunshine M, Cao H, Kane DW, Reimers M, Stephens RM, Bryant D, Burt SK. High-Throughput GoMiner, an 'industrial-strength' integrative gene ontology tool for interpretation of multiple-microarray experiments, with application to studies of Common Variable Immune Deficiency (CVID). BMC Bioinformatics. 2005;6:168. [PubMed] |
| 8. | Sun W, Dong WW, Mao LL, Li WM, Cui JT, Xing R, Lu YY. Overexpression of p42.3 promotes cell growth and tumorigenicity in hepatocellular carcinoma. World J Gastroenterol. 2013;19:2913-2920. [PubMed] [DOI] |
| 9. | Schena M, Shalon D, Davis RW, Brown PO. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science. 1995;270:467-470. [PubMed] |
| 10. | Jeanmougin M, de Reynies A, Marisa L, Paccard C, Nuel G, Guedj M. Should we abandon the t-test in the analysis of gene expression microarray data: a comparison of variance modeling strategies. PLoS One. 2010;5:e12336. [PubMed] [DOI] |
| 11. | 孙 伟, 高 芳, 王 晓龙, 王 嵘, 顾 存林, 沈 国平, 许 博林, 耿 排力. 胃癌弱差异基因表达谱中TINF2基因的表达和生物学作用. 青海医学院学报. 2012;33:235-258. |
| 12. | Wang YA, Kamarova Y, Shen KC, Jiang Z, Hahn MJ, Wang Y, Brooks SC. DNA methyltransferase-3a interacts with p53 and represses p53-mediated gene expression. Cancer Biol Ther. 2005;4:1138-1143. [PubMed] |
| 13. | Li H, Rauch T, Chen ZX, Szabó PE, Riggs AD, Pfeifer GP. The histone methyltransferase SETDB1 and the DNA methyltransferase DNMT3A interact directly and localize to promoters silenced in cancer cells. J Biol Chem. 2006;281:19489-19500. [PubMed] |
| 14. | Zhao Z, Li C, Song Y, Wu Q, Qiao F, Fan H. Association of the DNMT3A -448A>G polymorphism with genetic susceptibility to colorectal cancer. Oncol Lett. 2012;3:450-454. [PubMed] |
| 15. | He S, Wang F, Yang L, Guo C, Wan R, Ke A, Xu L, Hu G, Xu X, Shen J. Expression of DNMT1 and DNMT3a are regulated by GLI1 in human pancreatic cancer. PLoS One. 2011;6:e27684. [PubMed] [DOI] |
| 16. | Wu Q, Lu S, Wang L, Hu J, Qiao F, Qiu X, Zhao C, Lao Y, Song Y, Fan H. DNMT3A rs36012910 A& gt; G polymorphism and gastric cancer susceptibility in a Chinese population. Mol Biol Rep. 2012;39:10949-10955. [PubMed] [DOI] |
| 17. | Matsui C, Kaieda S, Ikegami T, Mimori-Kiyosue Y. Identification of a link between the SAMP repeats of adenomatous polyposis coli tumor suppressor and the Src homology 3 domain of DDEF. J Biol Chem. 2008;283:33006-33020. [PubMed] [DOI] |
| 18. | Coutinho-Camillo CM, Salaorni S, Sarkis AS, Nagai MA. Differentially expressed genes in the prostate cancer cell line LNCaP after exposure to androgen and anti-androgen. Cancer Genet Cytogenet. 2006;166:130-138. [PubMed] |
| 19. | Clayton A, Harris CL, Court J, Mason MD, Morgan BP. Antigen-presenting cell exosomes are protected from complement-mediated lysis by expression of CD55 and CD59. Eur J Immunol. 2003;33:522-531. [PubMed] |
| 20. | Li B, Gao MH, Chu XM. Molecular mechanism of a novel CD59-binding peptide sp22 induced tumor cells apoptosis. J Cell Biochem. 2012;113:3810-3822. [PubMed] [DOI] |
| 21. | Cui W, Zhao Y, Shan C, Kong G, Hu N, Zhang Y, Zhang S, Zhang W, Zhang Y, Zhang X. HBXIP upregulates CD46, CD55 and CD59 through ERK1/2/NF-κB signaling to protect breast cancer cells from complement attack. FEBS Lett. 2012;586:766-771. [PubMed] [DOI] |
| 22. | Varela JC, Atkinson C, Woolson R, Keane TE, Tomlinson S. Upregulated expression of complement inhibitory proteins on bladder cancer cells and anti-MUC1 antibody immune selection. Int J Cancer. 2008;123:1357-1363. [PubMed] [DOI] |
| 23. | Ravindranath NM, Shuler C. Cell-surface density of complement restriction factors (CD46, CD55, and CD59): oral squamous cell carcinoma versus other solid tumors. Oral Surg Oral Med Oral Pathol Oral Radiol Endod. 2007;103:231-239. [PubMed] |