修回日期: 2013-05-12
接受日期: 2013-05-19
在线出版日期: 2013-06-28
目的: 检测微小RNA-130b(miR-130b)在食管鳞癌(esophageal squamous cell carcinoma, ESCC)组织中的表达并探讨其对ESCC细胞增殖、迁移的影响.
方法: microRNA(miRNA)芯片筛选ESCC组织中异常表达的miRNAs, TaqMan MGB探针法定量PCR检测19例ESCC组织及配对癌旁组织标本中miR-130b的表达; 通过脂质体转染模拟物miR-130b mimics(miR-130bm)促进ESCC细胞Eca109中miR-130b的表达, 转染抑制物miR-130b inhibitor(miR-130bi)抑制Eca109细胞中miR-130b的表达; 进一步采用CCK-8法和Transwell迁移实验检测ESCC细胞增殖、迁移的变化; 生物学信息预测miR-130b的靶基因, 双荧光素酶报告基因验证其靶向作用; 采用SYBR Green定量PCR和Western blot检测靶基因mRNA和蛋白表达.
结果: miR-130b在ESCC组织中的表达明显高于癌旁组织(P<0.01); 转染miR-130bm可有效增加ESCC细胞Eca109中miR-130b的表达, 进而促进Eca109细胞的增殖和迁移, 平均迁移细胞数明显多于对照(112.9±2.4 vs 54.3±1.8, P<0.01); 转染miR-130bi则降低细胞中miR-130b的表达; 进而抑制细胞的增殖和迁移, 平均迁移细胞数较对照明显减少(33.9±2.3 vs 56.2±1.9, P<0.01). miR-130b可作用于PTEN3'非翻译区抑制其表达. miR-130b可负向调控PTEN蛋白表达, 并促进Akt磷酸化, 但对PTEN mRNA表达无明显影响.
结论: miR-130b在ESCC组织中表达上调, 增加其表达促进ESCC细胞Eca109增殖和迁移, 降低其表达则抑制Eca109细胞增殖和迁移; miR-130b可在转录后水平负向调控PTEN的表达并促进Akt磷酸化. 提示miR-130b有望成为ESCC治疗的新靶点.
核心提示: 本研究初步证实了miR-130b在食管鳞癌(esophageal squamous cell carcinoma, ESCC)组织中表达上调, 具有促进ESCC细胞Eca109增殖和迁移的作用, 且可在转录后水平靶向调控PTEN蛋白的表达并促进Akt磷酸化. 本实验为ESCC发病机制的研究提供了新靶点, 为miR-130b用于ESCC的靶向治疗提供了初步的理论依据.
引文著录: 郁婷婷, 李硕, 傅敏根, 曹日昇, 朱宏, 施瑞华. miR-130b在食管鳞癌中的表达及对食管鳞癌细胞增殖和迁移的影响. 世界华人消化杂志 2013; 21(18): 1685-1692
Revised: May 12, 2013
Accepted: May 19, 2013
Published online: June 28, 2013
AIM: To detect the expression of microRNA 130b (miR-130b) in esophageal squamous cell carcinoma (ESCC) and explore its effects on the proliferation and migration of ESCC cells in vitro.
METHODS: MicroRNA (miRNA) microarray was used to select dysregulated miRNAs in ESCC. The expression of miR-130b in ESCC tissue specimens and matched tumor-adjacent tissue specimens was detected by real-time PCR. After the ESCC cell line Eca109 was transfected with miR-130b mimic (miR-130bm) or miR-130b inhibitor (miR-130bi), cell proliferation and migration were determined by cell counting kit-8 (CCK-8) assay and transwell migration assay. The target genes of miR-130b were predicted by bioinformatics and verified by dual luciferase reporter gene assay. The mRNA and protein expression of target genes were detected by real-time PCR and Western blot.
RESULTS: The expression of miR-130b was higher in ESCC than in matched tumor-adjacent tissue (P < 0.01). The expression of miR-130b was effectively enhanced in Eca109 cells transfected with miR-130bm and down-regulated in those transfected with miR-130bi. Transfection with miR-130bm promoted the proliferation and migration of Eca109 cells, and the number of migrating cells was significantly more in miR-130bm-transfected cells than in controls (112.9 ± 2.4 vs 54.3 ± 1.8, P < 0.01). In contrast, transfection with miR-130bi suppressed cell proliferation and migration, and the average number of migrating cells was significantly less in miR-130bi-transfected cells than in controls (33.9 ± 2.3 vs 56.2 ± 1.9, P < 0.01). MiR-130b could interact with 3′-untranslated region of PTEN and down-regulate its expression. MiR-130b could negatively regulate the expression of PTEN protein and promote Akt phosphorylation, but had no effect on the expression of PTEN mRNA.
CONCLUSION: MiR-130b is overexpressed in ESCC. Overexpression of miR-130b promotes the proliferation and migration of Eca109 cells, and inhibition of miR-130b expression suppresses cellular proliferation and migration. MiR-130b can regulate the expression of PTEN at the translational level and promote Akt phosphorylation. MiR-130b may be a potential target for the therapy of ESCC.
- Citation: Yu TT, Li S, Fu MG, Cao RS, Zhu H, Shi RH. Expression of miR-130b in esophageal squamous cell carcinoma and effect of miR-130b transfection on cell proliferation and migration in an esophageal squamous cell carcinoma cell line. Shijie Huaren Xiaohua Zazhi 2013; 21(18): 1685-1692
- URL: https://www.wjgnet.com/1009-3079/full/v21/i18/1685.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i18.1685
食管癌是世界范围内高发的恶性肿瘤, 5年生存率为15%-25%, 在我国组织学类型以鳞癌为主, 加强对食管鳞癌(esophageal squamous cell carcinoma, ESCC)发病机制的研究能更好地指导临床诊断和治疗[1]. 研究发现超过50%的microRNAs(miRNAs)位于肿瘤相关的染色体区域或染色体脆性位点, 可调节30%-60%人类基因的表达, 参与增殖、迁移等多种细胞生物学功能的调控, 在肿瘤的发生发展中具有重要意义[2,3]. miR-130b是新近发现的一种miRNA, 已有相关报道其在子宫内膜癌、肝癌、胃癌等恶性肿瘤中表达上调[4-6]. 目前尚未关于miR-130b在ESCC中的表达及功能的研究报道. 本实验旨在检测ESCC组织中异常表达的miRNAs, 进一步研究miR-130b对ESCC细胞株Eca109生物学功能的影响及对相关基因的调节作用, 以期为ESCC的靶向治疗提供新的理论基础.
收集22例ESCC组织及22例配对癌旁组织(南京医科大学第一附属医院胸外科手术标本, 2011-09/2012-05), 患者均为Ⅰ-Ⅲ期原发性ESCC患者, 且术前未接受放、化疗, 所有组织标本均经病理学检查确认. ESCC细胞株Eca109购自中国科学院细胞库, RPMI 1640培养基、胎牛血清(南京凯基), TaqMan反转录试剂盒、TaqMan定量PCR试剂盒、SYBR Green定量PCR试剂盒、hsa-miR-130b、U6探针(Applied Biosystems), 转染物(上海吉玛, 表1), CCK-8试剂盒(碧云天), Transwell小室(Millipore), PTEN、Akt、pAkt抗体(cell signalling technology), GAPDH抗体(bioworld), 二抗(Santa Cruz), TRIzol、PTEN、GAPDH引物、报告基因质粒(Invitrogen, 表1), 双荧光素酶报告基因检测试剂盒E1910( Promega), 其他试剂(Sigma).
| 名称 | 引物序列 |
| 模拟物对照 | 5'-UUCUCCGAACGUGUCACGUTT-3' |
| 模拟物miR-130bm | 5'-CAGUGCAAUGAUGAAAGGGCAU-3' |
| 抑制物对照 | 5'-CAGUACUUUUGUGUAGUACAA-3' |
| 抑制物miR-130bi | 5'-AUGCCCUUUCAUCAUUGCACUG-3' |
| GAPDH | |
| 上游引物 | 5'-AGCCTCAAGATCATCAGCAATG-3' |
| 下游引物 | 5'-TGTGGTCATGAGTCCTTCCACG-3' |
| PTEN | |
| 上游引物 | 5'-TTTGAAGACCATAACCCACCAC-3' |
| 下游引物 | 5'-ATTACACCAGTTCGTCCCTTTC-3' |
1.2.1 miRNA芯片检测: 用Agilent人miRNA检测芯片(产品编号: G4870A)同时对3例ESCC组织及其配对癌旁组织进行检测, 筛选出ESCC组织中异常表达的miRNAs, 此项工作由北京博奥公司代理完成.
1.2.2 细胞转染: ESCC细胞株Eca109用含10%胎牛血清的RPMI 1640培养液在37 ℃、5%CO2恒温培养箱中培养. 取对数生长期的Eca109细胞接种于6孔板中, 用脂质体LipofectaminTM2000试剂按照说明书分别进行转染: (1)NC组: 模拟物对照; (2)miR-130bm组: 模拟物miR-130b mimics; (3)NCi组: 抑制物对照; (4)miR-130bi组: 抑制物miR-130b inhibitor. 前2组终浓度为100 nmol/L, 后2组为200 nmol/L. 转染后在培养箱中继续孵育5 h, 换成含10%胎牛血清的RPMI 1640培养液继续培养48 h, 收集各组细胞进行相关试验.
1.2.3 实时荧光定量PCR检测: 采用TaqMan MGB探针法定量PCR检测19例ESCC组织及配对癌旁组织和各转染组细胞中miR-130b的表达; 用SYBR Green定量PCR检测各转染组细胞中PTEN mRNA的表达. TRIzol法提取总RNA, 采用TaqMan反转录试剂盒合成cDNA, 反转录反应条件为: 16 ℃, 30 min; 42 ℃, 30 min; 85 ℃, 5 min; 4 ℃, ∞. 以合成的cDNA作为模板, U6(TaqMan)、GAPDH(SYBR Green)作为内参, 进一步进行PCR反应, 扩增条件如下: 50 ℃, 2 min, 95 ℃, 10 min, 1个循环; 95 ℃, 15 s, 60 ℃, 60 s, 40个循环. 结果以2-△△Ct值表示.
1.2.4 CCK-8法检测细胞增殖能力: 将各转染组细胞按3000个/孔接种于96孔细胞培养板中, 每孔100 μL培养液, 分别于培养24、48、72、96 h按10 μL/孔加入CCK-8检测试剂, 在培养箱中继续孵育2 h, 450 nm测定吸光度(A)值, 绘制增殖曲线.
1.2.5 Transwell细胞迁移实验: 用不含血清的培养基饥饿细胞12 h同步化, 各转染组分别取200 μL细胞悬液(含1×105个细胞)加入底膜微孔直径为0.8 μm的Transwell小室底部, 在下室(24孔板底部)加入600 μL含10%胎牛血清的RPMI 1640培养液, 常规培养24 h后, 75%乙醇固定、结晶紫染液染色, 显微镜观察拍照, 随机取5个视野, 计算平均迁移细胞数.
1.2.6 靶基因预测: 用6.2版本TargetScan(http://www.targetscan.org/)和miRDB(http://mirdb.org/miRDB/)两个常用的miRNA靶基因预测数据库搜索miR-130b的靶基因, 并整合分析生物学信息.
1.2.7 Western blot实验: 提取各组细胞总蛋白, 10%SDS-PAGE凝胶电泳, 恒压100 V电泳, 恒流300 mA转膜, 室温封闭, 一抗PTEN(1:1000)、Akt(1:1000)、pAkt(1:1000), 4 ℃孵育过夜, 二抗(1:5000)室温孵育, 曝光、显影.
1.2.8 双荧光素酶报告基因实验: 合成携带PTEN-3'非翻译区(3'UTR)目的片段的质粒pGL3-control-PTEN-3'UTR. 按1.0×104个/孔将Eca109细胞接种于24孔板, 每孔100 μL液体, 生长融合至80%左右, pGL3-control-PTEN-3'UTR质粒和内参pRL-TK质粒(按质量比1000:1), miR-130bm及NC分别共转染细胞, 每组设3个平行孔, 转染24 h后收集细胞, 用双荧光素酶报告基因检测试剂盒检测, 计算各组相对荧光素酶活性.
统计学处理 实验数据使用SPSS17.0进行统计分析, 数据以mean±SD表示, 采用配对t检验比较ESCC组织和癌旁组织中miR-130b表达的差异, 两组指标间比较采用LSD-t检验; P<0.05为有显著性差异.
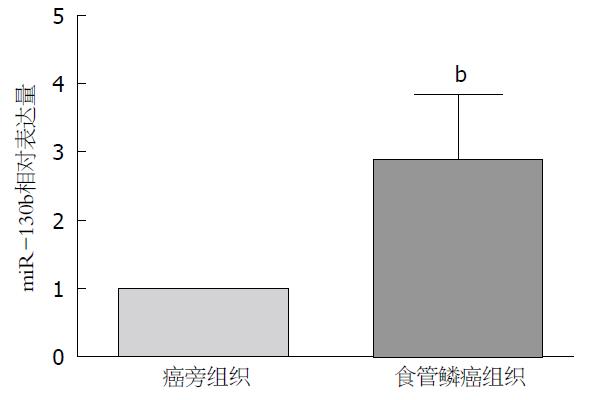
miRNA芯片分析结果以t检验显示有显著统计学差异(P<0.05), 表达量变化>2.0作为筛选条件, 与癌旁组织相比, 共有51个miRNAs在ESCC组织中表达上调, 其中miR-130b上调5.18倍. 定量PCR进一步证实miR-130b在ESCC组织中表达上调, 其相对表达量为2.87±0.97(P<0.01, 图1). miR-130bm组细胞中miR-130b表达量较NC组明显升高; 130bi组较NCi组则明显下降(P<0.01).
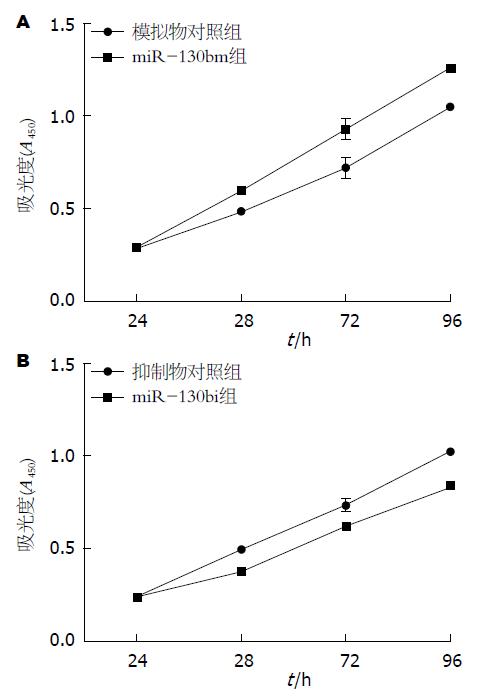
CCK-8检测结果示NC组和miR-130bm组、NCi组和miR-130bi组A值分别在24 h处达同步化, 无统计学差异(表2). 在48、72、96 h各时间点, 转染miR-130b mimics均可促进Eca109细胞的增殖, 而转染miR-130b inhibitor则抑制Eca109细胞的增殖(P<0.05, 表2, 图2).
| 分组 | 24 h | 48 h | 72 h | 96 h |
| 模拟物对照组 | 0.287±0.003 | 0.486±0.004 | 0.723±0.057 | 1.049±0.064 |
| miR-130b模拟物组 | 0.293±0.003 | 0.601±0.008 | 0.932±0.056 | 1.263±0.015 |
| 抑制物对照组 | 0.249±0.009 | 0.499±0.010 | 0.738±0.033 | 1.026±0.010 |
| miR-130b抑制物组 | 0.246±0.005 | 0.383±0.014 | 0.627±0.007 | 0.841±0.023 |
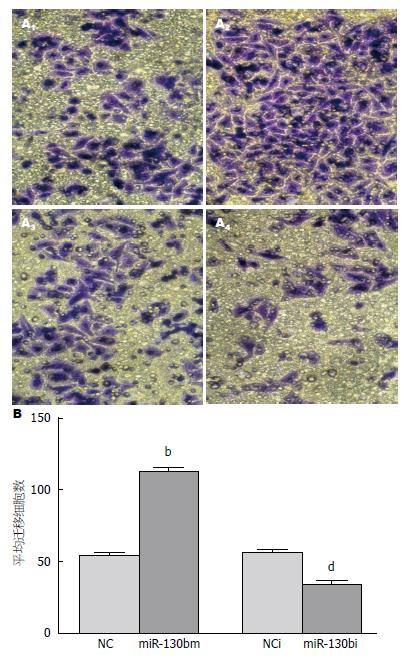
Transwell检测结果示各转染组细胞在24 h后均能穿过滤过膜(图3); miR-130bm组平均迁移细胞数明显多于对照NC组(112.9±2.4 vs 54.3±1.8); miR-130bi组平均迁移细胞数则少于对照NCi组(33.9±2.3 vs 56.2±1.9); 差异均有统计学意义(P<0.01, 图3).
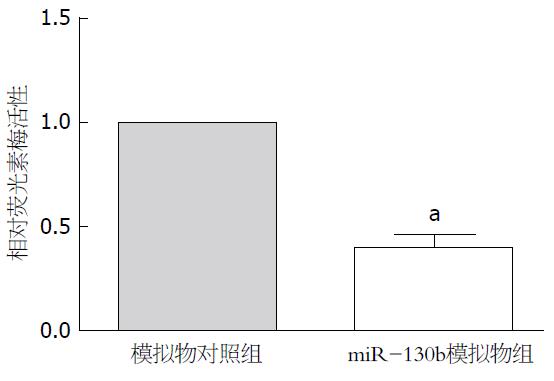
TargetScan和miRDB预测结果均显示PTEN可能为miR-130b靶基因之一, TargetScan提示miR-130b与PTEN mRNA 3'UTR碱基不完全互补配对. 双荧光素酶报告基因检测结果示miR-130bm组的相对荧光素酶活性较对照NC组下降达50%以上(P<0.05, 图4).
Western blot结果示miR-130bm组PTEN蛋白表达明显受抑制, 其相对表达量为0.63±0.01(P<0.05), 而miR-130bi组PTEN蛋白表达量增加, 其相对表达量为1.41±0.08(P<0.05, 图5); SYBR Green定量PCR结果示miR-130bm组和NC组、miR-130bi组和NCi组分别相比, PTEN mRNA表达均无统计学差异.
Western blot结果示转染miR-130b mimics促进Akt磷酸化, 其相对表达量为1.84±0.13(P<0.05), 转染miR-130b inhibitor则抑制Akt磷酸化, 其相对表达量为0.74±0.01(P<0.05, 图5); 而总Akt蛋白表达量变化无统计学差异.
ESCC的发生发展是一个多阶段、多因素相互作用的过程, 可能与原癌基因的激活、抑癌基因的失活、肿瘤转移抑制基因异常等有关[7,8], 但其发病机制仍未完全阐明. miRNA是1993年首次发现的一类非编码内源性小分子RNA[9], 研究表明在多种肿瘤中均存在miRNA的表达异常, 通过形成庞大的调控网络, 在肿瘤的发生发展过程中起着至关重要的调节作用[10,11]. 目前miRNA在ESCC方面的研究开展仍较少[12]. 本研究首先采用miRNA芯片分析技术筛选出在ESCC组织中表达异常的miRNA, 其中表达显著上调的miR-21、miR-223等在ESCC方面已有相关研究报道[13,14], 而表达上调5.18倍的miR-130b在ESCC中的表达及功能未有相关报道.
Wu等[15]报道ESCC组织中33个miRNAs表达上调, 40个表达下调, 与本研究miRNA芯片分析结果存在一定的差异. 由此考虑到miRNA芯片结果可能具有一定的假阳性, 本研究扩大组织样本量, 采用TaqMan实时荧光定量PCR进一步证实miR-130b在ESCC组织中的表达上调. 肿瘤细胞的异常增殖和迁移是肿瘤发生发展过程中的重要环节, 我们通过转染miR-130b mimics和miR-130b inhibitor研究miR-130b在ESCC细胞Eca109中表达升高和降低对细胞增殖和迁移的影响, 结果显示过表达miR-130b促进Eca109细胞的增殖和迁移, 抑制miR-130b的表达使Eca109细胞的增殖和迁移能力减弱, 提示miR-130b在ESCC中具有促癌倾向. 已有研究报道miR-130b在子宫内膜癌、肝癌、胃癌等肿瘤中表达上调, 可促进肿瘤细胞增殖、迁移侵袭和增加对化疗药物的耐药性[3-5]; 而在卵巢癌、甲状腺乳头状癌等肿瘤组织中表达下调, 与肿瘤的侵袭性及对化疗药物的敏感性呈负相关[16,17]. 总之, miR-130b在不同的肿瘤中存在表达差异, 可能与肿瘤的组织特异性有关. 目前普遍认为在肿瘤中表达上调的miRNA通过抑制抑癌基因或调控细胞分化、凋亡的相关基因促进肿瘤生长, 发挥促癌作用, 而低表达的miRNA则抑制促癌基因发挥抑癌作用[18], 对miR-130b靶基因的研究有助于进一步明确其在ESCC中的作用机制. 根据靶基因预测结果, 作为重要抑癌基因之一的人第10号染色体缺失的磷酸酶及张力蛋白同源的基因(phosphatase and tensin homologue, PTEN)引起了我们的注意.
PTEN是迄今为止发现的第一个具有双特异性磷酸酶活性的抑癌基因, 位于染色体10q23区域, 其结构和功能异常存在于人类多种恶性肿瘤中[19-22]. PTEN作为Akt重要的上游因子, 可使磷酸酰肌醇三磷酸(phosphatidyl-inositol 3,4,5 trisphosphate, PIP3)脱磷酸化, 减少PIP3在浆膜上的积聚, 进而抑制Akt家族蛋白的磷酸化, 阻碍下游通路的激活, 最终抑制肿瘤细胞增殖、迁移及血管生成等[23]. Chang等[24]采用免疫组织化学方法检测PTEN蛋白在64例ESCC组织和64例配对正常食管上皮组织中的表达, 结果显示PTEN蛋白在ESCC组织中的表达量显著低于正常食管上皮组织. 而后Zhou等[25]采用腺病毒转染技术, 通过体内外试验证实PTEN在ESCC中可抑制细胞增殖, 促进凋亡, 减缓肿瘤的生长, 发挥抑癌基因的作用. 研究表明PTEN在多种人类恶性肿瘤中均存在基因突变或缺失, 但在ESCC中的发生率较低, 仅表现为蛋白表达的普遍降低[26]. 由此推断, PTEN在ESCC中的表达可能存在其他调控机制. Meng等[27]在肝癌中首次证实miR-21可与PTEN mRNA 3'UTR直接结合, 靶向抑制PTEN促进肝癌细胞的增殖和迁移侵袭. 随后相继有研究发现miR-221/222, miR-22可靶向作用于PTEN激活Akt信号通路发挥促癌作用[28,29]. 近期研究发现miR-216a/217可靶向结合PTEN和SMAD7增加肝癌复发率及对化疗药物的耐药性[30]. 这些研究报道提示在某些特定的恶性肿瘤中, miRNA的表达异常参与了PTEN表达的调控. 因此, 我们选择PTEN作为研究miR-130b在ESCC中作用机制的靶点分子. 由于靶基因预测存在很高的假阳性率, 本研究采用双荧光素酶报告基因实验证实miR-130b可作用于PTEN mRNA的3'UTR调控其表达. 大量研究表明miRNA可与靶基因mRNA 3'UTR序列完全或不完全互补配对结合, 进而促进靶基因的mRNA降解或抑制其蛋白表达, 在转录或转录后水平调控靶基因的表达[31]. 本实验结果表明miR-130b在转录后水平调控PTEN的表达, 并促进Akt磷酸化,这一过程可能是miR-130b促进ESCC细胞增殖、迁移的作用机制之一.
总之, 本研究初步证实了miR-130b在ESCC组织中表达上调, 具有促进ESCC细胞Eca109增殖和迁移的作用; 且可在转录后水平靶向调控PTEN的表达并促进Akt磷酸化. 本实验为ESCC发病机制研究提供了新靶点, 为miR-130b用于ESCC的靶向治疗提供了初步的理论依据. 但miR-130b靶向抑制PTEN, 促进Akt磷酸化后具体激活的信号通路, 及其在ESCC病变进展过程中的作用, 有待进一步研究阐明.
食管鳞癌(esophageal squamous cell carcinoma, ESCC)发病率高, 5年生存率低, 加强其发病机制的研究有助于指导诊断和治疗. miRNAs参与调控细胞各项生物学功能, 在多种肿瘤的发生发展中具有重要的意义.
唐世刚, 教授, 湖南省人民医院
微小RNA-130b(miR-130b)是新近发现的一种miRNA, 已有相关研究报道在子宫内膜癌、肝癌、胃癌等恶性肿瘤中表达上调, 目前尚未有miR-130b在ESCC中的表达及功能的相关研究报道.
Li等研究发现miR-130b在子宫内膜不典型增生组织中表达上调, 在子宫内膜癌组织中上调更明显; 体内外实验证实miR-130b可靶向作用于人类DICER基因(DICER1)促进子宫内膜癌的侵袭.
本研究采用miRNA芯片分析技术筛选出ESCC组织中异常表达的miRNAs, 选取miR-130b进一步研究, 通过上调和下调ESCC细胞中miR-130b的表达探讨其对细胞增殖和侵袭的影响及可能的作用机制.
本实验为ESCC发病机制研究提供了新靶点, 为miR-130b用于ESCC的靶向治疗提供了初步的理论依据.
本研究总体设计合理, 方法可靠, 结论可信, 有一定的实际指导意义.
编辑: 田滢 电编:闫晋利
| 1. | Pennathur A, Gibson MK, Jobe BA, Luketich JD. Oesophageal carcinoma. Lancet. 2013;381:400-412. [PubMed] [DOI] |
| 2. | Song JH, Meltzer SJ. MicroRNAs in pathogenesis, diagnosis, and treatment of gastroesophageal cancers. Gastroenterology. 2012;143:35-47. e2. [PubMed] [DOI] |
| 3. | Bhatti I, Lee A, Lund J, Larvin M. Small RNA: a large contributor to carcinogenesis? J Gastrointest Surg. 2009;13:1379-1388. [PubMed] [DOI] |
| 4. | Li BL, Lu C, Lu W, Yang TT, Qu J, Hong X, Wan XP. miR-130b is an EMT-related microRNA that targets DICER1 for aggression in endometrial cancer. Med Oncol. 2013;30:484. [PubMed] [DOI] |
| 5. | Ma S, Tang KH, Chan YP, Lee TK, Kwan PS, Castilho A, Ng I, Man K, Wong N, To KF. miR-130b Promotes CD133(+) liver tumor-initiating cell growth and self-renewal via tumor protein 53-induced nuclear protein 1. Cell Stem Cell. 2010;7:694-707. [PubMed] [DOI] |
| 6. | Lai KW, Koh KX, Loh M, Tada K, Subramaniam MM, Lim XY, Vaithilingam A, Salto-Tellez M, Iacopetta B, Ito Y. MicroRNA-130b regulates the tumour suppressor RUNX3 in gastric cancer. Eur J Cancer. 2010;46:1456-1463. [PubMed] [DOI] |
| 7. | Radojicic J, Zaravinos A, Spandidos DA. HPV, KRAS mutations, alcohol consumption and tobacco smoking effects on esophageal squamous-cell carcinoma carcinogenesis. Int J Biol Markers. 2012;27:1-12. [PubMed] |
| 8. | Hongo M, Nagasaki Y, Shoji T. Epidemiology of esophageal cancer: Orient to Occident. Effects of chronology, geography and ethnicity. J Gastroenterol Hepatol. 2009;24:729-735. [PubMed] [DOI] |
| 9. | Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;75:843-854. [PubMed] |
| 10. | Esquela-Kerscher A, Slack FJ. Oncomirs - microRNAs with a role in cancer. Nat Rev Cancer. 2006;6:259-269. [PubMed] |
| 11. | Bandyopadhyay S, Mitra R, Maulik U, Zhang MQ. Development of the human cancer microRNA network. Silence. 2010;1:6. [PubMed] [DOI] |
| 12. | Gu J, Wang Y, Wu X. MicroRNA in the pathogenesis and prognosis of esophageal cancer. Curr Pharm Des. 2013;19:1292-1300. [PubMed] |
| 13. | Ma WJ, Lv GD, Tuersun A, Liu Q, Liu H, Zheng ST, Huang CG, Feng JG, Wang X, Lin RY. Role of microRNA-21 and effect on PTEN in Kazakh's esophageal squamous cell carcinoma. Mol Biol Rep. 2011;38:3253-3260. [PubMed] [DOI] |
| 14. | Li S, Li Z, Guo F, Qin X, Liu B, Lei Z, Song Z, Sun L, Zhang HT, You J. miR-223 regulates migration and invasion by targeting Artemin in human esophageal carcinoma. J Biomed Sci. 2011;18:24. [PubMed] [DOI] |
| 15. | Wu BL, Xu LY, Du ZP, Liao LD, Zhang HF, Huang Q, Fang GQ, Li EM. MiRNA profile in esophageal squamous cell carcinoma: downregulation of miR-143 and miR-145. World J Gastroenterol. 2011;17:79-88. [PubMed] [DOI] |
| 16. | Yang C, Cai J, Wang Q, Tang H, Cao J, Wu L, Wang Z. Epigenetic silencing of miR-130b in ovarian cancer promotes the development of multidrug resistance by targeting colony-stimulating factor 1. Gynecol Oncol. 2012;124:325-334. [PubMed] [DOI] |
| 17. | Yip L, Kelly L, Shuai Y, Armstrong MJ, Nikiforov YE, Carty SE, Nikiforova MN. MicroRNA signature distinguishes the degree of aggressiveness of papillary thyroid carcinoma. Ann Surg Oncol. 2011;18:2035-2041. [PubMed] [DOI] |
| 18. | Babashah S, Soleimani M. The oncogenic and tumour suppressive roles of microRNAs in cancer and apoptosis. Eur J Cancer. 2011;47:1127-1137. [PubMed] [DOI] |
| 19. | Song MS, Salmena L, Pandolfi PP. The functions and regulation of the PTEN tumour suppressor. Nat Rev Mol Cell Biol. 2012;13:283-296. [PubMed] [DOI] |
| 20. | Waniczek D, Snietura M, Młynarczyk-Liszka J, Pigłowski W, Kopeć A, Lange D, Rudzki M, Arendt J. PTEN expression profiles in colorectal adenocarcinoma and its precancerous lesions. Pol J Pathol. 2013;64:15-20. [PubMed] |
| 21. | Panagiotou I, Tsiambas E, Lazaris AC, Kavantzas N, Konstantinou M, Kalkandi P, Ragkos V, Metaxas GE, Roukas DK, Vilaras G. PTEN expression in non small cell lung carcinoma based on digitized image analysis. J BUON. 2012;17:719-723. [PubMed] |
| 22. | Squarize CH, Castilho RM, Abrahao AC, Molinolo A, Lingen MW, Gutkind JS. PTEN Deficiency Contributes to the Development and Progression of Head and Neck Cancer. Neoplasia. 2013;15:461-471. [PubMed] |
| 23. | Xu M, Mo YY. The Akt-associated microRNAs. Cell Mol Life Sci. 2012;69:3601-3612. [PubMed] [DOI] |
| 24. | Chang D, Wang TY, Li HC, Wei JC, Song JX. Prognostic significance of PTEN expression in esophageal squamous cell carcinoma from Linzhou City, a high incidence area of northern China. Dis Esophagus. 2007;20:491-496. [PubMed] |
| 25. | Zhou YA, Zhang T, Zhao JB, Wang XP, Jiang T, Gu ZP, Wang XN, Li XF. The adenovirus-mediated transfer of PTEN inhibits the growth of esophageal cancer cells in vitro and in vivo. Biosci Biotechnol Biochem. 2010;74:736-740. [PubMed] |
| 26. | Hou G, Lu Z, Liu M, Liu H, Xue L. Mutational analysis of the PTEN gene and its effects in esophageal squamous cell carcinoma. Dig Dis Sci. 2011;56:1315-1322. [PubMed] [DOI] |
| 27. | Meng F, Henson R, Wehbe-Janek H, Ghoshal K, Jacob ST, Patel T. MicroRNA-21 regulates expression of the PTEN tumor suppressor gene in human hepatocellular cancer. Gastroenterology. 2007;133:647-658. [PubMed] |
| 28. | Garofalo M, Di Leva G, Romano G, Nuovo G, Suh SS, Ngankeu A, Taccioli C, Pichiorri F, Alder H, Secchiero P. miR-221& amp; 222 regulate TRAIL resistance and enhance tumorigenicity through PTEN and TIMP3 downregulation. Cancer Cell. 2009;16:498-509. [PubMed] [DOI] |
| 29. | Bar N, Dikstein R. miR-22 forms a regulatory loop in PTEN/AKT pathway and modulates signaling kinetics. PLoS One. 2010;5:e10859. [PubMed] [DOI] |
| 30. | Xia H, Ooi LL, Hui KM. MiR-216a/217-induced epithelial-mesenchymal transition targets PTEN and SMAD7 to promote drug resistance and recurrence of liver cancer. Hepatology. 2013; Mar 7. [Epub ahead of print]. [PubMed] [DOI] |
| 31. | Filipowicz W, Bhattacharyya SN, Sonenberg N. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight? Nat Rev Genet. 2008;9:102-114. [PubMed] [DOI] |