修回日期: 2013-04-20
接受日期: 2013-05-12
在线出版日期: 2013-06-18
目的: 探讨小干扰RNA(small interfering RNA, siRNA)对人食管癌裸鼠移植瘤细胞凋亡影响.
方法: 使用Smo siRNA转染人食管癌EC9706细胞, 设无关序列组和空白对照组为对照, 将转染好的细胞注射到裸鼠肩胛旁区, 4 wk后取瘤组织. 采用免疫组织化学SP法、Western blot方法检测裸鼠移植瘤组织中Smo、Gli1蛋白的表达; 应用原位杂交、半定量RT-PCR方法检测裸鼠移植瘤组织中Smo、Gli1 mRNA的表达; 运用TUNEL法和透射电镜等方法观察裸鼠移植瘤细胞的凋亡情况.
结果: 转染siRNA后, 免疫组织化学和原位杂交结果显示, 3组组织中siRNA干扰组Smo蛋白(8.37±1.73)及mRNA(2.32±0.63)、Gli1蛋白(3.53±0.37)及mRNA(3.35±0.87)阳性表达细胞数均低于对照组(无关对照组Smo蛋白及mRNA分别为8.42±1.49、9.61±0.85, 空白对照组Smo蛋白及mRNA 8.37±1.73、9.82±0.63; 无关对照组Gli1蛋白及mRNA分别为0.89±0.06、8.41±1.64, 空白对照组Gli1蛋白及mRNA 0.91±0.05、8.53±1.38), 且差异均具有统计学意义(P<0.05); Western blot及RT-PCR结果显示, 与对照组相比(无关对照组Smo蛋白及mRNA分别为9.61±0.85、0.89±0.06; 空白对照组Smo蛋白及mRNA为0.91±0.05、0.96±0.07; 无关对照组Gli1蛋白及mRNA分别为0.87±0.08、0.89±0.07, 空白对照组Gli1蛋白及mRNA分别为0.84±0.06、0.87±0.06; siRNA干扰组中的Smo(0.33±0.06)及mRNA(0.35±0.07)、Gli1(0.29±0.05)及mRNA(0.29±0.05)的表达量明显下调, 组间两两相比差异具有统计学意义(P<0.05); TUNEL结果显示, siRNA干扰组凋亡率(apoptosis rate, AI)明显升高, 组间两两比较AI差异具有统计学意义(P<0.05); 透射电镜检测结果显示, 实验组与对照组相比凋亡细胞数明显增多. 细胞胞质固缩、电子致密度增高, 可见凋亡早期细胞, 染色质固缩并凝结成形态不同的块状, 凋亡晚期细胞可见, 细胞核裂解为碎块状, 产生凋亡小体. 两对照组超微结构无明显差异, 细胞膜完整, 线粒体等细胞器正常, 细胞核基本正常.
结论: Smo siRNA可通过下调裸鼠食管癌细胞中Smo基因的表达, 进而体内诱导裸鼠食管癌移植瘤细胞的凋亡.
核心提示: Hedgehog(HH)信号通路与食管癌密切相关, 设计合成的Smo siRNA可以沉默Smo基因, 抑制HH信号通路, 促进食管癌细胞的凋亡, 为分子靶向阻断HH信号通路治疗食管癌奠定了基础.
引文著录: 孙海斌, 张虎, 丁胭脂, 张红, 陈奎生. Smo siRNA对裸鼠食管癌移植瘤细胞凋亡的影响. 世界华人消化杂志 2013; 21(17): 1579-1588
Revised: April 20, 2013
Accepted: May 12, 2013
Published online: June 18, 2013
AIM: To investigate the inhibitory effect of small interfering RNA (siRNA)-mediated silencing of the Smoothened (Smo) gene on tumor cell apoptosis in a nude mouse model bearing human esophageal carcinoma.
METHODS: An Smo siRNA or a non-relevant siRNA was used to transfect human esophageal carcinoma EC9706 cells. Non-transfected cells were used as a blank control. The transfected cells were injected into the parascapular region of nude mice. After 4 wk, tumor tissue samples were taken to detect the expression of Smo and Gli1 proteins and mRNAs by immunohistochemistry, in situ hybridization, Western blot, semi quantitative RT-PCR, and to determine cell apoptosis by TUNEL assay and transmission electron microscopy.
RESULTS: Immunohistochemistry and in situ hybridization analyses showed that the expression levels of Smo protein (8.37 ± 1.73 vs 8.42 ± 1.49, 8.37 ± 1.73) and mRNA (2.32 ± 0.63 vs 9.61 ± 0.85, 9.82 ± 0.63) and Gli1 protein (3.53 ± 0.37 vs 0.89 ± 0.06, 0.91 ± 0.05) and mRNA (3.35 ± 0.87 vs 8.41 ± 1.64, 8.53 ± 1.38) were significantly lower in the Smo siRNA group than in the non-relevant group and blank control group (all P < 0.05). Western blot and RT-PCR results also revealed that the expression levels of Smo protein (0.33 ± 0.06 vs 9.61 ± 0.85, 0.91± 0.05) and mRNA (0.35 ± 0.07 vs 0.89 ± 0.06, 0.96 ± 0.07) and Gli1 protein (0.29 ± 0.05 vs 0.87 ± 0.08, 0.84 ± 0.06) and mRNA (0.29 ± 0.05 vs 0.89 ± 0.07, 0.87 ± 0.06) were significantly lower in the Smo siRNA group than in the non-relevant group and blank control group (all P < 0.05). TUNEL results showed that the apoptosis rate increased significantly in the Smo siRNA group compared with two control groups (both P < 0.05). Transmission electron microscopy analysis revealed cytoplasmic pyknosis, increased electron density, chromatin condensation and condensation, nuclear fragmentation, and presence of apoptotic bodies in the Smo siRNA group. Two control groups had no such histopathological changes. The number of apoptotic cells increased significantly in the Smo siRNA group compared to control groups.
CONCLUSION: SiRNA-mediated Smo gene silencing induces tumor cell apoptosis in a nude mouse model bearing human esophageal carcinoma.
- Citation: Sun HB, Zhang H, Ding YZ, Zhang H, Chen KS. SiRNA-mediated Smoothened gene silencing induces tumor cell apoptosis in a nude mouse model bearing human esophageal carcinoma. Shijie Huaren Xiaohua Zazhi 2013; 21(17): 1579-1588
- URL: https://www.wjgnet.com/1009-3079/full/v21/i17/1579.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i17.1579
恶性肿瘤细胞与胚胎前体细胞有许多共同的特征, 许多胚胎期起作用的信号通路在肿瘤的形成和转移过程中起重要作用[1]. Hedgehog(HH)信号通路就是其中之一, 该信号通路的异常激活与多种肿瘤, 如基底细胞癌[2,3]、髓母细胞瘤[4]、横纹肌肉瘤[5]、小细胞肺癌[6]、胰腺癌[7]、结肠癌[8]、食管癌[9]等的发病有关. HH信号通路主要包括4个部分: Hedgehog配体(Hh)、Patched受体(Ptc)、Smoothened(Smo)蛋白、核转录因子(Gli)及下游目的基因构成[10-12]. 该通路异常与多种恶性肿瘤的发生发展及肿瘤细胞的凋亡密切相关[13]. 本研究设计、合成特异性Smo小干扰RNA(small interfering RNA, siRNA), 转染食管癌EC9706细胞后构建裸鼠移植瘤, 以探讨Smo基因靶向性siRNA对裸鼠食管癌移植瘤细胞凋亡的影响.
人食管癌EC9706细胞株由中国医学科学院肿瘤研究所分子肿瘤学国家重点实验室惠赠; Balb/c裸鼠购自上海斯莱克实验动物有限公司. RPMI 1640、LipofectamineTM 2000和TRIzol购自美国Invitrogen公司; Smo siRNA购自上海吉玛制药技术有限公司; SP9000免疫组织化学试剂盒购自北京中杉金桥生物技术有限公司; Smo、Gli1 cDNA探针购自上海生工生物技术有限责任公司; cDNA反转录试剂盒、PCR扩增试剂盒购自北京天根生化科技有限公司; 蛋白提取试剂盒购自宝生物工程(大连)有限公司; 人Smo、Gli1和β-actin引物购自上海生工生物技术有限责任公司; BCA蛋白定量试剂盒购自北京康为世纪生物科技有限公司; 兔抗人Smo、Gli多克隆抗体购自美国SAB公司; 兔抗人β-actin多克隆抗体购自北京博奥森生物技术有限公司; TUNEL试剂盒购自瑞士Roche公司.
1.2.1 细胞转染及裸鼠移植瘤的构建: 人食管癌EC9706细胞于含青霉素100 U/mL、链霉素100 U/mL和10%胎牛血清的RPMI 1640培养基中, 在37 ℃、5%CO2的培养箱中贴壁培养. 取对数生长期细胞镜下计数后以2×105/mL接种到六孔板, 待细胞密度达到85%-90%融合时严格按照LipofectamineTM 2000操作说明分别转染3条Smo siRNA, 设24、48和72 h 3个转染时间段, 每个时间段均设150、200和250 ng/µL 3个转染剂量, 将特异性siRNA-1、siRAN-2及siRAN-3分别转染EC9706细胞, 筛选最佳siRNA、最佳浓度、最佳作用时间(预实验结果显示250 ng/µL的siRNA-1处理72 h抑制效应最强, 后续实验采用250 ng/µL的siRNA-1处理EC9706细胞72 h), 同时设立无关序列组和空白对照组.
1.2.2 裸鼠食管癌移植瘤的构建: 将裸鼠随机分为3组, 分别为siRNA干扰组、无关序列组和空白对照组, 每组5只, 为区别动物, 采用剪耳缘法对其进行标记, 在相同的条件下对3组动物进行分笼饲养. 为保证动物健康, 木屑、标准食物及饮用水均采用高压灭菌、隔天更换. 待体外转染完成后, 将裸鼠背部肩胛旁区皮下选为注射部位, 对其进行接种, 注射剂量为1×107/mL, 0.2 mL/只.
1.2.3 免疫组织化学: (1)免疫组织化学步骤: 免疫组织化学染色步骤严格按照说明书进行, DAB底物工作液显色, 苏木素复染. 以PBS代替一抗作阴性对照; (2)结果判定以阳性细胞百分比结合染色强度统计积分. 细胞质染成棕黄色或棕褐色为阳性, 随机观察10个高倍视野, 每个视野记数200个细胞, 计算阳性细胞数.
1.2.4 原位杂交: (1)原位杂交步骤: 原位杂交步骤严格按照说明书进行, BCIP/NBT染色, 核固红复染. 采用0.1 mg/mL RNase, 37 ℃预处理组织切片60 min及用无探针的杂交液孵育组织切片作阴性对照; (2)结果判定[14]: 以阳性细胞百分比结合染色强度统计积分. 细胞质染成紫蓝色为阳性, 随机观察10个高倍视野, 每个视野记数200个细胞, 计算阳性细胞数.
1.2.5 RT-PCR: 取新鲜肿瘤组织, 按TRIzol操作说明, 提取各组总RNA. 逆转录反应合成cDNA第一链后以β-actin为内参照进行PCR反应, Smo扩增产物为306 bp, 上游引物为: 5'- CGCTACCCTGCTGTTATTCTCT-3', 下游引物为: 5'-CAG-GTGGAAGTAGGAGGTCTTG-3'; Gli1扩增产物为201 bp, 上游引物为: 5'-TTGGAGAAGCCGAGCCGAGTATC-3', 下游引物为: 5'-GAGTAGACAGAGGTTGGGAGGTAAGG-3'; β-actin扩增产物为480 bp, 上游引物为: 5'-CATCCTGCGTCTGGACCT-3', 下游引物为: 5'-TCAGGAGGAGCAATGATCTTG-3'(引物序列设计合成均由上海生工完成). 所有操作严格按照RT-PCR试剂盒说明书进行, PCR反应条件: (Smo)94 ℃预变性5 min, 94 ℃变性30 s, 59 ℃退火45 s, 72 ℃延伸60 s, 35个循环; 最后72 ℃延伸5 min至4 ℃; (Gli1)94 ℃预变性5 min, 94 ℃变性30 s, 58 ℃退火45 s, 72 ℃延伸60 s, 35个循环, 最后72 ℃延伸5 min至4 ℃. 取10 μL产物1%琼脂糖凝胶电泳, 用Alpha View软件进行图像分析, 以Smo mRNA与β-actin电泳条带积分光密度值之比表示Smo mRNA的相对表达量.
1.2.6 Western blot: 取新鲜组织适量剪碎, 加入预冷的裂解液匀浆, 加入抽提试剂, 低温离心30 min, 上清即总蛋白. 用BCA法测定蛋白质浓度后, 取50 μg蛋白上样, 通过12%分离胶后转至PVDF膜上, 脱脂奶粉封闭; 加入兔抗人Smo抗体(1:500), 4 ℃过夜; 用TBS洗膜10 min×3次, 加入羊抗兔二抗, 室温微摇2 h, TBS 洗膜10 min×3次; 曝光后照相, 用Alpha View软件进行图像分析, 以目的条带与β-actin积分光密度值之比表示目的蛋白的相对表达量.
统计学处理 应用SPSS17.0统计软件进行统计学处理, 计量资料采用mean±SD表示; 两组均数的比较用t检验; 多组均数的比较用方差分析; 方差不齐时先进行变量转换. 显著性水准a = 0.05.
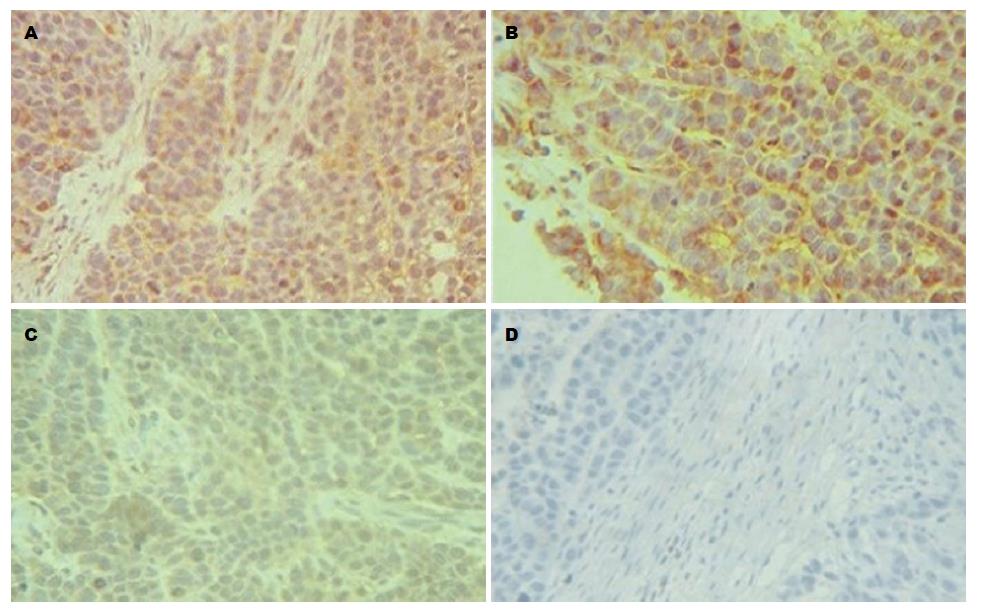
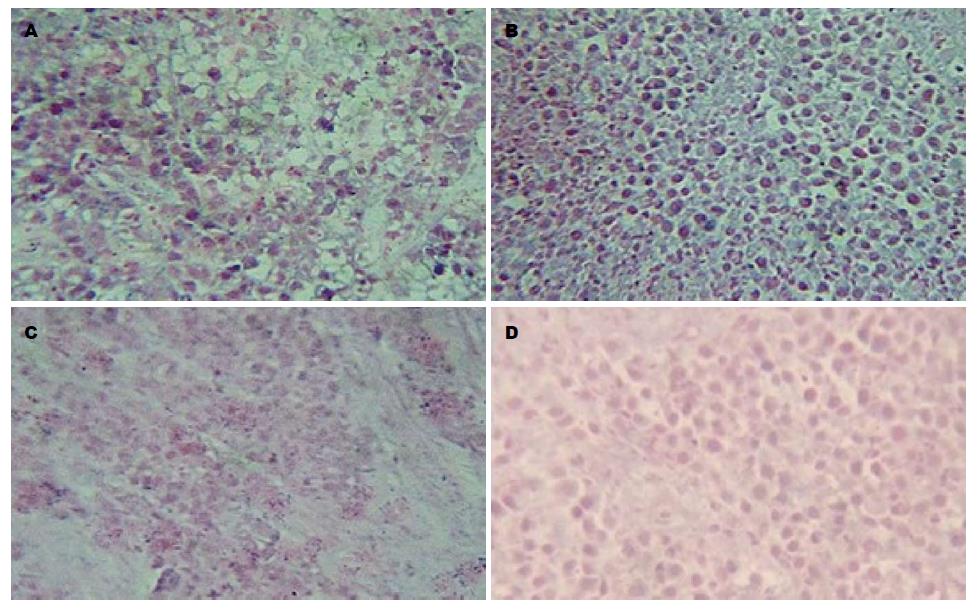
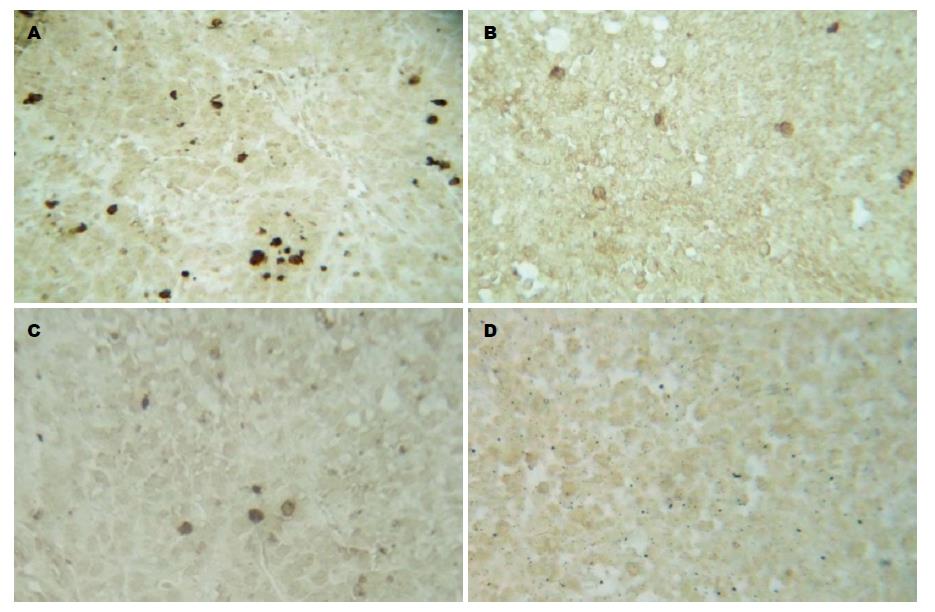
2.1.1 免疫组织化学检测各组移植瘤组织内Smo蛋白的表达: 免疫组织化学结果显示, Smo蛋白阳性表达信号主要定位于细胞质中, 呈浅黄色或棕黄色颗粒. SiRNA干扰组、空白对照组、无关序列组Smo蛋白表达分别为: 2.63±0.57、8.42±1.49和8.37±1.73, 实验组与对照组两两比较Smo蛋白的表达差异有统计学意义(P<0.05), 无关序列组与空白对照组比较差异无统计学意义(P>0.05)(表1, 图1).
| 分组 | Smo蛋白表达 | F 值 | P值 |
| siRNA干扰组 | 2.63±0.57 | ||
| 无关序列组 | 8.42±1.49 | 715.28 | 0.000 |
| 空白对照组 | 8.37±1.73 |
2.1.2 免疫组织化学检测各组移植瘤组织中Gli1蛋白的表达: 免疫组织化学结果显示, Gli1蛋白阳性表达信号主要定位于细胞核中, 胞质次之, 呈浅黄色或棕黄色颗粒. SiRNA干扰组与对照组两两比较, Gli1蛋白的表达差异有统计学意义(P<0.05), 无关序列组与空白对照组比较差异无统计学意义(P>0.05)(表2, 图2).
| 分组 | Gli1蛋白表达 | F 值 | P值 |
| siRNA干扰组 | 2.32±0.63 | ||
| 无关序列组 | 7.37±1.57 | 713.52 | 0.000 |
| 空白对照组 | 7.29±1.49 |
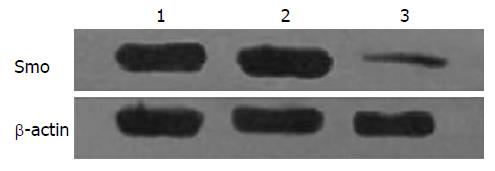
2.2.1 Western blot检测移植瘤组织中Smo蛋白的表达: 实验组与对照组相比, Smo蛋白表达量明显下降差异有统计学意义(P<0.05), 两对照组Smo蛋白表达量差异无统计学意义(P>0.05)(表3, 图3).
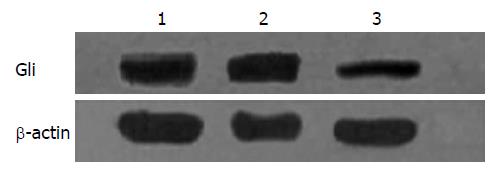
2.2.2 Western blot检测移植瘤组织中Gli1蛋白的表达: 实验组与对照组相比Gli1蛋白表达量明显下降, 差异有统计学意义(P<0.05), 两对照组Gli1蛋白表达量差异无统计学意义(P>0.05)(表4, 图4).
2.3.1 原位杂交检测移植瘤组织中Smo mRNA的表达: Smo mRNA阳性表达信号主要定位于细胞质中, 呈紫蓝色颗粒. siRNA干扰组、空白对照组、无关序列组Smo mRNA表达分别为: 3.53±0.37、9.61±0.85和9.82±0.63. 实验组与对照组两两比较Smo mRNA表达差异有统计学意义(P<0.05), 无关序列组与空白对照组比较差异无统计学意义(P>0.05)(表5, 图5).
| 分组 | Smo mRNA表达 | F 值 | P值 |
| siRNA干扰组 | 3.53±0.37 | ||
| 无关序列组 | 9.61±0.85 | 736.20 | 0.000 |
| 空白对照组 | 9.82±0.63 |
2.3.2 原位杂交检测移植瘤组织中Gli1 mRNA的表达: Gli1 mRNA阳性表达信号主要定位于细胞质中, 呈紫蓝色颗粒. 实验组与对照组两两比较Smo mRNA表达差异有统计学意义(P<0.05), 无关序列组与空白对照组比较差异无统计学意义(P>0.05)(表6, 图6).
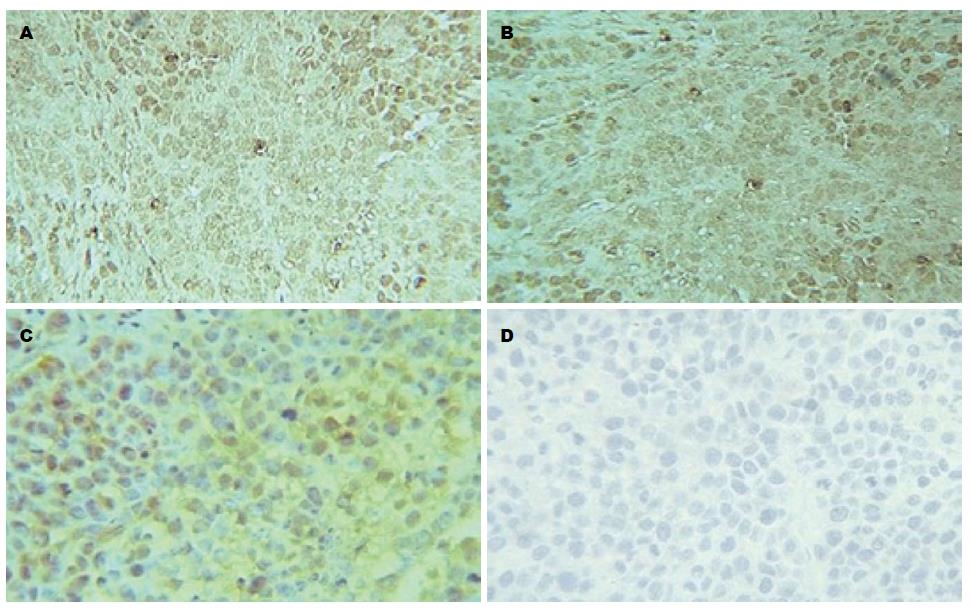
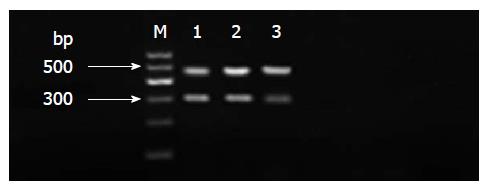
2.4.1 RT-PCR检测移植瘤组织中Smo mRNA的表达: Smo和β-actin扩增产物经电泳后, 结果显示扩增片段大小与设计的大小完全一致, 分别为306和480 bp. 与空白对照组及无关序列组相比, siRNA干扰组中的Smo mRNA表达量明显下降差异有统计学意义(P<0.05), 两对照组中Smo mRNA差异无统计学意义(P>0.05)(表7, 图7).
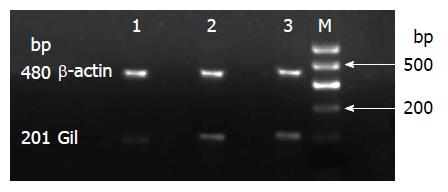
2.4.2 RT-PCR检测移植瘤组织中Gli1 mRNA的表达: Gli1和β-actin扩增产物经电泳后, 结果显示扩增片段大小与设计的大小完全一致, 分别为201和480 bp. 与空白对照组及无关序列组相比, siRNA干扰组中的Gli mRNA表达量明显下降, 差异有统计学意义(P<0.05), 两对照组Gli1 mRNA表达差异无统计学意义(P>0.05)(表8, 图8).
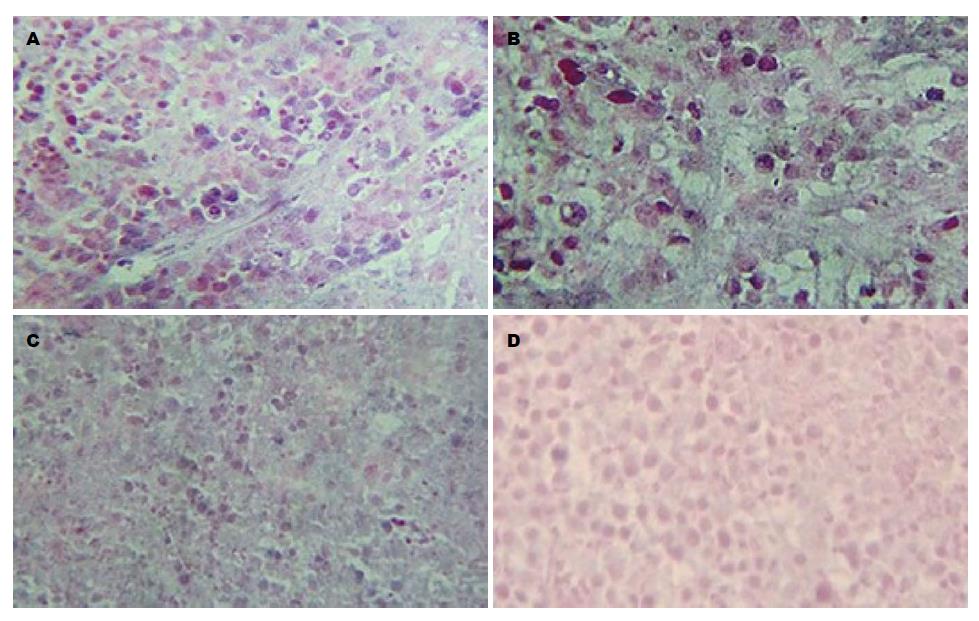
实验组与对照组相比凋亡细胞数增多, 实验组与各对照组AI差异有统计学意义(均P<0.05); 空白对照组与无关序列组AI差异无统计学意义(均P>0.05)(表9, 图9).
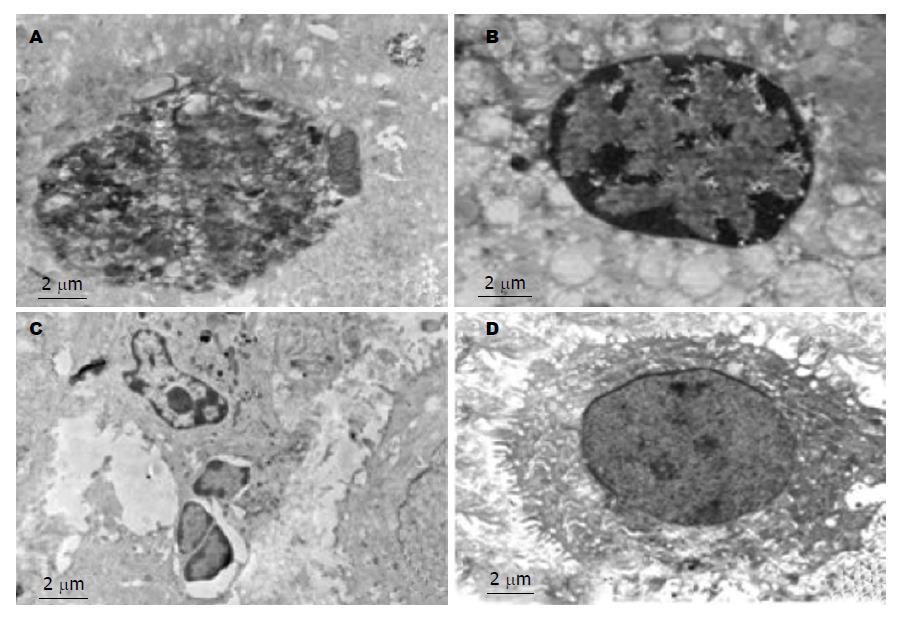
透射电镜下实验组凋亡细胞数明显增加. 凋亡细胞微绒毛减少、胞质固缩、细胞体积变小、胞质电子致密度增高, 可见凋亡早期细胞, 其表现为染色质固缩并凝结成形态不同的块状, 聚集于核内不同部位; 内质网疏松化并与胞膜融合形成一个个空泡; 凋亡晚期细胞可见, 细胞核裂解为碎块状, 产生数量不等体积不同的凋亡小体. 图10A可见大量空泡及凋亡小体形成; 图10B可见细胞表面的微绒毛结构明显减少并可见到线粒体; 图10C典型的凋亡小体形成; 图10D未发生凋亡的移植瘤细胞, 细胞表面有大量微绒毛结构.
HH信号通路在哺乳动物胚胎发育、分化及肿瘤形成中发挥着重要的作用[15,16]. 至今为止在哺乳动物中至少发现3种Hh同源基因配体: Sonic Hedgehog(SHH)、Indian Hedgehog(IHH)和Desert Hedgehog(DHH), 分别编码3种相应的蛋白: SHH、IHH和DHHl[17,18]. Hedgehog信号通路中的主要成员有Hh配体、Ptch受体、Smo蛋白和Gli转录因子. 此通路的转导顺序是, Hh配体结合跨膜受体Patched(Ptch)后, 释放Ptch介导抑制的Smo受体[19,20]. Smo阻遏解除使GLI锌指转录因子激活并转入核内, 促进Hh靶基因的转录[21,22]. 恶性肿瘤中Hh信号通路的异常激活有两种方式: 基因突变(非配体依赖性)或配体高表达(配体依赖性)[23,24]. 非配体依赖性主要是指Ptch受体功能缺失性突变和Smo蛋白功能获得性突变; 配体依赖性是指Hh配体的过表达. 在Hedgehog信号通路中作为信号转换蛋白的Smo是一种原癌基因[25,26], 在整个信号转导通路中起到"枢纽"的作用, 当其发生功能获得性突变(非配体依赖性激活)或Hedgehog解除了Ptch对其的抑制作用(配体依赖性激活)时, 会引起此信号通路的激活[18]. 因此本课题选用Smo作为靶基因, 采用siRNA技术对裸鼠食管癌移植瘤细胞凋亡进行研究.
Ma等[27]报道在22例食管癌病例中有24例存在Shh和Ptch受体的高表达. Litingtung等[28]通过将小鼠的Shh基因剔除后, 发现小鼠食管闭锁、狭窄且分化差, 这些结果提示Shh配体在食管的生长分化中是不可缺少的. 本实验室近期研究结果显示[29], Smo、Gli蛋白及mRNA在食管鳞癌细胞中高表达; Smo高表达与食管鳞癌的浸润深度、淋巴结转移密切相关; Smo siRNA可抑制食管鳞癌的浸润和转移. 可见Smo与食管癌的发生发展有关. 这与孙波等[30]的研究是一致的.
本文应用Western blot、免疫组织化学、原位杂交及RT-PCR检测Smo蛋白及其mRNA, 结果发现裸鼠食管癌移植瘤组织中Smo mRNA高表达, 该结果提示食管癌EC9706细胞的发生与Smo基因的高表达有关; 应用RNAi原理降解Smo mRNA的表达结果显示Smo siRNA能有效降低EC9706细胞内的Smo mRNA降解效果达到65%, 达到下调EC9706细胞Smo基因表达的预期. TUNEL结果显示, Smo mRNA被降解后EC9706细胞的凋亡率增加, 结果显示实验组与对照组相比凋亡率显著升高差异具有统计学意义(P<0.05), 结果提示EC9706细胞内Smo基因表达的下调可诱导EC9706细胞的凋亡; Smo基因的高表达与EC9706细胞的凋亡异常有关; 透射电镜观察结果显示, siRNA干扰组细胞微绒毛减少、胞质固缩、细胞体积变小、胞质电子致密度增高, 可见凋亡早期细胞, 其表现为染色质固缩并凝结成形态不同的块状, 聚集于核内不同部位; 内质网疏松化并与胞膜融合形成一个个空泡; 凋亡晚期细胞可见, 细胞核裂解为碎块状, 产生数量不等体积不同的凋亡小体. 两对照组超微结构无明显差异, 细胞膜完整, 线粒体等细胞器正常, 细胞核正常, 与实验组相比, 实验组凋亡率明显增高. 结果提示, 下调Smo基因的表达可诱导EC9706细胞的凋亡.
Hedgehog(HH)信号通路是在胚胎期起作用的信号通路之一, 该信号通路的异常激活与多种恶性肿瘤的发生发展密切相关. Smoothened(Smo)是该通路重要组成部分, 研究表明, Smo在食管癌等恶性肿瘤中高表达, 且与肿瘤的浸润转移密切相关, 可见Smo是肿瘤靶向治疗的有效靶点.
李苏宜, 教授, 主任医师, 东南大学附属中大医院肿瘤科
靶向阻断HH信号通路治疗肿瘤成为当前研究的热点. Smo基因在该通道中起着"枢纽"作用, 所以沉默Smo的表达, 抑制HH信号通路成为研究的重点. 寻求沉默Smo的有效抑制剂或分子生物学技术是当前亟待解决的问题.
Li等剔除小鼠的Shh基因, 发现Shh配体在食管发生及演变过程中起着重要作用; 特别是Ma等研究发现食管癌组织中Shh和Ptch受体均高表达. 国内孙波等的研究结果表明Smo与食管癌的发生及浸润转移密切相关.
本研究利用RNA干扰技术沉默Smo基因, 体外研究Smo基因与食管癌的关系, 并用电镜观察肿瘤细胞凋亡情况.
RNA干扰技术具有高效、高特异性及廉价等特点, 其应用前景广阔. 利用RNA干扰技术沉默Smo基因, 抑制HH信号通路治疗食管癌是可行的.
本文从多角度多层次体外探讨HH信号通路中Smo、Gli1基因的异常表达对食管癌裸鼠模型荷载肿瘤病灶凋亡的影响, 具有一定的创新性, 设计严谨, 逻辑性强.
编辑: 田滢 电编:鲁亚静
| 1. | Bailey JM, Singh PK, Hollingsworth MA. Cancer metastasis facilitated by developmental pathways: Sonic hedgehog, Notch, and bone morphogenic proteins. J Cell Biochem. 2007;102:829-839. [PubMed] [DOI] |
| 2. | Sekulic A, Mangold AR, Northfelt DW, LoRusso PM. Advanced basal cell carcinoma of the skin: targeting the hedgehog pathway. Curr Opin Oncol. 2013;25:218-223. [PubMed] |
| 3. | Lupi O. Correlations between the Sonic Hedgehog pathway and basal cell carcinoma. Int J Dermatol. 2007;46:1113-1117. [PubMed] [DOI] |
| 4. | Archer TC, Weeraratne SD, Pomeroy SL. Hedgehog-GLI pathway in medulloblastoma. J Clin Oncol. 2012;30:2154-2156. [PubMed] [DOI] |
| 5. | Li F, Shi W, Capurro M, Filmus J. Glypican-5 stimulates rhabdomyosarcoma cell proliferation by activating Hedgehog signaling. J Cell Biol. 2011;192:691-704. [PubMed] [DOI] |
| 6. | Park KS, Martelotto LG, Peifer M, Sos ML, Karnezis AN, Mahjoub MR, Bernard K, Conklin JF, Szczepny A, Yuan J. A crucial requirement for Hedgehog signaling in small cell lung cancer. Nat Med. 2011;17:1504-1508. [PubMed] [DOI] |
| 7. | Hao K, Tian XD, Qin CF, Xie XH, Yang YM. Hedgehog signaling pathway regulates human pancreatic cancer cell proliferation and metastasis. Oncol Rep. 2013;29:1124-1132. [PubMed] |
| 9. | Mori Y, Okumura T, Tsunoda S, Sakai Y, Shimada Y. Gli-1 expression is associated with lymph node metastasis and tumor progression in esophageal squamous cell carcinoma. Oncology. 2006;70:378-389. [PubMed] [DOI] |
| 10. | Milla LA, González-Ramírez CN, Palma V. Sonic Hedgehog in cancer stem cells: a novel link with autophagy. Biol Res. 2012;45:223-230. [PubMed] [DOI] |
| 11. | Robbins DJ, Fei DL, Riobo NA. The Hedgehog signal transduction network. Sci Signal. 2012;5:re6. [PubMed] [DOI] |
| 12. | Kubota K, Yamauchi N, Matsumoto K, Watanabe R, Oozono S, Aramaki S, Wood C, Soh T, Hattori MA. Expression of hedgehog family genes in the rat uterus during early pregnancy. J Reprod Dev. 2008;54:340-345. [PubMed] [DOI] |
| 13. | Park MY, Park YS, Nam JH. RNA interference against granulin-epithelin precursor prevents hepatocellular carcinoma growth: its application as a therapeutic agent. Int J Oncol. 2011;39:853-861. [PubMed] |
| 14. | 张 威, 陈 玉, 亚 国伟, 陈 奎生. RNA干扰对EC9706细胞mTOR表达及细胞生长与凋亡的影响. 郑州大学学报(医学版). 2010;45:11-15. |
| 15. | Medina V, Calvo MB, Díaz-Prado S, Espada J. Hedgehog signalling as a target in cancer stem cells. Clin Transl Oncol. 2009;11:199-207. [PubMed] [DOI] |
| 16. | Jiang J, Hui CC. Hedgehog signaling in development and cancer. Dev Cell. 2008;15:801-812. [PubMed] [DOI] |
| 17. | Zarogoulidis P, Zarampouka K, Huang H, Darwiche K, Huang Y, Sakkas A, Zarogoulidis K. Hedgehog signaling pathway: the must, the maybe and the unknown. J Thorac Dis. 2013;5:195-197. [PubMed] |
| 18. | Choy SW, Cheng SH. Hedgehog signaling. Vitam Horm. 2012;88:1-23. [PubMed] [DOI] |
| 19. | Coni S, Infante P, Gulino A. Control of stem cells and cancer stem cells by Hedgehog signaling: pharmacologic clues from pathway dissection. Biochem Pharmacol. 2013;85:623-628. [PubMed] [DOI] |
| 20. | Katayama S, Ashizawa K, Gohma H, Fukuhara T, Narumi K, Tsuzuki Y, Tatemoto H, Nakada T, Nagai K. The expression of Hedgehog genes (Ihh, Dhh) and Hedgehog target genes (Ptc1, Gli1, Coup-TfII) is affected by estrogenic stimuli in the uterus of immature female rats. Toxicol Appl Pharmacol. 2006;217:375-383. [PubMed] [DOI] |
| 21. | Hynes M, Stone DM, Dowd M, Pitts-Meek S, Goddard A, Gurney A, Rosenthal A. Control of cell pattern in the neural tube by the zinc finger transcription factor and oncogene Gli-1. Neuron. 1997;19:15-26. [PubMed] [DOI] |
| 22. | Li SH, Fu J, Watkins DN, Srivastava RK, Shankar S. Sulforaphane regulates self-renewal of pancreatic cancer stem cells through the modulation of Sonic hedgehog-GLI pathway. Mol Cell Biochem. 2013;373:217-227. [PubMed] [DOI] |
| 23. | Szkandera J, Kiesslich T, Haybaeck J, Gerger A, Pichler M. Hedgehog signaling pathway in ovarian cancer. Int J Mol Sci. 2013;14:1179-1196. [PubMed] [DOI] |
| 24. | Che L, Yuan YH, Jia J, Ren J. Activation of sonic hedgehog signaling pathway is an independent potential prognosis predictor in human hepatocellular carcinoma patients. Chin J Cancer Res. 2012;24:323-331. [PubMed] [DOI] |
| 25. | Ruiz i Altaba A, Sánchez P, Dahmane N. Gli and hedgehog in cancer: tumours, embryos and stem cells. Nat Rev Cancer. 2002;2:361-372. [PubMed] [DOI] |
| 26. | Kim PG, Albacker CE, Lu YF, Jang IH, Lim Y, Heffner GC, Arora N, Bowman TV, Lin MI, Lensch MW. Signaling axis involving Hedgehog, Notch, and Scl promotes the embryonic endothelial-to-hematopoietic transition. Proc Natl Acad Sci U S A. 2013;110:E141-E150. [PubMed] [DOI] |
| 27. | Ma X, Sheng T, Zhang Y, Zhang X, He J, Huang S, Chen K, Sultz J, Adegboyega PA, Zhang H. Hedgehog signaling is activated in subsets of esophageal cancers. Int J Cancer. 2006;118:139-148. [PubMed] [DOI] |