修回日期: 2013-03-10
接受日期: 2013-04-15
在线出版日期: 2013-05-08
目的: 分析消化不良患者的正常胃黏膜菌群特征.
方法: 收集2例正常胃黏膜病理学消化不良患者的胃窦黏膜, 提取黏膜组织基因组DNA,应用PCR方法扩增16S rRNA, 进行二代454焦磷酸测序. 测序数据应用操作单元(operational taxonomic units, OTU)聚类分析、多样性分析和分类学分析等生物信息技术分析胃黏膜菌群的结构、多样性和丰度.
结果: 患者A和B胃黏膜菌群的高通量测序分析分别得到20565条和17487条优化序列, 测序覆盖深度>0.98. 经过97%相似度归并后分别得到638个OTUs和667个OTUs; 菌群多样性分析显示正常胃黏膜菌群的chao指数和shannon指数均低于粪便菌群. 患者A的胃黏膜菌群由58个属的细菌构成, 归属于14个门, 以变形菌门为主(99.49%), 其次为放线菌门、硬壁菌门和拟杆菌门等. 患者B胃黏膜菌群构成与患者A相似, 由64个属的细菌构成, 归属于19个门. 粘质沙雷(氏)菌为正常胃黏膜菌群的优势主导菌.
结论: 正常胃黏膜菌群多样性丰富, 以变形菌门为主, 沙雷(氏)菌为优势主导菌.
核心提示: 本研究运用高通量测序技术和生物信息学分析方法, 对两例胃黏膜组织学正常个体的胃菌群的结构、丰度、组成进行了分析. 结果发现正常胃黏膜菌群的丰度低于肠道菌群, 但具有高度的多样性. 胃菌群的组成以变形菌门为主, 沙雷(氏)菌属为优势主导菌, 这为研究胃黏膜菌群在慢性胃病发生中的作用奠定了基础.
引文著录: 王莉莉, 董开芯, 周建华, 于新娟, 贾盛佼, 董全江. 正常胃黏膜菌群特征分析2例. 世界华人消化杂志 2013; 21(13): 1250-1255
Revised: March 10, 2013
Accepted: April 15, 2013
Published online: May 8, 2013
AIM: To characterize the normal gastric microbiota.
METHODS: Genomic DNA was extracted from antral biopsies obtained from two patients who had histologically normal gastric mucosa. The 16S rRNA gene was amplified using PCR. The barcoded 454-pyrosequencing method was used to analyze the amplified products. To investigate the community structure, diversity and abundance of gastric microbiota, sequence analysis was performed using bioinformatic methods including OTU hierarchical clustering and alpha diversity analysis.
RESULTS: High-throughout sequencing of the gastric microbiota from patients A and B obtained 20565 and 17487 sequences, respectively. The coverage was greater than 0.98. Gastric microbiota was composed of 638 and 667 OTUs, respectively. Alpha diversity analysis revealed that Chao and Shannon indices of gastric microbiota were slightly lower than those of fecal microbiota. For patient A, the gastric microbiota was composed of 58 bacterial genera belonging to 14 phyla. Proteobacteria, Actinomycetes, Firmicutes and Bacteroides were dominant in the gastric microbiota from patient A. The gastric microbiota from patient B had a similar bacterial composition, consisting of 64 bacterial genera belonging to 19 phyla. Serratia was the predominant genus in both patients.
CONCLUSION: Normal gastric microbiota shows a high diversity. Proteobacteria is the most abundant phylum, while Serratia is the predominant genus.
- Citation: Wang LL, Dong KX, Zhou JH, Yu XJ, Jia SJ, Dong QJ. 454-pyrosequencing analysis of normal gastric mucosal microbiota. Shijie Huaren Xiaohua Zazhi 2013; 21(13): 1250-1255
- URL: https://www.wjgnet.com/1009-3079/full/v21/i13/1250.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i13.1250
人胃肠道内定居着大量的细菌, 他们构成维持人体正常生理功能所必需的胃肠道菌群. 目前, 研究[1]发现健康人群胃肠道菌群的细菌种类超过1000种, 数量约为1014. 胃肠道菌群能够促进人体肠道发育、营养吸收和病原体抵抗等, 并且可以直接参与调节小肠上皮细胞增殖、脂肪储存和炎症应答等机制[2,3]. 健康状态下, 胃肠道菌群与人体和胃肠道微环境保持着一个平衡状态. 当机体受到某些内因或外因的作用如不合理饮食、感染、应激等, 引起胃肠道菌群的结构、数量、比例、定位和代谢特征的变化等, 从而导致疾病的发生[4]. 人体胃肠道微生物群落是一个复杂的系统, 传统的培养法无法提供全面的信息来探究其群落结构的组成[5]. 本研究利用第二代454焦磷酸测序技术, 通过提取2例胃黏膜病理学检查正常的消化不良患者的胃黏膜菌群基因组DNA并扩增其16S rRNA, 进行高通量测序和生物信息学分析检测正常胃黏膜菌群的结构组成、多样性和丰度, 探讨生理状态下胃黏膜菌群的结构和优势主导菌, 为研究胃黏膜菌群与慢性胃部疾病的相关性提供理论依据.
胃窦黏膜活检标本取自青岛市市立医院胃镜室行胃镜检查的2例消化不良患者: (1)有消化不良的上消化道症状; (2)病理检测胃窦黏膜无异常发现. 患者A, 男, 86岁; 患者B, 男, 37岁. 检查前1 mo内均未服用任何抗生素. 标本分别编号为A、B, 放入脑心浸出液(BHI)培养基中, -80 ℃保存备用.
1.2.1 胃黏膜菌群的基因组DNA提取: 采用E.Z.N.A DNA试剂盒(美国OMEGA公司)抽提胃黏膜菌群的基因组DNA, 步骤按说明书进行. 1%琼脂糖凝胶电泳检测抽提的基因组DNA完整性.
1.2.2 PCR扩增: 根据合成区域, 合成带有"5' 454 A、B接头-特异引物3'"的融合引物, 细菌16S rRNA引物序列为: 8F: 5'-AGAGT TTGATCCTGGCTCAG-3'; 533R: 5'-TTACCGCGGCTGCTGGCAC-3'(测序端). PCR反应体系为: 5 × FastPfu 缓冲液4 μL, 正向引物5 μmol/L, 反向引物5 μmol/L, DNA 20 ng, dNTP 2.5 mmol/L, FastPfu 多聚酶0.4 μL, 加ddH2O至总体积20 μL. PCR 反应条件为: 95 ℃ 2 min; 25个循环包括95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 30 s; 72 ℃ 5 min. 2%琼脂糖凝胶电泳检测PCR产物.
1.2.3 高通量测序: PCR产物定量均一化后采用454焦磷酸高通量测序仪(瑞士Roche公司)进行高通量测序.
1.2.4 生物信息学分析[6,7]: (1)优化序列数据统计: 去除序列末端的后引物和接头序列、多碱基N、polyA/T尾巴及低质量碱基; 去除所得序列的barcode标签序列、前引物序列; 丢弃长度短于200 bp、模糊碱基数>0、序列平均质量低于25的序列; (2)操作单元(operational taxonomic units, OTU)聚类分析: 提取非重复序列, 与silva数据库[8]中已比对的(16S/18S, SSU)核糖体序列数据库进行比对, 去杂后生成分类操作单元(operational taxonomic units, OTU); (3)菌群多样性分析: 根据OTU聚类分析结果, 采用ace, chao, simpson, shannon, 评估指数分别对样本进行多样性分析[9]. 其中ace, chao指数是对菌群丰度进行评估; simpson, shannon指数对菌群多样性进行评估; (4)稀释性曲线分析: 采用mothur软件[10]在97%相似性水平绘制标本稀释性曲线, 比较测序数量不同的样本物种的丰富度及样本的取样大小是否合理; (5)菌群分类学分析: 采用mothur软件将OTU中全部序列与silva数据库进行比对, 找出其最相近且可信度达80%以上的种属信息. 并将每一个OTU中的所有序列进行类比, 找出同一OTU中的不同序列的最近祖先的种属信息. 根据silva库中的参考序列对OTU进行种属鉴定; (6)菌群群落结构分析: 根据分类学分析比对结果, 在属水平上对样品中群落结构进行菌群种类和丰度分析.
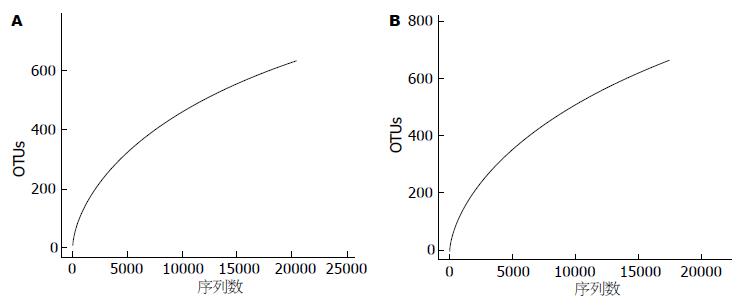
运用PCR方法扩增细菌16S rRNA基因进行高通量测序, 患者A的胃黏膜菌群共获得23810条有效序列, 优化序列为20565条, 测序覆盖深度(coverage指数)为0.99, 经过97% 相似度归并后得到638个OTUs; 患者B的胃黏膜菌群得到20245条有效序列, 经优化为17487条序列, coverage指数为0.98, 经过97% 相似度归并后得到667个OTUs(表1). 稀释性曲线分析可见, 尽管每个样品的多样性存在差异, 但所有的曲线均趋于平缓、指数达到饱和(图1). 表明本实验每个样品的测序量足以完整描述该样品菌群的组成.
| 测序量(条) | 97%相似性水平 | ||||||
| OTU | ace指数 | chao指数 | simpson指数 | shannon指数 | coverage指数 | ||
| 患者A | 20565 | 638 | 1209 | 994 | 0.22 | 2.93 | 0.99 |
| 患者B | 17487 | 667 | 1529 | 1232 | 0.21 | 2.96 | 0.98 |
患者A和B的胃黏膜菌群在97%相似性水平多样性指数详见表1. 患者A胃黏膜菌群多样性的ace指数为1209, chao指数为994, shannon指数为2.93, simpson指数为0.22;患者B胃黏膜菌群多样性的ace指数为1529, chao指数为1232, shannon指数为2.96, simpson指数为0.21. 两样本的chao指数和shannon指数均低于已有研究的粪便菌群多样性指数[11].
分类学分析结果显示, 患者A胃黏膜菌群主要由58个属的细菌构成, 归属于14个门, 以变形菌门为主(99.49%), 其次为放线菌门(0.32%)、硬壁菌门(0.04%)和拟杆菌门(0.03%)等. 患者B胃黏膜菌群构成与患者A相似, 由64个属的细菌构成, 归属于19个门, 以变形菌门为主(99.03%), 其次为硬壁菌门(0.33%)、拟杆菌门(0.23%)和放线菌门(0.12%)等.
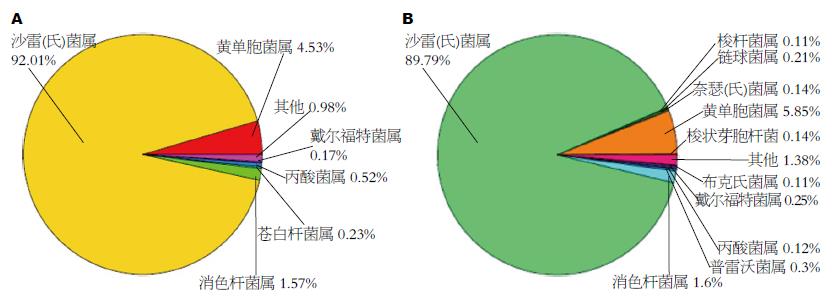
进一步在属水平上进行群落结构分析可见, 患者A的胃黏膜菌群主要由沙雷(氏)菌属(Serratia, 92.01%)、黄单胞菌属(Stenotrophomonas, 4.53%)、消色杆菌属(Achromobacter, 1.57%)、丙酸菌属(Propionibacterium, 0.52%)、苍白杆菌属(Ochrobactrum, 0.23%)、戴尔福特菌属(Delftia, 0.17%)等组成. 患者B的胃黏膜菌群主要由沙雷(氏)菌属(89.79%)、黄单胞菌属(5.85%)、消色杆菌属(1.60%)、普雷沃菌属(Prevotella, 0.3%)、戴尔福特菌属(0.25%)、链球菌属(Streptococcaceae Streptococcus, 0.21%)、梭状芽胞杆菌(Clostridium, 0.14%)、奈瑟(氏)菌属(Neisseria, 0.14%)、丙酸菌属(0.12%)、布克氏菌属(Burkholderia, 0.11%)和梭杆菌属(Fusobacterium, 0.11%)等组成(图2).
过去人们认为胃内由于胃酸和其他抗菌因子的存在, 处于"相对无菌"的状态[12-14]. 而胃内幽门螺杆菌的发现, 打破了传统观念. 目前研究表明, 胃内存在细菌群落, 并已经适应胃内环境[15]. 尽管胃内菌群对人体健康和疾病极其重要, 但目前研究较少, 而且多是依靠传统培养技术和一代测序技术手段为基础, 对研究胃内菌群的多样性和基因多态性有较大偏差[16,17]. 随着基因组学和现代分子生物技术的飞速发展, 以454测序技术为代表的高通量测序技术更能完全覆盖微生物群落, 并能发现罕见的少数肠道微生物, 使人类可以更清晰地研究和了解人体微生物菌群的多样性和基因多态性[18]. 既往二代高通量测序研究主要聚焦于皮肤[19]、口腔[20]、食管[21]和肠道[22,23]等部位的菌群分析, 对国内正常胃黏膜菌群的二代高通量测序分析目前尚无报道.
本研究通过提取两例正常胃黏膜的菌群基因组DNA并扩增其16S rRNA, 并进行高通量测序, 样本A得到20565条优化序列, 经过97%相似度归并后得到638个OTUs; 样本B得到17487条优化序列和667个OTUs. 两样本的coverage指数均在0.98以上, 可以认为样本中至少有98%微生物被检测到, 测序深度较为合理. 2005年, 学者Bik等[24]从23例人胃黏膜活检标本提取细菌基因组DNA, 构建16S rDNA文库, 采用Sanger法一代测序技术只克隆出1833条序列和193.4个OTUs. 本研究每个样品得到的优化序列, 均远远高于Bik EM等克隆出的序列, 从而保证了微生物菌群多样性分析的深度和广度, 为后续的研究打下了坚实的基础. 通过菌群多样性指数比较分析, 样本A和B的chao指数高于Bik EM等的研究约50倍, chao指数是用chao1算法估计群落中含OTU数目的指数, 数值越大代表物种总数越多, 因此可以认为本研究鉴定的正常胃黏膜中微生物菌群中的物种总数远高于Bik EM等的研究. Simpson指数用来估算样品中微生物的多样性指数之一, 在生态学中常用来定量的描述一个区域的生物多样性, Simpson指数值越大, 说明群落多样性越低. Bik EM等研究Simpson指数为5.2, 高于本研究结果, 因此可以说明本研究测序的微生物群落多样性较为丰富. chao指数和Simpson指数有利的证明了采用二代测序能够比一代测序更能深入了解人体微生物菌群的种类和多样性.
Nam等[11]通过高通量测序技术研究正常的肠道粪便菌群并分析菌群多样性, chao平均指数为1500, shanno平均指数为4.99, 均高于本研究多样性结果. 粪便菌群代表了整个消化道菌群, 证明胃肠道菌群物种总数和多样性高于胃黏膜.
通过菌群分类学分析可见, 患者A和B的胃黏膜菌群在门水平上分析结果相似, 均以变形菌门为主, 分别占99.49%和99.03%, 放线菌门、硬壁菌门和拟杆菌门所占比例略有差异. 2011年学者Maldonado-Contreras等[25]提取了12例人胃黏膜细菌基因组DNA, 采用基因芯片技术对16S rRNA进行原位杂交, 并通过G2芯片分类法在门水平上分析, 按照比例从高到低依次为: 变形菌门、硬壁菌门、放线菌门和拟杆菌门. 香港学者Li等[26]采用一代克隆测序分析5例健康人胃黏膜和5例胃炎患者胃黏膜菌群, 在正常胃黏膜菌群中鉴定出硬壁菌门、变形菌门、拟杆菌门、放线菌门和梭杆菌门等8个门, 其中变形菌门所占比例最高(37%), 其次为硬壁菌门(22%), 而胃窦炎患者硬壁菌门(41%)所占比例最高, 其次为拟杆菌门(25%), 变形菌门(20%). 以上二位学者在用不同方法对正常胃黏膜菌群的研究结果与本研究结果大体一致, 变形菌门细菌为正常胃黏膜菌群的优势主导菌. 然而本研究变形菌门所占比例高于上述学者的研究, 主要原因可能与受试对象所在的地域、自然环境因素和饮食等相关.
Hong等[27]经高通量测序技术研究, 认为正常结肠黏膜菌群主要由硬壁菌门(50.9%±21.3%), 拟杆菌门(40.2%±23.8%)和变形菌门(8.6%±4.7%)等组成; 属水平上分析含量最丰富前3位为拟杆菌属, 明串珠菌属和魏斯氏菌属, 但是不同个体和不同部位(左结肠、右结肠和直肠)比例差异较大. 通过比较发现, 本研究正常胃黏膜菌群种类和结肠黏膜菌群大体相似, 但是所占比例不同; 属水平上的细菌差异较大, 优势菌各有不同. 其原因可能是胃内和结肠生理环境和功能不一样, 导致不同的优势菌定植, 继而发挥各自的生理作用.
本研究中经属水平上进行群落结构分析, 患者A和B的胃黏膜菌群以沙雷(氏)菌为优势主导菌. 沙雷(氏)菌包括9个种, 临床上最常见, 也是生化特征最明显的是粘质沙雷氏菌[28]. 粘质沙雷菌为革兰氏阴性菌, 周身鞭毛、能运动、无荚膜、无芽胞, 广泛分布于自然界, 是水和土壤中的常居菌群, 亦是临床上常见的条件致病菌, 在机体免疫功能降低时可引起肺部和尿道感染以及败血症[29,30]. 按Bergey氏分类法, 属于肠道细菌类.
有研究表明, 胃肠道微生物的过度缺失或者过度繁殖, 都会导致炎症性肠病、甚至癌症[31]. 因此, 胃肠道菌群与疾病的关系非常密切. 胃部病理状态下如胃炎或者胃癌患者胃黏膜菌群是否与正常的胃黏膜菌群的结构、多样性和丰度有差别以及幽门螺杆菌存在是否对胃黏膜菌群是否有影响, 仍需要深入研究. 随着上述问题的解决, 将进一步对慢性胃部疾病的发病机制、诊断和临床治疗有重要的指导意义.
目前研究发现正常人的胃肠道菌群的细菌种类超过1000种, 数量约为1014. 胃肠道菌群能够促进人体肠道发育、营养吸收和病原体抵抗等, 并且可以直接参与调节小肠上皮细胞增殖、脂肪储存和炎症应答等机制. 健康状态下, 胃肠道菌群与人体和胃肠道微环境保持着一个平衡状态. 胃肠道微生物的过度缺失或者过度繁殖, 都会导致炎症性肠病、甚至癌症.
谭周进, 教授, 湖南中医药大学
人体胃肠道微生物群落是一个复杂的系统, 传统的培养法无法提供全面的信息来探究其群落结构的组成. 既往二代高通量测序研究主要聚焦于皮肤、口腔、食管和肠道等部位的菌群分析, 对国内正常胃黏膜菌群的二代高通量测序分析目前尚无报道.
Li等采用一代克隆测序分析5例健康人胃黏膜和5例胃炎患者胃黏膜菌群, 在正常胃黏膜菌群中鉴定出硬壁菌门、变形菌门、拟杆菌门、放线菌门和梭杆菌门等8个门, 其中变形菌门所占比例最高(37%), 其次为硬壁菌门(22%), 而胃窦炎患者硬壁菌门(41%)所占比例最高, 其次为拟杆菌门(25%), 变形菌门(20%).
本研究采用高通量测序和生物信息学分析检测正常胃黏膜菌群的结构组成、多样性和丰度, 探讨生理状态下胃黏膜菌群的结构构成和优势主导菌, 为研究胃黏膜菌群与慢性胃部疾病的相关性提供理论依据.
高通量测序技术: 又称为"下一代"测序技术, 是对传统测序一次革命性的改变, 以能一次并行对几十万到几百万条DNA分子进行序列测定和一般读长较短等为标志. 高通量测序技术因其高通量、低成本、高精度和易操作的优点, 已被广泛应用于菌群的结构分析.
肠道微生物是目前研究的热点, 本文对2例病理检测正常的胃黏膜的菌群特征进行了研究, 具有一定科研价值. 选题正确, 实验设计合理, 数据可靠.
编辑: 李军亮 电编: 鲁亚静
| 1. | Palmer C, Bik EM, DiGiulio DB, Relman DA, Brown PO. Development of the human infant intestinal microbiota. PLoS Biol. 2007;5:e177. [PubMed] [DOI] |
| 2. | Bäckhed F, Ding H, Wang T, Hooper LV, Koh GY, Nagy A, Semenkovich CF, Gordon JI. The gut microbiota as an environmental factor that regulates fat storage. Proc Natl Acad Sci USA. 2004;101:15718-15723. [PubMed] [DOI] |
| 3. | Rakoff-Nahoum S, Paglino J, Eslami-Varzaneh F, Edberg S, Medzhitov R. Recognition of commensal microflora by toll-like receptors is required for intestinal homeostasis. Cell. 2004;118:229-241. [PubMed] [DOI] |
| 4. | Noverr MC, Huffnagle GB. Does the microbiota regulate immune responses outside the gut? Trends Microbiol. 2004;12:562-568. [PubMed] [DOI] |
| 5. | Moore WE, Holdeman LV. Human fecal flora: the normal flora of 20 Japanese-Hawaiians. Appl Microbiol. 1974;27:961-979. [PubMed] |
| 6. | Turnbaugh PJ, Hamady M, Yatsunenko T, Cantarel BL, Duncan A, Ley RE, Sogin ML, Jones WJ, Roe BA, Affourtit JP. A core gut microbiome in obese and lean twins. Nature. 2009;457:480-484. [PubMed] [DOI] |
| 7. | Kennedy DM, Stanton JA, García JA, Mason C, Rand CJ, Kieser JA, Tompkins GR. Microbial analysis of bite marks by sequence comparison of streptococcal DNA. PLoS One. 2012;7:e51757. [PubMed] [DOI] |
| 8. | Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner FO. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools. Nucleic Acids Res. 2013;41:D590-D596. [PubMed] [DOI] |
| 9. | Lin YT, Whitman WB, Coleman DC, Chih-Yu C. Molecular characterization of soil bacterial community in a perhumid, low mountain forest. Microbes Environ. 2011;26:325-331. [PubMed] [DOI] |
| 10. | Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appl Environ Microbiol. 2009;75:7537-7541. [PubMed] [DOI] |
| 11. | Nam YD, Jung MJ, Roh SW, Kim MS, Bae JW. Comparative analysis of Korean human gut microbiota by barcoded pyrosequencing. PLoS One. 2011;6:e22109. [PubMed] [DOI] |
| 12. | Marshall BJ, Warren JR. Unidentified curved bacilli in the stomach of patients with gastritis and peptic ulceration. Lancet. 1984;1:1311-1315. [PubMed] [DOI] |
| 13. | Higuchi K, Fujiwara Y, Tominaga K, Watanabe T, Shiba M, Nakamura S, Oshitani N, Matsumoto T, Arakawa T. Is eradication sufficient to heal gastric ulcers in patients infected with Helicobacter pylori? A randomized, controlled, prospective study. Aliment Pharmacol Ther. 2003;17:111-117. [PubMed] [DOI] |
| 14. | Sherwood PV, Wibawa JI, Atherton JC, Jordan N, Jenkins D, Barrett DA, Shaw PN, Spiller RC. Impact of acid secretion, gastritis, and mucus thickness on gastric transfer of antibiotics in rats. Gut. 2002;51:490-495. [PubMed] [DOI] |
| 15. | Merrell DS, Goodrich ML, Otto G, Tompkins LS, Falkow S. pH-regulated gene expression of the gastric pathogen Helicobacter pylori. Infect Immun. 2003;71:3529-3539. [PubMed] [DOI] |
| 16. | Adamsson I, Nord CE, Lundquist P, Sjöstedt S, Edlund C. Comparative effects of omeprazole, amoxycillin plus metronidazole versus omeprazole, clarithromycin plus metronidazole on the oral, gastric and intestinal microflora in Helicobacter pylori-infected patients. J Antimicrob Chemother. 1999;44:629-640. [PubMed] [DOI] |
| 17. | Zoetendal EG, Collier CT, Koike S, Mackie RI, Gaskins HR. Molecular ecological analysis of the gastrointestinal microbiota: a review. J Nutr. 2004;134:465-472. [PubMed] |
| 18. | Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008;26:1135-1145. [PubMed] [DOI] |
| 19. | Roberson ED, Liu Y, Ryan C, Joyce CE, Duan S, Cao L, Martin A, Liao W, Menter A, Bowcock AM. A subset of methylated CpG sites differentiate psoriatic from normal skin. J Invest Dermatol. 2012;132:583-592. [PubMed] [DOI] |
| 20. | Goldman CG, Matteo MJ, Loureiro JD, Almuzara M, Barberis C, Vay C, Catalano M, Heredia SR, Mantero P, Boccio JR. Novel gastric helicobacters and oral campylobacters are present in captive and wild cetaceans. Vet Microbiol. 2011;152:138-145. [PubMed] [DOI] |
| 21. | Yang L, Francois F, Pei Z. Molecular pathways: pathogenesis and clinical implications of microbiome alteration in esophagitis and Barrett esophagus. Clin Cancer Res. 2012;18:2138-2144. [PubMed] [DOI] |
| 22. | Brignardello J, Morales P, Diaz E, Romero J, Brunser O, Gotteland M. Pilot study: alterations of intestinal microbiota in obese humans are not associated with colonic inflammation or disturbances of barrier function. Aliment Pharmacol Ther. 2010;32:1307-1314. [PubMed] [DOI] |
| 23. | Manges AR, Labbe A, Loo VG, Atherton JK, Behr MA, Masson L, Tellis PA, Brousseau R. Comparative metagenomic study of alterations to the intestinal microbiota and risk of nosocomial Clostridum difficile-associated disease. J Infect Dis. 2010;202:1877-1884. [PubMed] [DOI] |
| 24. | Bik EM, Eckburg PB, Gill SR, Nelson KE, Purdom EA, Francois F, Perez-Perez G, Blaser MJ, Relman DA. Molecular analysis of the bacterial microbiota in the human stomach. Proc Natl Acad Sci USA. 2006;103:732-737. [PubMed] [DOI] |
| 25. | Maldonado-Contreras A, Goldfarb KC, Godoy-Vitorino F, Karaoz U, Contreras M, Blaser MJ, Brodie EL, Dominguez-Bello MG. Structure of the human gastric bacterial community in relation to Helicobacter pylori status. ISME J. 2011;5:574-579. [PubMed] [DOI] |
| 26. | Li XX, Wong GL, To KF, Wong VW, Lai LH, Chow DK, Lau JY, Sung JJ, Ding C. Bacterial microbiota profiling in gastritis without Helicobacter pylori infection or non-steroidal anti-inflammatory drug use. PLoS One. 2009;4:e7985. [PubMed] [DOI] |
| 27. | Hong PY, Croix JA, Greenberg E, Gaskins HR, Mackie RI. Pyrosequencing-based analysis of the mucosal microbiota in healthy individuals reveals ubiquitous bacterial groups and micro-heterogeneity. PLoS One. 2011;6:e25042. [PubMed] [DOI] |
| 28. | Dauga C, Grimont F, Grimont PA. Nucleotide sequences of 16S rRNA from ten Serratia species. Res Microbiol. 1990;141:1139-1149. [PubMed] [DOI] |
| 29. | Arroyo JC, Milligan WL, Postic B, Northey J, Parker E, Bryan CS. Clinical, epidemiologic and microbiologic features of a persistent outbreak of amikacin-resistant Serratia marcescens. Infect Control. 1981;2:367-372. [PubMed] |
| 30. | Arslan U, Erayman I, Kirdar S, Yuksekkaya S, Cimen O, Tuncer I, Bozdogan B. Serratia marcescens sepsis outbreak in a neonatal intensive care unit. Pediatr Int. 2010;52:208-212. [PubMed] [DOI] |
| 31. | Grahn N, Hmani-Aifa M, Fransén K, Söderkvist P, Monstein HJ. Molecular identification of Helicobacter DNA present in human colorectal adenocarcinomas by 16S rDNA PCR amplification and pyrosequencing analysis. J Med Microbiol. 2005;54:1031-1035. [PubMed] [DOI] |