修回日期: 2013-03-14
接受日期: 2013-03-29
在线出版日期: 2013-04-18
目的: 应用microRNA(miRNA)芯片筛选及qRT-PCR技术检测可作为遗传性非息肉性大肠癌生物标志物的血清miRNAs.
方法: 选取4个遗传性非息肉性大肠癌患者及3个无家族史正常人的血清miRNAs进行miRNA芯片检测, 再用q-PCR方法对于芯片结果进行验证.
结果: miRNA芯片筛查出57个上调及30个下调miRNAs, 在这些差异性表达的miRNAs中选出8个较为理想的miRNAs作进一步研究, 运用3个靶基因预测软件预测靶基因后取交集, 得到294个靶基因, 均是属于mir-20a-5p, mir-548b-5p和mir-548as-3p的靶基因. 用qPCR的方法在标本中进行验证发现mir-548as-3p的表达情况符合芯片结果-在遗传性非息肉性大肠癌患者血清中表达上调.
结论: 血清miRNAs在遗传性非息肉性大肠癌患者中的差异性表达, mir-548as-3p可能为遗传性非息肉性大肠癌非侵入性的生物标志物.
核心提示: 应用微小RNA(microRNA, miRNA)芯片筛选出可作为遗传性非息肉性大肠癌生物标志物的血清miRNA-mir-548as-3p, 在遗传性非息肉性大肠癌患者血清中显著表达上调. 通过qRT-PCR技术在样本中验证miRNA芯片结果的准确性.
引文著录: 田晰晰, 珠珠, 黄鉴, 任俊宇, 王跃, 张楠, 陈明清, 董坚. 遗传性非息肉性大肠癌生物标志物的血清miRNAs的筛选. 世界华人消化杂志 2013; 21(11): 1040-1045
Revised: March 14, 2013
Accepted: March 29, 2013
Published online: April 18, 2013
AIM: To screen and identify serum microRNAs (miRNAs) that might be used as promising biomarkers for hereditary nonpolyposis colorectal cancer using miRNA array and qRT-PCR.
METHODS: Four serum samples from patients with hereditary nonpolyposis colorectal cancer and three serum samples from healthy controls were used to identify potential markers by miRNA array. The results of miRNA array were confirmed by qRT-PCR.
RESULTS: We found 57 up-regulated miRNAs and 30 down-regulated miRNAs by miRNA array, and 8 miRNAs were chosen for further analysis. Three target gene prediction programs were used to predict target genes of these 8 miRNAs, and 294 genes were predicted, all of which were target genes of mir-20a-5p, mir-548b-5p and mir-548as-3p. qPCR analysis confirmed that serum mir-548as-3p was significantly higher in patients with hereditary nonpolyposis colorectal cancer.
CONCLUSION: Our study demonstrates that serum miRNAs are differentially expressed in patients with hereditary nonpolyposis colorectal cancer, and that mir-548as-3p can potentially serve as a noninvasive biomarker for this disease.
- Citation: Tian XX, Zhu Z, Huang J, Ren JY, Wang Y, Zhang N, Chen MQ, Dong J. Serum microRNAs as promising novel biomarkers for hereditary nonpolyposis colorectal cancer. Shijie Huaren Xiaohua Zazhi 2013; 21(11): 1040-1045
- URL: https://www.wjgnet.com/1009-3079/full/v21/i11/1040.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v21.i11.1040
大肠癌为我国最常见的恶性肿瘤之一, 正以年增长率4.2%的速度逐渐接近欧美发达国家水平(位居第2位), 而遗传性非息肉性大肠癌(hereditary non-polyposis colorectal cancer, HNPCC), 又称Lynch综合征在大肠癌中占10%-15%, 具有肿瘤的遗传性、高发型、多发性和肿瘤发生的多器官性等特点, 其主要的发病机制是错配修复基因(MMR)的突变, 是危害人类健康的主要疾病之一[1-3]. 微小RNA(microRNA, miRNA)是一类长度大约为22个核苷酸的内源性高度保守的非编码单链小RNA分子, 通过与靶基因转录体mRNA互补来降解靶基因或对靶基因进行翻译抑制, 具有癌基因和抑癌基因样作用, 是参与肿瘤发生发展的重要分子. 近年来研究发现[4-7]肿瘤患者的血清血浆中确实存在肿瘤来源的表达特异性的miRNA, 由于血清血浆中miRNA能够逃逸内源性核糖核酸酶的消化, 并且在强酸、强碱、高温、反复冻融、过夜核糖核酸酶消化等条件下都能较稳定地表达, 故研究人员普遍认为血清血浆miRNA比一般蛋白质更适合作为肿瘤标志物. Valeri等[8]研究发现HNPCC患者MMR基因突变频率最高的3个基因-hMLH1、hMSH2、hMSH6是mir-155的靶基因, 说明HNPCC与miRNA之间存在联系.本研究通过miRNA芯片检测HNPCC患者血清与正常人血清miRNA表达谱, 初步探讨血清miRNA作为遗传性非息肉性大肠癌生物标志物的可行性.
共25例血清样本: 来自6个HNPCC家系的10例HNPCC患者血清男6例, 女4例和9例家系正常人血清(有3例结肠镜检查发现有1-2个息肉; 男5例, 女4例), HNPCC患者与家系正常人来自相同的6个家系, 还有6例无HNPCC家族史正常人血清(男3例, 女3例). 本研究所选6个HNPCC家系均符合ACⅡ标准[2]. 研究用人血清样本均获得昆明医科大学附属第一医院伦理委员会的批准, 并取得受试者的知情同意.
1.2.1 miRNA芯片检测: 采用丹麦Exiqon公司的miRCURYTM LAN miRNA芯片, miRNA芯片检测的样本: 实验组: HNPCC患者vs对照组: 无家族史正常人. 分别取200 μL的血清, 按照说明书用TRIzol(Invitrogen) and miRNeasy mini kit (QIAGEN)提取miRNA并用NanoDrop 1000进行miRNA浓度及纯度检测, 采用miRCURY™ Array Power标志试剂盒, 用标记酶Hy3™或Hy5™ 荧光集团标记miRNA, 可以得到用于与芯片杂交的荧光探针, 在标准条件下使用MAUI杂交仪, 将标记好的探针和miRCURY™芯片进行杂交, 重复4次实验. 使用Axon GenePix 4000B芯片扫描仪扫描芯片的荧光强度, 并将实验结果转化成数字型数据保存.
1.2.2 miRNA提取: 取200 μL血清应用miRNeasy mini kit(QIAGEN)按照说明书进行miRNA提取, 用NanoDrop 1000进行miRNA浓度及纯度检测, 于-80 ℃保存.
1.2.3 逆转录反应: 按照One Step PrimeScript miRNA cDNA Synthesis Kit(TAKARA)说明书对miRNA样本进行反转录. 反应条件: 37 ℃ 60 min, 85 ℃ 5 s.
1.2.4 qRT-PCR: 用购自Takara hsa-mir-548as-3p引物, 以cDNA为模版, hsa-mir16[9]为内参, 进行qPCR, 设置3个复孔.按照SYBR® Premix Ex Taq™Ⅱ(TAKARA)说明书操作, 反应条件: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 30 s共40个循环; 95 ℃ 15 s; 60 ℃ 1 min; 95 ℃ 15 s. 荧光定量qPCR仪为StepOnePlus(ABI).
统计学处理 数据处理及统计分析microRNA芯片结果转化成数字型数据保存, 使用GenePix Pro 6.0 software软件对原始数据进行分析运算. 运用Miranda、Targetscan、Mirbase 3个预测软件进行靶基因预测. qRT-PCR实验结果采用SPSS13.0统计软件进行分析, 图表以mean±SD的形式表示.
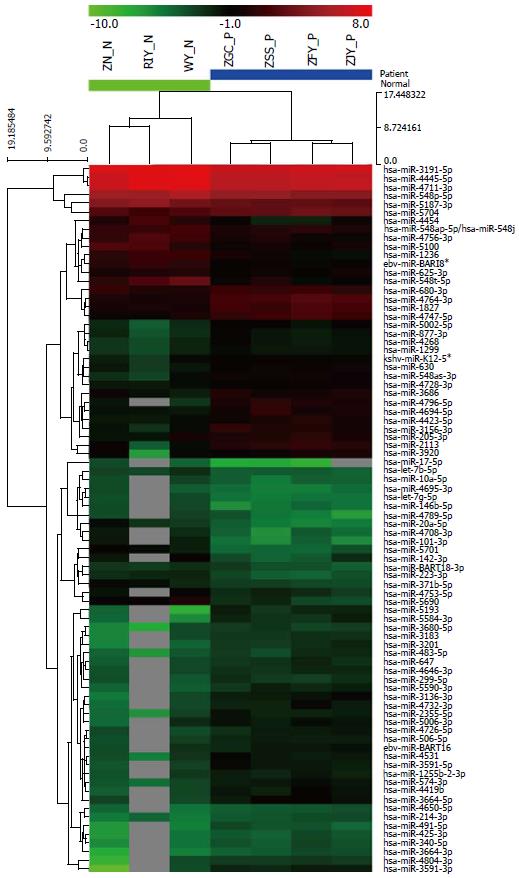
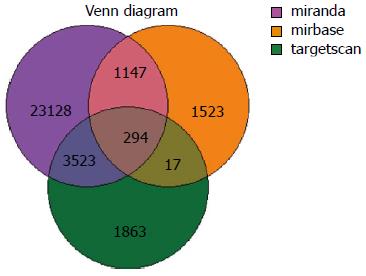
miRNA芯片检测出HNPCC患者血清中较正常人血清有57个上调及30个下调特异性表达的miRNAs(图1). 根据筛选原则: 1 Fold Change值、P值及ForeGround值(理想值>60)取在正常人组、患者组内数值较为平均的miRNA; 2在两组间平均数值差异性明显的miRNA. 筛选出7个较为理想的miRNAs(表1)进行靶基因预测, 分别用Miranda、Targetscan、Mirbase 3个预测软件进行靶基因预测后取交集得到294个靶基因-是mir-20a-5p, mir-548b-5p和mir-548as-3p的靶基因(图2).
| 名称 | 上调 | 名称 | 下调 | ||
| 改变倍数 | P值 | 改变倍数 | P值 | ||
| mir-548as-3p | 3.7 | 0.001 | mir-20a-5p | 0.13 | 0.04 |
| mir-4747-5p | 3.15 | 0.009 | mir-548t-5p | 0.19 | 0.048 |
| mir-1827 | 2.9 | 0.004 | mir-548b-5p | 0.53 | 0.029 |
| mir-5187-3p | 0.40 | 0.021 | |||
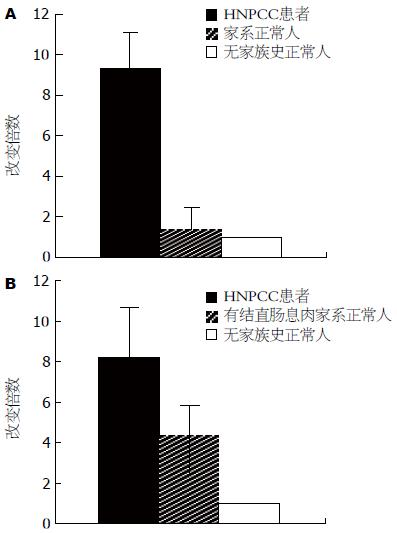
HNPCC患者、家系正常人和无家族史正常人血清中mir-548-as-3p表达量的差异性比较. 10例HNPCC患者mir-548-as-3p在血清中的表达量是6例无家族史正常人的9.30倍±3.47倍(P<0.01, t = 6.72 vs无家族史正常人); 6例家系正常人mir-548-as-3p在血清中的表达量是6例无家族史正常人的1.37倍±1.1倍(P>0.05), 故说明家系正常人与无家族史正常人血清mir-548as-3p表达无差异性, 而HNPCC患者血清mir-548-as-3p的表达量相较于家系正常人及无家族史正常人均显著升高(图3A).
3例有结直肠息肉的家系正常人血清mir-548-as-3p表达量是无家族史正常人的4.43倍±1.46倍(P<0.05, t = 4.89 vs无家族史正常人), 而来源相同家系的3例HNPCC患者mir-548-as-3p血清中表达量是无家族史正常人的8.18倍±2.54倍(P<0.05, t = 4.06 vs无家族史正常人), 故有结直肠息肉家系正常人mir-548-as-3p在血清中的表达量高于无家族史正常人略低于HNPCC患者(图3B).
目前对于HNPCC的临床诊断一般参照ACⅡ标准[2]: (1)1个家族中至少有3例以上患HNPCC相关癌(结直肠癌、子宫内膜癌、小肠癌、输尿管和肾盂癌); (2)其中1例是另外2例的直系亲属; (3)必须累及到连续两代人; (4)至少有1人诊断时<50岁; (5)除外家族性腺瘤; (6)肿瘤经病理证实. 而该诊断标准对于HNPCC家系成员的早期筛查和诊断的意义不大. 文献报道, 对于MLH1和MSH2基因免疫组织化学的检测能发现90%以上的HNPCC患者, 但这是一种有创、不便、令人感觉不适的检测方法, 并且仍然有一定的漏检率, 因此寻找一种新型的, 无创的生物标志物辅助诊断就尤为重要. Chen等[7]研究发现, 血清中含有大量稳定的miRNA, 他可能比一般蛋白质更适合作为生物标志物, 并且发现取等量的血清不提取miRNA直接进行semi-quantitative RT-PCR与血清中提取miRNA后进行semi-quantitative RT-PCR的表达量无明显差异, 若今后作为生物标志物应用于临床则是一种方便快捷准确的临床检验方法.
通过miRNA芯片检测到87个差异性表达的miRNAs, 从中筛选出7个较为可靠的差异性表达的血清miRNAs-mir-548as-3p、mir-4747-5p、mir-1827、mir-20a-5p、mir-548t-5p、mir-548b-5p、mir-5187-3p. 其中mir-1827有一篇与小细胞肺癌相关文献[10], 而关于mir-548家族有一篇详尽的关于其功能等的深入研究报道[11], mir-20a有很多相关文献报道过其与结直肠癌的关系[12], 但是本研究所筛选出的miRNAs并不是那些为人熟知的, 功能及对肿瘤的作用研究透彻的miRNAs. 有文献报道过的miRNAs也是数量有限的, 为我们今后继续HNPCC的血清miRNA表达谱的研究提供了广泛空间.
利用qPCR在25例血清样本中验证了miRNA芯片结果-mir-548-as-3p在HNPCC患者血清中的表达量明显高于无家族史正常人和家系正常人. 值得注意的是来自3个不同家系有结直肠息肉的家系正常人血清mir-548as-3p的表达量虽然低于相应家系中HNPCC患者, 但是较无息肉家系正常人及无家族史的正常人来说表达量高4倍. 这一实验结果可否说明血清mir-548-as-3p可以作为HNPCC早期筛查的生物标志物?设想若可以作为辅助HNPCC早期筛查诊断的一种生物标志物, 当有HNPCC家族史的家系成员除做肠镜筛查排除患HNPCC的可能外, 在通过血清mir-548as-3p检测发现表达量升高时, 便可通过改变生活习惯或做预防性治疗措施来降低HNPCC的发病率, 对提高家系成员的生活质量有重大意义.
通过miranda、mirbase、targetscan 3个靶基因预测软件预测找到了101个mir-548as-3p的靶基因. 有较为常见的癌基因-BAG2、BAX、BCL3、GLI3、MLL、RAD51、TGFBR3等. 这些基因在肿瘤的发生发展中都发挥了一定的作用, 其中BAG2、BAX、BCL3、GLI3有文献报道是结直肠癌相关基因[13-16], MLL主要为急性淋巴细胞白血病和急性髓细胞样白血病相关基因. TGFBR3也为多种癌症相关基因. RAD51参与DNA/RNA损伤修复的基因, 与遗传性乳腺癌的相关[17], 而HNPCC主要致病因素就是错配修复基因的突变引起, 所以值得思考的是RAD51与HNPCC是否相关?HNPCC又称Lynch综合征(癌症家族综合征), 他不仅指结直肠癌还是与众多癌症相关的综合征, 故也正与我们预测的miRNAs靶基因是与多种肿瘤相关基因这一结果相符. 而这些基因参与的通路及细胞功能作用等问题都需要我们在今后的进行更进一步的研究来解释.
本文对血清miRNAs与遗传性非息肉性大肠癌进行了初步的研究, 找到了血清miRNAs与HNPCC间的联系, 而关于这种联系的机制还需在今后做大量的科学研究工作-扩大样本量来进一步验证血清miRNA的芯片结果以及关于miRNA与靶基因, 靶基因与HNPCC间进行更深入的研究以期望发现对于HNPCC早期筛查、诊断、治疗有价值的生物标志物来提高病患的生活质量及生存率.
遗传性非息肉性大肠癌具有肿瘤的遗传性、高发性、多发性和肿瘤发生的多器官性等特点. miRNA是参与肿瘤发生发展的重要分子, 血清miRNA具有比一般蛋白质更适合作为肿瘤标志物的特点, 作为遗传性非息肉性大肠癌生物标志物的研究对于疾病的早期筛查诊断有一定的临床意义.
顾国利, 副主任医师, 中国人民解放军空军总医院普通外科; 王娅兰, 教授, 重庆医科大学基础医学院病理教研室
血清miRNA作为生物标志物在疾病中的临床应用已经成为近年来国内外研究的热点, 并且已经发现多种疾病潜在的miRNA生物标志物, 但若真正成为对临床有指导意义的肿瘤标志物还需要更多实验研究支持.
Huang等研究发现血浆mir-29a和mir92a可作为早期散发性结直肠癌的潜在生物标志物, 并且发现这两个miRNA在早期结直肠癌术前和术后的表达量也不一致, 这说明血清血浆miRNA不仅可以作为早期诊断结直肠癌的生物标志物还有可能作为判断预后的重要指标.
国内外关于血清miRNA在遗传性非息肉性大肠癌的筛查鲜有报道, 而HNPCC又是具有遗传性的肿瘤, 若能找到作为早期诊断的优良生物标志物对于提高患者生活质量及生存率有重大意义.
血清miRNA作为遗传性非息肉性大肠癌生物标志物的可能性并提供一定的依据. 若要应用于临床还需更为精细的研究设计, 更大的样本量反复验证.
本文的研究方法和技术具有一定创新性、系统性. 结果基本可靠, 统计学处理方法的使用恰当. 文章的科学性、创新性较好, 能够反映HNPCC基础研究的先进水平.
编辑: 田滢 电编: 鲁亚静
| 3. | Rodriguez-Bigas MA, Boland CR, Hamilton SR, Henson DE, Jass JR, Khan PM, Lynch H, Perucho M, Smyrk T, Sobin L. A National Cancer Institute Workshop on Hereditary Nonpolyposis Colorectal Cancer Syndrome: meeting highlights and Bethesda guidelines. J Natl Cancer Inst. 1997;89:1758-1762. [PubMed] [DOI] |
| 4. | Mitchell PS, Parkin RK, Kroh EM, Fritz BR, Wyman SK, Pogosova-Agadjanyan EL, Peterson A, Noteboom J, O'Briant KC, Allen A. Circulating microRNAs as stable blood-based markers for cancer detection. Proc Natl Acad Sci U S A. 2008;105:10513-10518. [PubMed] [DOI] |
| 5. | Taylor DD, Gercel-Taylor C. MicroRNA signatures of tumor-derived exosomes as diagnostic biomarkers of ovarian cancer. Gynecol Oncol. 2008;110:13-21. [PubMed] [DOI] |
| 6. | Tsujiura M, Ichikawa D, Komatsu S, Shiozaki A, Takeshita H, Kosuga T, Konishi H, Morimura R, Deguchi K, Fujiwara H. Circulating microRNAs in plasma of patients with gastric cancers. Br J Cancer. 2010;102:1174-1179. [PubMed] [DOI] |
| 7. | Chen X, Ba Y, Ma L, Cai X, Yin Y, Wang K, Guo J, Zhang Y, Chen J, Guo X. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res. 2008;18:997-1006. [PubMed] [DOI] |
| 8. | Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A. Modulation of mismatch repair and genomic stability by miR-155. Proc Natl Acad Sci U S A. 2010;107:6982-6987. [PubMed] [DOI] |
| 9. | Lawrie CH, Gal S, Dunlop HM, Pushkaran B, Liggins AP, Pulford K, Banham AH, Pezzella F, Boultwood J, Wainscoat JS. Detection of elevated levels of tumour-associated microRNAs in serum of patients with diffuse large B-cell lymphoma. Br J Haematol. 2008;141:672-675. [PubMed] [DOI] |
| 10. | Xiong F, Wu C, Chang J, Yu D, Xu B, Yuan P, Zhai K, Xu J, Tan W, Lin D. Genetic variation in an miRNA-1827 binding site in MYCL1 alters susceptibility to small-cell lung cancer. Cancer Res. 2011;71:5175-5181. [PubMed] [DOI] |
| 11. | Liang T, Guo L, Liu C. Genome-wide analysis of mir-548 gene family reveals evolutionary and functional implications. J Biomed Biotechnol. 2012;2012:679563. [PubMed] [DOI] |
| 12. | Chai H, Liu M, Tian R, Li X, Tang H. miR-20a targets BNIP2 and contributes chemotherapeutic resistance in colorectal adenocarcinoma SW480 and SW620 cell lines. Acta Biochim Biophys Sin (Shanghai). 2011;43:217-225. [PubMed] [DOI] |
| 13. | Kambara T, Sharp GB, Nagasaka T, Takeda M, Sasamoto H, Nakagawa H, Isozaki H, MacPhee DG, Jass JR, Tanaka N. Allelic loss of a common microsatellite marker MYCL1: a useful prognostic factor of poor outcomes in colorectal cancer. Clin Cancer Res. 2004;10:1758-1763. [PubMed] [DOI] |
| 14. | Ji H, Kumm J, Zhang M, Farnam K, Salari K, Faham M, Ford JM, Davis RW. Molecular inversion probe analysis of gene copy alterations reveals distinct categories of colorectal carcinoma. Cancer Res. 2006;66:7910-7919. [PubMed] [DOI] |
| 15. | Van Waes C. Nuclear factor-kappaB in development, prevention, and therapy of cancer. Clin Cancer Res. 2007;13:1076-1082. [PubMed] [DOI] |
| 16. | Kang HN, Oh SC, Kim JS, Yoo YA. Abrogation of Gli3 expression suppresses the growth of colon cancer cells via activation of p53. Exp Cell Res. 2012;318:539-549. [PubMed] [DOI] |
| 17. | Kato M, Yano K, Matsuo F, Saito H, Katagiri T, Kurumizaka H, Yoshimoto M, Kasumi F, Akiyama F, Sakamoto G. Identification of Rad51 alteration in patients with bilateral breast cancer. J Hum Genet. 2000;45:133-137. [PubMed] [DOI] |